磺胺类药物pKa 值的QSPR 研究
2015-07-13李美萍张生万
李美萍,张生万
(山西大学生命科学学院,太原030006)
1 引 言
磺胺类药物是一种典型的作为饲料添加剂和疾病防治的兽药,在国内外养殖业中广泛使用.在动物养殖的应用过程中很大部分会随粪便排出体外而进入环境. 磺胺类药物一旦进入环境会分布到土壤、水和空气中,即开始在水体、悬浮物、土壤等环境介质中发生迁移、转化、吸附等一系列过程[1]. 而在环境介质中的吸附和迁移行为是决定环境中磺胺类药物分布和残留的重要过程,其吸附行为与其电离常数pKa 有关,尽管常见磺胺类药物的pKa 值可以通过实验确定或从相关手册上查到,但是通常实验得到的pKa 值的方法比较繁琐,不仅如此,在某些情况下,实验测定值还存在较大不确定性. 因此,研究磺胺类化合物的结构和pKa 值之间的定量关系以及从理论和计算上寻找有效和可靠的预测pKa 值的方法十分重要. 李玉琴[2]曾报道了基于启发式和基因表达式编程法预测磺胺类药物的pKa 值,但关于三维全息原子场作用矢量(three-dimensional holographic vector of atomic interaction field,3D-HoVAIF)描述子用于磺胺类药物的pKa 值预测还未见有关文献报道. 最近,我们从磺胺类药物的分子结构出发,提出了一个简单而有效的方法来计算其pKa 值.通过使用定量结构- 性质相关(quantative-structure property relationonship,QSPR)方法,并结合三维全息原子场作用矢量参数,对31 个磺胺类化合物的pKa 值进行了预测,获得比较满意的结果.此研究不但可以对该类化合物的pKa 值进行预测,还有助于对其在环境中的作用机理的研究.
2 基本原理及方法
三维全息原子场作用矢量(three-dimensional holographic vector of atomic interaction field,3DHoVAIF)[3]从原子之间相对距离和原子的自身性质出发,基于三种原子间非键作用方式—静电、立体和疏水作用,将有机分子中常见的氢、碳、氮、磷、氧、硫、氟、氯、溴、碘原子分别按其所处元素周期表中不同主族划分为5 大类,为了能充分表达分子的细微结构特征,又将不同主族中原子按其杂化状态分为10 类,这样分类后一个分子内部不同类别的原子之间,相互作用情况就有55 种. 由于3D-HoVAIF 分别采用静电、立体和疏水三种势能来表达原子间不同的作用形式,所以,对于一个有机分子而言,用于表征分子结构信息的原子作用项就有3 ×55 =165 个. 这些三维矢量描述子可以不借助任何实验参数得到. 其具体的描述公式参见文献[3-6],在此不再赘述.
2.1 数据采集及结构表征
本文研究的31 种磺胺类药物及其pKa 值来自于文献[2],其名称见表1. 使用Chemoffice8.0 绘制31 种磺胺类药物的分子结构图,用Chem3D 自带的MOPAC 半经验量子化学软件在AM1 水平上最终优化得到分子空间结构(截断值0.001KJ/mol),并采用Mulliken 布居分析法以单点(singlepoint)形式计算出原子的净电荷数量,将分子中每个原子的空间位置及电荷分别以笛卡儿坐标和净电子数目的形式输入 C 语言应用程序3DHoVAIF. exe 加以处理,得到分子的3D-HoVAIF 描述子.

表1 磺胺类药物名称及pKa 值Table 1 Name and pKa values of sulfonamides
2.2 模型建立
本文以R (建模相关系数),Rcv (交互检验相关系数),RMSEF (拟合误差)和RMSEP (预测误差)作为衡量模型预测能力的标准. 关于模型建立过程中训练集和测试集的样本选取,我们按照参考文献[2]从31 个样本中选取10 个样本作为测试集,其余的21 个样本作为训练集,选取的测试集在表1 中用“* ”号标出.
为了研究3D-HoVAIF 描述子与磺胺类药物pKa 值的内在联系,本文使用三维全息原子场作用矢量165 个描述子进行逐步回归 (Stepwise Multiple Regression,SMR),选出显著性变量,分别采用多元线性回归(Multiple Lines Regression,MLR)和偏最小二乘回归 (Partial Least Square Regression,PLS)进行建模. 并采用留一法交互检验对模型的稳定性和预测能力进行评估.
2.2.1 多元线性回归
在多元线性回归过程中,为了消除变量间偶然相关的影响,剔除影响模型稳定性的自变量,使回归方程始终显著,需要在多元回归分析中引入逐步回归的方法. 本文采用SPSS 统计学软件对变量进行逐步回归分析,得到如下方程:
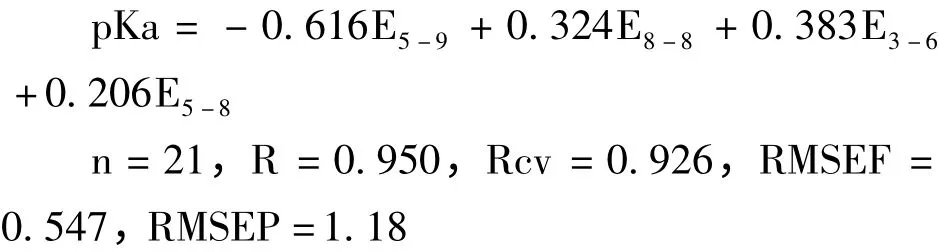
其中,E 表示静电作用. 如,E5-9表示第5 类N 原子与第9 类S 原子的静电作用项,即sp3杂化的N原子与sp2杂化的S 原子之间的静电作用;E8-8表示第8 类S 原子与第8 类S 原子的静电作用项,即sp3杂化的S 原子与sp3杂化的S 原子之间的静电作用;以此类推. 根据本实验建立的模型能够看出,sp3杂化的N 原子与sp2杂化的S 原子之间的静电作用对磺胺类药物的pKa 影响较大,且呈负相关. 使用上述模型对31 个磺胺类化合物的pKa值进行估计与预测,并将计算结果与实验值相关情况绘于图1 中.
2.2.2 偏最小二乘回归
为了检验模型的稳定性,继而用偏最小二乘回归方法进行建模. 为保证变量单位空间尺度一致,首先采用自定标法对原始变量标准化处理,随后用化学计量学软件Simca -p10.0 对训练集样本建模,并以交叉检验最高值(交叉检验Y 的解释方差)所对应的主成分数2 作为模型的复杂程度. 由4 个变量提取2 个主成分解释了Y 变量89.6%方差,交叉检验解释Y 的方差为78.3%,其结果见表2. 同时将pKa 值实验值与预测值对比结果绘于图1 中. 从图1 和表2 可见PLS 计算结果与多元线性回归结果基本吻合,说明所选模型具有较好的稳定性和预测能力.
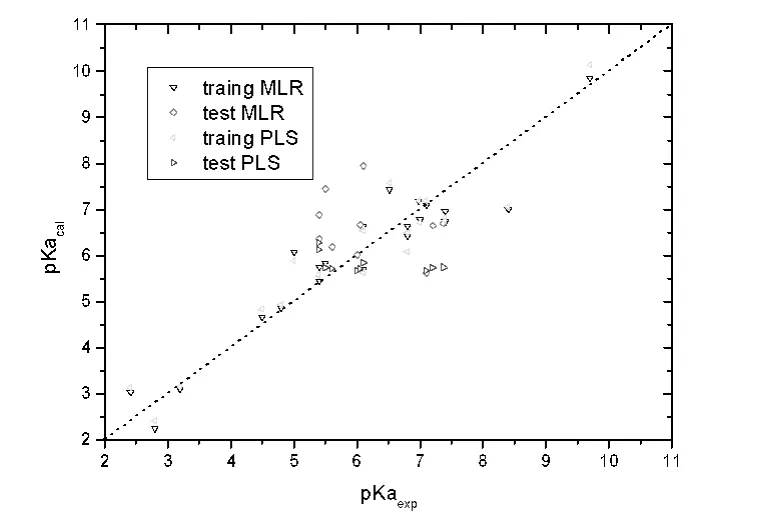
图1 pKa 实验值与预测值相关Fig.1 Plot of calculated value and experimental value
文献1 运用启发式方法(HM)和基因表达式编程(GEP)方法建立了磺胺类药物pKa 值预测模型,为了便于比较将其结果与本文基于3D -HoVAIF 描述子建模的相关数据列表2.

表2 磺胺类药物pKa 值QSPR 模型比较Table 2 Contrast of QSPR models for pKa values of sulfonamides
从表2 可看出,采用3D-HoVAIF 所建模型和文献2 建模所得相关系数和交互检验复相关系数相当,但3D-HoVAIF 计算简便,结构参数的取值完全来自分子本身的结构,描述子的物理意义明确且所建的线性模型可以识别和提供对这些化合物的pKa 值起作用的描述符.
3 结 论
研究结果表明了静电作用是影响磺胺类药物pKa 值的主要因素,尤其是sp3杂化的N 原子与sp2杂化的S 原子之间的静电作用对磺胺类药物的pKa 值影响较大,且呈负相关. 同时从本文的结果看出3D-HoVAIF 能够较好地表征磺胺类药物分子的结构信息,具有计算简便、物理意义明确的优点,并且避免了当今大多3D 分子设计技术在应用前需要对分子进行叠合操作等方面缺点,将其应用在其他抗癌药物活性[7-10]研究方面也取得了一定的效果,显示出3D-HoVAIF 较为广阔的应用前景.
[1] He J H,Qiu J R,He D C,et al. Environmental behavior and related control technologies of sulfonamides[J]. Guangdong Agri. Sci.,2012,7:225(in Chinese)[何金华,丘锦荣,贺德春,等. 磺胺类药物的环境行为及其控制技术研究进展[J]. 广东农业科学,2012,7:225][2] Li Y Q,Si H Z,Xiao Y L,et al. Quantitative structure activity relationship models based on heuristic method and gene expression programming for the prediction of the pKa values of sulfa drugs[J]. Acta Pharm. Sin.,2009,44(5):486(in Chinese)[李玉琴,司宏宗,肖玉良,等.基于启发式和基因表达式编程法预测磺胺类药物的pKa 值[J]. 药学学报,2009,44 (5):486]
[3] Zhou P,Tian F F,Li Z L. Three dimensional holographic vector of atomic interaction field(3D - HoVAIF)[J]. Chem. Intell. Lab. Syst.,2007,87:88.
[4] Tong J B,Zhou P,Zhang S W,et al. QSAR studies of anti-HIV drug HEPT using (3D -HoVAIF)[J].Acta Phys. Chim. Sin.,2006,22:721(in Chinese)[仝建波,周鹏,张生万,等.三维全息原子场作用矢量用于HEPT 类抗艾滋病药物的QSAR 研究[J].物理化学学报,2006,22:721]
[5] Zhou P,Mei H,Tian F F,et al. Applying novel three dimensional holographic vector of atomic interaction field to QSAR studies for several classical drug systems[J]. Chem. J. Chin. Univ.,2007,28(6):1027(in Chinese)[周鹏,梅虎,田菲菲,等. 新型三维原子场全息相互作用矢量用于几类典型药物体系的QSAR 研究[J]. 高等学校化学学报,2007,28(6):1027]
[6] Li M P,Zhang S W. QSAR studies of 5'-methylthioadenosine nucleosidase inhibitors using three -dimensional holographic vector of atomic interaction field[J]. J. Anal. Sci.,2011,27(5):566 (in Chinese)[李美萍,张生万. 三维全息原子场作用矢量用于5' -甲硫腺苷核苷酶抑制剂的QSAR 研究[J]. 分析科学学报,2011,27(5):566]
[7] Tong J B,Wang P,Li Y F,et al. Quantitative structure activity relationship studies of 5 -pheny-1 -phebylamino-1H - imidazole of Anti - HIV drug using three-dimensional descriptors[J]. J.At.Mol.Phys.,2011,28(1):41(in Chinese)[仝建波,王平,李云飞,等. 咪唑衍生物类抗艾滋病药物3D -QSSR 研究[J]. 原子与分子物理学报,2011,28(1):41]
[8] Tong J B,Chen Y,Che T,et al. Quantitative structure activity relationship studies of anti - tuberculosis drug using three - dimensional structure descriptors[J]. J. At. Mol. Phys.,2012,29(3):382(in Chinese)[仝建波,陈洋,车挺,等. 酰肼类抗结核病药物的3D - QSAR 研究[J]. 原子与分子物理学报,2012,29(3):382]
[9] Xu L N,Liang G Z,Li Z L,et al. Three-dimensional holographic vector of atomic interaction field for quantitative structure - activity relationship of Aza -bioisosteres of anthrapyrazoles (Aza - APs)[J]. J.Mol. Graph. Model.,2008,26:1252.
[10] Zhu W P,Liang G Z,Liao L M,et al. Chemical structural characterization and design of influenza neuram inidase inhibitors using three - dimensional holographic vector of atomic interaction field through quantitative structure-activity relationship studies[J]. Chinese J.Anal.Chem.,2008,36(6):799(in Chinese)[朱万平,梁桂兆,廖立敏,等. 三维全息原子场作用矢量(3D-HoVAIF)用于神经氨酸酶抑制剂的结构表征与设计及定量构效关系(QSAR)研究[J]. 分析化学,2008,36(6):799]
