纳米材料和蛋白质相互作用的表征方法
2015-07-13张谐天陶玉贵杨良军郑述翔梁敏东
张谐天,陶玉贵,杨良军,郑述翔,梁敏东
(安徽工程大学 生物与化学工程学院,安徽 芜湖 241000)
纳米材料在生物学领域的应用范围十分广泛,如药物输送、成像、治疗、诊断以及接下来更进一步的发展,包括激活/失活、仿制和实现生物分子系统的功能[1]。这些功能都是基于纳米材料独特的物理和化学性质、尺寸大小等特点。但是纳米粒子一旦进入生物系统中,就会与蛋白质之间相互反应,在纳米粒子表面形成所谓的“蛋白质冠状物”[2]。
近期研究中,纳米粒子表面所形成的蛋白质冠状物对于纳米粒子的相关生物学影响已经被讨论。目前,纳米粒子与蛋白质的相互作用可以分为两大类[3]:①纳米粒子与单一的蛋白质的相互反应,即结合到纳米粒子上的蛋白质种类已知;②纳米粒子与数以百计的蛋白质组之间的相互反应。一旦纳米材料与蛋白质相互作用之后,其表面的性质都会发生不定向性的改变[4]。
因此寻找到一个对蛋白质与纳米粒子复合物的特性进行准确分析的技术方法和仪器尤其重要。Manmoudi 等[4]罗列出了研究影响蛋白质冠状物形成因素的基本表征方法,Li 等[5]总结了纳米粒子与不同蛋白质类型反应的动力学和热力学表征方法。然而,针对蛋白质冠状物的厚度、构象等方面的表征方法的总结相对较少。在此,将目前对蛋白质冠状物各种参数的不同表征方法及其主要特点总结于表1 中,同时下文将会进一步详细的讨论每种表征方法。

表1 纳米粒子与蛋白质相互作用的分析方法Table 1 Overview of analytical strategies to monitor NP-protein interaction
1 蛋白质冠状物的物理参数
1.1 蛋白质冠状物的密度与厚度
1.1.1 透射电子显微镜(TEM) TEM 以波长远小于可见光的电子束作为光源,且电子束的波长与发射电子束的电压平方根成反比,也就是说在高电压的条件下,波长就会相对较短,所以其分辨率,就会相对较高,能够观察到光学显微镜下无法看清的小于0.2 nm 的细微结构。由于电子束的穿透能力非常微弱,所以当采用透射电子显微镜技术时,必须要注意电镜标本的制作要求,即可在不损伤试样原始组织的条件下[6]直接从纳米或微米尺度的试样中切取厚度为50 nm 左右的薄膜。如图1[7]所示,N-琥珀酰壳聚糖与蛋白质的复合物的直径通常为100~200 nm。

图1 N-琥珀酰壳聚糖吸附BSA 之后的TEMFig.1 TEM morphology of NSCS loaded BSA N-琥珀酰壳聚糖浓度为0.5 mg/mL,BSA/N-琥珀酰壳聚糖的摩尔比30 ∶1
1.1.2 原子力显微镜(AFM) 原子力显微镜是一种可用来研究包括绝缘体在内的固体材料表面结构的分析仪器。由于其对工作环境的适应性非常强,如既可以在常温常压的条件下工作,也可以在液体环境中工作,所以其可以用来观察SEM 和TEM 所不能观察的样品,如反应液中大分子的整体结构、具有生命特性的活体组织。AFM 可以提供纳米粒子与蛋白质复合物的三维表面图,且不需要对纳米粒子与蛋白质复合物进行任何特殊处理,这样就不会对复合物造成不可逆的转变[8]。但是其运行的速度较慢,虽然能够呈现出三维立体的样品轮廓,但是其成像范围却非常有限。
1.2 蛋白质冠状物的数量
1.2.1 离心分离 离心分离,是通过高速旋转所产生的离心力对吸附到纳米粒子上的蛋白质组进行分离的最基本方法。在离心过程中,必须注意以下几点:①溶液体积的变化;②样品的洗涤步骤;③样品的洗涤时间。因为所形成的蛋白质冠状物的厚度,表面形貌都取决于这些因素。但是在离心高分子量蛋白质或者蛋白质复合物时,通常会分不清蛋白质冠状物在离心管中的位置[9]。
1.2.2 色谱法 尺寸排阻色谱技术(SEC),也被称为凝胶过滤层析法。由于待分离物质自身重量和体积的大小的不同,通过凝胶孔隙的时间也随之不同,从而对其进行分离、纯化。因此,SEC 也能够检测到蛋白质结合到纳米粒子上的解离和吸附的程度。采用SEC 对碳富勒烯-蛋白质,二氧化硅纳米粒子-蛋白质以及高分子蛋白质聚合物进行分离的已经报道出来了。除了分离蛋白质,SEC 也能够检测到在纳米粒子与蛋白质相互反应过程中最佳的结合率和交换率[10]。离子交换色谱(IEC)包括阳离子交换色谱法和阴离子交换色谱法[11],根据待测物自身所带的净电荷,不断调整pH 值或者流动相的离子浓度来分别进行蛋白质、纳米粒子以及纳米粒子与蛋白质复合物的分离[12]。
1.2.3 电泳 毛细管电泳可以根据蛋白质的电荷和摩擦力分离蛋白质,分离的程度与结果通常可以用紫外线或者荧光检测器来检测[13],不仅可以对吸附到纳米粒子上的不同种类的蛋白质进行研究,如血浆蛋白、白蛋白等,而且可以使吸附蛋白质不需要经历解吸过程就可以直接量化。然而,在分离过程中,一定要注意毛细管的内表面不能吸附有蛋白质,这样就会严重影响到检测的灵敏度[14]。凝胶电泳法是根据不同大小的蛋白质,其在电泳中的迁移率也随之不同,所以垂直通过具有网状结构的高分子聚丙烯酰胺凝胶的速度不一样,从而得到分离,再将凝胶中的蛋白质用考马斯蓝或者硝酸银等染料进行染色,通过密度检测法来量化蛋白质丰度[15]。在纳米粒子颗粒和单个的蛋白质相互作用反应体系中,1-CE 既可以用来纯化纳米粒子-蛋白质复合物,还可用来评估纳米粒子与蛋白质的结合率[16]。而双向凝胶电泳(2-DE)则可以根据不同蛋白质等电点[17]和分子量都不相同[18]的特点分离出大量(5 000)不同种类的蛋白质[13]。这两种方法常用于测定纳米粒子与蛋白质复合物的数量。
1.2.4 动态光散射(DLS) DLS 是一项基于纳米粒子的光散射进行工作的,广泛应用于待测样品流体力学直径测定的技术[19]。尺寸相似的蛋白质和纳米粒子产生的散射光比较难以区分,因此,只有保证NPs 尺寸明显大于蛋白质的尺寸(10 nm)[20]才能避免蛋白质和纳米粒子的散射光相互混淆。DLS可以清楚的观察到蛋白质冠状物的形成过程,确定纳米粒子与蛋白质复合物的流体动力学半径的大小及其变化[21]。当蛋白质结合到纳米粒子的表面上时,纳米粒子的粒径就会增大,当其表面结合的蛋白质达到饱和时,这种增大就会停止。因此,可以通过纳米粒子尺寸增大的比例来间接测定蛋白质与纳米粒子的结合率[22],而且DLS 还可以通过Einstein-Stokes 方程测定出复合物的扩散常数:
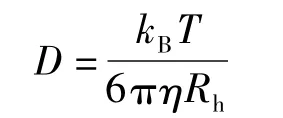
式中,D 为扩散常数;kB为玻尔兹曼常数;T 为绝对温度;η 为粘度;Rh为颗粒流体动力学半径。
1.2.5 紫外吸收可见光谱(UV-vis spectroscopy)
紫外光谱见图2。
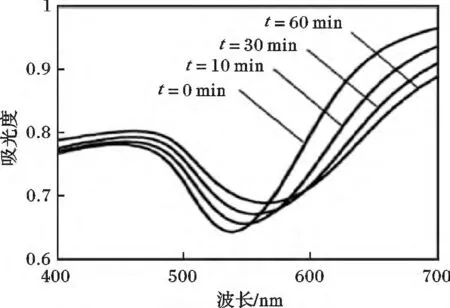
图2 NPs 与BSA 进行反应的紫外可见光谱的透射范围Fig.2 Transmission spectra of gold NPs incubated with BSA measured by UV-Vis spectroscopy
紫外吸收可见光谱是基于待测样品在紫外和可见光波长范围内入射光(I0)与透射光(I1)比率的度量对其进行分析的,具有反应速度灵敏,操作过程简单,且对样品的制备要求较低等特点。纳米粒子表面吸附的蛋白质诱导了吸收光谱发生如宽度和吸收峰的移动等变化,这些变化可以用来检测纳米粒子与蛋白质的结合率。但是样品的吸收范围会受到不同参数的影响而使量化难度增大,如反应物、浓度、pH 值以及环境的介电常数等,所以其量化程度一般。然而,对于那些符合朗伯-比尔定律的样品,特别是浓度很低的时候,通过与单个成分相重叠的光谱范围,可以得到吸附蛋白质的浓度。这个方法已经被用来研究碳纳米管吸附BSA 的实验中[23]。在AuNPs 吸附BSA 的研究中[22],等离子峰出现了红移和宽度扩大,证明了BSA 的吸附量发生了一定的转变。
2 蛋白质冠状物的化学参数
2.1 蛋白质冠状物的种类
2.1.1 基质辅助激光解析电离飞行时间质谱分析
(MALDI-TOF-MS) 基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)是一个通过检测磷蛋白降解产生的磷酸肽来分析蛋白质磷酸化作用的有效工具[20],确定蛋白质与NPs 是否结合。蛋白质通过一个蛋白酶被切除和消化,碎片可以用MALDI-TOFMS 分析出来,然后在蛋白质数据库中搜寻肽质量指纹谱来确定蛋白质的种类。对于结合到纳米粒子与上的蛋白质的确定,通常采用以下两种方法:首先,使蛋白质从纳米粒子表面释放出来,如使用表面活性剂等[24],然后通过电泳将其分离出来。第二种方法是就地纯化的纳米粒子与蛋白质复合物,然后通过LC/MS 分离和分析。如图3 所示[25],MALDITOF-MS 成功的发现了样品中独特的多肽。

图3 基质辅助激光解析电离飞行时间质谱(A)核纤层蛋白Alelta10;(B)核纤层蛋白AFig.3 MALDI-TOF spectrum of (A)lamin Adelta10,(B)lamin A
2.1.2 N 端微量测序 N 端微量测序是从蛋白质/多肽N-末端按顺序切割下来,通常为含有10 ~30个N 端氨基酸残基,且易形成稳定的多肽序列用于分析鉴定。同时还可以通过蛋白质数据库来识别结合到纳米粒子上的蛋白质[26]。双向凝胶电泳中的蛋白质可以通过自动气相测序器来测定自身氨基酸序列[27]。该技术的主要缺点就是当氨基酸的N 端被化学修饰或者是它隐藏在蛋白质内部的时候D端微量测定技术就不会起作用了。此外,由于该方法需要蛋白质纯度的识别效率较高,所以要与双向凝胶电泳一起使用[28]。
2.2 蛋白质冠状物的构象
蛋白质和纳米粒子之间的相互作用可能会引起蛋白质构象的变化,而这些变化又可能会产生未知抗原以及蛋白质功能的转变。所以,检测当纳米粒子与蛋白质相互反应时的蛋白质的构象的变化是生物学中一个重要的研究方向。分析的方法通常使用傅里叶变换红外光谱(FTIR)、拉曼光谱、荧光光谱和圆二色性光谱(CD)。
2.2.1 红外(FTIR)和拉曼光谱 红外光谱和拉曼光谱在测定蛋白质构象变化的过程中是相辅相成的,不仅可以确定蛋白质是否结合到纳米粒子的表面,还可以检测纳米粒子与蛋白质复合物的表面特性。对于红外光谱,蛋白质的二级结构可以通过酰胺键的吸收率来计算。在酰胺Ⅰ,Ⅱ和Ⅲ带中,酰胺Ⅰ振动谱带(1 700 ~1 600 cm-1)是最敏感的区域,这个区域最适合确定蛋白质的构象。Noinville等[29]课题组利用BSA 吸附在载体表面时构象改变对自身亲水性、疏水性的影响。如图4 所示[30],从红外光谱中可以看出不同摩尔比的BSA 与AgNP 反应时,酰胺Ⅰ带和酰胺Ⅱ带振动峰值的区别。
Samir 等[31]利用拉曼光谱分别测定了溶菌酶和α-乳白蛋白吸附在反相作用色谱介质上的构象变化。蛋白质的拉曼光谱中展现的吸收带主要由三部分构成:①多肽主链;②芳香族侧链;③含硫的侧链。拉曼光谱较红外光谱有两大优势:①拉曼光谱可以测量水溶液中纳米粒子与蛋白质的复合物;②操作简单易懂,测量光谱区域不受局限。
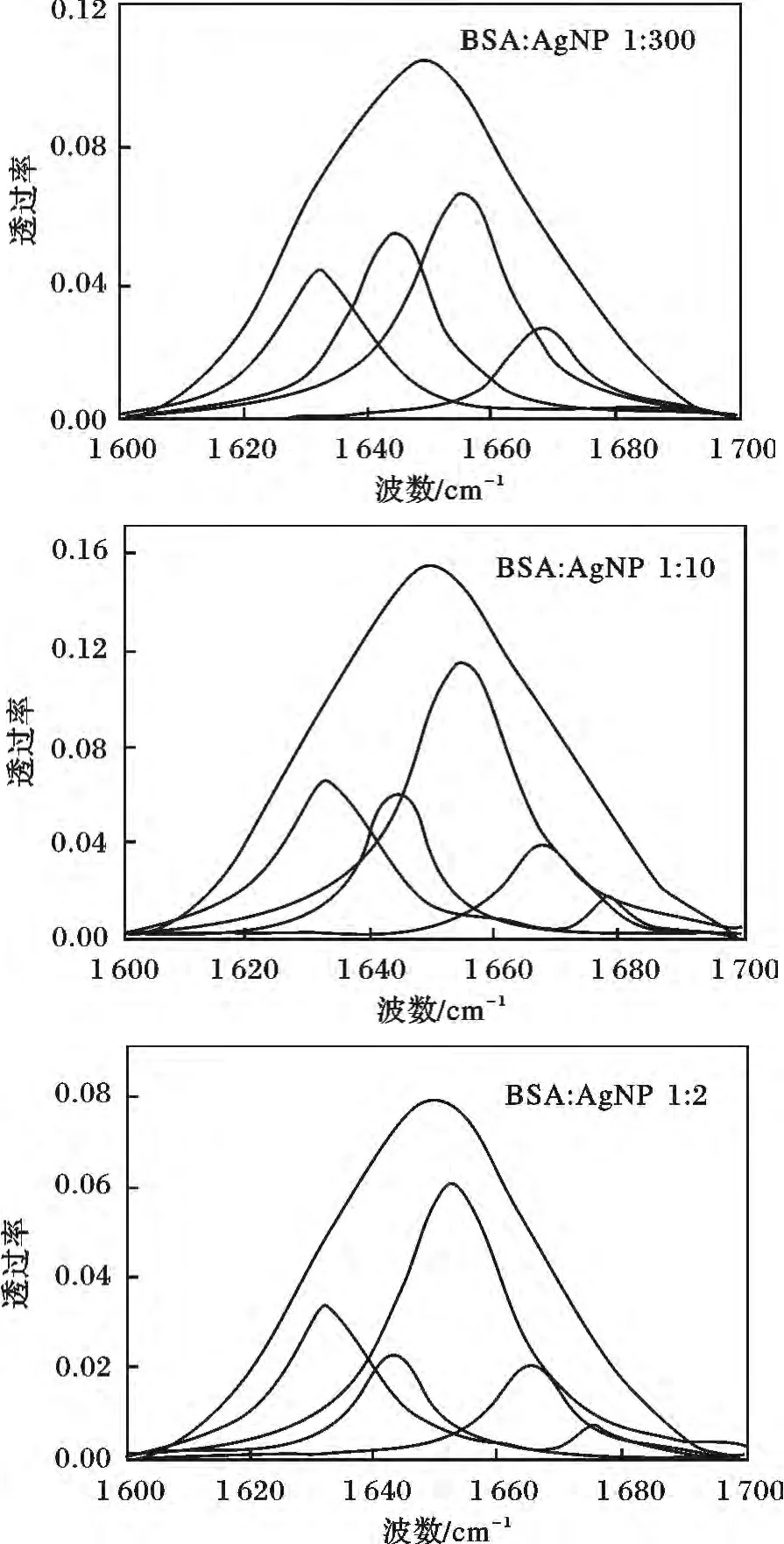
图4 4 种不同摩尔比的BSA 和AgNP 在25 ℃条件下的FTIR 光谱Fig.4 FTIR spectra of BSA at four different molar ratio of BSA to AgNP at 25 ℃temperature
2.2.2 荧光光谱 因为许多重要的生物反应过程如蛋白质构象的变化、蛋白质侧链的旋转运动以及分子之间的结合的时间仅仅只需要几秒钟时间,然而这短短几秒中也正好是被激发的荧光状态维持的时间[32],所以荧光光谱不仅可以测量结合位点的数量、结合常数以及蛋白颗粒结合的协同程度(希尔常数)[33],同时对蛋白质结构变化的测定也非常灵敏。
蛋白质是由氨基酸构成的大分子结构,在构成蛋白质的众多氨基酸种类中,部分氨基酸自身就含有荧光素,如络氨酸、色氨酸以及苯丙氨酸。在纳米粒子与蛋白质相互作用的过程中,NPs 或者蛋白质至少其中一个必须含有内在荧光素。如若不然,则必须向该反应体系中加入外源荧光素。因为纳米粒子与蛋白质之间的相互反应通常会改变荧光素本身的化学环境,甚至是自身的结构,这就会改变蛋白质的荧光性,导致蛋白质发生动态/静态荧光猝灭。一旦蛋白质发生荧光猝灭,光谱中的荧光强度就会减弱,原本的发射峰就会发生偏移,同时也可以根据荧光个向异性法(FA)[34]和荧光共振能量转移[35]来共同证明纳米粒子与蛋白质发生了相互作用。如果荧光猝灭效应是由碰撞引起的,荧光团的生命周期也将减少[29]。荧光强度猝灭的周期遵循Stern-Volmer 的关系:
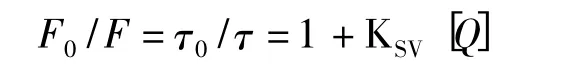
式中,F0,F 分别代表初始和修饰之后的荧光强度;τ0代表周期;Ksv是Stern-Volmer 常数;[Q]是猝灭浓度[36]。
在多功能蛋白质与多壁碳纳米管相互作用的实验中,此方程被用来表明亲和力是取决于纳米管的直径、表面化学性质和蛋白质类型,Shang 等[37]课题组通过不同pH 值的反应体系中BSA 的荧光发射光谱的不同证明了BSA 的构象会受到反应体系中不同pH 值的影响,见图5。
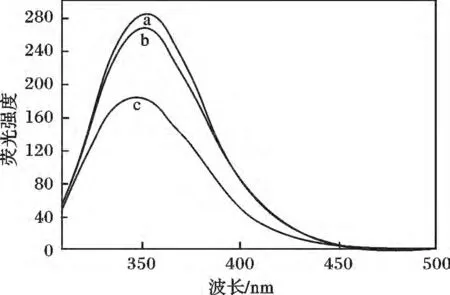
图5 BSA 的荧光发射光谱Fig.5 Fluorescence emission spectra of BSA
2.2.3 圆二色性光谱(CD) 不同的蛋白质二级结构(α-螺旋,β-折叠)在CD 光谱的紫外区域都有着自己的特点,所以圆二色性光谱法已经广泛用于检测结合到纳米粒子上的蛋白质的构象变化[38]。然而,CD 通常反应的是整个分子的平均值,所以即使CD 可以测定出一个蛋白质含有50 个α-螺旋,但是也不能确定哪个特定的氨基酸残基参与了α-螺旋的形成。另一个不足就是在200 nm 左右的高吸收能量区域可能会降低测量的准确度。因此,蛋白质二级结构百分比的计算通常是采用在长波长(>200 nm)区域内收集的数据[39]。Su 等[40]通过CD光谱,在不同pH 值的条件下,研究了碳纳米管表面不同的多肽的构象变化,发现在接近中性pH 值的范围内,结合到碳纳米管上的UW-1 的β 折叠结构发生变形,而蛋白质B3的α 螺旋构象并没有改变。
3 结束语
特殊化学和物理性能的纳米材料在生物医学领域的研究飞速增长,但对于其自身毒性以及应用的安全性研究仍处于起步阶段,而且目前的研究大部分都是集中在蛋白质与纳米颗粒之间短时的相互作用,这些相互作用包括蛋白质冠状物的形成,细胞直接的接触,粒子在细胞表面的结合、内吞以及进入细胞内的反应过程等方面。因此仍然需要大量的科学研究以及更多的分析技术和方法来更好的了解纳米材料与生物系统相结合的界面发生的变化,从而深入了解纳米材料-蛋白质相互作用的机理,全面认识纳米材料毒性问题,客观评价纳米材料-蛋白质之间复杂的相互作用。
[1] Bergese P,Hamad-Schifferli K.Nanomaterial Interfaces in Biology[M].New York:Humana Press,2013.
[2] Hu Z,Zhao L,Zhang H,et al. The on-bead digestion of protein corona on nanoparticles by trypsin immobilized on the magnetic nanoparticle[J].Journal of Chromatography A,2014,1334:55-63.
[3] Cedervall T,Lynch I,Lindman S,et al.Understanding the nanoparticle-protein corona using methods to quantify exchange rates and affinities of proteins for nanoparticles[J]. Proceedings of the National Academy of Sciences,2007,104(7):2050-2055.
[4] Mahmoudi M,Lynch I,Ejtehadi M R,et al.Protein-nanoparticle interaction:opportunities and challenges[J].Chemical Reviews,2011,111(9):5610-5637.
[5] Li L,Mu Q,Zhang B,et al.Analytical strategies for detecting nanoparticle-protein interaction[J]. Analyst,2010,135(7):1519-1530.
[6] Cui M,Liu R,Deng Z,et al.Quantitative study of protein coronas on gold nanoparticles with different surface modifications[J].Nano Research,2014,7(3):345-352.
[7] Zhu A,Yuan L,Chen T,et al.Interactions between N-succinyl-chitosan and bovine serum albumin[J]. Carbohydrate Polymers,2007,69(2):363-370.
[8] Mao S,Lu G,Yu K,et al.Specific protein detection using thermally reduced graphene oxide sheet decorated with gold nanoparticle-antibody conjugates[J]. Advanced Materials,2010,22(32):3521-3526.
[9] Aggarwal P,Hall J B,McLeland C B,et al. Nanoparticle interaction with plasma proteins as it relates to particle biodistribution,biocompatibility and therapeutic efficacy[J]. Advanced Drug Delivery Reviews,2009,61(6):428-437.
[10]Chithrani B D,Chan W C W.Elucidating the mechanism of cellular uptake and removal of protein-coated gold nanoparticles of different sizes and shapes[J].Nano Letters,2007,7(6):1542-1550.
[11]Okuda M,Eloi J C,Jones S E W,et al.Fe3O4nanoparticles:protein-mediated crystalline magnetic superstructures[J].Nanotechnology,2012,23(41):1-7.
[12] Aubin-Tam M E,Hamad-Schifferli K. Gold nanoparticlecytochrome c complexes:the effect of nanoparticle ligand charge on protein structure[J].Langmuir,2005,21(26):12080-12084.
[13] Kim H R,Andrieux K,Delomenie C,et al. Analysis of plasma protein adsorption onto PEGylated nanoparticles by complementary methods:2-DE,CE and Protein Labon-chip system[J].Electrophoresis,2007,28(13):2252-2261.
[14] Lynch I,Dawson K A. Protein-nanoparticle interactions[J].Nano Today,2008,3(1):40-47.
[15] Monopoli M P,Walczyk D,Campbell A,et al. Physicalchemical aspects of protein corona:relevance to in vitro and in vivo biological impacts of nanoparticles[J]. Journal of the American Chemical Society,2011,133(8):2525-2534.
[16] Bhattacharya J,Choudhuri U,Siwach O,et al. Interaction of hemoglobin and copper nanoparticles:implications in hemoglobinopathy[J].Nanomedicine:Nanotechnology,Biology and Medicine,2006,2(3):191-199.
[17]Karlsson H,Ljunggren S,Ahrén M,et al.Two-dimensional gel electrophoresis and mass spectrometry in studies of nanoparticle-protein interactions[J].Gel Electrophoresis,2012,133(8):2525-2534.
[18]Lee I S,Lee N,Park J,et al.Ni/NiO core/shell nanoparticles for selective binding and magnetic separation of histidine-tagged proteins[J].Journal of the American Chemical Society,2006,128(33):10658-10659.
[19]Jamison J A,Bryant E L,Kadali S B,et al.Altering protein surface charge with chemical modification modulates protein-gold nanoparticle aggregation[J].Journal of Nanoparticle Research,2011,13(2):625-636.
[20]del Pino P,Pelaz B,Zhang Q,et al.Protein corona formation around nanoparticles-from the past to the future[J].Materials Horizons,2014,1(3):301-313.
[21]Hühn D,Kantner K,Geidel C,et al.Polymer-coated nanoparticles interacting with proteins and cells:focusing on the sign of the net charge[J]. ACS Nano,2013,7(4):3253-3263.
[22]Tessier P M,Jinkoji J,Cheng Y C,et al. Self-interaction nanoparticle spectroscopy:a nanoparticle-based protein interaction assay[J].Journal of the American Chemical Society,2008,130(10):3106-3112.
[23] Edri E,Regev O. pH effects on BSA-dispersed carbon nanotubes studied by spectroscopy-enhanced composition evaluation techniques[J]. Analytical Chemistry,2008,80(11):4049-4054.
[24] Belgorodsky B,Fadeev L,Ittah V,et al. Formation and characterization of stable human serum albumin-tris-malonic acid [C60] fullerene complex[J]. Bioconjugate Chemistry,2005,16(5):1058-1062.
[25] Zhu K,Kim J,Yoo C,et al. High sequence coverage of proteins isolated from liquid separations of breast cancer cells using capillary electrophoresis-time-of-flight MS and MALDI-TOF MS mapping[J]. Analytical Chemistry,2003,75(22):6209-6217.
[26] Webel R,Milbradt J,Auerochs S,et al. Two isoforms of the protein kinase pUL97 of human cytomegalovirus are differentially regulated in their nuclear translocation[J].Journal of General Virology,2011,92(3):638-649.
[27] Aggarwal P,Hall J B,McLeland C B,et al. Nanoparticle interaction with plasma proteins as it relates to particle biodistribution,biocompatibility and therapeutic efficacy[J]. Advanced Drug Delivery Reviews,2009,61(6):428-437.
[28]Kim H R,Andrieux K,Gil S,et al. Translocation of poly(ethylene glycol-co-hexadecyl)cyanoacrylate nanoparticles into rat brain endothelial cells:role of apolipoproteins in receptor-mediated endocytosis[J].Biomacromolecules,2007,8(3):793-799.
[29]Servagent-Noinville S,Revault M,Quiquampoix H,et al.Conformational changes of bovine serum albumin induced by adsorption on different clay surfaces:FTIR analysis[J]. Journal of Colloid and Interface Science,2000,221(2):273-283.
[30] Banerjee V,Das K P. Interaction of silver nanoparticles with proteins:A characteristic protein concentration dependent profile of SPR signal[J]. Colloids and Surfaces B:Biointerfaces,2013,111:71-79.
[31]Sane S U,Cramer S M,Przybycien T M.Protein structure perturbations on chromatographic surfaces[J]. Journal of Chromatography A,1999,849(1):149-159.
[32]Mu Q,Liu W,Xing Y,et al.Protein binding by functionalized multiwalled carbon nanotubes is governed by the surface chemistry of both parties and the nanotube diameter[J].The Journal of Physical Chemistry C,2008,112(9):3300-3307.
[33]Xie X,Wang Z,Zhou X,et al.Study on the interaction of phthalate esters to human serum albumin by steady-state and time-resolved fluorescence and circular dichroism spectroscopy[J]. Journal of Hazardous Materials,2011,192(3):1291-1298.
[34]Song L,Wang H,Wang S,et al.Memory effect of reversibly thermo switchable self-assembly-competent recombinant TMV coat protein with multi-binding moieties with potential applications in nanoparticle purification[J].Journal of Materials Science,2014,49(7):2693-2704.
[35] Li H,Wang M,Wang C,et al. Silver nanoparticle-enhanced fluorescence resonance energy transfer sensor for human platelet-derived growth factor-BB detection[J].Analytical Chemistry,2013,85(9):4492-4499.
[36] Kathiravan A,Renganathan R,Anandan S. Interaction of colloidal AgTiO2nanoparticles with bovine serum albumin[J].Polyhedron,2009,28(1):157-161.
[37] Shang L,Wang Y,Jiang J,et al. pH-dependent protein conformational changes in albumin:gold nanoparticle bioconjugates:a spectroscopic study[J]. Langmuir,2007,23(5):2714-2721.
[38]Xiao Q,Huang S,Qi Z D,et al. Conformation,thermodynamics and stoichiometry of HSA adsorbed to colloidal CdSe/ZnS quantum dots[J].Biochimicaet Biophysica Acta (BBA)-Proteins and Proteomics,2008,1784 (7):1020-1027.
[39]Whitmore L,Wallace B A.Protein secondary structure analyses from circular dichroism spectroscopy:methods and reference data bases[J].Biopolymers,2008,89(5):392-400.
[40] Su Z,Leung T,Honek J F. Conformational selectivity of peptides for single-walled carbon nanotubes[J].The Journal of Physical Chemistry B,2006,110 (47):23623-23627.
