盐穗木HcDmc1基因的克隆和表达分析
2015-07-04张富春
杜 驰,张富春
(新疆大学 生命科学与技术学院,新疆生物资源基因工程重点实验室,乌鲁木齐830046)
植物生长过程中会因为温度、紫外照射、高盐等外界胁迫因素而产生DNA 损伤,并诱导许多DNA损伤修复基因表达[1]。DNA 损伤有多种形式,其中最严重的DNA 损伤形式是DNA 双链断裂(doublestand break,DSB),DSB损伤及时修复会导致染色体断裂及细胞死亡[2]。DSB修复需要双链断裂修复基因的参与,而DNA 损伤修复基因Dmc1(disrupted meiotic cDNA)则发挥重要的作用。例如植物在外界胁迫下会出现减数分裂障碍,联会复合体相关基因表达发生变化时,联会复合体形成受阻[3],这与DNA 损伤修复基因dmc1突变体的表型相似[4-5]。
研究发现,损伤修复蛋白Dmc1定位于第一次减数分裂前期Ⅰ的偶线期,是同源染色体配对和交叉重组所必需的[6]。Dmc1聚合到断裂双链中的单链DNA 3′末端尾巴上,结合非断裂同源双链DNA,通过同源重组帮助同源染色体联会配对,参与双链断裂修复重组[7]。近年来,已从酵母、人、鼠、拟南芥等生物中克隆获得Dmc1基因,人与酵母Dmc1基因功能相似[8],Dmc1 缺失会阻止同系物之间的相互作用,dmc1突变体丧失重组功能;鼠Dmc1是减数分裂同源染色体联会所必需的[9];拟南芥dmc1突变体的同源染色体不能配对形成二价体[10]。这说明Dmc1是修复DNA 损伤断裂,维持双链正常复制、减数分裂的有序进行,抵御外界逆境胁迫,维持植物正常生长的重要调控因子。
盐穗木(Halostachyscaspica)隶属于藜科(Chenopodiaceae)盐穗木属(Halostachys),生长于干旱荒漠盐碱地,对盐分适应性极强,是新疆主要的盐土荒漠植物之一,其茎、叶发育成肉质化同化枝,以减少水分的散失,成为典型的耐盐植物[11-12]。根据实验室以RNA-seq测序技术进行的盐穗木盐胁迫响应的转录组测序[13]分析,发现在盐胁迫条件下,盐穗木同化枝和根中DNA 损伤应激通路中相关基因表达差异明显,提示DNA 损伤修复的相关基因可能与盐胁迫具有密切相关性。本研究从盐胁迫下转录组数据库中,筛选到了DNA 损伤修复基因HcDmc1,并对其进行克隆和生物信息学分析,探讨不同盐浓度胁迫下盐穗木DNA 损伤修复基因HcDmc1在不同时间点和不同组织的表达变化规律,为深入研究DNA 损伤修复基因HcDmc1在盐穗木的耐盐调控机理中的作用奠定基础。
1 材料和方法
1.1 实验材料
盐穗木种子采于五家渠103 团野外盐碱地(87.31°E,44.29°N)。播种于铺有基质(珍珠岩∶蛭石∶花土=1∶1∶3,用双子叶植物营养液拌湿)花盆中,在(24±2)℃,光照350μmol·m-2·s-1,昼/夜16h/8h,相对湿度为40%~60%条件下,并施以1/4 MS培养液培养120d左右。
1.2 盐穗木的盐胁迫处理
选取生长4个月左右的盐穗木幼苗,置于100、300、500和700mmol/L NaCl溶液进行处理,处理前48~72h在托盘中加自来水去除干旱胁迫因素。待托盘中自来水吸净无多余水分,将不同浓度NaCl溶液分别淋浇在基质上,直至溶液充满基质淋出于托盘。将花盆置于处理前的培养环境,分别于各处理浓度下0、24和72h,以及7 和14d采样。
1.3 总RNA 提取和cDNA 合成
称量NaCl各胁迫处理后的盐穗木根和同化枝各200 mg,分别用百泰克总RNA 提取试剂盒(RP3402,北京)提取总RNA,方法参照试剂盒说明书。用ND-2000 和15%变性PAGE 胶检测RNA浓度和质量,每份3个重复。以1μg总RNA 为模板进行逆转录(20μL 反应体系),以Takara Oligo(dT)作为引物用M-MLV 反转录酶合成cDNA。反应程序:反转录反应液30℃静置10min;移至42℃水浴锅孵育60 min;70℃热击15 min;冰浴2 min。以此cDNA 为模板进行PCR 扩增。
1.4 HcDmc 1扩增
根据本课题组前期对盐穗木转录组测序获得的盐穗木EST 序列,克隆并分析DNA 损伤修复基因(HcDmc1)开放阅读框。设计上游引物(5′-CTCGTGGAATTCTCTCTCT TTCTCTATCTC-3′)和下游引物(5′-CCAGTAACTAGCTACATAATGCACGGTACTC-3′),以无胁迫处理状态下的盐穗木cDNA 为模板,扩增HcDmc1开放阅读框。PCR 反应扩增参数为94℃预变性5min;94℃变性30s,58℃退火30s;72℃延伸90s,30个循环;72℃延伸10min。反应产物进行琼脂糖凝胶电泳检测。
按PCR 产物纯化试剂盒(Omega)说明书进行PCR 产物回收。回收的片段与pEASY-T5载体(北京全式金公司)连接,操作按试剂盒说明书进行。连接产物转化E.coliDH5α感受态细胞(北京全式金公司),酶切鉴定出阳性克隆后,送生工生物工程(上海)股份有限公司测序。
1.5 序列分析
利用DNASTAR 软件对已获得的HcDmc1的核酸序列进行分析并推导其氨基酸序列;利用Prot-Param(http://web.expasy.org/protparam/)分析氨基酸序列的理化性;利用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)分析蛋白质的跨膜结构;利用Protscale(http://web.expasy.org/protscale/)分析蛋白质亲疏水性;利用NCBI的保守结构域数据库CDD(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi?RID=1K7GNVX3015&mode=all)分析预测氨基酸序列的保守结构域;利用PSORT ⅡPrediction(http://psort.hgc.jp/form2.html)和WoLF PSORT(http://wolfpsort.org/)预测蛋白质的亚细胞定位;利用TargetP(http://www.cbs.dtu.dk/services/TargetP/)分析蛋白潜在信号肽;利用NetPhos(http://www.cbs.dtu.dk/services/NetPhos/)分析蛋白质磷酸化位点;利用MEGA 6.0 和DNAman软件分析其同源性及构建系统发育树。
1.6 盐胁迫下HcDmc 1基因表达
根据HcDmc1的cDNA 序列设计qRT-PCR上游引物(5′-ACTCCTCGGCAATCTTCATCAAA-3′)和下游引物(5′-GAACGGTTTGGGATGGATGCT-3′),以Hcβ-Actin基因作为内参基因,设计上游引物(5′-CCAAAGGCCAACAGAGAGAAGA-3′)和下游引物(5′-TGAGACACACC ATCACCAGA-3′)。以NaCl各胁迫后盐穗木的根和同化枝的cDNA为模板,取1.0μL cDNA,参照QIAGEN(Invitrogen公司)试剂盒说明书进行qRT-PCR,反应参数为:95℃预变性30s;95℃变性15s;58℃退火30s;72℃延伸40s(实时荧光信号采集);40个循环。采用ABI PRISM 7500实时定量PCR 仪进行检测,数据采用2-ΔΔCT法进行分析[14]。
2 结果与分析
2.1 盐穗木Dmc 1基因的克隆及序列分析

图1 HcDmc 1核酸序列及其氨基酸序列Fig.1 HcDmc 1nucleotide acid sequence and amino acid sequence
利用PCR 扩增获得盐穗木DNA 双链断裂铰链蛋白基因HcDmc1,开放阅读框为1 035bp,编码344个氨基酸(图1)。通过NCBI保守结构域数据库,分析HcDmc1的保守结构域,结果显示该蛋白含有2个超家族,H3TH(helix-3-turn-helix)和ABC-ATPase超家族。H 3TH超家族是一个带正电的DNA 活性位点,结合5′核酸酶,主要包含了FEN1(Flap Endonuclease-1)、EXO1(Exonuclease-1)、Mkt1、GEN1(Gap Endonuclease 1)、XPG(Xeroderma pigmentosum complementation group G)等核酸内切酶,参与DNA 复制、修复、重组。而ABC-ATPase超家族,有ATP 运输核苷酸的结合位点和核苷酸水解酶ABC 转运蛋白,包含2 个DNA结合域loop1 和loop2,2 个嘌呤核苷酸结合位点(motif A and B),说明HcDmc1可能具有通过这2个结构域发挥运输核苷酸参与DNA 损伤修复的功能。
图2显示,盐穗木Dmc1氨基酸序列与马铃薯、番茄、山柳菊、黄瓜等的氨基酸序列相似性较高。运用MEGA 6.0软件对18种植物的Dmc1氨基酸序列构建系统进化树(图2),结果表明盐穗木Dmc1为独立的一枝。
2.2 HcDmc1蛋白的结构特征、功能分析
通 过ExPASy ProtParam 预测HcDmc1基因编码的氨基酸序列的组成和理化性质。该基因编码344个氨基酸,其编码蛋白相对分子量为37 694.2 Da,理论等电点为5.46,酸性氨基酸残基总数(Asp+Glu)为40个,碱性氨基酸残基总数(Arg+Lys)为47个,原子总数是5 339,分子式为C1669H2693N455O509S13,疏水性平均值GRAVY((Grand average of hydropathicity)为-0.125,表明其为亲水性蛋白。该蛋白质不稳定指数是31.72,为稳定的蛋白。应用Psort、WoLF PSORT 两种工具预测该蛋白的亚细胞定位,该蛋白定位于细胞质、细胞核、线粒体、叶绿体、细胞骨架和过氧化物酶体。两种预测软件结果一致,推测该蛋白定位于细胞质的概率较高,同时线粒体、叶绿体中也有所分布。结合Signal P 4.0 Server软件对信号肽的预测结果显示,HcDmc1的N 端至第70位氨基酸之间信号肽分值和综合剪切分值D=0.101<D-cutoff=0.450,没有明显峰值,推测HcDmc1编码的蛋白不存在信号肽。通过Expasy软件中的TMHMM Server v.2.0工具预测,HcDmc1氨基酸序列中不存在跨膜螺旋区,344 个氨基酸残基全部在膜外,为非跨膜蛋白。
2.3 HcDmc1的磷酸化位点和活性位点的分析
磷酸化是生物体内一种普遍的调节方式,在细胞信号转导和蛋白调控过程中起着重要的作用。磷酸化位点分析表明,HcDmc1有5个丝氨酸、3个苏氨酸、3个酪氨酸磷酸化位点。利用NPS(Network Protein Sequence Analysis)的Prosite Scan对Hc-Dmc1进行活性位点分析,结果(表1)显示,HcDmc1 有7 类活性位点,分别是N-糖基化位点、cAMP-和cGMP-依赖性蛋白激酶磷酸化位点、蛋白酶C磷酸化位点、酪蛋白酶Ⅱ磷酸化位点、酪氨酸激酶磷酸化位点、N-十四酰化位点、酰胺化位点、ATP/GTP-结合位点。这是HcDmc 1发挥其催化作用所必须的。
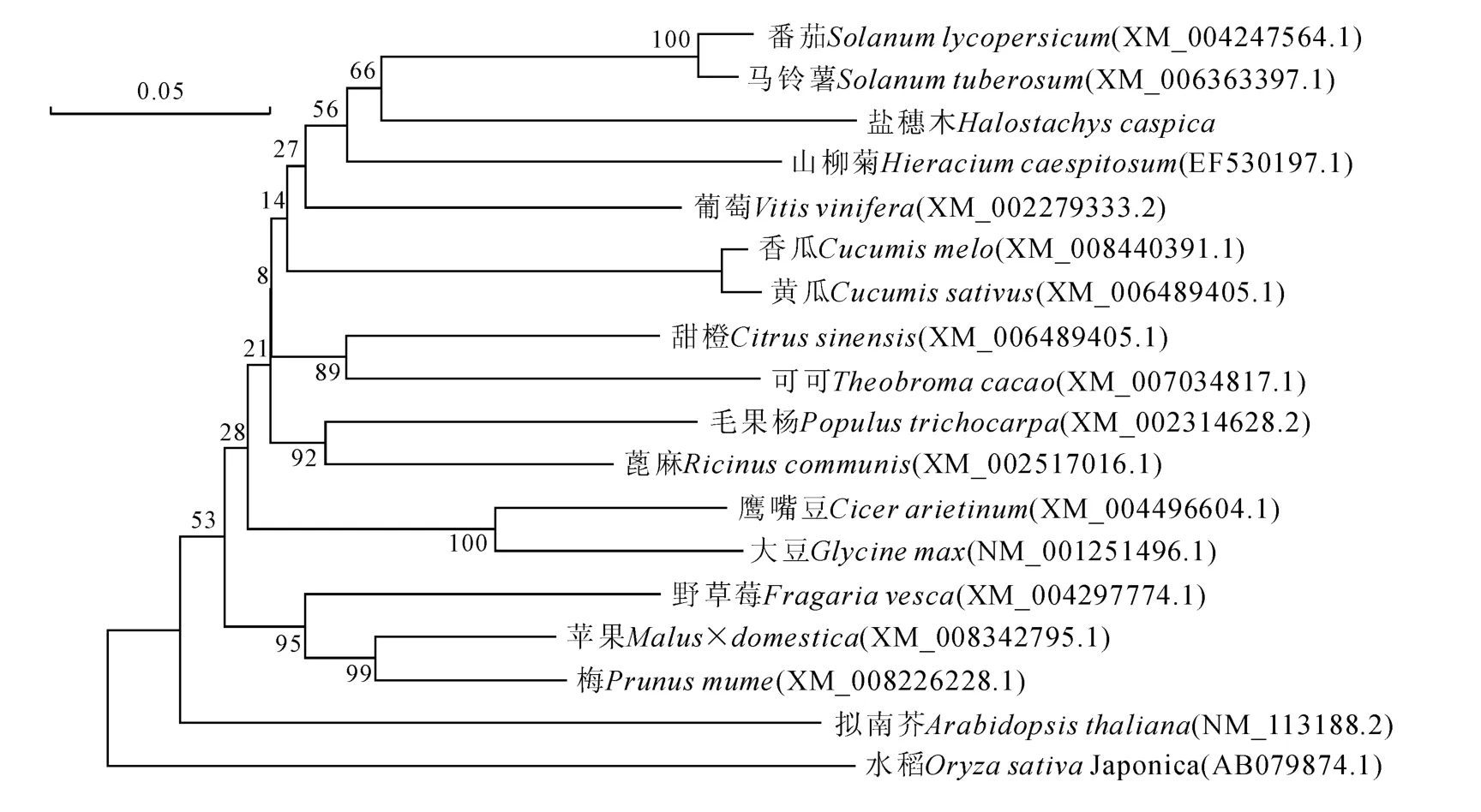
图2 不同植物基于Dmc1氨基酸序列的系统进化树Fig.2 Phylogenetic tree of different plants based on amino acid of Dmc1
2.4 盐胁迫下HcDmc 1基因的表达分析
采用qRT-PCR方法,以β-actin作为内参,分别检测不同浓度盐胁迫下盐穗木根和同化枝HcDmc1转录水平变化(图3)。首先以HcDmc1和Hcβ-Actin实时定量引物建立标准曲线,HcDmc1熔解曲线所得的TM 值为(82.77±0.1)℃,峰值单一,没有引物二聚体和非特异性条带。在8个10n倍系列稀释条件下得到的标准曲线方程为y=-3.295x+29.772,相关系数(R2)为0.995,扩增效率(Efficiency)为101.132。经过扩增条件、程序的优化,HcDmc1扩增效率达到了相对定量公式2-△△CT 的使用标准,进而继续进行相对定量实验。
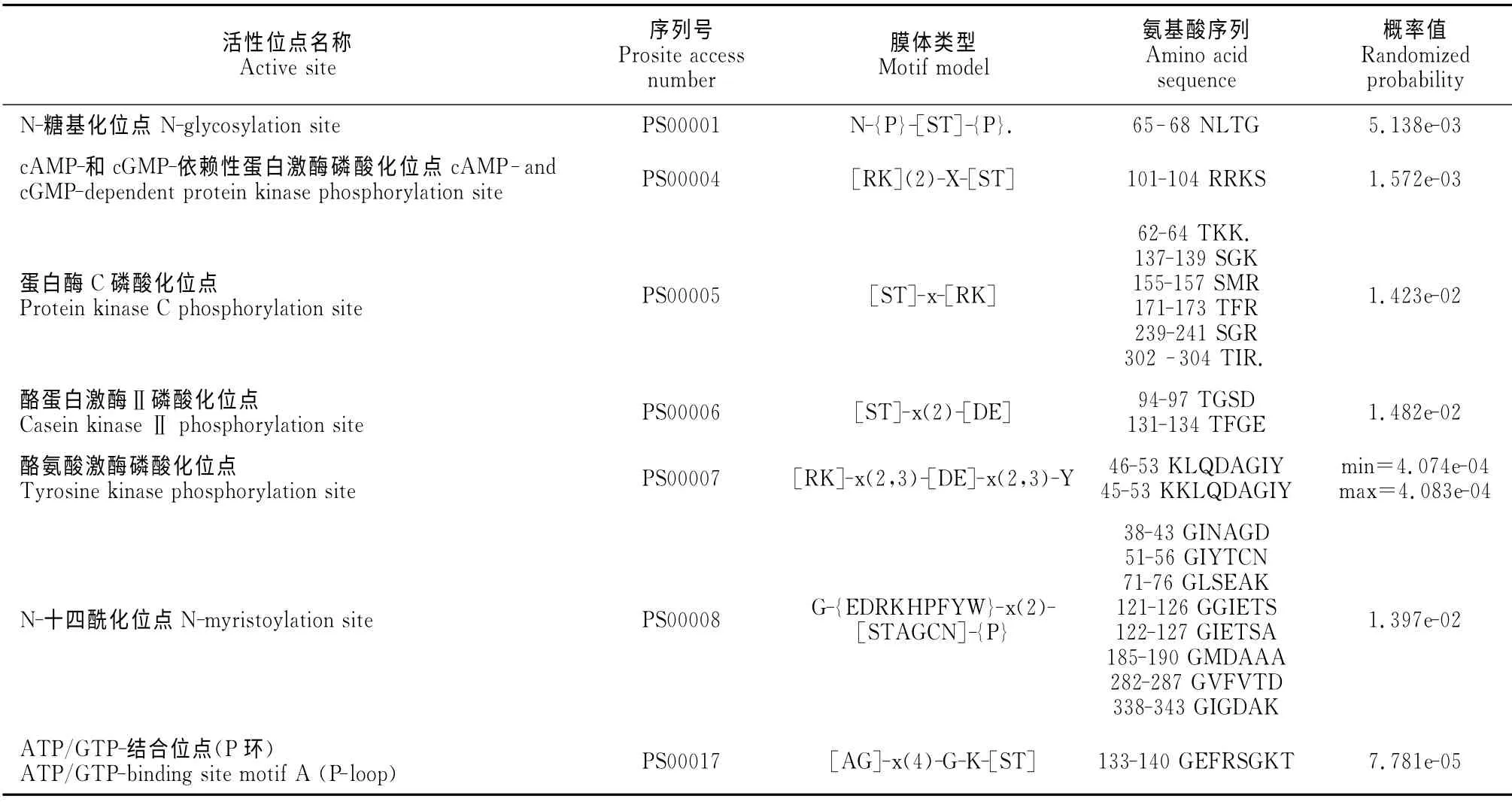
表1 HcDmc1在NPS中的活性位点预测分析Table 1 Scanning of HcDmc1for site/signatures against proscan database in NPS
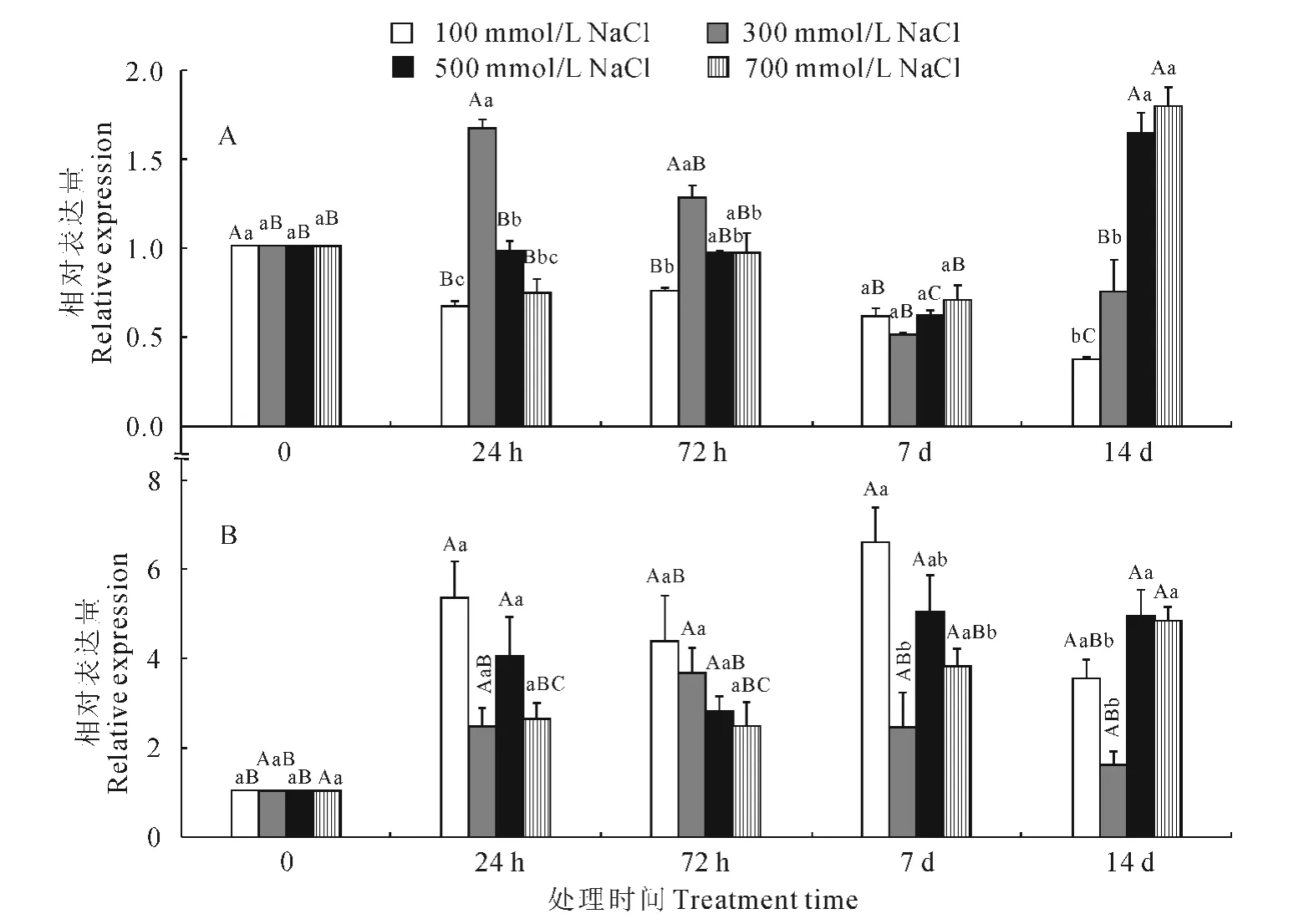
图3 盐穗木根(A)和同化枝(B)中HcDmc1相对表达Fig.3 Relative expression of HcDmc1response to salt-stress in root(A)and branch(B)
相对定量计算结果表明,300mmol/L NaCl胁迫处理后,HcDmc1 在根中的表达明显上调。在100mmol/L NaCl胁迫下,根中HcDmc1表达无明显变化,但在300mmol/L NaCl胁迫24h后表达显著上升,约为处理0h的盐穗木组织的1.66倍,在500和700mmol/L NaCl胁迫下,均为胁迫14d达到最高,约为0h的1.63和1.79倍(图3,A)。
而同化枝中HcDmc1的表达量在100mmol/L NaCl胁迫24h后开始剧增,在胁迫7d时达到最高,约为0h 的6.58 倍(图3,B)。在500 和700 mmol/L NaCl胁迫下,表达略有下降,但仍然维持在较高水平。
3 讨 论
减数分裂是植物有性生殖的重要过程,也是遗传信息传递的基本方式。正常状况下,第一次减数分裂前期的粗线期的染色体是二价体形态,然而拟南芥dmc1突变体中染色体以单体状态存在,说明AtDmc1基因对减数分裂交叉结的形成是必需的,维持染色体正常配对和联会[15]。此外,Dmc1蛋白作为大肠杆菌RecA 蛋白的同源蛋白[16],能够聚合到断裂的单链DNA 上,形成核蛋白纤维[17],通过非同源末端连接途径参与联会复合体配对,重组修复断裂的双链[18]。
对Dmc1基因编码氨基酸序列分析发现其在植物中具有较高的相似性(80% 以上),说明植物Dmc1的氨基酸序列在进化上是高度保守的,其编码的氨基酸序列长度和顺序变异较小,其特定基序对于Dmc1蛋白发挥活性结合双链、单链DNA 具有重要作用,尤其是基序GEFRSGKT 是ATPbinding结合位点,广泛存在于Dmc1及其同源蛋白Rad51和DNA 重组修复蛋白RecA、RadA 中[19]。对HcDmc1 磷酸化位点和活性位点的分析可知蛋白激酶可能参与了HcDmc1活性功能调节,而蛋白激酶是植物抵御逆境胁迫信号传导链的重要组分,HcDmc1在减数分裂中的作用可能受到了特定信号调节,并参与到植物抗逆途径中。对HcDmc1蛋白进行信号肽预测、跨膜结构域预测、疏水性/亲水性分析和亚细胞定位都显示该蛋白主要存在于细胞质,不与细胞膜结合,是稳定的亲水性蛋白,这与动物中Dmc1基因序列的分析结果相同[20]。
已有研究表明,植物在外界非生物胁迫下,DNA 会造成不同程度损伤变异,甚至断裂,同源重组和联会复合体是影响减数分裂的发生和配子的形成的主要因素[21-23]。Deng[24]对水稻的Dmc1基因进行RNAi干扰之后发现其具有同源配对缺陷,不能形成二价体,同时出现花粉不育现象,McHale等[25]研究发现,在两种显著不同温度环境中,相同基因型的马铃薯(Solanumtuberosum)产生未减数配子的频率相差很大。这说明,DNA 损伤修复基因Dmc1在逆境下具有保持染色体完整,减数分裂中染色体正常配对,同时修复断裂的双链的功能。本研究发现,盐穗木减数分裂同源重组蛋白基因Hc Dmc1受盐胁迫诱导表达,尤其是在其繁殖器官同化枝中表达量显著升高,推测其可能是盐穗木通过高表达HcDmc1蛋白,对高盐胁迫造成的断裂缺口的DNA 单链进行同源依赖性修复,以缓冲重组而导致的新型遗传表现,提高盐穗木盐胁迫环境适应性和生殖生长能力。
目前关于Dmc1基因表达模式研究的较少,本研究与拟南芥Dmc1和水稻Dmc1基因的表达分析结果相似,表现为在生殖器官中表达活性最高,在叶和根中表达量略低[26]。通过对HcDmc1在盐穗木盐胁迫下不同组织表达定量分析,发现随着盐浓度和胁迫时间的增加,盐穗木根中表达量升高,暗示HcDmc1在根中可能后期响应盐胁迫。而在同化枝中的响应明显提前于根,说明盐穗木同化枝中Hc-Dmc1对盐胁迫更为敏感,并在长时间高盐胁迫下维持较高的表达水平,以保证繁殖器官减数分裂、复制等生命周期的正确性,进而确保盐胁迫下物种的稳定遗传。
本研究对从盐穗木中克隆获得的HcDmc1 基因进行了生物信息学分析,探讨了HcDmc1响应盐胁迫基因的表达变化规律,证明了HcDmc1能够参与盐穗木盐胁迫应答过程。而HcDmc1 在盐穗木缓解盐胁迫过程中如何形成联会复合体和参与减数分裂重组,有待深入研究。盐穗木HcDmc1基因的克隆和盐胁迫下不同组织的表达分析,有助于阐明DNA 损伤修复在盐穗木耐盐调控机理中的作用。
[1]ZHU J K.Plant salt tolerance[J].TrendsinPlantScience,2001,6(2):66-71.
[2]BOYKO A,FILKOWSKI J,KOVALCHUK I,etal.Homologous recombination in plants is temperature and day-length dependent[J].MutationResearch,2005,572(1-2):73-83.
[3]ZHANG Q B,LI Q F,LI J H,etal.b-DAZL:A novel gene in bovine spermatogenesis[J].ProgressinNaturalScience,2008,18(10):1 209-1 218.
[4]COUTEAU F,BELZILE F,HORLOW C,etal.Random chromosome segregation without meiotic arrest in both male and female meiocytes of a dmc1mutant ofArabidopsis[J].PlantCell,1999,11(9):1 623-1 634.
[5]DENG Z Y,WANG T.OsDMC1is required for homologous pairing inOryzasativa[J].PlantMolecularBiology,2007,65(1-2):31-42.
[6]BISHOP D K.RecA homologs Dmc1and Rad51interact to form multiple nuclear complexes prior to meiotic chromosome synapsis[J].Cell,1994,79(6):1 081-1 092.
[7]LI J,HARPER LC,GOLUBOVSKAYA I,etal.Functional analysis of maize RAD51in meiosis and Double-Strand Break repair[J].Genetics,2007,176(3):1 469-1 482.
[8]SATO S,SEKI N,HOTTA Y,etal.Expression profiles of a human gene identified as a structural homologue of meiosis-specific recA-like genes[J].DNAResearch,1995,2(4):183-186.
[9]KAGAWA W,KURUMIZAKA H.From meiosis to postmeiotic events:uncovering the molecular roles of the meiosis-specific recombinase Dmc1[J].FEBSJournal,2010,277(3):590-598.
[10]KLIMYUK VI,JONES JD G.AtDMC1,theArabidopsishomologue of the yeast DMC1gene:characterization,transposon-induced allelic variation and meiosis-associated expression[J].ThePlantJournal,1997,11(1):1-4.
[11]ZHANG L X(张立新),LI SH X(李生秀).Research progress on relationships betaine and drought/salt resistance of plants[J].ActaBot.Boreal.-Occident.Sin.(西北植物学报),2004,24(9):1 765-1 771(in Chinese).
[12]CHANG D(常 丹),ZHANG X(张 霞),ZHANG F CH(张富春).Cloning and expression analysis ofHcPEAMTgene fromHalostachyscaspica[J].ActaBot.Boreal.-Occident.Sin.(西北植物学报),2014,34(8):1 522-1 528(in Chinese).
[13]ZHAO H(赵 航),JIA F Q(贾富强),ZHANG F CH(张富春),etal.The transcriptome information analysis of differentially expressed genes ofHalostachyscaspicaundersalt stress[J].ChineseJournalofBioinformatics(生物信息学),2014,12(2):90-98(in Chinese).
[14]LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J].Methods,2001,25(4):402-408.
[15]YOSHIDA K,KONDOH G,MATSUDA Y,etal.The mouse RecA-like gene Dmc1is required for homologous chromosome synapsis during meiosis[J].MolecularCell,1998,1(5):707-718.
[16]LI Z F,GOLUB E I,GUPTA R,etal.Recombination activities of HsDmc1protein,the meiotic human homolog of RecA protein[J].PNAS,1997,94(21):11 221-11 226.
[17]MATSUDA Y,HABU T,HORI T,etal.Chromosome mapping of the mouse homologue of DMC1,the yeast meiosis-specific homologous recombination gene[J].ChromosomeResearch,1996,4(3):249-250.
[18]PITTMAN D L,COBB J,SCHIMENTI K J,etal.Meiotic prophase arrest with failure of chromosome synapses in mice deficient for Dmc1,agermline-specific RecA homolog[J].MolecularCell,1998,1(5):697-705.
[19]STORY R M,WEBER I T,STEITZ T A.The structure of theE.coliRecA protein monomer and polermer[J].Nature,1992,355(6 358):318-325.
[20]LI X(李 贤),LI Q F(李齐发),ZHAO X B(赵兴波)etal.Sequence analysis and study on the expression level of Dmc1mRNA in yak and cattle-yak testis[J].ScientiaAgriculturaSinica(中国农业科学),2010,43(15):3 221-3 229(in Chinese).
[21]KLECKNER N.Meiosis:how could it work[J].PNAS,1996,93(16):8 167-8 174.
[22]ZICKIER D,KLECKNER N.The leptotene-zygolene transistion of meiosis[J].AnnualReviewofGenetics,1998,32(1):619-697.
[23]CHEN Z J.Genetic and epigenetic mechanisms for gene expression and phenotypic variation in plant polyploids[J].AnnualReviewOf PlantBiology,2007,58:377-406.
[24]DENG Z Y,WANG T.OsDMC1is required for homologouspairing inOryzasativa[J].PlantMolecularBiology,2007,65(1-2):31-42.
[25]SOLTIS D E,SOLTIS P S.Polyploidy:recurrent formation and genome evolution[J].TrendsEcologyEvolution,1999,14(9):348-352.
[26]DING Z J,WANG T,CHONG K,etal.Isolation and characterization of OsDMC1,the rice homologue of the yeast DMC1gene essential for meiosis[J].SexualPlantReproduction,2001,13(5):285-288.
