西藏牦牛mtDNA ATP8全序列测定及遗传多样性分析
2015-07-01柴志欣姬秋梅张成福信金伟宋乔乔钟金城
杨 琴, 柴志欣, 姬秋梅, 张成福, 信金伟, 宋乔乔, 钟金城
(1.西南民族大学,四川 成都 610041;2.西藏自治区农牧科学院畜牧兽医研究所,西藏 拉萨 850000)
西藏牦牛mtDNAATP8全序列测定及遗传多样性分析
杨 琴1, 柴志欣1, 姬秋梅2, 张成福2, 信金伟2, 宋乔乔1, 钟金城1
(1.西南民族大学,四川 成都 610041;2.西藏自治区农牧科学院畜牧兽医研究所,西藏 拉萨 850000)
对西藏16 个牦牛类群共367 头个体的mtDNAATP8(Adenosine Triphosphate 8)基因进行克隆及序列分析。结果表明,西藏牦牛mtDNAATP8基因全长201~203 bp,T、C、A和G 4种核苷酸的平均比例分别为29.3%、23.0%、41.8%和6.0%,A+T含量明显高于G+C,表现出一定的碱基偏倚性;在367 头牦牛中,共检测到19 个变异位点,其中单一信息位点15 个,简约信息位点4 个,存在转换和插入2 种变异类型,碱基替换中存在转换73 次,以A/G、T/C为主,占98.63%;在插入变异类型中以A碱基插入为主;367 头牦牛共捡出20 种单倍型,单倍型多样性和核苷酸多样性指数分别为0.332和0.001 89,说明西藏牦牛具有较贫乏的遗传多样性;聚类分析显示,西藏牦牛可分为2 类,其中桑日牦牛、类乌齐牦牛和桑日牦牛为1 类,其余牦牛类群为另1 类。20 种单倍型可以分为2 个聚类簇(I和Ⅱ),其中聚类簇I包含17 种单倍型,占全部单倍型数的77.27%,包含了本次研究中的所有西藏牦牛类群;聚类簇Ⅱ中有3 种单倍型,囊括了除错那、嘉黎、康布和帕里类群外的12 个类群,显示西藏牦牛存在2 个母系起源。
西藏牦牛;线粒体DNA;58基因;遗传多样性
拥有“高原之舟”美称的牦牛(Bosgrunniens)是分布于海拔3 000 m的青藏高原及其毗邻的高山、亚高山地区的珍稀牛种,在长期的自然选择和人工选择作用下,不管是其形态结构还是生理功能都能很好地适应高寒、低氧以及强紫外辐射的环境。全世界现有牦牛1 420余万头,其中中国有1 330多万头,占牦牛总量的93.67%;西藏作为牦牛的主产区之一,现有牦牛492.3 万头,占中国牦牛总数的37%,位居全国第2[1,2]。牦牛能够为牧民提供肉、奶等生产、生活必需品,是当地畜牧业经济中不可缺少的畜种,在遗传上是一个极为宝贵的基因库。遗传多样性是生物多样性的重要组成部分,是生态多样性和物种多样性的基础。动物线粒体DNA(mitochondrial DNA, mtDNA)因具有母系遗传、多态性丰富、结构简单等特点,被认为是研究物种起源、演化和分类最好的分子遗传标记[3]。国内学者张成福、赵上娟、姬秋梅和姜雪鸥等[4-7]通过测定西藏11个牦牛类群的D-loop区、COⅢ、Cytb、12SrRNA基因的核苷酸序列对西藏牦牛的遗传多样性、类群间的亲缘关系和系统进化关系进行了研究。但利用ATP8基因对西藏牦牛的遗传多样性及其类群间的系统进化关系研究尚属空白。哺乳动物编码ATP合成酶F0亚基部分结构的ATP8基因位于线粒体基因组上,是线粒体内膜呼吸链基因的重要成员之一[8,9]。本研究以西藏16个牦牛类群为研究对象,通过测定mtDNAATP8全基因序列,探讨牦牛遗传多样性及其类群间的亲缘关系,以期为进一步的保护牦牛遗传资源提供理论依据。
1 材料与方法
1.1 样品采集
从16 个西藏牦牛类群中,选取健康成年的牦牛367头(表1),采集其耳组织,用体积分数75%的乙醇保存带回实验室,存于-20 ℃的冰箱中备用。
1.2 基因组DNA的提取及检测
运用动物组织基因组提取试剂盒(TianGen生物技术公司)提取基因组DNA,用1%的琼脂糖凝胶电泳和紫外分光光度计分别检测DNA的纯度和浓度,-20 ℃保存备用。
1.3 引物设计与序列扩增
根据GenBank中公布的牦牛ATPase8基因的核苷酸序列(GenBank:AY684273.2),用Primer Premier 5.0设计1对引物,引物序列为:F:GTTAGAGATTGAGAGCAGTATGCT;R:AAACCACATGGCTAAGCCGG,由英潍捷基(上海)生物科技有限公司合成。
PCR反应体系为25 μL;其中,上、下游引物各1 μL,DNA 模板1 μL,2×long Taq DNA预混酶12.50 μL(0.625 U),无菌ddH2O 9.50 μL。
PCR扩增条件:94 ℃预变性2 min;94 ℃变性15 s,55 ℃退火15 s,72 ℃延伸30 s,30个循环;72 ℃延伸3 min,4 ℃保存。
1.4 目的基因克隆测序
扩增产物用1%琼脂糖凝胶电泳检测目的片段大小,然后用DNA胶回收试剂盒(TianGen)进行分离纯化,再连接到pMDTM19-T载体(TaKaRa公司)上,并转化到高效感受态细胞DH5α中,LB平板培养基(含有2%X-Gal,1%IPTG和1%Amp)上37 ℃培养12~16 h,筛选阳性克隆在700 μL LB液体培养基(含1%Amp)中37 ℃振荡培养6~7 h。重组子经菌液PCR鉴定后,用ABI3730XL型DNA测序仪进行正反双向测序。
1.5 数据处理
用DNAMAN 5.2.2软件对所测序列进行拼接,比对并进行人工校对;用MEGA5.05软件统计序列的长度、碱基组成、氨基酸组成,以及计算类群间的Kimura双参数距离并以邻接法(Neighbor-Joining,NJ) 构建品种间聚类关系;用DnaSP5.10软件进行变异位点、核苷酸多样性以及单倍型多样性分析;用Network4.6.1.1(http://www. fluxus-engineering.com/sharenet.htm)构建单倍型网络关系图。
2 结果与分析
2.1 西藏牦牛mtDNAATP8基因的核苷酸和氨基酸组成
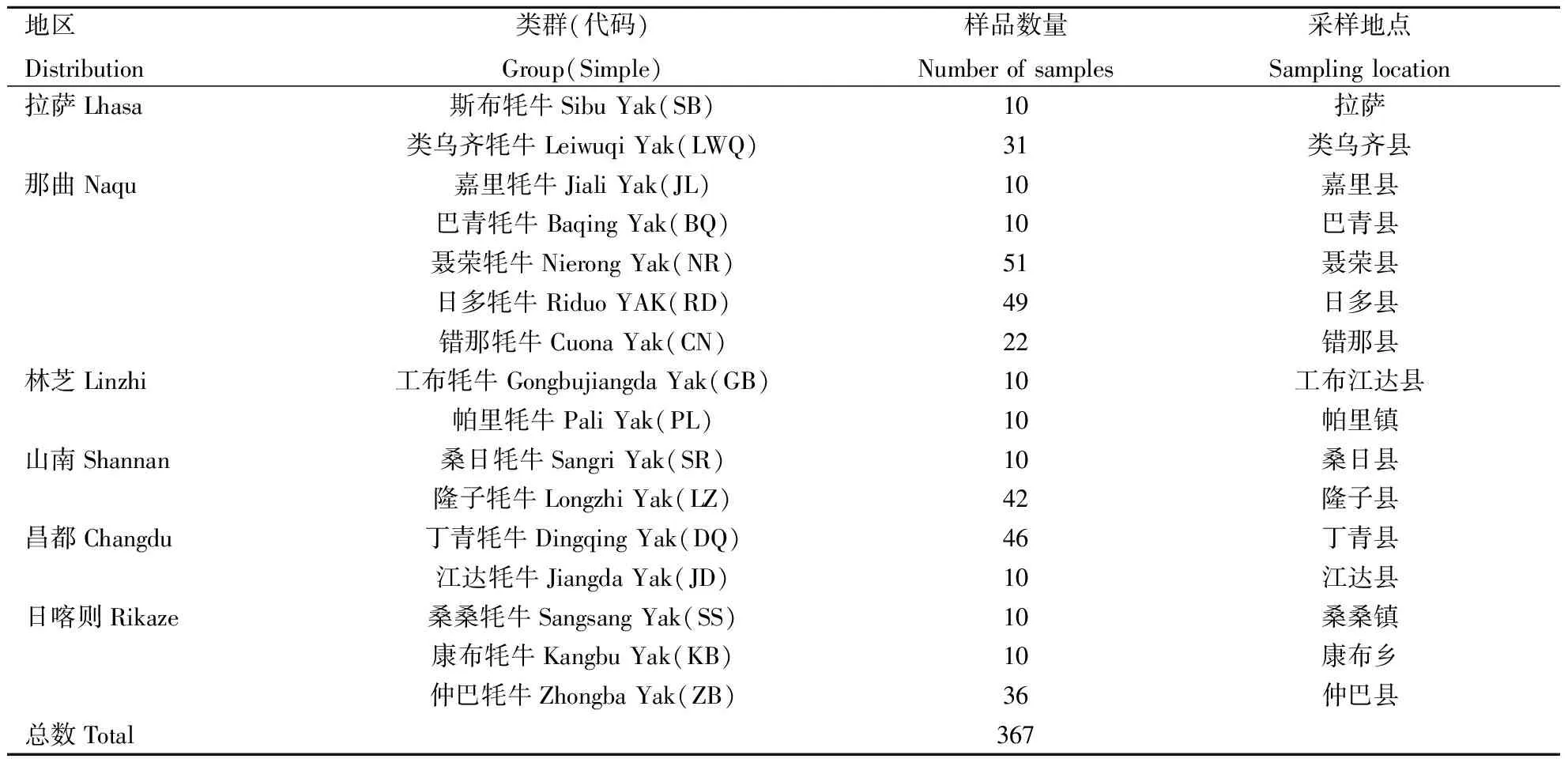
表1 供试牦牛类群、数量及采样地点
经测序得到西藏16 个牦牛类群367 个个体的ATP8基因全序列,基因全长201~203 bp,起始密码子为AUG(ATG),终止密码子为UAA(TAA),编码66个氨基酸。T,C,A和G 4种核苷酸的平均比例分别为29.3%、23.0%、41.8%和6.0%;A+T 含量71.1%,G+C 含量29.0%,表明西藏牦牛ATP8基因具有明显的碱基偏倚性,这与线粒体蛋白编码基因的典型特征相符。根据脊椎动物线粒体中密码子编码规律[10],统计发现:ATP8基因所编码各氨基酸平均含量差异较大,不含半胱氨酸(Cys)、精氨酸(Arg)和缬氨酸(Val),而亮氨酸和丝氨酸的平均含量较高,分别为15.64%和11.31%。
2.2 西藏牦牛ATP8基因的遗传多样性
在367 头牦牛中,全序列共有19 个可变位点,其中单一信息位点(Singleton variable sites)15 个,简约信息位点(Parsimony informative sites)4 个。所测序列与下载的参考序列(GenBank:AY684273.2)进行比对,发现存在转换和插入2 种变异类型,其中转换74 次,以A->G,T->C为主,占98.64%;存在4 个插入位点(SS5:2223;DQ38:30

表2 西藏牦牛ATP8基因单倍型多样性和核苷酸多样性
根据西藏牦牛ATP8全序列19 个变异位点,从367 条序列中共捡出20 种单倍型(表3),其中单倍型1和单倍型2所占比例最多,分别达73%和13%以上。西藏牦牛总体的单倍型多样性为0.332(表2),各类群的平均单倍型多样性值在0.000~0.711,其中桑桑牦牛的单倍型多样性最高。在西藏牦牛ATP8单倍型序列变异位点的基础上,运用NetWork4.6.1.1构建单倍型网络关系图(图1)。结果显示,20 种单倍型被分为2 个聚类簇(I和II),其中聚类簇I包含17 种单倍型,占全部单倍型的90%,包含了本研究中的所有西藏牦牛类群;聚类簇II中有3种单倍型,囊括了除错那、嘉黎、康布和帕里类群外的12 个类群。
2.3 西藏牦牛ATP8基因单倍型序列同源性比较分析
运用DNAMAN5.2.2软件对西藏牦牛ATP8基因20 种单倍型序列进行同源性比较,其结果如表4所示。同源性范围值98.48%~99.50%,单倍型Hap-1序列与其他单倍型序列相比,其与单倍型Hap-2和单倍型Hap-11、Hap-13的同源性要明显低于其他序列;而单倍型Hap-2序列与Hap-1和Hap-11、Hap-13的同源性明显高于其他序列,这说明单倍型Hap-11、Hap-13是在单倍型Hap-2的基础上演变来的,而Hap-2又起源于单倍型Hap-1。
2.4 西藏牦牛类群间的遗传距离及聚类分析
运用Mega软件对西藏16 个牦牛类群的分子遗传距离进行计算(表5),表明西藏16 个牦牛类群间的遗传距离变异范围在0.0000~0.0034,其中康布(KB)、嘉黎(JL)和帕里(PL)之间任意2 个的遗传距离均为0.0000,桑日(SR)、类乌齐(LWQ)和桑桑(SS)牦牛与其他类群间的平均遗传距离较其他类群间的距离大,其中桑日牦牛与其他类群间的遗传距离最远,可作为选育的优先考虑对象。根据ATP6基因的遗传距离,用邻接法(NJ)对西藏牦牛16 个类群进行聚类分析(图2),结果显示桑日、类乌齐和桑桑牦牛为一类,其他牦牛类群则为另一类。
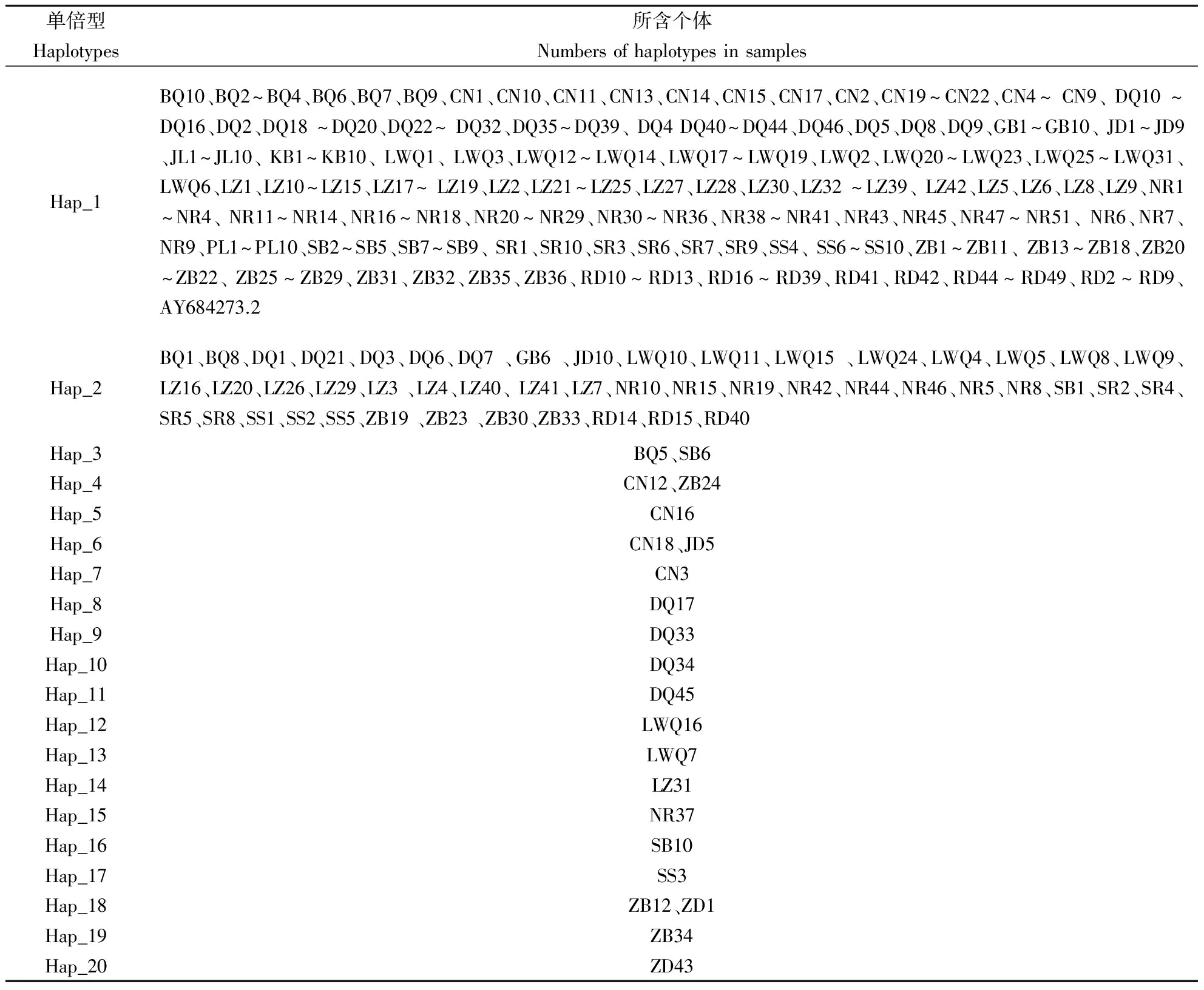
表3 西藏牦牛16个类群ATP8基因的单倍型分布
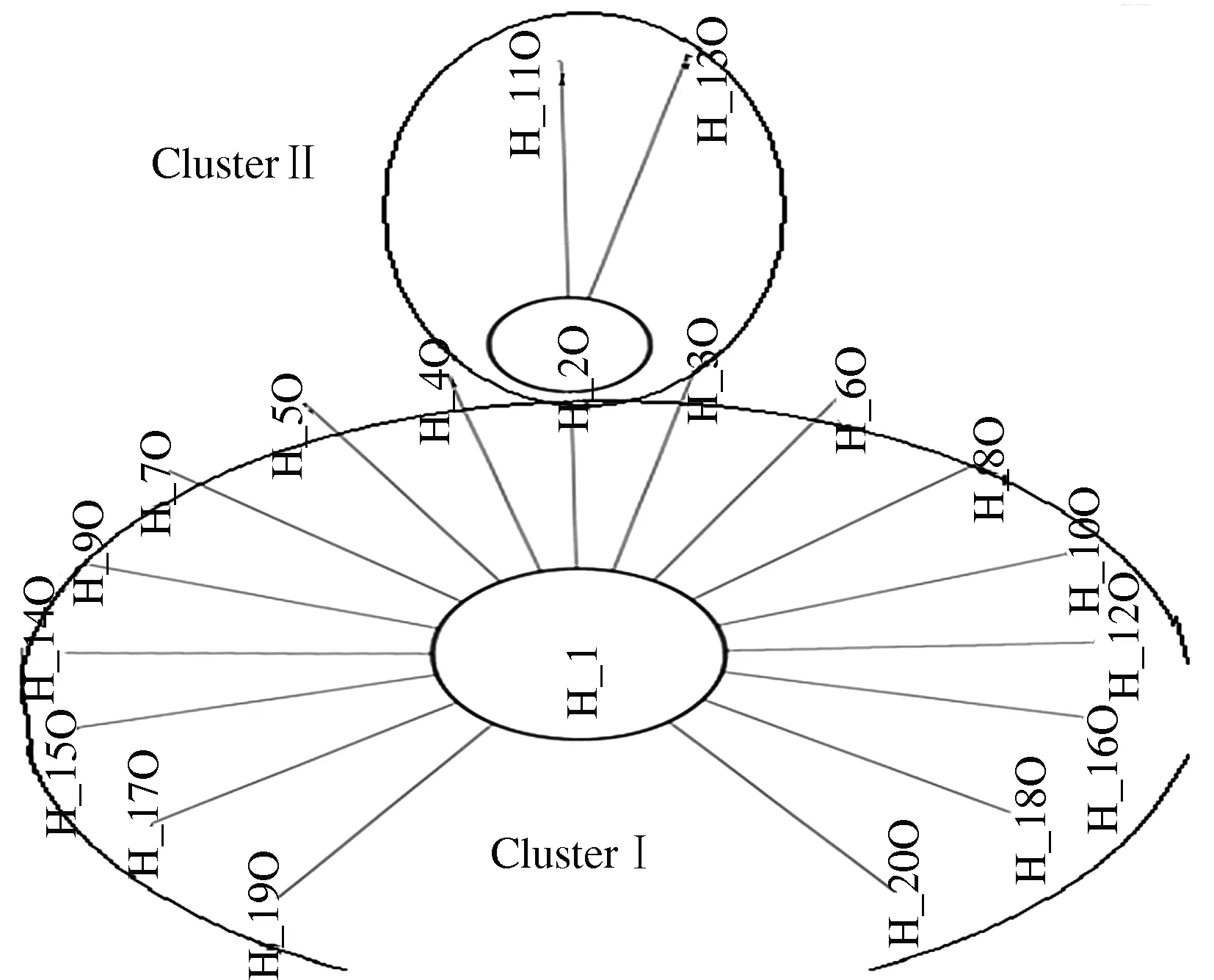
注:每个圆圈代表一种单倍型,其大小与该种单倍型出现的频率成正比。
Note: Each circle represents one kind of haploid type, its size and the frequency is proportional to the haploid form.
图1 西藏牦牛mtDNAATP8基因20种单倍型网络关系图
Fig.1 Networt relationship chart of 22 haplotypes of mtDNAATP8 in Tibetan yak
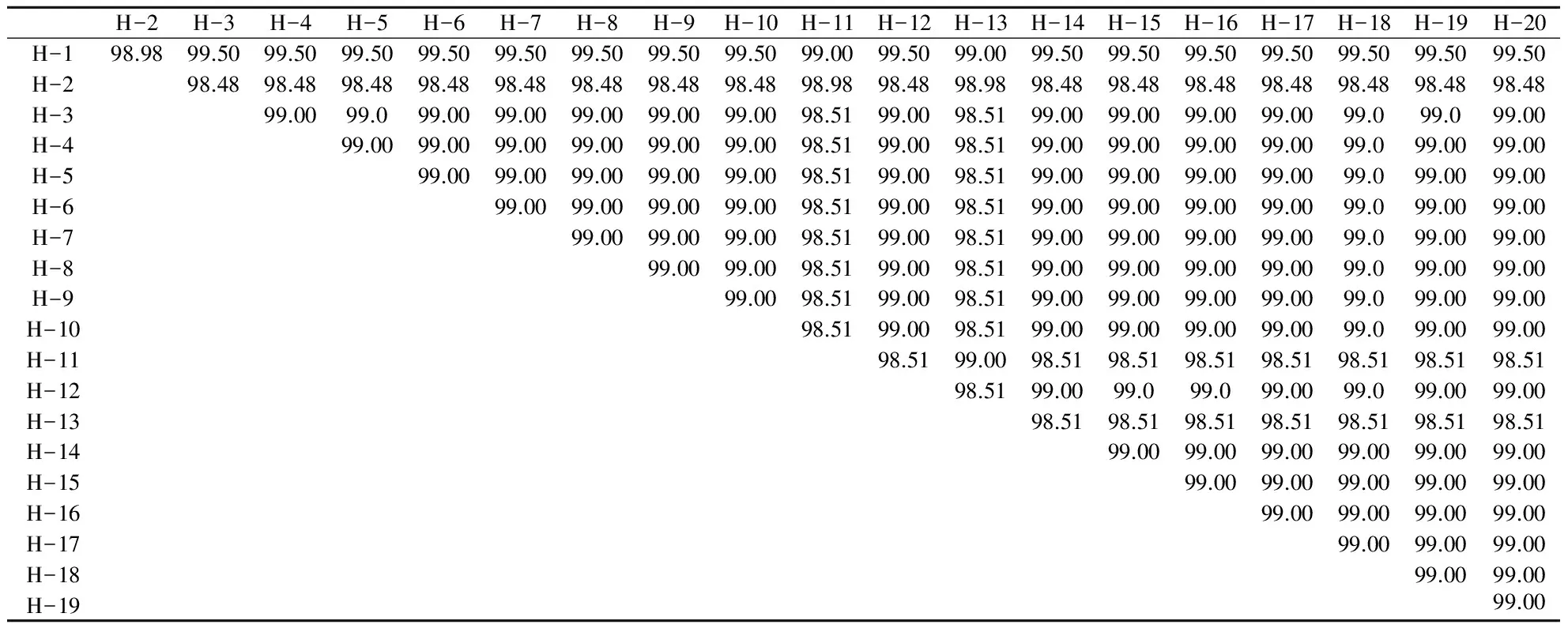
表4 西藏牦牛ATP8基因20种单倍型序列的同源性比较
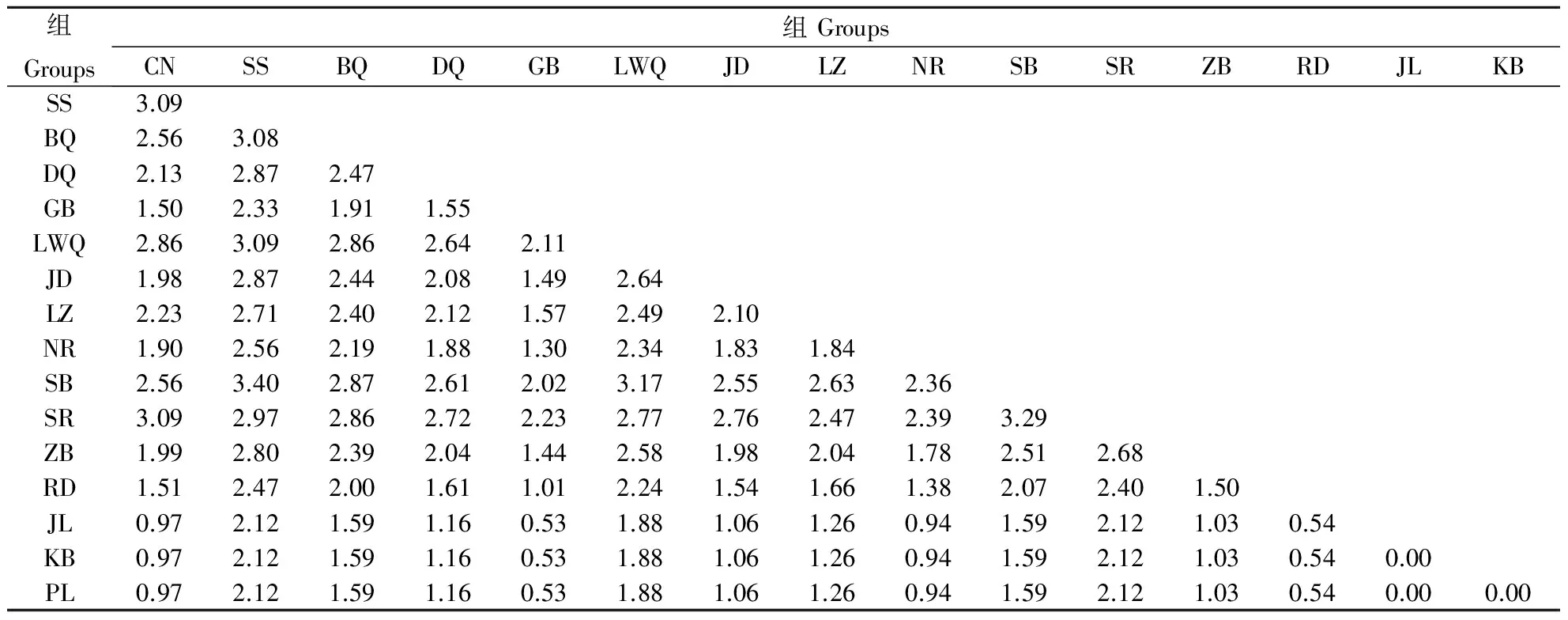
表5 西藏牦牛16个类群间的双参数遗传距离
注:表中数据均要乘以10-3。
Note: Data in the table have to be multiplied by 10-3.

图2 基于Kimura 双参数距离的西藏牦牛类群间的NJ 聚类关系
3 结论与讨论
3.1 西藏牦牛ATP8基因单倍型及核苷酸多样性分析
由单倍型分析可知,不仅73%的个体属于单倍型Hap-1,而且每个群体中都含有单倍型Hap-1,由此推断单倍型Hap-1应是西藏牦牛类群的主要单倍型,其他单倍型是在此基础上演变而来,这与图1所表示的结果一致。
单倍型多样性和核苷酸多样性常作为衡量群体DNA水平上遗传多样性的重要指标,其值越小,群体的多态程度就越低,遗传多样性就越贫乏。动物的遗传多样性是动物持续进化和适应外界环境变化的物质基础,群体的遗传多样性越丰富,则其适应环境变化的能力就越强;反之,不稳定的外界环境将成为群体灭绝的最大威胁,因此,对西藏牦牛类群之间的进化关系和遗传多样性分析,不仅有利于保持各品种的优势基因,还对牦牛资源的开发和管理有着十分重要的现实意义。对平均核苷酸的多样性分析发现,桑桑牦牛在16 个类群中相对较高,其单倍型多样性达到0.711,说明类乌齐牦牛的遗传多样性较其他类群丰富;而帕里牦牛、康布牦牛和嘉黎牦牛无论是核苷酸多样性还是单倍型多样性都为零,说明以上3 种牦牛类群是很纯的类群。同时发现,西藏16 个牦牛类群的总体单倍型多样性(Hd)和核苷酸多样性(Pi)分别为0.332和0.001 89,位于被认为线粒体多态低范围(0.15%~0.47%)[11]。由此可见,西藏牦牛总类群线粒体遗传多样性较为贫乏。这一结果较张成福等[4,12,13]对D-loop区,姬秋梅等[5]对cytb基因,及赵上娟等[6]对COIII基因和姜雪欧等[7]对12SrRNA的研究结果均要低;其核苷酸多样性与D-loop区和12SrRNA相比较低,却稍高于COIII和cytb,这表明,西藏牦牛具有较贫乏的遗传多样性。
3.2 西藏牦牛类群的系统进化关系
近年来,西藏牦牛的遗传多样性、系统分化以及品种的分类一直是研究的热点。赵上娟等[6]研究认为,西藏11 个牦牛类群(111头)可分为三大类,即帕里牦牛系、巴青牦牛系、斯布牦牛系;姬秋梅等[5]认为西藏11 个牦牛类群(110头)可以分为五大类,既帕里牦牛系,江达牦牛系,巴青牦牛系,桑日牦牛系和类乌齐牦牛系。本研究结果表明,西藏牦牛可以分为两大类,桑日牦、类乌齐牦牛和桑桑牦牛为1 类,其余13 个牦牛类群为另一类。这与文献[5]和[6]的研究结果有很大的不同,究其原因可能是:1)本研究所涉及的样本类群多,总体数量大,对于遗传多样性的研究更具有可靠性;2)试验研究的目的基因不同。这一结果与陈智华等[14]根据西藏牦牛染色体及血液蛋白多态性将西藏牦牛划分为藏东南山地牦牛与藏西北草地牦牛的结果基本一致;结合本次试验,牦牛类群样品的地理分布与类群间的聚类关系图进行分析发现,分布在藏北的嘉黎牦牛与分布在藏南的康布、帕里牦牛为1类,而藏东北的类乌齐牦牛和藏西的桑桑牦牛为1类,说明了人类的干预将会提升品种间的基因交流概率,从而逐渐降低牦牛遗产分化受地理隔离的影响程度。
西藏牦牛的20种单倍型网络关系图分为Ⅰ和Ⅱ2个聚类簇,其中聚类簇Ⅰ包含有17 种单倍型,囊括了本研究的所有牦牛类群;聚类簇Ⅱ中有3种单倍型,囊括了除错那、嘉黎、康布和帕里类群外的12个类群,这说明了西藏牦牛可能具有2 个母系起源,这与张成福等[4]的研究结果相一致。
从西藏牦牛ATP8基因的单倍型多样性和核苷酸多样性来看,其具有较贫乏的遗传多样性;聚类分析表明,西藏牦牛16个类群可以分为两大类,既桑日牦牛、类乌齐牦牛和桑桑牦牛为1类,其余12个类群为另1类;单倍型分析表明,西藏牦牛具有2个母系起源。
[1] WIENER G, HAN J L, LONG R J. The Yak[M]. 2nded.Bangkok,Thailand:regional office for Asia and the Pacific of the food and agriculture organization of the United Nations, 2003.
[2] 钟金城,赵素君,陈智华,等. 牦牛品种的遗传多样性及其分类研究[J]. 中国农业科学,2006,39(2):389-397.
[3] BRUFORD M W, BRADLEY D G, LUIKART G. DNA markers reveal the complexity of livestock domestication[J]. Nature Reviews Genetics, 2003,4(11): 900-910.
[4] 张成福,徐丽娟,姬秋梅,等. 西藏牦牛mtDNA D-loop区的遗传多样性及遗传分化[J]. 生态学报,2012,5(32):1387-1395.
[5] 姬秋梅,唐懿挺,张成福,等. 西藏牦牛mtDNA cytb基因的序列多态性及其系统进化分析[J].畜牧兽医学报,2012,43(11):1723-1732.
[6] 赵上娟,陈智华,姬秋梅,等. 西藏牦牛mtDNA COIII全序列测定及系统进化关系[J]. 中国农业科学,2011,23:4902-4910.
[7] 姜雪欧,钟金城. 西藏牦牛12SrRNA 基因全序列测定及系统进化关系[J]. 家畜生态学报,2013,34(6):8-12.
[8] 王镜岩,朱圣庚,徐长法. 生物化学[M].3版. 北京:高等教育出版社,2007.
[9] GARESSE R. Drosophila melanogaster mitochondrial DNA: gene organization and evolutionary considerations genetics[J]. Genetics, 1988,118:649-663.
[10]NEI M, KUMAR S. Molecular Evolution and phylogenetics [M]. New York: Oxford University Press, 2000.
[11]LAN H,SHI L M.The origin and genetic differentiation of native breeds of pigs in southwest China: An approach from mitochondrial DNA polymorphism [J].Biochem Genet, 1993,31,51-60.
[12]郭松长,祈得林,赵新全,等. 家牦牛线粒体DNA (mtDNA) 遗传多样性及其分类[J]. 生态学报,2008,28(9):4286-4294.
[13]赖松家,王 玲,刘益平,等. 中国部分牦牛品种线粒体DNA遗传多态性研究[J].遗传学报. 2005, 32(5):463-470.
[14]陈智华,钟金城,胡欧明,等.西藏牦牛和黄牛遗传多态性的研究[J]. 西南民族学院学报:自然科学版,1999,25(3):274-277.
(责任编辑:朱秀英)
Sequence analysis and genetic diversity of mtDNAATP8 of Tibet yaks
YANG Qin1, CHAI Zhixin1, JI Qiumei2, ZHANG Chengfu2, XIN Jinwei2,SONG Qiaoqiao1, ZHONG Jincheng1
(1.Key Laboratory of Animal Genetics and Breeding of State Ethnic Affairs Commission and Ministry of Education, Southwest University for Nationalities, Chengdu 610041,China; 2.Institute of Animal Science and Veterinary, Tibet Academy of Agricultural and Animal Husbandry Sciences, Lhasa 850000,China)
In order to investigate the genetic diversity and clustering relationships of Tibet yak, the mitochondrialATP8 (Adenosine Triphosphate 8) gene from 367 yaks in 16 populations were sequenced and analyzed. The results showed that the length of yak groups’ATP8 complete sequences were 201 bp~203 bp, and the average nucleotide composition of all sequences was 29.3%T, 23.0%C, 41.8%A and 6.0%G, and the average nucleotide content of A+T (71.1%) was obviously higher than that of G+C (28.9%). There were 19 polymorphic sites within sequences. Among these polymorphic sites, 15 were singleton variable sites and 4 were parsimony-informative sites. Conversion and insertions were detected in nucleotide sequences. A total of 20 haplotypes from 70 polymorphic sites were identified, and the nucleotide diversities and the haplotypes diversities were 0.00189 and 0.332, indicating a relatively poor genetic diversity in Tibetan yaks. The NJ phylogenetic tree revealed that the Tibet yak could be divided into two types. Network relationship chart of 20 haplotypes presented two clusters, indicating that Tibet yaks may be derived from two separate maternal lineages.
Tibet yak; mitochondrial DNA;ATP8 gene; genetic diversity
2014-06-17
国家科技支撑计划项目(2012BAD03B02)、西南民族大学研究生学位点建设项目(2013XWDS071007)
杨 琴(1987-),女,四川遂宁人,硕士研究生,主要从事基因组与生物信息学方面的研究。
钟金城(1963-),男,四川成都人,教授,博士。
1000-2340(2015)03-0368-06
S 513
A
