灯盏花ITS2基因克隆及RNA二级结构预测
2015-06-24赵春艳杨青松王锦琳
熊 勇,赵春艳,杨青松,王锦琳
(1.云南民族大学 民族药资源化学国家民委-教育部重点实验室,云南 昆明 650500;2.昆明食用菌研究所,云南 昆明 650221)
灯盏花ITS2基因克隆及RNA二级结构预测
熊 勇1,赵春艳2,杨青松1,王锦琳1
(1.云南民族大学 民族药资源化学国家民委-教育部重点实验室,云南 昆明 650500;2.昆明食用菌研究所,云南 昆明 650221)
利用PCR方法扩增灯盏花ITS2基因并克隆,获得ITS2基因序列.用生物软件对ITS2序列进行分析并构建系统进化树和RNA二级结构,得到灯盏花的ITS2核苷酸序列为205 bp,基于ITS序列飞蓬属11种共分为5支,分支与地理分布情况基本一致.ITS2 RNA二级结构在飞蓬属种间表现基本一致,而TS2区结构表现出属间差异.利用rDNA ITS区序列一结构和二级结构相结合达到鉴定药用植物灯盏花分类的目的.
灯盏花;基因克隆;ITS2;系统树;RNA二级结构
灯盏花学名为短葶飞蓬Erigeronbreviscapus(Vant.)Hand.-Mazz,为菊科(Composttiae)紫苑族(Trib.AstereaeCass.)飞蓬属(Ergeiorn)多年生草本植物,主要产于我国云南、贵州、四川等省,其中95%以上的野生资源分布于云南[1],作为传统民族药,在云南少数民族苗族和白族地区的运用已有600多年历史[2].从20世纪70年代开始,以灯盏花总黄酮为原料的灯盏花制剂广泛应用于临床,具有多种疗效且疗效好、安全性高,尤其是在治疗心脑血管疾病方面,其总有效率达88.3%[3-4],目前野生资源日益减少[5].为解决灯盏花药材的资源短缺,在云南省很多地州灯盏花已经大面积种植并推广[6],对当地的经济发展起到重要的作用.由于灯盏花野生种值资源长期的自然选择和生境的改变,以及栽培品种经过长期的人工杂交选育等原因,导致部分基因流失[7],栽培品种的遗传多样性丰富度在降低,因此利用有效的DNA分子方法对该药用植物的鉴定具有重要的作用.DNA分子鉴定技术拥有传统鉴定不可比拟的一些优势,已成功运用于多种中药材鉴定[8],其中核糖体DNA第二内转录间隔区(rDNA ITS2)序列长度小于 300 bp,适合进行各种分子操作,而且变异较快,能够提供较丰富的信息位点,已在药用植物鉴别系统发育和分类研究中发挥重要作用[9].本文以云南灯盏花为材料,对其rDNA ITS2序列进行了克隆及序列测定,并将其与相关植物的序列进行比较分析,以此来鉴定药用植物灯盏花,同时为我国药材市场安全提供保障.
1 材料与方法
1.1 试验材料
1.1.1 植物材料
供试灯盏花来自云南省剑川县沙溪镇,实验室栽种,新鲜灯盏花植物叶片作为材料.
1.1.2 试剂
2×PCR Master mix,DNA MarKer,PCR产物快速胶回收试剂盒,质粒puM-T,大肠杆菌感受态细胞,上述试剂及试剂盒均购于北京泰克生物技术有限公司.
1.1.3 引物
根据相关文献设计引物[9],设计引物由北京梓熙生物科技有限公司合成,灯盏花rDNA ITS2区域的引物的序列如表1:

表1 设计的引物序列
1.2 试验方法
1.2.1 灯盏花总DNA的提取
灯盏花总DNA提取方法参照SDS法[10],用琼脂糖凝胶电泳检测DNA质量.
1.2.2 灯盏花的ITS2基因的PCR扩增
PCR扩增反应在Eppedorf PCR仪上进行,反应体系总体积50 μL:25 μL 2×Mixture,引物2R和引物2F各2 μL,DNA模板1 μL,ddH2O 20 μL.
反应条件:94 ℃预变性5 min,94 ℃变性1 min,50 ℃退火1 min,72 ℃延伸2 min,32次循环,72 ℃再延伸10 min,最后4 ℃保存.扩增产物用1.5%琼脂糖凝胶电泳检测,在Bio-Rad凝胶成像系统观察结果.
1.2.3 灯盏花ITS2基因克隆
取PCR产物于1.5%胶进行琼脂糖凝胶电泳检测,进行胶回收.将回收的DNA片段连接到puM-T载体上,于16 ℃反应3 h;将连接后的puM-T载体转化到DH5α感受态宿主中培养;再将菌液涂布在含有X-gal底物、IPTG诱导物和氨苄青霉素的固体培养基平板中,利用蓝白斑遗传学筛选法筛选;挑选白色单一菌落,LB液体培养基培养,提取质粒,将构建的重组质粒通过PCR法转化子鉴定,将重组质粒送到生物公司进行测序.
1.2.4 数据分析
用生物软件DNAMAN对正向、反向测序结果进行拼接获得灯盏花ITS2基因一致序列,并在NCBI上进行同源性比较,查看克隆结果的可靠性,去除5.8S和28S区段获得ITS2间隔区序列.用Mega 6.0[11]中的 Kimura-2-parameter 模型计算遗传距离,构建距离矩阵.以菊科飞蓬属Erigeron为聚类基础,用邻接 (neighbor-joining method,N-J) 法构建系统进化树,再用DNAstar[12]、ClassyTK1.0、RnaViz2.0[13]等绘制RNA二级结构.
2 结果与分析
2.1 灯盏花ITS2基因PCR扩增产物
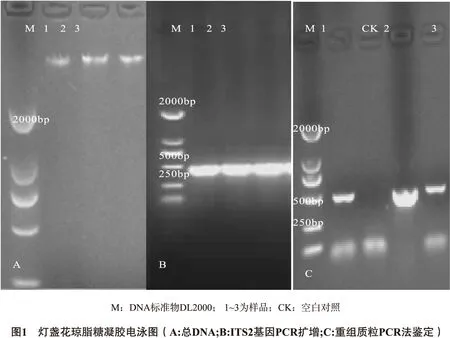
灯盏花不同居群ITS2基因PCR扩增序列长度约在250~500 bp之间,PCR产物均出现清晰明显条带(图1).
2.2 测序结果分析
用DNAMAN对正向、反向测序结果进行拼接获得灯盏花ITS2基因一致序列,使用基于隐马尔可夫模型的HMMer注释方法去除两端5.8S和28S区段获得ITS2间隔区序列205 bp,灯盏花ITS2基因序列如图2.
2.3 遗传距离
以自测的灯盏花Erigeronbreviscapu为探针,在NCBI库进行blast获得飞蓬属11个种,利用生物软件Mega6.0中Kimura 2-parameter model分析飞蓬属基于ITS2序列的种间遗传距离并构建距离矩阵(表2).飞蓬属种间的遗传距离为0.000~0.051,Erigeronkarvinskianus[14]与其他种的遗传距离均较大为 0.036~0.051;本研究获得的灯盏花ITS2序列与NCBI库中Erigeronbreviscapus(GU213433)遗传距离最近,而与加勒比飞蓬Erigeronkarvinskianus最远,与地理分布距离一致.

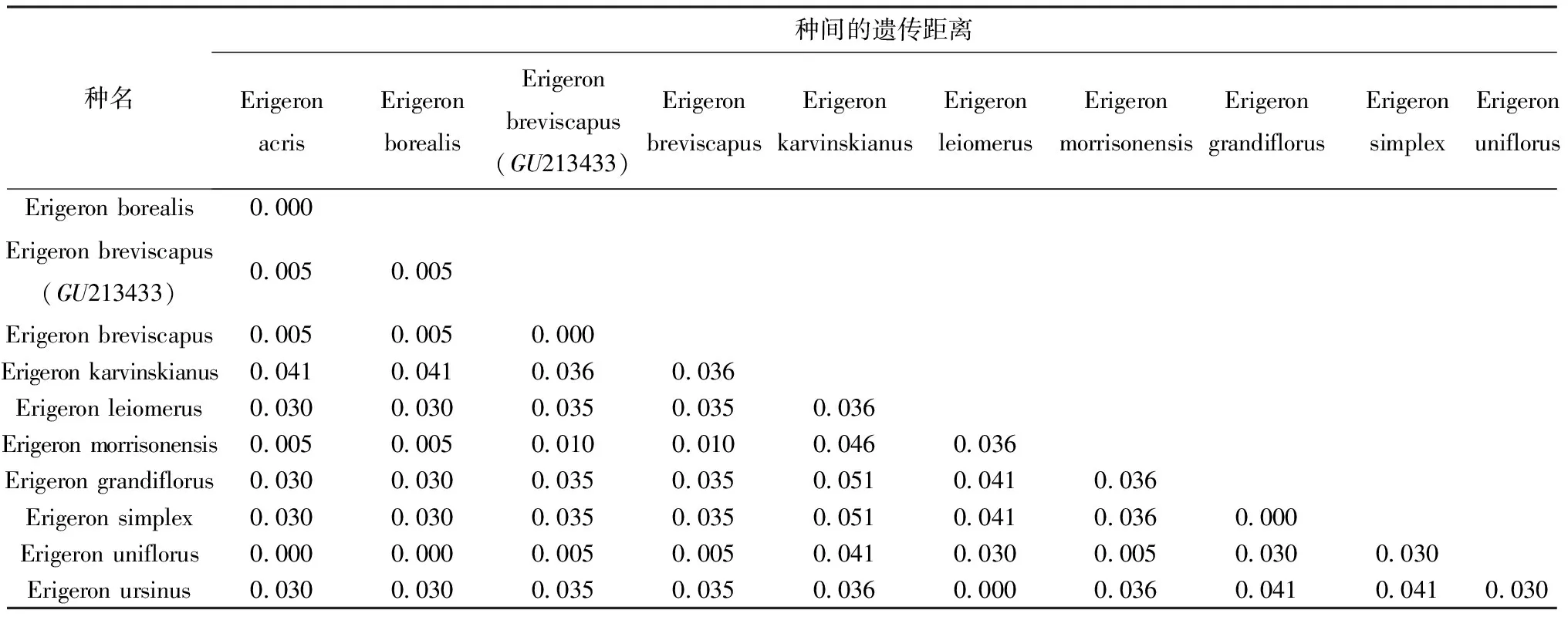
表2 飞蓬属种间的遗传距离
2.4 系统发育分析
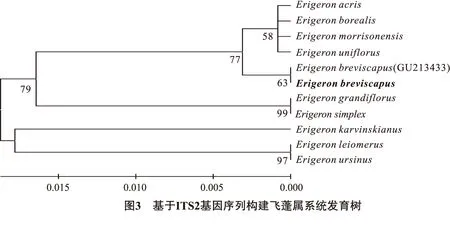
将飞蓬属的11个物种进行序列对比,并采用Mega软件构建其N-J系统发育树图3(黑体为本研究获得的灯盏花ITS2序列).系统发育树共分成5支,Erigeronmorrisonensis、Erigeronborealis、Erigeronacris与Erigeronunifloru先聚在一起,然后再与Erigeronbreviscapus聚类,作为整个进化树的核心.实验所要研究的目的样品Erigeronbreviscapus与GenBank库中Erigeronbreviscapus(GU213433)聚在一起,说明本实验结果准确,而Erigeronkarvinskianus[15]单独聚在一起.灯盏花与飞蓬属的其他物种有一定的差异,其他的物种聚在不同的分支,这可能与物种的地理分布有关系,如Erigeronbreviscapus(短葶飞蓬)主要生长在云南、西藏;Erigeronmorrisonensisvar(玉山飞蓬)等分布在台湾,Erigeronursinus(一年蓬)等分布在四川、西藏、安徽等省;Erigeronkarvinskianus(加勒比飞蓬)主要生长在北美,而台湾也有分布.每个分支的物种基本与地理分布相符合.
2.5 灯盏花ITS2基因RNA二级结构分析
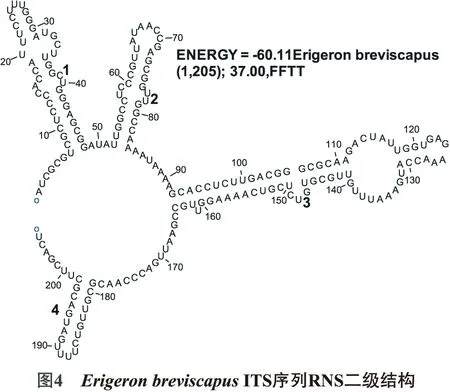
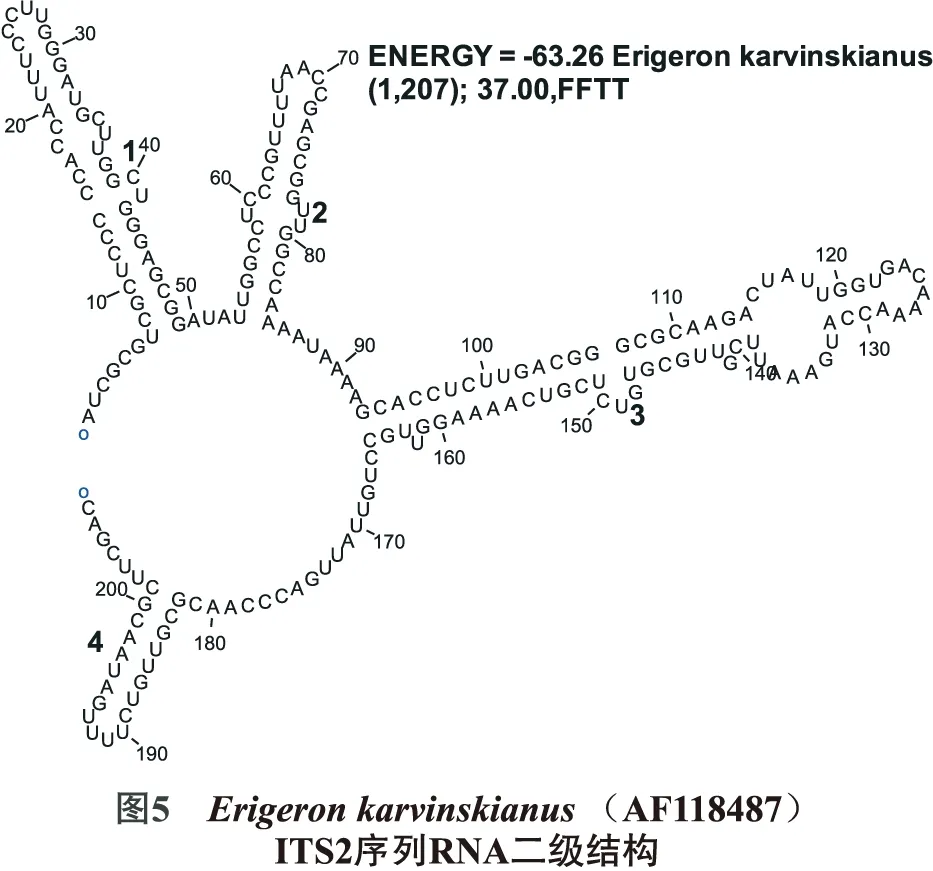
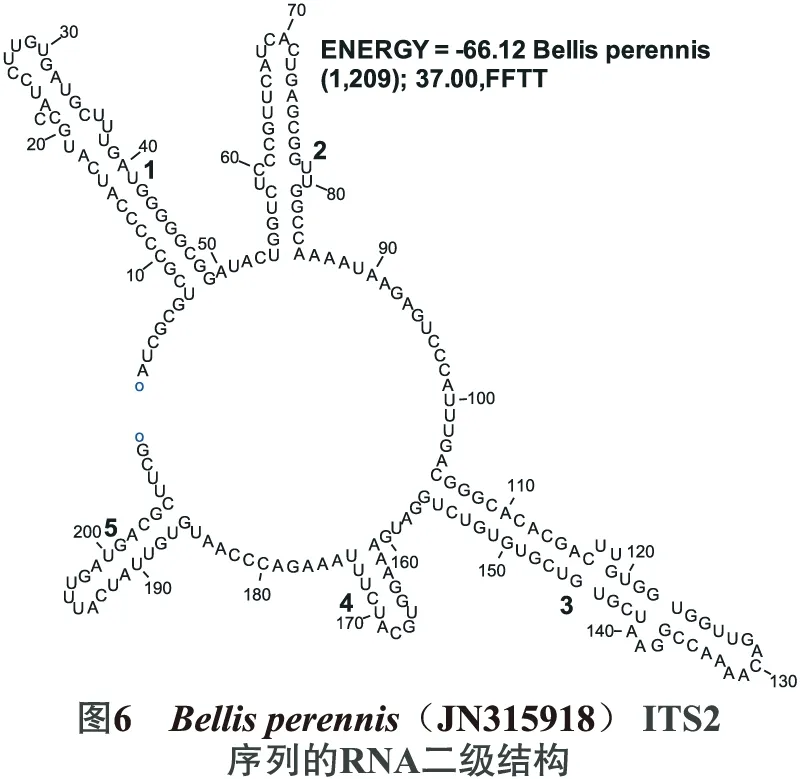
利用DNAstar中GenQuset、RnaViz2.0等软件构建本研究获得的Erigeronbreviscapus、GeneBank下载的Erigeronkarvinskianus(AF118487)以及菊属雏菊Bellisperennis(JX065158)[15]rDNA ITS2区序列二级结构,并分析结构上的差异,结果见图4~6.
短葶飞蓬(灯盏花)Erigeronbreviscapus与加勒比飞蓬Erigeronkarvinskianus在一级结构上具有一定的差异,基于ITS2核苷酸序列在11个飞蓬属的种间距离也较远,但从从图4~5 可以看出,Erigeronbreviscapus与Erigeronkarvinskianu预测的RNA二级结构得到的茎环数量一样都是4,结构也相似,而菊属雏菊Bellis perennis ITS2二级结构茎环数为5,结构也相差较大,表现为种内基本一致,属间差异较大.
3 结果与讨论
ITS2二级结构的核心区域具有高度的保守性,在研究物种的起源及高级阶元的系统进化关系中起着很重要的作用[16].相对于DNA序列和氨基酸序列,RNA二级结构能够提供结构差异方面的信息,对于灯盏花的分子分类具有一定的参考价值,可以作为一种研究物种分子系统学的重要手段.本研究发现,飞蓬属种间rDNA 的ITS2核苷酸序列具有一定的差异,表现出遗传距离与地理分布项一致,但是构建的rDNA 的ITS2 RNA二级结构在飞蓬属种间基本一致,表现为在菊科不同属间二级结构有差异.
本研究所克隆测序的灯盏花ITS2序列长度约为205bp,与GeneBank中的Erigeron breviscapus(GU213433)相比较没有差异,结果正确.用Mega中Kimura 2-parameter model计算并构建遗传距离图,以及用N-J法构建系统进化树,所分析的飞蓬属11个种分为五支,加勒比飞蓬Erigeron karvinskianu与其他种遗传距离较大,每个分支的物种基本与地理分布相符合,通过遗传距离以及系统发育树,可以为灯盏花的杂交育种提供一定的理论基础.
[1] 吴征镒,陈书坤.云南植物志:种子植物[M].北京:科学出版社,2004:129-131.
[2] 赵新杰,夏华玲.灯盏花的研究进展[J].时珍国医国药,2000,213(9):566.
[3] 宫碧琴.治疗瘫痪新药灯盏花素鉴定会在昆明召开[J].中草药,1980,11(10):480.
[4] 马宇辉,罗国安,王义明.灯盏花研究近况[J].中成药,2004,26(1):63.
[5] 王平理,杨生超,杨建文,等.云南灯盏花种质资源的考察与采集[J].现代中药研究与实践,2007,22(2):25.
[6] 杨生超,吴道聪,王平理,等.红河灯盏花GAP基地环境质量评价[J].现代中药研究与实践,2006(1):9-11.
[7] 熊勇,赵春艳,高兴艳,等.药用植物灯盏花rbcL基因的克隆、生物信息学及适应性进化分析(英文)[J].生物技术,2014,24(3):25-31.
[8] 邵棚柱,曹晖.中药分子鉴定[M].上海:复旦大学出版社,2004:73-95.
[9] 赵春艳,桂明英,桑兰,等.黑木耳菌株ITS 序列分析及二级结构预测[J].生物技术,2013,23(6):46-51
[9] MATSUMOTO T,OBATAKE Y,FUKUMASA-NAKAI Y,et al.Phylogenetic position of Pholiota nameko in the genus Pholiota inferred from restriction analysis of ribosomal DNA.Mycoscience,2003,44:197-202.
[10] RUSSO P,JUUTI J T,RAUDASKOSKI M.Cloning,sequence and expression of aβ-tubulin-encoding gene in the homobasidiomycete Schizophyllum commune[J].Gene,1992,119:175-182.
[11] 许忠能.生物信息学[M].北京:清华大学出版社, 2008.
[12] 李军.生物软件选择与使用指南[M].北京:化学工业出版社,2008.
[13] 吴祖建,高芳銮,沈建国.生物信息学分析实践[M].北京:科学出版社,2010.
[14] NOYES R D.Biogeographically and evolutionary insights on Erigeron and allies(Asteraceae)from ITS sequence data[J].Plant Systematics and Evolution 2000,220(2):93-114.
[15] LOONEY B P,BIRKEBAK J M,MATHENY P B.Systematics of the genus Auricularia with an emphasis on species from the southeastern United States[J].N Am Fungi,2013,8(6):1-25.
[16] 钟立强,张成锋,蔡生力,等.黑龙江野鲤核糖体内转录间隔区1( ITS-1)的克隆及二级结构的预测与分析[J].江苏农业学报,2010,26( 4):778-783.
(责任编辑 王 琳)
Cloning of ITS2 gene and analysis of its RNA secondary structural characteristics inErigeronbreviscapus
XIONG Yong1,ZHAO Chun-yan2,YANG Qing-song1,WANG Qing-lin1
(1.Key Laboratory of Chemistry in Ethnic Medicinal Resources,State Ethnic Affairs Commission and Ministry of Education of China,Yunnan Minzu University,Kunming 650500,China; 2. Kunming Edible Fungi Institute,Kunming 650221,China)
The rDNA ITS2 region in theErigeronbreviscapuswas amplified by PCR and cloned,and its sequence was obtained. The biological software was used to analyze rDNA ITS2 sequence and modeled its RNA secondary structure and phylogenetic tree. The length of ITS sequence was 205 bp. The phylogenetic tree(N-J) showed 11Erigeronspecies clustered into 5 branches based on ITS2 gene sequences. The ITS homology ofErigeronwas consistent with that of geographic distribution. A comparison of the secondary structures of rDNA ITS2 sequences revealed that the different species inErigeronhad no diversity. The secondary structure of the ITS2 regions was quite different among these genus species. Nucleotide sequence combined with the secondary structure of rDNA ITS region may achieve some identification of the classification ofErigeronbreviscapus.
Erigeronbreviscapus; ITS2 gene cloning; Phylogenetic tree; RNA secondary structure
2014-11-24.
云南民族大学民族药资源化学国家民委-教育部重点实验室开放基金(MZY1116).
熊勇(1977-),男,硕士,副教授.主要研究方向:生物化学.
Q7
A
1672-8513(2015)02-0151-05
