浴场海水中大肠杆菌耐药性及其耐药基因研究
2015-06-01王玥苏洁明红霞石岩赵莎关道明樊景凤
王玥,苏洁,明红霞,石岩,赵莎,关道明,樊景凤*
(1.国家海洋环境监测中心,辽宁大连 116023;2.国家海洋局近岸海域生态环境重点实验室,辽宁大连 116023;3.大连海洋大学水产与生命学院,辽宁大连 116023)
浴场海水中大肠杆菌耐药性及其耐药基因研究
王玥1,3,苏洁1,2,明红霞1,2,石岩1,3,赵莎1,3,关道明1,2,樊景凤1,2*
(1.国家海洋环境监测中心,辽宁大连 116023;2.国家海洋局近岸海域生态环境重点实验室,辽宁大连 116023;3.大连海洋大学水产与生命学院,辽宁大连 116023)
为了解海水浴场中大肠杆菌耐药性状况及其耐药基因分布,以大连星海浴场近岸海水样品为研究对象,分离鉴定得到41株大肠杆菌,采用K-B药敏纸片法检测其对9种常用抗生素的耐药性,并对耐药菌株中的耐药基因进行检测。结果表明:41株受试大肠杆菌菌株对四环素的耐药率最高(46%),而对氨曲南的耐药率最低(5%),大肠杆菌的多重耐药率达到34%。采用PCR方法对41株分离菌株进行四环素耐药基因检测,结果显示,仅四环素耐药基因tetA和tetB被检出,其他4种类型四环素耐药基因未被检出。大肠杆菌基因组与质粒DNA中tetA的检出率分别为34%和29%;tetB的检出率分别为2%和12%。相关性分析结果表明大肠杆菌的四环素耐药表型与四环素耐药基因的阳性检出率显著相关。
大肠杆菌;浴场海水;耐药性;耐药基因
1 引言
随着海洋环境与资源的不断开发与利用,海洋也逐步成为耐药性微生物及其耐药基因的重要储存库。耐药细菌也会随雨水冲刷、潮汐和海流带入近岸海滨旅游区,通过菌种间遗传物质的传递,耐药基因有可能被传入海水环境中的人体病原菌。由于海滩娱乐水域承载了大量的游客,这些耐药性致病菌可通过海水吞咽引发游泳人群致命性肠道疾病,因而大大增加了人体耐药病原菌病害发生和流行的可能性[1—3]。
大肠杆菌(Escherichiacoli)作为海产品及海洋环境粪便污染的重要指示微生物,是临床上引起感染最常见的肠杆菌科细菌之一,致病性的大肠杆菌会引发严重的腹泻和败血症等疾病[4—6]。2013年中国CHINET细菌耐药性监测结果显示,在各大医院采集临床分离菌的8万多株菌中耐药性大肠杆菌的检出率最高[7]。作为引起人类感染最常见的肠杆菌科细菌,大肠杆菌也是耐药基因的主要储存库,在耐药基因的传播中发挥着重要作用[8]。此外,临床上发现,大肠杆菌的多重耐性现象较严重,并且多重耐药性的大肠杆菌已被发现于近岸海水中[9—10]。耐药性大肠杆菌及其耐药性基因会通过海产品或海水吞咽等途径传递给人类,使抗菌药对人类疾病的治疗效果下降或消失,成为威胁人类健康的重大公共卫生问题。
目前,国外仅有少数关于海水浴场中大肠杆菌耐药性的报道,如Turgeon等人通过检测比较2004年与2005年在魁北克南部近200个浴场中采集的海水样品发现海水浴场中耐药性大肠杆菌的检出率升高了10%[1]。国内仅在北黄海近岸海域开展了抗磺胺类大肠杆菌的分布调查,并且采用的是化学分析方法[11],而对娱乐海水浴场中耐药性大肠杆菌的研究还未见报道。此外,目前国外的研究仅局限于海水浴场中大肠杆菌耐药性状况的检测层面,对其在海水浴场中的传播扩散因子——耐药基因的分布研究尚未开展。因此,我们对大连星海浴场中大肠杆菌耐药性及其耐药基因进行研究,通过药敏实验分析了海水浴场中大肠杆菌的耐药性状况,根据抗生素敏感性检测结果采用PCR方法对受试大肠杆菌四环素耐药基因进行分析。本研究的开展将有助于了解娱乐海水对人体健康的影响,并为相关管理部门进行的海水浴场风险评价提供数据支持,对浴场中耐药基因污染的深入研究具有重要意义。
2 材料与方法
2.1 材料
2.1.1 样品采集
于2014年8月进行样品采集,水样采自大连星海浴场距岸边约30 m游泳人群密集处,无菌玻璃瓶采集表层(10~15 cm)海水,低温保存运至实验室。
2.1.2 抗生素及生化鉴定管
选取临床上治疗大肠杆菌感染常用的9种抗生素:四环素(TCY)、哌拉西林(PIP)、氨曲南(ATM)、左氧氟沙星(LYX)、头孢噻吩(CEP)、复方新诺明(SXT)、链霉素(STR)、庆大霉素(GEN)、氯霉素(CHL),这9种抗生素均购自杭州天和微生物试剂有限公司。实验中所选肠杆菌科细菌生化编码微量鉴定管及参照的肠杆菌科细菌生化编码鉴定手册由杭州天和微生物试剂有限公司提供。
2.1.3 菌株及培养基
实验用大肠杆菌质控菌株为标准菌株大肠杆菌ATCC 25922,购自广东环凯微生物科技有限公司。实验所需的培养基有4种:m-TEC,用于大肠杆菌分离培养;LB琼脂,用于菌种的纯化;EC肉汤,用于大肠杆菌生化鉴定;水解酪蛋白琼脂(Mueller-Hinton,MH),用于药敏实验。
2.2 大肠杆菌的分离鉴定
2.2.1 大肠杆菌的分离
使用滤膜法对大连星海浴场表层海水中的大肠杆菌进行过滤,水样过滤后滤膜置于m-TEC琼脂培养基上,于(35±1)℃培养2 h,再以(44.5±0.5)℃培养16~18 h。挑取黄色和黄棕色菌落初步鉴定为疑似大肠杆菌。于LB琼脂培养基上划线分离纯化。37℃培养过夜,直至平板上长出单一相同的菌落。
2.2.2 生理生化鉴定
用接种针在LB琼脂培养基上挑取单菌落,接种于肠杆菌科细菌生化编码微量鉴定管,进行以下15种试验项目:硫化氢实验、苯丙酸盐实验、葡萄糖酸盐实验、吲哚实验、葡磷胨水实验、枸橼酸盐实验、尿素实验、半固体实验、葡萄糖产气实验、赖氨酸实验、鸟氨酸实验、棉子糖实验、山梨醇实验、侧金盏花醇实验、木胶糖实验。将鉴定管置于37℃恒温培养24 h后,依据生化反应结果,参照细菌鉴定编码册进行判断并编码,再按照编码值检索生化编码表,即可获得对应的结果。
2.3 抗生素敏感性检测
采用CLSI推荐的K-B药敏纸片扩散法研究大肠杆菌对9种常用抗生素的耐药性[12]。细菌药敏实验结果参照CLSI公布的标准以敏感、中敏、耐药3种形式对抑菌圈大小作出解释。采用UHONET5.6软件对药敏结果进行分析。抗生素敏感性检测过程中采用大肠杆菌ATCC 25922作为质控菌株。
2.4 四环素耐药基因检测
为检测大肠杆菌中四环素耐药基因的分布,选取海水环境中常见的6种四环素耐药基因(tetA,tetB,tetC,tetD,tetE,tetG)为检测对象,以大肠杆菌基因组DNA和质粒DNA为模板进行四环素耐药基因的PCR扩增。PCR反应条件及引物信息参照文献[13]。25μL的PCR反应体系:2×Taq Master Mix 12.5μL,上下游引物各0.25μL,DNA模板1μL,dd H2O 11μL。基因组DNA和质粒DNA的提取均参照试剂盒(天根生物公司的基因组DNA和质粒DNA提取试剂盒)说明书。
2.5 统计学分析
应用SPSS软件,根据对大肠杆菌分离菌株体外四环素药物敏感性和四环素耐药基因PCR检测结果,将四环素耐药基因阳性菌株数与四环素耐药菌株数进行χ2检验,对四环素耐药基因阳性菌株中的耐四环素菌株数与四环素耐药基因阴性菌株数进行比较,分析大肠杆菌的四环素耐药性与四环素耐药基因阳性检出率的相关性。
3 结果
3.1 生理生化鉴定
对在m-TEC琼脂培养基上初筛得到的138株疑似大肠杆菌菌株进行生理生化鉴定,结果如表1所示。由表1可知,其中41株为大肠杆菌,分离率为30%,其余77株为非肠杆菌科细菌。这说明使用m-TEC琼脂培养基应用于海水中大肠杆菌的分离率较低。
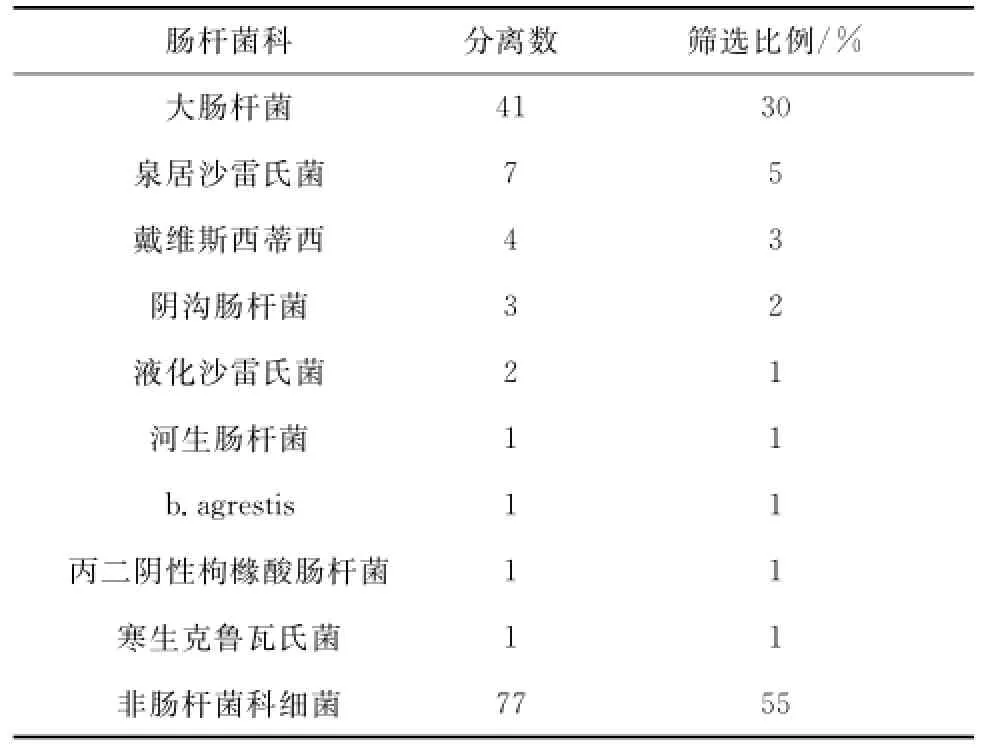
表1 138株筛选细菌种属分布Tab.1 The bacterial species identification of 138 isolates
3.2 药敏实验
41株大肠杆菌对9种常用抗生素的药敏实验结果显示,66%的受试大肠杆菌被检测到耐药性。其中受试菌株对四环素的耐药率最高(46%),对头孢噻吩和复方新诺明也呈现较高耐药性,耐药率达到30%以上。受试菌株对哌拉西林、左氧氟沙星、链霉素、庆大霉素和氯霉素的耐药率也达到了近20%。而对氨曲南耐药率最低(5%)(表2)。41株大肠杆菌的多重耐药率为34%,最高耐抗生素个数达到7(表3)。

表2 41株大肠杆菌对9种抗菌药物的耐药情况Tab.2 Antibiotic resistance of 41E.coliisolates against 9 antimicrobials

续表2
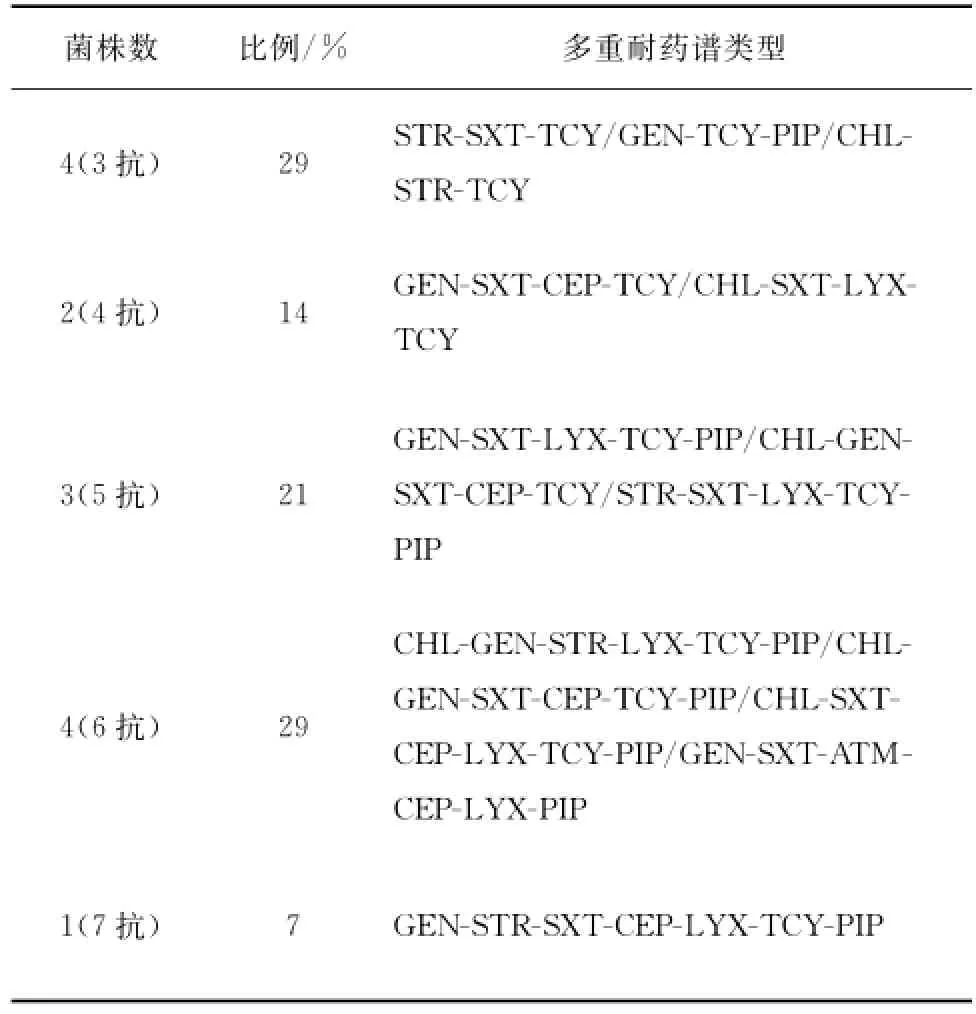
表3 41株大肠杆菌的多重耐药谱Tab.3 Multi-resistant pattern of 41E.coliisolates
3.3 四环素耐药基因的PCR检测
3.3.1 基因组DNA中四环素耐药基因检测
采用PCR方法对41株大肠杆菌基因组DNA中的四环素耐药基因tet(A,B,C,D,E,G)进行检测,结果显示,仅耐药基因tetA和tetB被检测到,检出率分别为34%和2%(表4)。四环素耐药菌株中14株(63%)检测到tetA基因,1株(5%)检测到tetB基因,2株四环素敏感菌株检测到tetA基因,推测其原因为携带四环素耐药基因但并未表达。其他四环素耐药基因检测结果均为阴性。实验结果中四环素耐药基因检测结果为阳性的菌株,四环素耐药菌株占了很大比率,tetA检测为阳性的菌株中86%为四环素耐药菌株,tetB检测为阳性的菌株中100%为四环素耐药菌株,这表明四环素耐药基因在基因组DNA中的携带情况与耐药菌株的四环素耐药表型可能存在一定的相关性。

表4 大肠杆菌基因组DNA中四环素耐药基因检出率Tab.4 Detection rate of tetracycline resistance gene ingenome DNA of 41E.coliisolates
3.3.2 质粒DNA中四环素耐药基因检测
大肠杆菌质粒DNA中四环素耐药基因检测结果显示,41株受试大肠杆菌中tetA的检出率为29%,而tetB的检出率为12%。四环素耐药基因tetA阳性菌株中91%为四环素耐药菌株,tetB阳性菌株中全部为四环素耐药菌株(表5),这说明四环素耐药基因在质粒DNA中的携带情况与耐药菌株的四环素耐药表型存在一定的相关性。

表5 大肠杆菌质粒DNA中四环素耐药基因检测结果Tab.5 Detection rate of tetracycline resistance gene in plasmid DNA of 41E.coliisolates
3.3.3 四环素耐药表型与四环素耐药基因的相关性分析
41株分离菌株中,有19株为四环素耐药菌株,耐药率为46%。大肠杆菌基因组DNA与质粒DNA中四环素耐药基因的检测结果显示,四环素耐药基因阳性菌株中四环素耐药菌株所占比例较高,分别为86.7%和94.1%。为进一步验证四环素耐药基因阳性菌株数与四环素耐药菌株数之间的相关性,将二者进行χ2检验,发现四环素耐药基因阳性菌株中的四环素耐药菌株数显著高于四环素耐药基因阴性菌株,表明大肠杆菌的四环素耐药性与四环素耐药基因阳性检出率相关(表6,表7)。
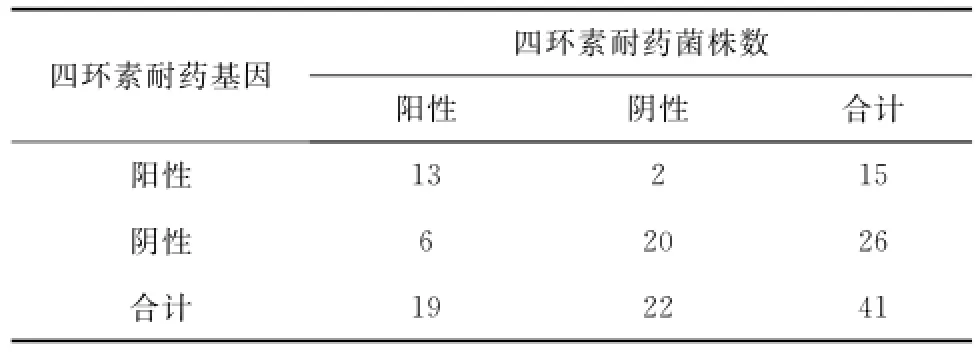
表6 大肠杆菌四环素耐药表型与基因组DNA中四环素耐药基因相关性分析Tab.6 Correlation among tetracycline resistant phenotype and tetracycline resistant gene in genome DNA ofE.coli
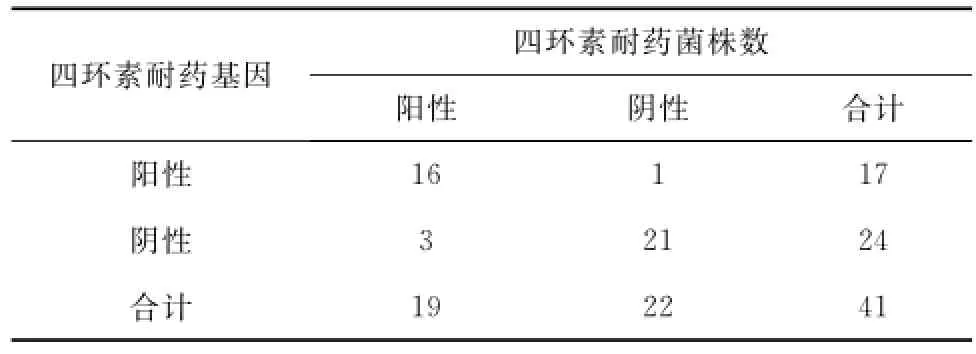
表7 大肠杆菌四环素耐药表型与质粒DNA中四环素耐药基因相关性分析Tab.7 Correlation among tetracycline resistant phenotype and tetracycline resistant gene in plasmid DNA ofE.coli
4 讨论
本文采用m-TEC琼脂培养基在大连星海浴场海水中初筛获得138株疑似大肠杆菌,经生理生化鉴定发现41株分离菌株为大肠杆菌,这说明应用于m-TEC琼脂培养基方法对海水中大肠杆菌的分离率较低。药敏试验结果显示,66%的受试大肠杆菌被检测到耐药性,受试大肠杆菌对9种抗生素中四环素的耐药率最高。这可能与四环素类药物(包括四环素、土霉素、金霉素、强力霉素等)在我国使用时间较长,应用范围较广有关。相反,由于氨曲南等药物的临床使用相对较少,在药物使用的选择压力下,反映为抗生素的耐药率相对较低,这与国外的相关研究结果一致。如在印度东南部杜蒂戈林海岸的海洋生物中筛选出的168株大肠杆菌中,其中对四环素的耐药率达到了26%[10];韩国学者对GOMSO海湾表层海水中筛选出的131株大肠杆菌进行耐药性研究发现,四环素耐药率为33.6%、链霉素11.5%、氯霉素9.9%、庆大霉素0.8%[14]。本研究发现大连星海浴场海水中大肠杆菌不但普遍存在抗生素抗性,而且其多重耐药率较高,这说明星海浴场中多重耐药情况明显,而多重耐药的产生会给人和动物疾病的治疗带来极大困难,因此海水浴场水质相关管理部门应引起重视。
在本研究中,大肠杆菌对四环素的耐药性最为普遍,为进一步了解我国海水浴场海水中大肠杆菌四环素耐药性传播机制,本文初步研究了大肠杆菌中四环素耐药基因的分布。前期研究发现,海水环境中的细菌至少有7种以上不同质粒编码的四环素耐药基因(tetA,tetB,tetC,tetD,tetE,tetG,tetY等)[15—19],并且世界上不同海域内主要四环素耐药基因的种类和组成有很大的差异,这种差异不但与地理分布有关,而且还与细菌耐药性的进化有关。普遍认为主动外排作用和核糖体保护作用是细菌对四环素产生耐药性的主要机制,其中大肠杆菌以主动外排机制为主[20],研究较多的主动外排基因分别为tetA,tetB和tetC等,其中tetA与tetC作为代表四环素类药物外排机制的基因广泛存在于很多细菌中[21]。本研究发现受试大肠杆菌无论是基因组DNA还是质粒DNA中仅四环素耐药基因tetA和tetB被检测到,而且tetA的检出率最高,因此推测海水浴场海水中大肠杆菌四环素耐药性可能主要由tetA基因传递,主动外排机制可能为本研究中试验菌株四环素耐药的主要机制。Fan W等[22]在世界范围内收集了107株耐四环素大肠杆菌分离株,用多重PCR方法检测四环素耐药基因,结果发现其中49.5%含tetA基因,35.5%含tetB基因,5.6%含tetC基因,1.9%含tetD基因。以上数据说明tetA基因和tetB基因可能在大肠杆菌中分布广泛,这两种基因尤其是tetA基因作为四环素外排泵基因,在四环素耐药性方面起到主要作用。
通过检测四环素耐药基因还发现,四环素耐药基因检测结果为阳性的菌株在耐四环素的大肠杆菌中占了很大比率,这说明四环素耐药基因的携带情况与耐药菌株的四环素耐药表型可能存在一定的相关性。将四环素耐药基因阳性菌株数与四环素耐药菌株数进行χ2检验发现,四环素耐药基因阳性菌株中的耐四环素菌株数显著高于四环素耐药基因阴性菌株数,这表明大肠杆菌的四环素耐药表型与四环素耐药基因阳性检出率相关,因此进一步推断在本研究中大肠杆菌四环素耐药性的产生可能是由tetA和tetB基因介导的。
5 结论
(1)采用K-B药敏纸片扩散法研究发现,大连星海浴场表层海水中大肠杆菌耐药情况严重,其中对四环素的耐药率最高,并且多重耐药状况明显。
(2)对分离的大肠杆菌进行四环素耐药基因检测发现,主动外排基因tetA与tetB被检测到,主动外排机制被推测为受试菌株四环素耐药的主要机制。
(3)海水中受试大肠杆菌的四环素耐药表型与四环素耐药基因阳性检出率显著相关,其耐药性的产生可能是由tetA和tetB基因介导的。
[1] Turgeon P,Michel P,Levallois P,et al.Antimicrobial-resistantEscherichiacoliin public beach waters in Quebec[J].Canadian Journal of Infectious Diseases and Medical Microbiology,2012,23(2):e20-e25.
[2] Schmitt H,Stoob K,Hamscher G,et al.Tetracyclines and tetracycline resistance in agricultural soils:Microcosm and field studies[J].Microbial E-cology,2006,51(3):267-276.
[3] Ash R J,Mauck B,Morgan M.Antibiotic resistance of Gram-negative bacteria in rivers,United States[J].Emerging Infectious Diseases,2002,8(7):713-716.
[4] Kaper J B.PathogenicEscherichiaColi[J].Int J Med Microbiol,2005,295(6/7):355-356.
[5] Hui Y H,Gorham J R,Murrell K D,et al.Foodborne disease handbook.Diseases caused by viruses,parasites,and fungi.Volume 2[M].New York:Marcel Dekker,Inc,1994,2:682.
[6] Adzitey F,Ali G R R,Huda N,et al.Antibiotic resistance and plasmid profile ofEscherichiacoliisolated from ducks in Penang,Malaysia[J].International Food Research Journal,2013,20(3):1473-1478.
[7] 胡付品,朱德妹,汪复,等.2013年中国CHINET细菌耐药性监测[J].中国感染与化疗杂志,2014,14(5):365-374.
Hu Fupin,Zhu Demei,Wang Fu,et al.CHINET 2013 surveillance of bacterial resistance in China[J].Chinese Journal of Infection and Chemotherapy,2014,14(5):365-374.
[8] Perreten V,Schwarz F,Cresta k,et a1.Antibiotic resistance spread in food[J].Nature,1997,389(6653):801-802.
[9] Alm E W,Zimbler D,Callahan E,et al.Patterns and persistence of antibiotic resistance in faecal indicator bacteria from freshwater recreational beaches[J].Journal of Applied Microbiology,2014,117(1):273-285.
[10] Jeyasanta K I,Aiyamperumal V,Patterson J.Prevalence of antibiotic resistantEscherichiacoliin sea foods of Tuticorin coast,Southeastern India[J].Adv Biological Research,2012,6:70-77.
[11] 张婉茹,那广水,陆紫皓,等.北黄海近岸海域磺胺类抗生素及其抗性Escherichiacoli分布[J].应用与环境生物学报,2014,20(3):401-406.
Zhang Wanru,Na Guangshui,Lu Zihao,et al.Distribution of sulfonamides and sulfonamide-resistant Escherichia coliin the coastal marine environment of northern Yellow Sea,China[J].Chin J Appl Environ Biol,2014,20(3):401-406.
[12] Clinical and Laboratory Standards Institute.Performance Standards for Antimicrobial Susceptibility Testing;Twenty Fourth Informational Supplement[Z].CLSI documents M100-S24.CLSI,2014.
[13] Changkaew K,Utrarachkij F,Siripanichgon K,et al.Characterization of antibiotic resistance inEscherichiacoliisolated from shrimps and their environment[J].Journal of Food Protection,2014,77(8):1394-1401.
[14] Kwon-Sam P.Antimicrobial resistance and virulence genes presence inEscherichiacolistrains isolated from Gomso Bay,Korea[J].Fisheries and Aquatic Sciences,2013,16(4):221-227.
[15] Adams C A,Austin B,Meaden P G,et al.Molecular characterization of plasmid-mediated oxytetracycline resistance inAeromonassalmonicida[J].Applied and Environmental Microbiology,1998,64(11):4194-4201.
[16] DePaola A,Peller J T,Rodrick A G E.Effect of oxytetracycline-medicated feed on antibiotic resistance of gram-negative bacteria in Catfish Ponds[J].Applied and Environmental Microbiology,1995,61(6):2335-2340.
[17] DePaola A,Roberts M C.Class Dand E tetracycline resistance determinants in gram-negative bacteria from catfish ponds[J].Molecular and Cellular Probes,1995,9(5):311-313.
[18] Rhodes G,Huys G,Swings J,et al.Distribution of oxytetracycline resistance plasmids between aeromonads in hospital and aquaculture environments:implication of Tn1721 in dissemination of the tetracycline resistance determinant Tet A[J].Applied and Environmental Microbiology,2000,66(9):3883-3890.
[19] Schmidt A S,Bruun M S,Dalsgaard I,et al.Incidence,distribution,and spread of tetracycline resistance determinants and integron-associated antibiotic resistance genes among motile aeromonads from a fish farming environment[J].Applied and Environmental Microbiology,2001,67(12):5675-5682.
[20] 张纯萍,宁宜宝,宋立.健康鸡猪体内大肠杆菌对四环素的耐药性及耐药基因分布[J].中国农业科学,2010,43(12):2578-2583.
Zhang Chunping,Ning Yibao,Song Li.Resistance to tetracycline and distribution of tetracycline resistance determinants in commensal Escherichia coli isolated from clinically healthy chickens and pigs[J].Scientia Agricultura Sinica,2010,43(12):2578-2583.
[21] 江婷婷.不同时期猪源大肠杆菌对抗生素耐药表型、耐药基因比较研究[D].雅安:四川农业大学,2008.
Jiang Tingting.Study on the Variance of antibiotic resistance ofEcoliof swine of different years on phenotype and genotype level[D].Ya'an:Sichuan Agricultural University,2008.
[22] Fan W,Hamilton T,Webster S.Multiplex real-time SYBR Green IPCRassay for detection of tetracyclineefflux genes of Gram-negative bacteria[J].Molecular&Cellular Probes,2007,21(4):245-256.
Study on antibiotic resistance and resistant related genes of Escherichia coli from seawater of bathing beach
Wang Yue1,3,Su Jie1,2,Ming Hongxia1,2,Shi Yan1,3,Zhao Sha1,3,Guan Daoming1,2,Fan Jingfeng1,2
(1.National Marine Environmental Monitoring Center,Dalian 116023,China;2.The Ecological Environment Key Laboratory of Coastal Waters of the State Oceanic Administration,Dalian 116023,China;3.College of Fisheries and Life Science,Dalian Ocean University,Dalian 116023,China)
To study antibiotic resistance pattern and resistant gene ofEscherichiacolifrom seawater recreational beach,41E.coliisolates from seawater in Dalian Xinghai bathing beach were identified and characterized using biochemical tests.Antimicrobial resistance ofE.coliisolates was determined by Kirby-Bauer disk diffusion method,the result showed highest resistance to tetracycline(46%)and lowest resistance against ammonia QuNa(5%).Multidrug resistance was observed in 34%of 41E.coliisolates.Only tetracycline resistant genetetA andtetB gene were detected among the 41 isolates using PCR method.The detection frequency oftetA gene was 34%in genome DNA and 29%in plasmid DNA ofE.coli.WhiletetB gene was detected in 2%ofE.coligenome DNA and in 12% ofE.coliplasmid DNA.χ2test suggest that tetracycline resistant phenotype ofE.coliis correlated with tetracycline resistant gene.
Escherichiacoli;seawater of bathing beach;antibiotic resistance;resistant gene
S945.4
A
0253-4193(2015)12-0123-06
王玥,苏洁,明红霞,等.浴场海水中大肠杆菌耐药性及其耐药基因研究[J].海洋学报,2015,37(12):123—128,
10.3969/j.issn.0253-4193.2015.12.013
Wang Yue,Su Jie,Ming Hongxia,et al.Study on antibiotic resistance and resistant related genes ofEscherichiacolifrom seawater of bathing beach[J].Haiyang Xuebao,2015,37(12):123—128,doi:10.3969/j.issn.0253-4193.2015.12.013
2015-05-22;
2015-09-27。
海洋公益性行业科研专项(201105007);全球变化与海气相互作用(GASI-03-01-02-05);人社部2015年度留学人员科技活动项目。
王玥(1988—),男,辽宁省本溪市人,主要从事海洋生物学研究。E-mail:15941175630@163.com
*通信作者:樊景凤(1972—),女,研究员,主要研究方向为海洋微生物学和海洋生态学。E-mail:jffan@nmemc.org.cn
