猪STARD3NL基因克隆和表达谱分析
2015-03-22邢晋祎贾坤航李艳平
邢晋祎,贾坤航,李艳平
(1.临沂大学生命科学学院,临沂 276005;2.山东农业大学动物科技学院,泰安 271018)
猪STARD3NL基因克隆和表达谱分析
邢晋祎1*,贾坤航1,李艳平2
(1.临沂大学生命科学学院,临沂 276005;2.山东农业大学动物科技学院,泰安 271018)
本研究旨在克隆猪STARD3NL(STARD3 N-terminal like protein) 基因,对其基因结构进行分析,并研究该基因在猪不同组织中的表达特性。采用5′和3′-RACE以及RT-PCR方法克隆猪STARD3NL基因,利用生物信息学方法预测蛋白结构,应用qRT-PCR技术检测组织表达特性。结果表明,猪STARD3NL基因的cDNA序列长度为1 319 bp(GenBank登录号:HQ634946),编码235个氨基酸,该氨基酸序列与其他物种相比一致性较高;进化树分析表明,猪STARD3NL氨基酸序列与牛和绵羊的关系较近,与斑马鱼的进化关系最远。STARD3NL氨基酸序列包含保守的MENTAL结构域,为疏水蛋白,包含4个跨膜螺旋区;二级结构主要是α-螺旋,占60%;该蛋白不存在信号肽,不是分泌蛋白;亚细胞定位显示,该蛋白存在于内质网的概率为39.1%。qRT-PCR分析表明,STARD3NL基因在所检测猪的14种组织均有表达,在肝中表达水平最高,在眼肌中的表达水平最低;莱芜猪和大长猪被圆环病毒感染后,STARD3NL基因在2种猪肺组织中的表达水平差异不显著(P>0.05)。研究结果将为进一步研究STARD3NL基因的结构和功能以及猪抗病育种提供资料。
猪;STARD3NL基因;克隆;猪圆环2型病毒
STARD3NL又称为MENTHO,是一种功能尚不完全明确的整连蛋白。该蛋白的N端包含MENTAL结构域,与STARD3(MLN64)有很高的同源性。与STARD3不同的是,STARD3NL没有START结构域,此结构域是STARD类蛋白的特有结构域;与STARD3相同的是,STARD3NL在个体不同组织中也存在广泛的表达,定位在细胞内的晚期核内体,可能与那里的STARD3相互作用[1]。有人认为STARD3NL与STARD3一起通过核内体途径调控胆固醇的胞内运输[1-2]。MENTAL结构域含有4个跨膜螺旋,这种结构域能介导MLN64和STARD3NL同源体和异源体的互作,使该蛋白定位在晚期内体膜上[1-2]。
对STARD3蛋白的功能已有初步了解,但STARD3NL蛋白的功能尚不太清楚。STARD3过表达会诱导巨型核内体的产生,该诱导作用可能由MENTAL结构域行使。STARD3NL的过表达会导致细胞内巨型核内体的积累[3]。编码GST-MLN64融合蛋白的质粒与编码MENTHO 或 MLN64的质粒共转染的Hela细胞,STARD3NL 和STARD3与GST-STARD3发生特异性的免疫共沉淀。这表明STARD3可以在细胞内与STARD3NL形成同源或异源低聚物[2-4]。
目前,猪的STARD3NL基因序列和表达水平尚未见报道。莱芜猪以其抗病强、繁殖率高、耐粗饲、肉质细嫩香醇等特点而著称,为了充分挖掘其优良的基因库资源,本研究根据GenBank上登录的人和小鼠的STARD3NL基因设计简并引物,采用RT-PCR技术,克隆莱芜猪STARD3NL基因序列,并对其结构和功能进行生物信息学分析;采用荧光定量RT-PCR(qRT-PCR)方法分析STARD3NL基因mRNA在猪不同组织中的表达谱和用圆环病毒(Porcine circovirus type 2,PCV2)攻毒后表达水平的变化。试验结果可望为进一步研究猪STARD3NL基因的结构和功能,以及利用莱芜猪抗病性强的特点进行抗病育种奠定基础。
1 材料与方法
1.1 试验材料
1.1.1 试验动物和样品采集 选用15头45日龄PCV2-抗体阴性莱芜猪和15头大长猪(大白猪♂×长白猪♀),随机分成4组,即莱芜猪空白对照组5头,大长猪空白对照组5头,攻毒莱芜猪10头、攻毒大长猪10头,分4个猪圈饲养,观察饲养1周后进行PCV2人工感染,肌肉注射3 mL毒株:PCV2(SD2株)103.8TCID 50·mL-1,对照组肌肉注射3 mL生理盐水。
样品采集:分别在感染35 d后,选取4头猪屠宰,攻毒猪和大长猪对照组采集肺组织,莱芜猪对照组分别采集肝、肺、肾、胃、背膘、脾、脊髓、膀胱、胆囊、大肠、小肠、骨骼肌、眼肌、心组织2 g左右,立即存入液氮保存,然后存放-80 ℃冰箱备用。
1.1.2 主要试剂 M-MLV反转录酶、EcoRⅠ和Hind Ⅲ限制性内切酶、T4连接酶、LATaqDNA聚合酶、SYBR®Green I定量PCR试剂盒、RNA酶抑制剂、dNTP Mix、Marker DL2000、pMD18-T vector、琼脂糖凝胶DNA回收试剂盒等,以上试剂均购自大连TaKaRa公司;Trizol试剂购自Invitrogen公司;E.coliDH5α购自生工生物工程(上海)股份有限公司。
1.2 试验方法
1.2.1组织总RNA的抽提和cDNA第一链的合成 采用Trizol法,按照Trizol试剂盒说明书进行。RNA用灭菌DEPC水充分溶解后,-80 ℃冰箱保存备用。
cDNA第一链的合成:(1)在0.2 mL PCR管中加入: 1.0 μL(约2 μg) RNA、2.0 μL Oligo(dT)18(约0.5 μg)、无RNase的超纯水加到10 μL,轻轻混匀;(2)70 ℃变性5 min,立即置于冰上3 min,使RNA变性;(3)轻轻离心PCR管数秒,再加入试剂:5 μL M-MLV 5×reaction buffer、5 μL dNTPs(2.5 mmol·L-1)、1 μL RNase 抑制剂、1 μL M-MLV RT(200 U)、3 μL无RNase的水,混匀,轻轻离心数秒;(4)42 ℃延伸60 min,85 ℃ 15 min终止反应,冰上冷却2 min,立即进行PCR反应或保存于-20 ℃备用。
1.2.2STARD3NL基因克隆
1.2.2.1 引物设计和RT-PCR扩增:根据GenBank上人(NM_032016)和小鼠(NM_024270)的STARD3NL基因序列设计猪STARD3NL基因简并引物Stard-F和Stard-R;根据测序得到的序列设计4条5′、3′-RACE引物(GS5′ P outer、GS5′P inner、GS3′ P outer、GS3′P inner)和2条荧光定量PCR基因表达引物(Expr-F、Expr-R);以猪管家基因GAPDH(GenBank:AF017079)为内标,设计引物GAP-F和GAP-R,由工生物工程(上海)股份有限公司合成(表1)。
表1 PCR引物序列、退火温度、扩增片段大小及用途
Table 1 Primer sequences,annealing temperature,sizes of PCR product and purpose

引物名称Primername引物序列(5′→3′)Primersequence退火温度/℃AnnealingtemperaturePCR扩增片段大小/bpSizesofPCRproduct用途UsageStard⁃FStard⁃RGGAACAACCCGATGAGGATTAAGAATACAGCACGAGACG551063PartialcDNAcloningGS5′PouterGS5′PinnerTGGAATGGACATCACGCAAGGATAAGGATGCATGCGAGCTCTGACT631905′cDNAcloningGS3′PouterGS3′PinnerCACTACTGACATGATTGAACGGCCCTGTGGCTGGTAAGATAATGTC632603′cDNAcloningExpr⁃FExpr⁃RCATCTCCTTCATCCTTGCCTATCAGAAAGCCCACCAGGT55142ExpressionGAP⁃FGAP⁃RACTCACTCTTCTACCTTTGATGCTTGTTGCTGTAGCCAAATTCA57100Internalcontrol
中间目的片段PCR扩增体系:第一链cDNA 1.0 μL (约50~100 ng),10×LA PCR buffer 2.5 μL,dNTP Mix(2.5 mmol·L-1) 2.0 μL,MgCl2(25 mmol·L-1) 2.0 μL,Stard-F(20 μmol·μL-1) 0.5 μL,Stard-R(20 μmol·μL-1) 0.5 μL,LATaqDNA聚合酶0.2 μL(1 U),ddH2O 加至25 μL,吹打混匀后稍离心片刻。反应条件:94 ℃ 4 min,(94 ℃ 35 s,55 ℃ 35 s,72 ℃ 1.5 min)×35个循环,72 ℃延伸10 min,4 ℃保存。
5′和3′-RACE片段扩增:使用表1中STARD3NL基因特异性引物GS5′ P outer、GS5′P inner、GS3′ P outer和GS3′P inner,按照3′-Full RACE Core Set with PrimeScriptTMRTase和5′-Full RACE Kit with TAP(TaKaRa)试剂盒说明书进行降落PCR扩增,最后将第二轮PCR产物进行电泳检测。
1.2.2.2 PCR产物的克隆转化与测序: PCR产物经琼脂糖凝胶DNA回收试剂盒纯化回收目的片段,纯化产物与pMD18-T vector(宝生物工程公司)于16 ℃连接过夜,连接产物转化DH5α感受态细胞(上海生工),然后涂布于含有Amp、IPTG和X-gal的平板上培养过夜,挑取白斑菌落于3 mL LB培养基中(Amp浓度为100 μg·mL-1),37 ℃摇12 h。提取质粒,经EcoRⅠ和Hind Ⅲ酶切鉴定,至少挑取3个阳性克隆菌液送生工生物工程(上海)有限公司测序。
1.2.3STARD3NL基因的生物信息学分析 将测序获得的序列用DNAMAN软件预测开放阅读框(Open read frame,ORF),推测氨基酸序列,并比较与其它动物STARD3NL的氨基酸序列同源性;在NCBI网站(http://www.ncbi.nlm.nih.gov)上分析保守域等;用MEGA 5.1 软件采用邻近法构建进化树。利用ExPASy(http://www.expasy.org)等网站相关程序预测蛋白质疏水性、亲水性、理化特性、二级结构、跨膜结构域,跨膜螺旋结构等信息。
1.2.4STARD3NL基因表达水平分析 分别以引物Expr-F、Expr-R和GAP-F、GAP-R在LightCycler 480荧光定量PCR仪(Roche公司)上进行qRT-PCR,以猪管家基因GAPDH为内标,分析STARD3NL基因mRNA表达水平。采用SYBR Green I 染料法,参照SYBR®Premix Ex TaqTMII(Tli RNaseH Plus)试剂盒(TaKaRa)说明书进行。
qRT-PCR反应体系:SYBR®Premix Ex TaqTMII(Tli RNaseH Plus)10 μL、cDNA(稀释10倍)1 μL、引物各0.4 μL,灭菌超纯水加至20 μL。
qRT-PCR反应程序:94 ℃ 30 s;94 ℃ 10 s,55(57) ℃ 10 s,72 ℃ 1 s,40个循环。每个循环后采集荧光生成扩增曲线。为了分析实时荧光定量PCR扩增的特异性,温度从65 ℃缓慢升温到95 ℃,连续测定样品的荧光强度以获取溶解曲线。试验对所有样本进行3个重复测定,并在每次试验时设阴性对照。根据荧光曲线的Ct值及标准曲线,以猪GAPDH基因mRNA的表达量作为内参,用2-△△Ct法计算STARD3NL基因mRNA相对表达水平。
1.2.5 统计分析 采用SAS version 8.2 统计软件分析STARD3NL基因表达水平,用单因子方差分析和邓肯氏多重比较分析平均数之间的差异显著性。当P<0.05时,为差异显著。
2 结 果
2.1STARD3NL基因cDNA序列和氨基酸序列分析
以莱芜猪(对照组)肝cDNA为模板,用Stard-F和Stard-R引物扩增出1条特异带,长度约为1 100 bp左右(图1A),用5′和3′-RACE引物各扩增出一条特异带,长度分别为250和275 bp左右(图1B)。用DNAMAN软件对序列拼接和分析,结果表明,所扩增的猪STARD3NL基因序列长度为1 319 bp (GenBank No.:HQ634946),包括STARD3NL基因开放阅读框(708 bp)、5′UTR(128 bp)和3′UTR(483 bp)序列。该序列与GenBank上登录的人(BC003074.2)、大鼠(BC086352.1)、小鼠(BC003334.1)、牛(BC120111.1)STARD3NL基因cDNA核苷酸序列有很高的相似性。猪STARD3NAL基因编码235个氨基酸,与人、黑猩猩、小鼠、大鼠、牛、绵羊、狗、鸡、非洲爪蟾和斑马鱼STARD3NL氨基酸序列的一致性分别为97.45%、97.45%、94.89%、95.74%、99.57%、99.15%、97.87%、82.63%、68.22%和65.25%。这些结果表明,STARD3NL蛋白在哺乳动物之间具有高度保守性。根据猪和其他动物STARD3NL氨基酸序列,使用MEGA 5.1 软件采用邻近法构建分子进化树。结果表明,猪与牛和绵羊的STARD3NL分子亲缘关系最近,与斑马鱼STARD3NL分子亲缘关系相对较远(图2)。
2.2 STARD3NL蛋白保守域分析和理化性质预测2.2.1 STARD3NL蛋白保守域分析 在NCBI网站上 Blast检索分析STARD3NL蛋白保守结构域。结果显示,莱芜猪STARD3NL氨基酸序列包含保守的MENTAL结构域,符合蛋白的特点(图3)。2.2.2 STARD3NL蛋白的理化性质和结构预测 在ExPASy服务器上使用SOSUI程序进行疏水性分析,结果显示,莱芜猪STARD3NL蛋白平均疏水度(Average of hydrophobicity)为0.101 7,该蛋白是疏水蛋白(图4)。ProtScale程序预测到STARD3NL蛋白的理论等电点(pI)为4.64,分子量为26 713.60 u。用TMHMM程序预测到STARD3NL蛋白的4个跨膜螺旋区(图5),4个跨膜螺旋的位置分别在STARD3NL蛋白序列中的第55~74、94~116、123~142、152~171位氨基酸残基处。用SOPMA 程序预测STARD3NL二级结构为:α-螺旋占60%,延伸链占11.91%,β-转角占4.26%,无规卷曲占23.83%。用SignaIP 4.1 Server预测信号肽发现该蛋白不是分泌蛋白,不存在信号肽。利用工具PSORTII预测STARD3NL蛋白的细胞器定位,其分布概率:内质网(Endoplasmic reticulum)39.1%,质膜(Plasma membrane)21.7%,线粒体(Mitochondrial)17.4%,细胞外(Extracellular)4.3%,囊泡分泌系统(Vesicles of secretory system)4.3%,细胞核(Nuclear)4.3%,液泡(Vacuolar)4.3%,高尔基体(Golgi)4.3%。
2.3STARD3NL基因mRNA表达水平分析2.3.1STARD3NL基因在不同组织的表达谱 以莱芜猪(对照组)14种组织的cDNA为模板,分别进行STARD3NL和GAPDH基因qRT-PCR分析。结果表明,STARD3NL基因mRNA在14种组织均有表达(图6)。STARD3NL基因mRNA在肝中的表达水平最高,在眼肌中的表达水平最低,在肺、肾、胃、背膘、脾、脊髓、胆囊、大肠、小肠组织中呈现中等表达水平;并且在肝中的表达水平显著高于在膀胱、心、骨骼肌和眼肌中的表达水平(P<0.05)。
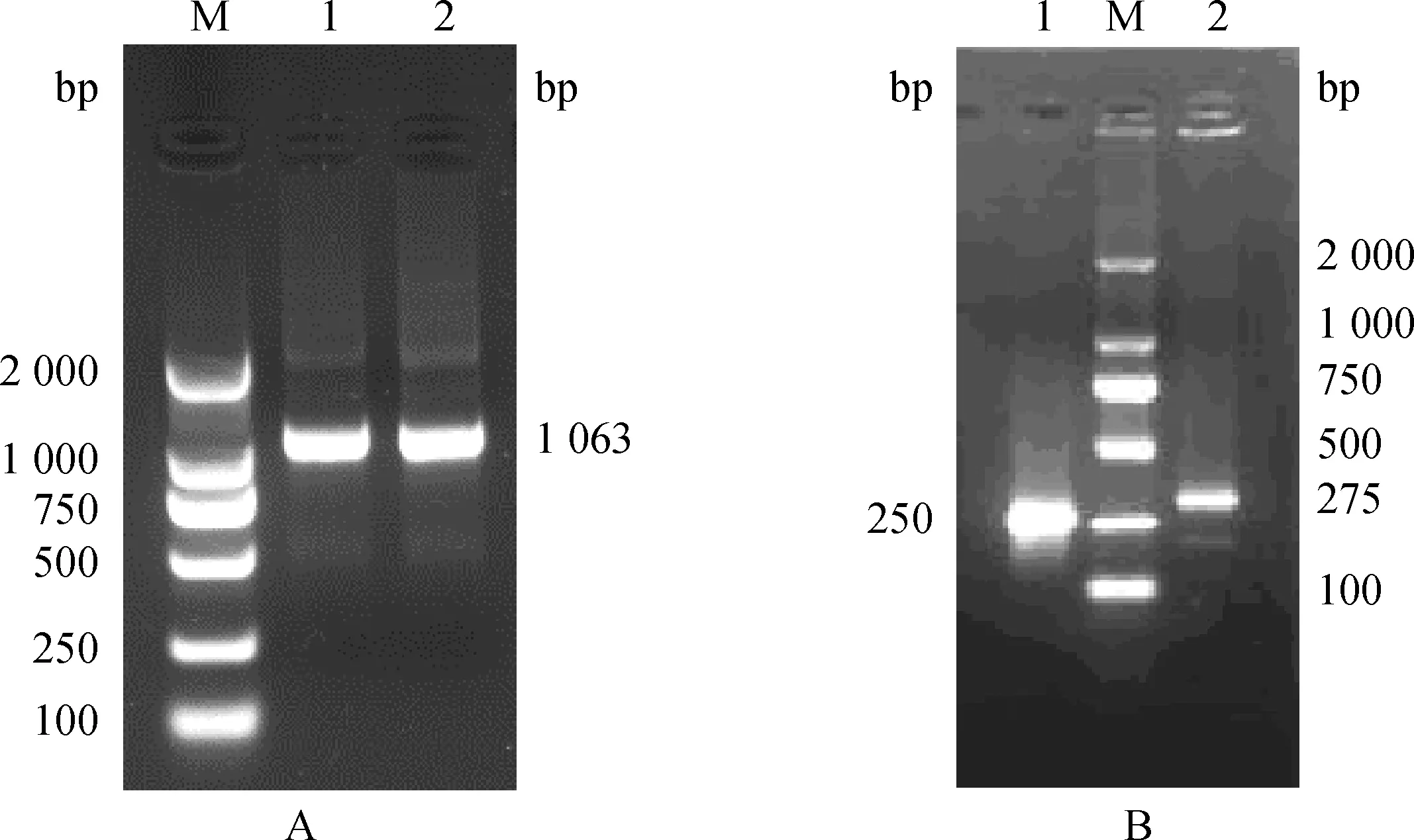
M.DNA 相对分子质量标准DL2000;A.Stard-F和Stard-R引物扩增产物(1和2泳道);B.5′(1泳道)和3′(2泳道)-RACE的第二轮PCR扩增产物 M.DL2000 DNA marker;A.Represents products of primer Stard-F and Stard-R(1 and 2 lane);B .Represents PCR products of second amplification according to 5′(1 lane) and 3′(2 lane) -RACE kit’s instructions图1 猪STARD3NL基因PCR产物电泳图Fig.1 Agarose gel electrophoresis patterns of the porcine STARD3NL gene PCR products

0.05代表遗传距离,每个节点的数字表示1 000次重复后的靴带百分比The scale bar is 0.05 representing genetic distance.The number at each node indicates the percentage of bootstrapping after 1 000 replications图2 邻近法构建的猪STARD3NL氨基酸系统发生树Fig.2 Phylogenetic tree of STARD3NL constructed by neighbor-joining(NJ) method using Mega 5.1

图3 STARD3NL蛋白包含保守的MENTAL结构域Fig.3 Conserved MENTAL domain of the porcine STARD3NL protein
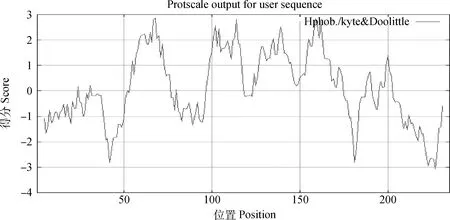
图4 猪STARD3NL蛋白的疏水性/亲水性预测Fig.4 Prediction of hydrophobicity/hydrophilicity of the porcine STARD3NL

图5 STARD3NL蛋白跨膜结构预测Fig.5 Transmembrane prediction of the porcine STARD3NL protein by TMHMM tool

数据代表“平均数±标准误”(n=4)。上标不同小写字母表示差异显著(P<0.05)Data are represented as “means ±SE”(n=4).The lowercase superscripts indicate statistical difference among different tissues(P<0.05)图6 猪STARD3NL基因mRNA表达谱Fig.6 The STARD3NL gene mRNA expression patterns in different tissues of porcine
2.3.2 猪圆环病毒对STARD3NL基因表达的影响 分别以对照组莱芜猪(LW)、大长猪(YL)和攻毒组莱芜猪(VLW)、大长猪(VYL)肺组织cDNA为模板,进行STARD3NL和GAPDH基因qRT-PCR分析。结果表明,不论是莱芜猪还是大长猪被感染圆环病毒后,STARD3NL基因的表达水平均高于不感染圆环病毒的同日龄猪(图7A、7B),但差异均不显著(P>0.05),说明猪在圆环病毒作用下STARD3NL基因上调。不同猪种之间比较发现,对照组莱芜猪肺组织STARD3NL基因的表达量高于对照组大长猪(图7C),同样,感染圆环病毒后也是莱芜猪高于大长猪(图7D)。尽管如此,差异均没达到显著水平(P>0.05)。
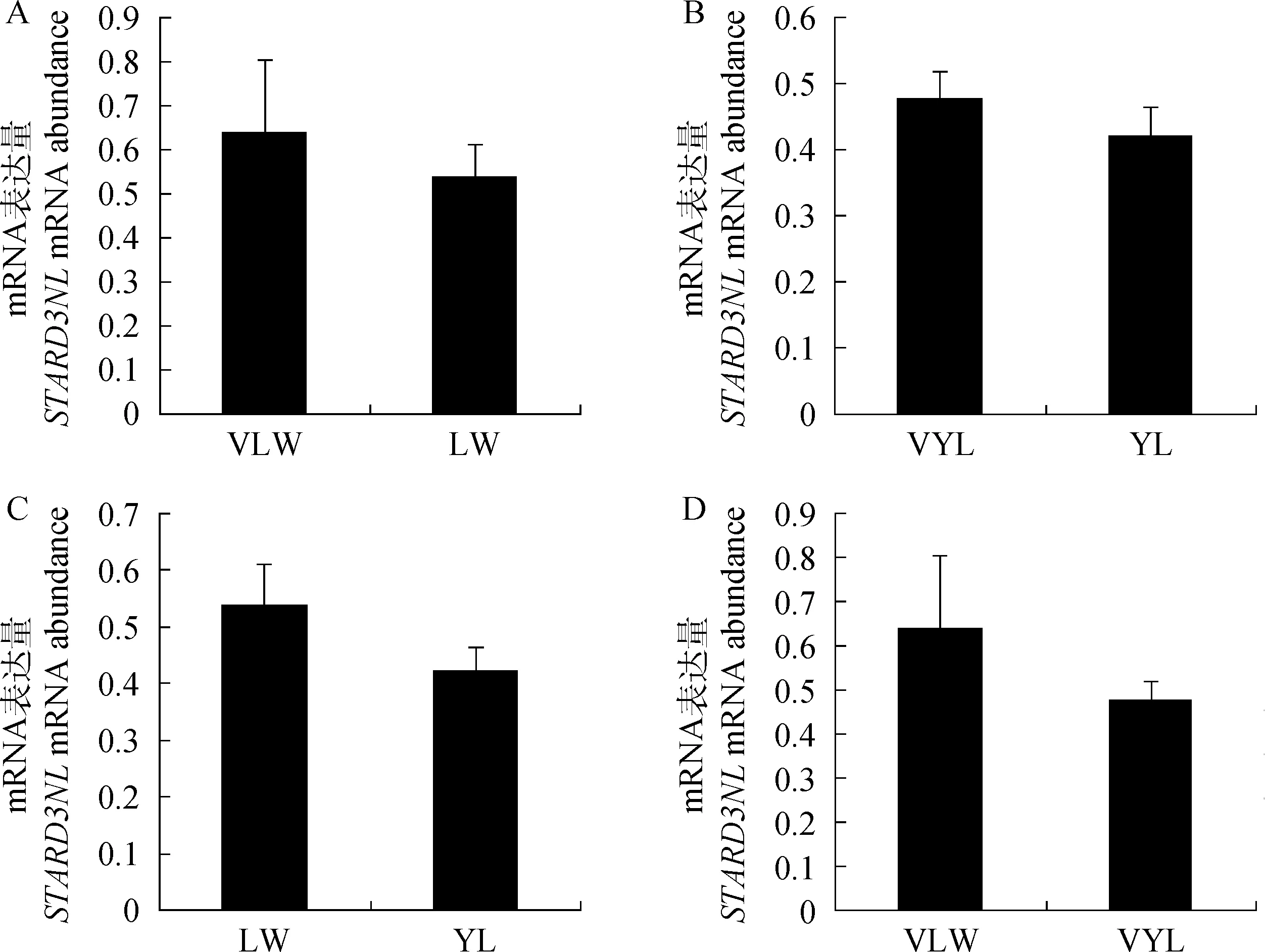
数据代表“平均数±标准误”(n=4)Data are represented as “means±SE”(n=4)图7 猪圆环病毒对STARD3NL基因mRNA表达水平的影响Fig.7 The effects of porcine circovirus type 2 on mRNA expression levels of STARD3NL gene
3 讨 论
本研究克隆得到了莱芜猪STARD3NL基因cDNA序列(GenBank登录号:HQ634946),长度为1 319 bp。此序列包含STARD3NL基因完整开放阅读框、5′UTR 和3′UTR序列。该基因核苷酸序列以及所编码的氨基酸序列与人、小鼠、大鼠、牛、狗等动物核苷酸序列和氨基酸序列具有很高的一致性,并且猪STARD3NL 蛋白序列符合其家族蛋白的典型特征。另外,在莱芜猪14种组织均检测到STARD3NL基因的表达,与STARD3NL基因在人体内组织具有广泛表达特性一致[2]。在细胞水平的研究表明,STARD3NL过表达可抑制晚期核内小体小管的产生,但不能调整胆固醇在纤维原细胞的积累[1,3,5]。研究发现STARD3NL基因可能与免疫系统有关[4],因此,为证明不同猪种被PCV2感染后是否存在STARD3NL基因表达水平的差异,本研究对莱芜猪和大长猪感染PCV2后肺组织中STARD3NL基因的表达水平也进行了分析,但差异不显著,推测STARD3NL基因可能与PCV2无关,其原因尚需进一步研究。总之,本研究丰富了猪基因组数据库,为进一步研究STARD3NL基因的功能和结构奠定了基础。
F.Alpy等研究发现,STARD3NL蛋白与STARD3蛋白在N端含有共同MENTAL的结构域[1],并以此给其命名。MENTAL结构域中的4个跨膜螺旋使它们定位在晚期核内体膜上,免疫双杂交试验也证明了该蛋白在细胞中的位置[1]。后来证实STARD3NL和STARD3可在生理条件下形成同源或异源低聚物[2],这可能是两者行驶功能的结构基础。最近发现,STARD3或者STARD3NL和VAP(突触小泡缔合性膜蛋白相关蛋白)在晚期核内体和内质网之间能形成一种新型的分子链[5]。
哺乳动物START类蛋白参与多种生理过程,如细胞内的脂质运输,脂质的代谢和信号的调节。START类蛋白的突变或缺失与遗传紊乱、自身免疫病和癌症等病理过程有联系[4]。K.Simons等研究证明,STARD3在胆固醇从低密度脂蛋白(LDL)到晚期核内体的运输过程中起转运蛋白的功能[6]。此外,STARD3还通过调控由肌动蛋白介导的细胞器的动态变化,来促使固醇积聚并锚定在膜蛋白上[7]。研究证明StAR及其相关结构域在细胞内脂质的运输机制中起重要作用[4,8-12]。由于STARD3在乳腺癌中存在过表达[13-16],所以STARD3的表达可作为乳腺癌诊断依据。
目前对STARD3NL蛋白的确切功能及其作用机制尚不清楚。有试验表明,STARD3NL的过表达会导致细胞内巨型的核内体的积累,但是这种积累对动物的生理生化以及发育的影响还不是太清楚[1]。STARD3的START结构域定点突变并未导致固醇代谢的明显变化[10],这说明START结构域在固醇代谢过程中并非不可或缺,但从另一角度证明C端缺少START结构域并不影响STARD3NL在固醇代谢过程中起重要作用。W.F.Li等人认为STARD3NL可作为检测骨质疏松症的候选基因[17]。尽管如此,猪STARD3NL基因研究刚刚起步,其功能尚需进一步研究。
[1] ALPY F,WENDLING C,RIO M C,et al.MENTHO,a MLN64 Homologue devoid of the START domain[J].JBiolChem,2002,277(52):50780-50787.[2] ALPY F,TOMASETTO C.MLN64 and MENTHO,two mediators of endosomal cholesterol transport[J].BiochemSocTrans,2006,34(Pt 3):343-345.
[3] ALPY F,LATCHUMANAN V K,KEDINGER V,et al.Functional characterization of the MENTAL domain[J].JBiolChem,2005,280(18):17945-17952.
[4] ALPY F,TOMASETTO C.Give lipids a START:the StAR-related lipid transfer(START) domain in mammals[J].JCellSci,2005,118(Pt 13):2791-2801.
[5] ALPY F,ROUSSEAU A,SCHWAB Y,et al.STARD3 or STARD3NL and VAP form a novel molecular tether between late endosomes and the ER[J].JCellSci,2013,126(Pt 23):5500-5512.
[6] SIMONS K,IKONEN E.How cells handle cholesterol[J].Science,2000,290(5497):1721-1726.
[8] REITZ J,GEHRIG-BURGER K,STRAUSS III J F,et al.Cholesterol interaction with the related steroidogenic acute regulatory lipid-transfer(START) domains of StAR(STARD1) and MLN64(STARD3)[J].FEBSJ,2008,275(8):1790-1802.
[9] NAKABAYASHI M,SHIBATA N,KOMORI H,et al.Structure of a conserved hypothetical protein,TTHA0849 from Thermus thermophilus HB8,at 2.4 a resolution:a putative member of the StAR-related lipid-transfer(START) domain superfamily[J].ActaCrystallogrF,2005,61:1027-1031.
[10] ROMANOWSKI M J,SOCCIO R E,BRESLOW J L,et al.Crystal structure of theMusmusculuscholesterol-regulated START protein 4(StarD4) containing a StAR-related lipid transfer domain[J].ProcNatlAcadSciUSA,2002,99(10):6949-6954.
[11] TSUJISHITA Y,HURLEY J H.Structure and lipid transport mechanism of a StAR-related domain[J].NatStructBiol,2000,7(5):408-414.
[12] KISHIDA T,KOSTETSKII I,ZHANG Z,et al.Targeted mutation of the MLN64 START domain causes only modest alterations in cellular sterol metabolism[J].JBiolChem,2004,279(18):19276-19285.
[13] ALPY F,BOULAY A,MOOG-LUTZ C,et al.Metastatic lymph node 64(MLN64),a gene overexpressed in breast cancers,is regulated by Sp/KLF transcription factors[J].Oncogene,2003,22(24):3770-3780.
[15] KAURANIEMI P,BRLUND M,MONNI O,et al.New amplified and highly expressed genes discovered in the ERBB2 amplicon in breast cancer by cDNA microarrays[J].CancerRes,2001,61(22):8235-8240.
[16] TOMASETTO C,RÉGNIER C,MOOG-LUTZ C,et al.Identification of four novel human genes amplified and overexpressed in breast carcinoma and localized to the q11-q21.3 region of chromosome 17[J].Genomics,1995,28(3):367-376.
[17] LI W F,HOU S X,YU B,et al.Genetics of osteoporosis:accelerating pace in gene identification and validation[J].HumGenet,2010,127(3):249-285.
(编辑 郭云雁)
Molecular Cloning and Expression Patterns Analysis of the Porcine STARD3 N-terminal like Protein(STARD3NL) Gene
XING Jin-yi1*,JIA Kun-hang1,LI Yan-ping2
(1.CollegeofLifeScience,LinyiUniversity,Linyi276005,China;2.CollegeofAnimalScienceandVeterinaryMedicine,ShandongAgriculturalUniversity,Tai’an271018,China)
The aim of this study was to clone porcineSTARD3NAL(STARD3 N-terminal like protein),and analyze the structure ofSTARD3NAL,and investigate its expression in porcine different tissues.The porcineSTARD3NALgene was cloned by 5′,3′-RACE and RT-PCR,the protein structure of porcine STARD3NL was analyzed by bioinformatics methods.The expression patterns in different tissues were detected by real-time RT-PCR.The results showed that the porcineSTARD3NLcDNA was 1 319 bp(GenBank accession No.:HQ634946) in length,which coded 235 amino acids and shared highly identity with those of other species.The phylogenetic tree analysis indicated the porcine STARD3NL was closely related to cattle and sheep,but distantly related to the zebrafish.The porcine STARD3NL protein contained conserved MENTAL domain and was hydrophobicity protein and there were 4 transmembrane helices in the STARD3NL protein.The secondary structure of STARD3NL protein showed that the STARD3NL protein fold for 60% into α-helix.This polypeptide contained no signal peptide in its amino acids,and was a nonsecretory protein.The residing probability of STARD3NL in the endoplasmic reticulum was 39.1%.By real-time PCR,mRNA expression of porcineSTARD3NLwere detected in all 14 tissues investigated,andSTARD3NLmRNA expression levels in liver were the highest and inlongissimusdorsiwere the lowest.In addition,no significant difference ofSTARD3NLmRNA expression levels in lung tissues was detected between Laiwu pigs(LW) and YL pigs(Yorshire♂×Landrace♀) in infected circovirus group(P>0.05).These results will provide the data for further research ofSTARD3NLgene structures and function,breeding for disease resistance.
porcine;STARD3NLgene;cloning;porcine circovirus type 2
10.11843/j.issn.0366-6964.2015.09.026
2014-09-28
山东省自然科学基金(ZR2013CL012);国家自然科学基金(31372333);临沂大学博士启动基金(BS08019)
邢晋祎(1968-),男,山东曹县人,博士,副教授,主要从事动物分子遗传学研究
*通信作者:邢晋祎,E-mail:xingjinyi@lyu.edu.cn
S828;S813.3
A
0366-6964(2015)09-1678-08
