京海黄鸡体组成性状的全基因组关联分析
2015-03-22樊庆灿张向前张跟喜王金玉顾玉萍
张 涛,樊庆灿,张向前,张跟喜,王金玉 *,顾玉萍
(1.扬州大学动物科学与技术学院,扬州 225009;2.江苏省动物遗传繁育与分子设计重点实验室,扬州 225009; 3.江苏京海集团,南通 226103)
京海黄鸡体组成性状的全基因组关联分析
张 涛1,2,樊庆灿1,2,张向前1,2,张跟喜1,2,王金玉1,2 *,顾玉萍3
(1.扬州大学动物科学与技术学院,扬州 225009;2.江苏省动物遗传繁育与分子设计重点实验室,扬州 225009; 3.江苏京海集团,南通 226103)
为了获得影响京海黄鸡体组成性状的SNPs标记及候选基因,为京海黄鸡的进一步遗传改良提供新的方法,本研究使用Illumina公司的鸡60K SNP芯片,对京海黄鸡13个体组成性状进行全基因组关联分析。共筛选出13个与体组成性状达到5%Bonferroni全基因组显著相关的SNPs(P<1.8E-6),130个达到5%Bonferroni全基因组潜在相关的SNPs(P<3.59E-5)。13个显著SNPs位于GRIK1、NCAPG、KCNIP4和CACNA2D2等12个基因的附近或内部,其中6个SNPs位于4号染色体上一个1.6 Mb区域(74.3~75.9 Mb)。130个潜在显著的SNPs中,有25个集中分布在4号染色体上的一个7.4 Mb(71.5~78.9 Mb)的区域内。共构建了5 650种单倍型,其中,14个与京海黄鸡6个体组成性状显著相关,14个单倍型中,9个位于4号染色体74.3~75.9 Mb区域内,该区域内包括LCORL、QDPR、KCNIP4、LDB2和FAM184B在内的多个功能基因。本研究结果表明,位于4号染色体的71.5~78.9 Mb区域以及该区域附近的GRIK1、NCAPG、KCNIP4、CACNA2D2、LCORL、QDPR、KCNIP4、LDB2和FAM184B基因对京海黄鸡的体组成有重要影响。
京海黄鸡;全基因组关联分析;体组成;单倍型
体组成性状是鸡的重要经济性状,直接影响鸡的产业化生产。通过传统育种对体组成进行的遗传改良取得了良好效果,然而,鸡体组成性状为复杂数量性状,传统育种方法已很难使体组成性状取得较大的遗传进展。近些年来,随着分子标记辅助育种的快速发展,其已成为改良遗传性状的新方法,体组成和肉质性状分子标记方面的研究已经取得令人瞩目的进展,发现了许多影响鸡体组成和肉质性状的候选基因和QTLs[1-5]。
以往主要采用PCR-RFLP和PCR-SSCP方法来鉴定与鸡不同性状相关的SNPs,但这两种方法存在明显的局限性,尤其是对于复杂数量性状而言。目前,全基因组关联分析研究(GWAS)在筛选人类和动物复杂性状相关候选基因的方面得到了广泛的应用[6-13]。X.Gu等[6]使用Illumina公司鸡的60K SNP芯片对隐性白羽肉鸡和丝羽乌骨鸡F2杂交群体的体重性状和日增重性状进行了全基因组关联分析,发现4号染色体上71.6~80.2 Mb是影响鸡生长性状SNPs的集中分布区域。L.Xie 等[7]使用鸡Illumina 60 K SNP芯片,对白洛克和杏花鸡的F2代杂交群体的体重性状和日增重进行了全基因组关联分析,发现鸡1号染色体的173.5~175.0 Mb是与鸡的体重和日增重性状关联显著的SNPs集中区域,该区域存在极为明显的连锁不平衡现象。W.Liu等[8]利用Illumina 60 K SNP芯片,以矮小型褐壳蛋鸡和白来杭为试验动物,研究鸡的繁殖性状和蛋品质,结果发现了8 个与鸡的繁殖性状和蛋品种性状关联显著的SNPs(P<0.05)和部分潜在显著的SNPs。除了以上研究找到的与生长繁殖等性状相关的SNPs外,鸡无尾、体组成及肉品质、抗马立克及新城疫等性状相关的一些位点也通过GWAS被找到[9-13]。
京海黄鸡是本课题组在长期开展我国地方鸡种质资源调查、评价与保护的基础上,经多年连续攻关成功培育而成的我国目前唯一通过国家畜禽遗传资源委员会审定的具有小型、优质、早熟和抗逆四大特点的新鸡种,京海黄鸡的育成,即采用了常规育种方法,又采用了分子辅助选择方法,是鸡育种中理论与实践相结合的一个范例。在本研究中,为了找到与京海黄鸡体组成性状显著相关的SNPs位点及影响体组成性状的候选基因,使用Illumina 公司的60K SNP 芯片对体组成性状进行全基因组关联分析。旨在发现与鸡体组成性状相关的候选基因和区域,为京海黄鸡标记辅助选择工作的开展奠定基础。
1 材料与方法
1.1 试验群体及表型检测
试验群体为212只来自同一批次19个半同胞家系的京海黄鸡,同一天孵化,笼养,自由采食和饮水,饲料符合[NRC]国际标准,所有鸡均健康。66周龄采集血液,并屠宰分割称重,记录其活重(BW)、屠体重(CW)、脚重(FW)、翅重(WW)、胸肌重(BMW)、腿肌重(LMW)、腹脂重(AW)、全净膛重(EW)和半净膛重(SEW),同时计算胸肌率(BMP)、腿肌率(LMP)、腹脂率(AWP)和全净膛率(EWP)。采用SPSS19.0软件对体组成性状进行统计分析(表1),数据使用Minitap(v16)软件中的Johnson法转换以符合正态分布。
1.2 试验方法
1.2.1 基因分型 使用上海生工生物工程(上海)股份有限公司的Dzup Genomic DNA Isolation Reagent(Blood) kit 提取基因组DNA,提取后经分光光度法检测浓度和质量,于-20 ℃保存,然后送至加拿大DNA LandMarks 股份有限公司使用60K SNP 芯片进行基因分型,具体步骤:将鸡DNA 浓度统一稀释为 50 ng·μL-1。保证基因组DNA在250 ng以上;加入 0.1 mol·L-1NaOH 使 DNA双链变为单链,加入中和剂中和,再加入全基因组扩增试剂, 37 ℃恒温过夜孵育;对扩增后产物进行过程利用终点式(End-point)片段化方法片段化;加入异丙醇使DNA 片段沉淀,4 ℃离心;将杂交缓冲液加入沉淀后的DNA中,使DNA完全溶解在杂交缓冲液中;将杂交缓冲液中的 DNA 与鸡60 K SNP芯片在杂交炉中杂交过夜;冲洗芯片以除去多余的DNA。以检测到的片段为模板,进行处理过的单碱基延伸反应,加入标签基团,以区分检测到的SNP 类型;使用XC4 试剂对反应后的芯片进行包被,真空条件下干燥1 h。iScan 芯片扫描仪扫描芯片的标签基因,使用 GenomeStudio 软件分析扫描后的图片,获得分型结果。
表1 体组成性状的描述性统计
Table 1 Descriptive statistics of body composition traits
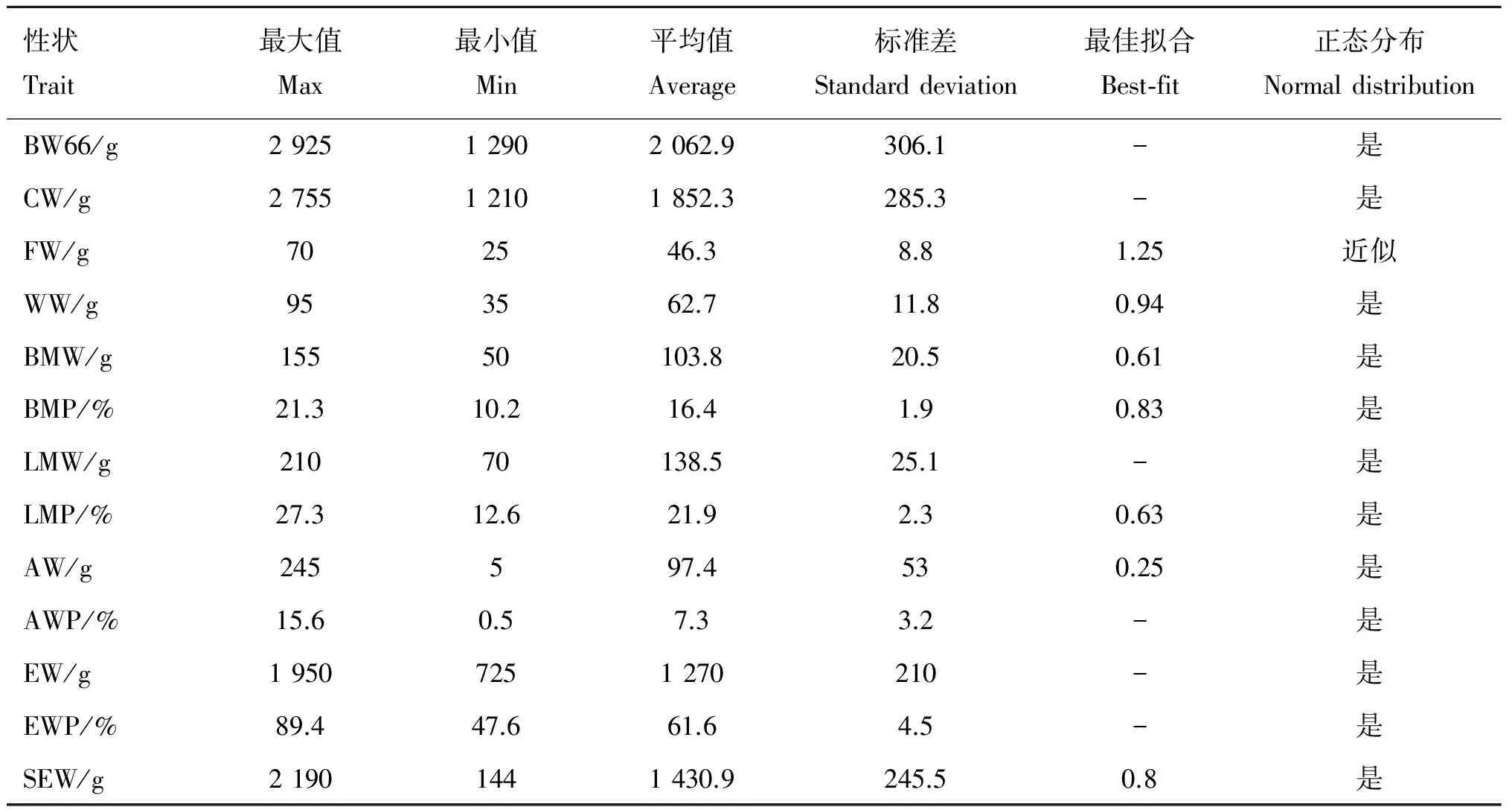
性状Trait最大值Max最小值Min平均值Average标准差Standarddeviation最佳拟合Best⁃fit正态分布NormaldistributionBW66/g292512902062.9306.1-是CW/g275512101852.3285.3-是FW/g702546.38.81.25近似WW/g953562.711.80.94是BMW/g15550103.820.50.61是BMP/%21.310.216.41.90.83是LMW/g21070138.525.1-是LMP/%27.312.621.92.30.63是AW/g245597.4530.25是AWP/%15.60.57.33.2-是EW/g19507251270210-是EWP/%89.447.661.64.5-是SEW/g21901441430.9245.50.8是
1.2.2 质量控制 使用Plink(v1.07)软件对数据进行质量控制[14],剔除最小检出率低于90%的个体以及最小检出频率低于95%、最小哈代温伯格平衡小于1.0E-6和最小等位基因频率小于3%的SNPs。1.2.3 群体结构 试验样本的群体结构使用Plink软件中的多维尺度分析法(Multidimensional scaling analysis,MDS)进行计算,具体方法是以常染色体上25个SNPs为一个SNP窗,计算内部成对SNPs的r2值,高于0.2则剔除一个标记,并每5个单位进行步移检测,最后得到12 877个独立的SNPs标记,利用找到的标记,在Plink中计算所有个体间成对的IBS(Identity-by-state distances)距离,再以IBS矩阵进行MDS分析,以第一和第二主成分作MDS图。MDS图使用R(2.15.1)软件绘制[15],使用GCTA软件进行主成分分析法(PCA),主成分PCA1和PCA2作为协变量代入模型中以减小群体分层效应[15-17]。采用Plink软件基于46 665个SNPs进行单倍型分析,具体方法为计算同一染色体上200 kb内的碱基对间的R2值,若大于0.8则认为两个SNPs为连锁。
1.3 统计分析
本研究使用一般线性回归模型(GLM)进行统计分析,模型:
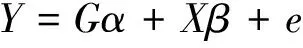
Y为观察值向量,G为遗传效应向量,X为包括第一、第二主成分(PCA1和PCA2)在内的固定效应矩阵,α和β为关联矩阵,e为随机残差向量。此外,采用GLM对单倍型和体组成性状进行关联分析。运用连锁不平衡修正的Bonferroni校正,对P值进行校正,此处独立标记计算r2值设为0.4[18],得到27 824个独立SNPs标记,因此Bonferroni校正后全基因组显著P值为1.80E-6(0.05/27 824),全基因组潜在显著P值为3.59E-5(1/27 824)。使用R(2.15.1)软件作QQ和曼哈顿图。
2 结 果
2.1 群体结构分析
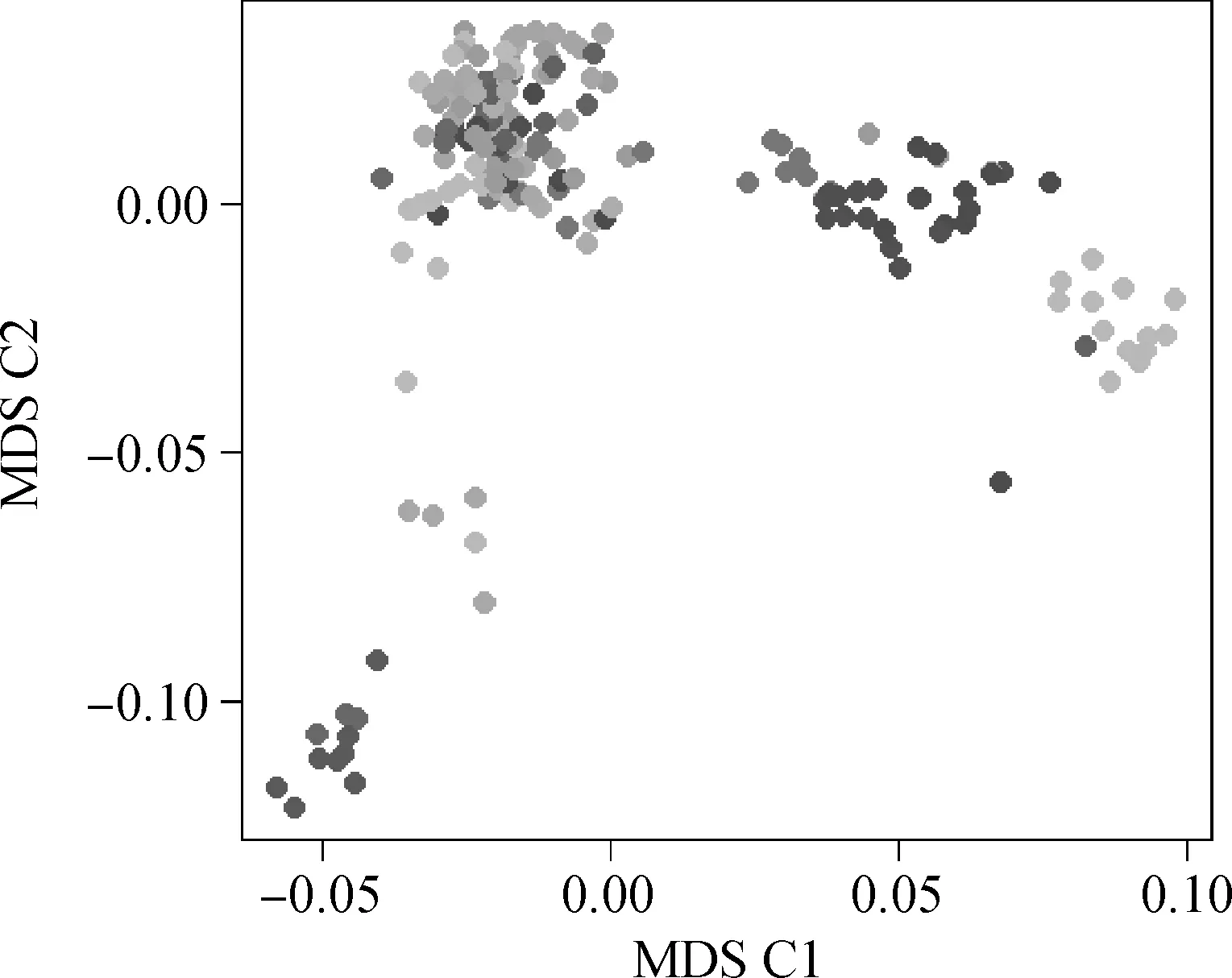
图1 多维等级分析显示的群体结构图Fig.1 Population structure identified by multidimensional scaling analysis
群体结构结果显示(图1),京海黄鸡19个半同胞家系的分布有一定的分层现象,分层现象是影响全基因组关联分析准确性的重要因素,若不考虑群体结构因素的影响,那么分层效应会被误认为是基因效应,影响结果的准确性,因此后续分析中必须使用某种方法来降低群体分层对分析结果的影响。本研究采用主成分分析法来校正群体分层,将主成分分析中的第一、第二主成分代入模型中来降低群体分层效应。
2.2 全基因组关联分析
经质控后,最终200个样本和46 665个SNPs可用于全基因组关联分析,质量控制后的SNPs分布见表2。关联分析结果显示,本研究共筛选出13个与体重、脚重、翅重、胸肌重、腿肌重、腹脂重、腹脂率、全净膛重和半净膛重达到全基因组水平显著关联(P<1.80E-6)的SNPs位点(表3)。其中6个SNPs位点位于4号染色体上74.3~75.9 Mb区域内,影响活体重、脚重、翅重、胸肌重、全净膛重和半净膛重6个体组成性状。另外一个位于4号染色体34.5 Mb处,显著影响脚重。另外3个影响脚重和翅重的SNPs 分别位于5号和7号染色体上。与胸肌重、腹脂重、腹脂率关联的6个SNPs分别位于1、2、12和15号染色体上。未筛选出与屠体重、胸肌率、腿肌率和全净膛率关联显著的位点,此外,还筛选出130个达到全基因组潜在显著的SNPs,其中,大部分位点主要位于2、4和13号染色体上,有25个位点集中分布在4号染色体71.5~78.9 Mb区域内。有显著相关位点的性状的曼哈顿图和QQ图见图2和图3。
2.3 与多个性状显著关联的SNPs位点
本研究筛选出的13个显著位点中,5个SNPs与多个性状显著相关,位于4号染色体75.5 Mb附近的rs14710787和rs16023603与7个体组成性状显著相关;而位于4号染色体75.3 Mb附近的rs14490981与活体重、脚重、全净膛重和半净膛重4个性状显著相关;此外,位于1号染色体上的rs15368284与胸肌重、腿肌重、全净膛重和半净膛重4个性状显著相关,位于2号染色体上的rs14210468与活体重、屠体重、全净膛重和半净膛重显著相关。
2.4 单倍型关联分析
本研究对5 650个单倍型和13个体组成性状进行关联分析,结果共发现了14个与活体重(1)、脚重(6)、翅重(2)、腹脂重(2)、腹脂率(2)和半净膛重(1)达到全基因组水平显著关联的单倍型(表5)。14个单倍型中,有9个单倍型分布于4号染色体74.3~75.9 Mb区域内,影响活体重、脚重、翅重和半净膛重。其他单倍型分布于13、15、21和Z染色体上,14个单倍型中,有7个单倍型是由基因组水平关联显著的SNPs组成。此外,笔者还发现与体组成性状潜在关联的单倍型58个(未列出)。
3 讨 论
全基因组关联分析(Genome-wide association study,GWAS)是以遍布于整个基因组的单核苷酸多态性为分子标记,以发现影响复杂性状发生的遗传标记和遗传标记的分布特征为目的,对复杂经济性状直接进行关联分析。全基因组关联分析被认为是一种确定影响重要经济性状或导致人类某些遗传疾病分子标记的有效方法。为了寻找影响京海黄鸡体组成性状的SNP位点,本研究采用全基因组关联分析的方法筛选与京海黄鸡体组成性状相关的SNPs标记,结果共筛选出13个与活体重、脚重、翅重、胸肌重、腿肌重、腹脂重、腹脂率、全净膛重和半净膛重达到全基因组水平显著关联(P<1.80E-6)的SNPs位点。其中,5个SNPs与多个性状显著相关,位于4号染色体75.5 Mb附近的rs14710787和rs16023603与7个体组成性状显著相关,两个SNPs均位于NCAPG基因内部。而位于4号染色体75.3 Mb附近的rs14490981与活体重、脚重、全净膛重和半净膛重4个性状显著相关。此外,位于1号染色体上的rs15368284与胸肌重、腿肌重、全净膛重和半净膛重4个性状显著相关,位于2号染色体上的rs14210468与活体重、屠体重、全净膛重和半净膛重显著相关。这些影响多个屠宰性状的SNPs也具有更高的研究价值,其所在基因可能为重要候选基因。
表2 质控后SNPs信息统计
Table 2 Basic statistics of SNPs after quality control
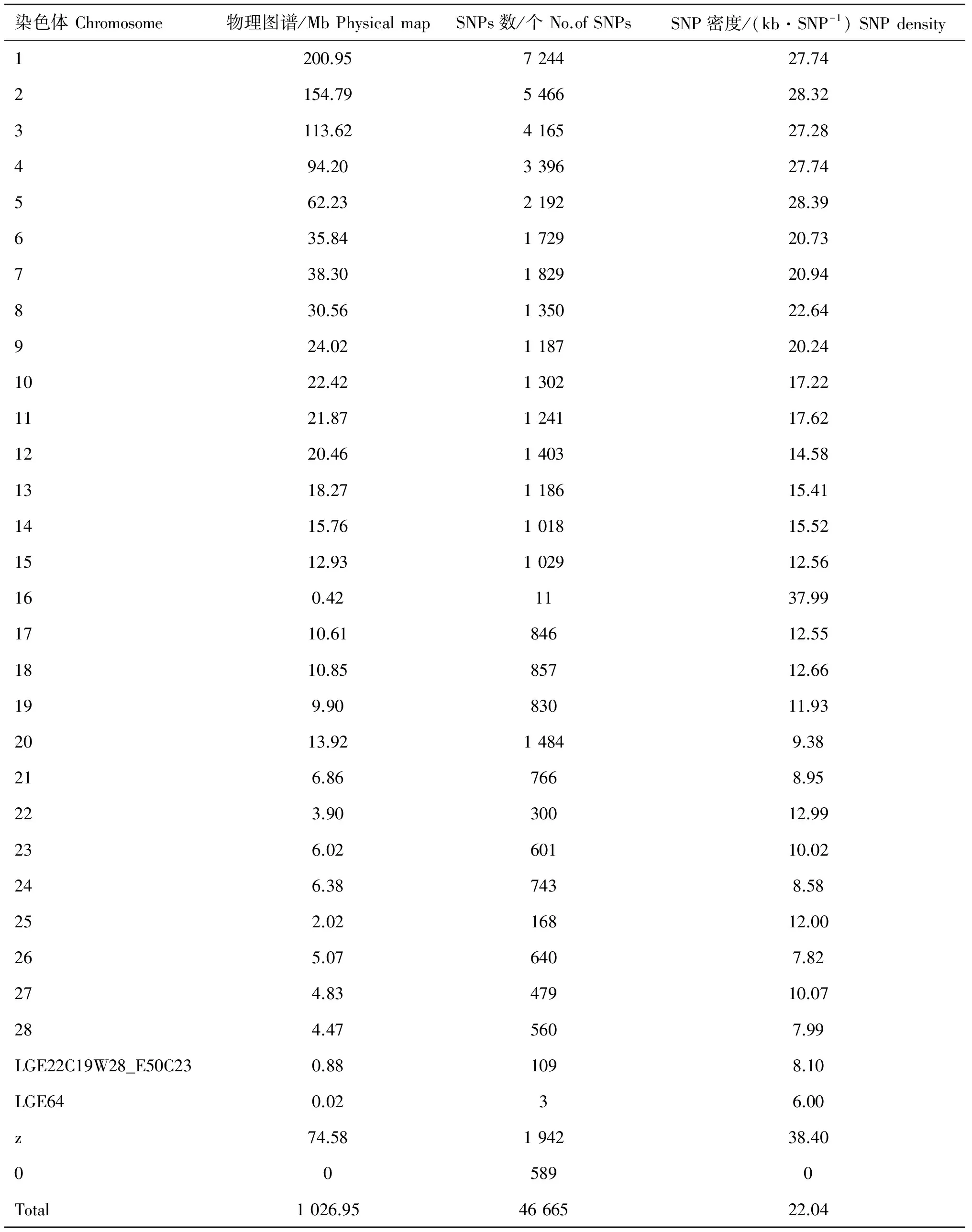
染色体Chromosome物理图谱/MbPhysicalmapSNPs数/个No.ofSNPsSNP密度/(kb·SNP-1)SNPdensity1200.95724427.742154.79546628.323113.62416527.28494.20339627.74562.23219228.39635.84172920.73738.30182920.94830.56135022.64924.02118720.241022.42130217.221121.87124117.621220.46140314.581318.27118615.411415.76101815.521512.93102912.56160.421137.991710.6184612.551810.8585712.66199.9083011.932013.9214849.38216.867668.95223.9030012.99236.0260110.02246.387438.58252.0216812.00265.076407.82274.8347910.07284.475607.99LGE22C19W28_E50C230.881098.10LGE640.0236.00z74.58194238.40005890Total1026.954666522.04
表3 京海黄鸡体组成性状的显著SNPs位点
Table 3 Significant SNPs for body composition traits in Jinghai Yellow chicken
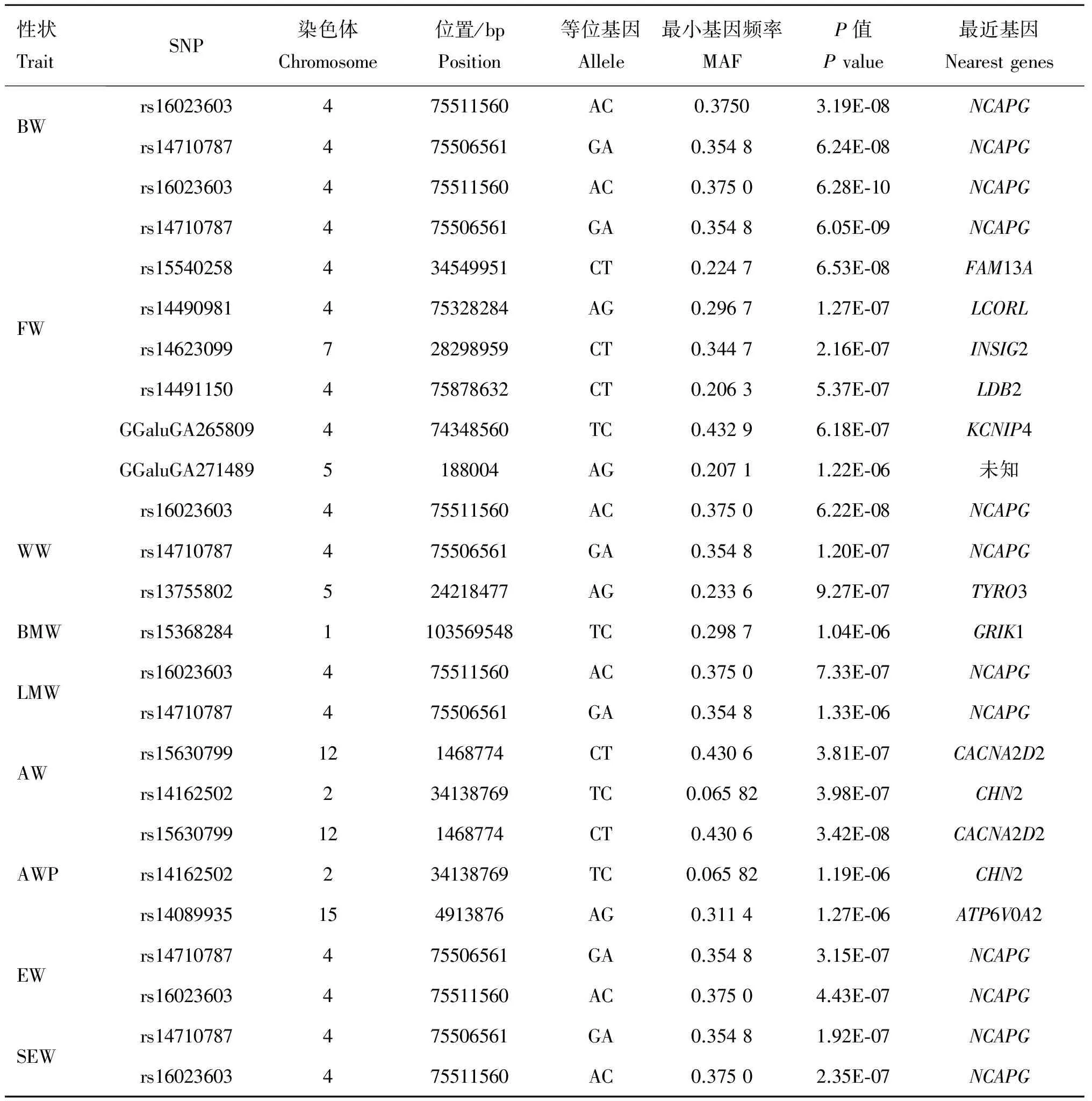
性状TraitSNP染色体Chromosome位置/bpPosition等位基因Allele最小基因频率MAFP值Pvalue最近基因NearestgenesBWrs16023603475511560AC0.37503.19E⁃08NCAPGrs14710787475506561GA0.35486.24E⁃08NCAPGFWrs16023603475511560AC0.37506.28E⁃10NCAPGrs14710787475506561GA0.35486.05E⁃09NCAPGrs15540258434549951CT0.22476.53E⁃08FAM13Ars14490981475328284AG0.29671.27E⁃07LCORLrs14623099728298959CT0.34472.16E⁃07INSIG2rs14491150475878632CT0.20635.37E⁃07LDB2GGaluGA265809474348560TC0.43296.18E⁃07KCNIP4GGaluGA2714895188004AG0.20711.22E⁃06未知WWrs16023603475511560AC0.37506.22E⁃08NCAPGrs14710787475506561GA0.35481.20E⁃07NCAPGrs13755802524218477AG0.23369.27E⁃07TYRO3BMWrs153682841103569548TC0.29871.04E⁃06GRIK1LMWrs16023603475511560AC0.37507.33E⁃07NCAPGrs14710787475506561GA0.35481.33E⁃06NCAPGAWrs15630799121468774CT0.43063.81E⁃07CACNA2D2rs14162502234138769TC0.065823.98E⁃07CHN2AWPrs15630799121468774CT0.43063.42E⁃08CACNA2D2rs14162502234138769TC0.065821.19E⁃06CHN2rs14089935154913876AG0.31141.27E⁃06ATP6V0A2EWrs14710787475506561GA0.35483.15E⁃07NCAPGrs16023603475511560AC0.37504.43E⁃07NCAPGSEWrs14710787475506561GA0.35481.92E⁃07NCAPGrs16023603475511560AC0.37502.35E⁃07NCAPG
本研究筛选出与体组成性状关联显著的13个位点主要位于4号染色体上74.3~75.5 Mb区域内,130个潜在显著地位点中有25个集中位于4号染色体71.4~78.9 Mb区域,这表明4号染色体的71.5~78.9 Mb区域对京海黄鸡体组成具有重要的调控作用,这与之前报道的生长和屠宰性状的GWAS结果是一致的。X.Gu等[6]对乌骨鸡-白洛克鸡组建的F2代资源群进行GWAS分析,发现4号染色体 71.6~80.2 Mb区域与7~14周龄体重和日增重显著相关。L.Xie等[7]对F2代资源群进行GWAS分析,在该区域内发现一些与生长性状潜在显著关联的位点,而R.Liu等[11]对北京油鸡的屠宰性状进行GWAS分析,发现78.4~79.5 Mb区域与屠体重和全净膛重关联显著。这也说明该区域对鸡生长和屠宰都具有重要的调节作用。

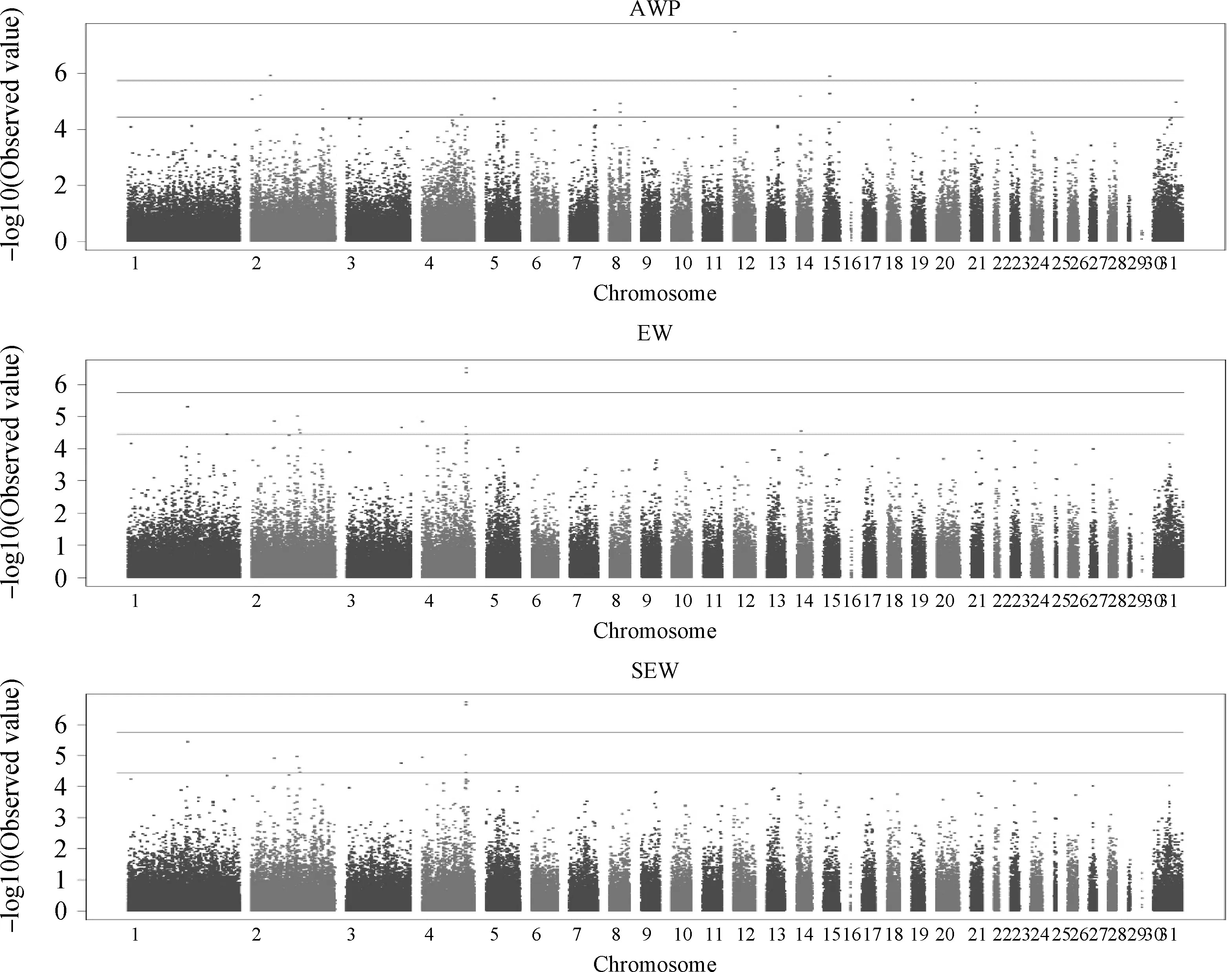
X轴1~28表示1~28号染色体,29、30和31分别代表LGE22、LGE64染色体片段和Z染色体;图中上线代表潜在显著的阈值(3.59E-5),下线代表全基因组显著的阈值(1.80E-06)1-28 on the x-axis indicate chromosomes 1-28,and 29,30 and 31 indicate LGE22,LGE64 and chromosome Z,respectively.The upper line in each figure shows the potential significant threshold:-log10(3.59E-5),and the lower one shows the genome-wide significant threshold:-log10(1.80E-06)图2 体组成性状全基因组显著位点的曼哈顿图Fig.2 Manhttan plots for the body composition triats with genome-wide significant SNPs
和半净膛重显著相关。这些影响多个屠宰性状的SNPs也具有更高的研究价值,其所在基因可能为重要候选基因。
本研究筛选出与体组成性状关联显著的13个位点主要位于4号染色体上74.3~75.5 Mb区域内,130个潜在显著地位点中有25个集中位于4号染色体71.4~78.9 Mb区域,这表明4号染色体的71.5~78.9 Mb区域对京海黄鸡体组成具有重要的调控作用,这与之前报道的生长和屠宰性状的GWAS的结果是一致的。X.Gu等[6]对乌骨鸡-白洛克鸡组建的F2代资源群进行GWAS分析,发现4号染色体 71.6~80.2 Mb区域与7~14周龄体重和日增重显著相关。L.Xie等[7]对F2代资源群进行GWAS分析,在该区域内发现一些与生长性状潜在显著关联的位点,而R.Liu[11]对北京油鸡的屠宰性状进行GWAS分析,发现78.4~79.5 Mb区域与屠体重和全净膛重关联显著。这也说明该区域对鸡生长和屠宰都具有重要的调节作用。
从NCBI和Ensembl获取每个显著SNP周围1 Mb区域内的最近基因作为影响体组成性状的可能候选基因。最终,共找到13个可能的候选功能基因,这些基因中,NCAPG基因对活重、脚重、翅重、腿肌重、全净膛重和半净膛重6个性状有显著影响,说明NCAPG基因可能为影响京海黄鸡体组成的重要候选基因。同时,GRIK1基因内的SNP对胸肌重有显著影响,CACNA2D2基因内的SNP对腹脂重和腹脂率有显著影响。以上结果表明,NCAPG、GRIK1和CACNA2D2基因可能为影响京海黄鸡体组成的重要候选基因。
NCAPG基因是编码凝缩蛋白复合体的一个亚基,该复合体在有丝分裂和减数分裂过程中,调节染色体的稳定和压缩。有报道显示,牛的NCAPG基因是影响牛体型和屠宰性状的一个新的候选基因,该基因内部的SNPs能够影响不同品种牛不同时期的体重、体型以及屠宰性状[19-24],目前该基因的研究在鸡上尚未见报道。GRIK1同时被称为GluR5,是哺乳动物主要的神经递质受体,参与多种日常神经的生理过程。GluR5和家族成员GluR6、GluR7的蛋白形成同聚体时可形成功能性的离子通道[25],该基因被认为是抗癫痫的重要候选基因[26-28]。CACNA2D2基因表达的蛋白属于L型钙离子通道复合体蛋白。L型钙离子通道复合体由α1、α2δ、β和γ亚基以1∶1∶1∶1比例组成。该复合体通过调节Ca离子转运,从而进行细胞调节[29-30]。CACNA2D2编码α2δ亚基,是α2δ蛋白的一个亚型,该基因被认为是多种肿瘤的抑制基因,此外,一些研究人员发现,另外一个亚型编码基因CACNA2D1是牛屠宰性状和肉品质性状的重要候选基因[31-33]。
图3 京海黄鸡体组成性状的QQ图Fig.3 QQ-plot of body composition traits of Jinghai Yellow chicken
表4 与超过两个体组成性状相关的SNPs
Table 4 SNPs associated with more than 2 body composition traits

名称SNPID染色体Chromosome位置/bpPosition最近基因Nearestgenes性状TraitP值Pvaluers153682841107693816GRIK1BMW1.04E⁃06LMW2.29E⁃06SEW3.71E⁃06EW5.12E⁃06rs14210468283654288LOC101750191CW2.20E⁃06EW9.81E⁃06SEW1.10E⁃05BW663.40E⁃05rs14490981478563545LOC101750905FW1.27E⁃07SEW9.59E⁃06EW2.05E⁃05BW662.07E⁃05rs14710787478797460FAM184BFW6.05E⁃09BW666.24E⁃08WW1.20E⁃07SEW1.92E⁃07EW3.15E⁃07LMW1.33E⁃06CW3.34E⁃05rs16023603478802461FAM184BFW6.28E⁃10BW663.19E⁃08WW6.22E⁃08SEW2.35E⁃07EW4.43E⁃07LMW7.33E⁃07CW1.74E⁃05BMW2.26E⁃05
本研究中,单倍型分析结果发现了14个与6个屠宰性状关联显著的单倍型,这些显著的单倍型中,有7个单倍型由显著的SNPs构建而成。其他6个关联显著的单倍型并不含有显著SNPs,这一方面证明了关联分析结果的准确性,另一方面还说明,单倍型分析综合考虑多个等位基因,能够发现SNP关联分析所不能发现的位点。此外,本研究发现的14个单倍型中,有9个单倍型位于4号染色体74.3~75.9 Mb区域内,影响活体重、脚重、翅重和半净膛重。说明4号染色体74.3~75.9 Mb是影响京海黄鸡体组成性状的一个重要区域。该区域内共发现了15个与体组成性状关联显著的SNPs,这进一步说明了该区域对鸡体组成性状的重要性。这一区域单倍型涉及到的基因有LDB2、LCORL、QDPR、KCNIP4和FAM184B。LDB2基因通过与多种转录因子结合,参与血管形成和脑的发育过程[34-35]。X.Gu等[6,36]对鸡生长性状的全基因组关联分析研究均报道了该基因。LCORL基因能够影响精子发生,该基因内的多态位点与成年人骨骼尺寸及身高关联显著[37]。吴丹[36]对鸡生长性状的全基因组关联分析中报道了该基因。QDPR基因编码二氢喋啶还原酶,该基因功能的报道较少。KCNIP4编码蛋白是一种K离子通道互作蛋白,K离子通道具有广泛的生理调节作用,包括神经递质释放、平滑肌收缩、心率调节和胰岛素分泌等[38-39]。FAM184B的研究较少,有研究表明,该基因与牛的采食、日增重和屠体重显著相关[40]。
表5 与京海黄鸡体组成性状显著相关的单倍型
Table 5 Significant haplotypes associated with body composition traits in Jinghai Yellow chicken
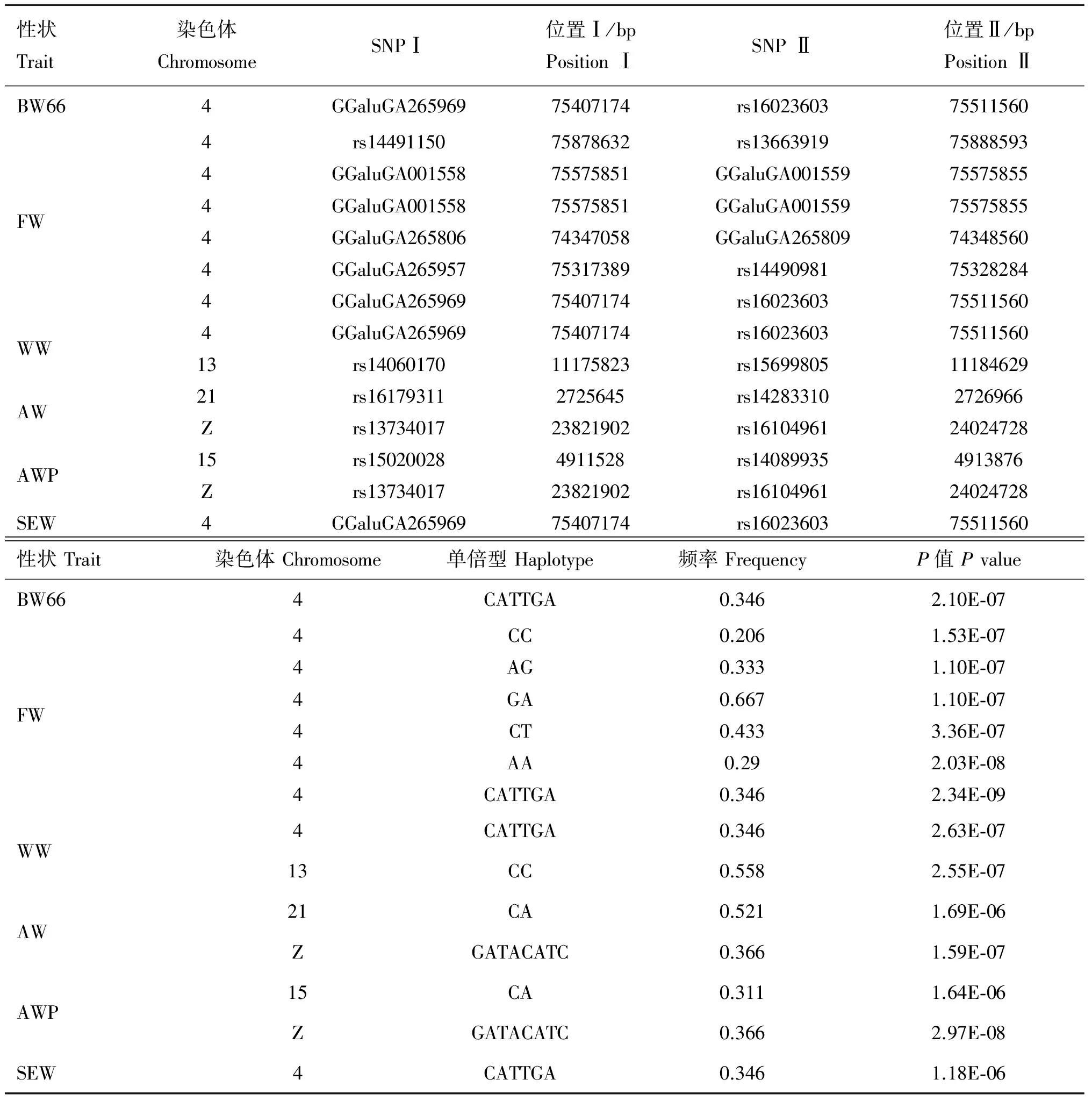
性状Trait染色体ChromosomeSNPⅠ位置Ⅰ/bpPositionⅠSNPⅡ位置Ⅱ/bpPositionⅡBW664GGaluGA26596975407174rs1602360375511560FW4rs1449115075878632rs13663919758885934GGaluGA00155875575851GGaluGA001559755758554GGaluGA00155875575851GGaluGA001559755758554GGaluGA26580674347058GGaluGA265809743485604GGaluGA26595775317389rs14490981753282844GGaluGA26596975407174rs1602360375511560WW4GGaluGA26596975407174rs160236037551156013rs1406017011175823rs1569980511184629AW21rs161793112725645rs142833102726966Zrs1373401723821902rs1610496124024728AWP15rs150200284911528rs140899354913876Zrs1373401723821902rs1610496124024728SEW4GGaluGA26596975407174rs1602360375511560性状Trait染色体Chromosome单倍型Haplotype频率FrequencyP值PvalueBW664CATTGA0.3462.10E⁃07FW4CC0.2061.53E⁃074AG0.3331.10E⁃074GA0.6671.10E⁃074CT0.4333.36E⁃074AA0.292.03E⁃084CATTGA0.3462.34E⁃09WW4CATTGA0.3462.63E⁃0713CC0.5582.55E⁃07AW21CA0.5211.69E⁃06ZGATACATC0.3661.59E⁃07AWP15CA0.3111.64E⁃06ZGATACATC0.3662.97E⁃08SEW4CATTGA0.3461.18E⁃06
4 结 论
本试验对京海黄鸡体组成性状进行了全基因组关联分析,共筛选出13个与体组成性状达到全基因组显著关联的SNPs,130个达到全基因组潜在显著地SNPs。位于4号染色体的71.5~78.9 Mb区域内有多个SNPs与多个屠宰性状关联显著,表明该区域可能为影响京海黄鸡体组成的重要候选区域,同时,在显著SNPs附近筛选出12个可能的候选基因。
[1] 程笃学.猪肉质性状的全基因组关联研究[D].北京:中国农业科学院,2012. CHENG D X.Genome-wide association study for meat trait in swine[D].Beijing:Chinese Academy of Agricultural Sciences,2012.(in Chinese)
[2] ABASHT B,LAMONT S.Genome-wide association analysis reveals cryptic alleles as an important factor in heterosis for fatness in chicken F2 population[J].AnimGenet,2007,38(5):491-498.
[3] ATZMON G,BLUM S,FELDMAN M,et al.QTLs detected in a multigenerational resource chicken population[J].JHeredity,2008,99(5):528-538.
[4] FANG M,Q NIE,C LUO,et al.Associations ofGHSRgene polymorphisms with chicken growth and carcass traits[J].MolBiolRep,2010,37(1):423-428.
[5] TANG S,OU J,SUN D,et al.A novel 62‐bp indel mutation in the promoter region of transforming growth factor‐beta 2(TGFB2) gene is associated with body weight in chickens[J].AnimGenet,2011,42(1):108-112.
[6] GU X,FENG C,MA L,et al.Genome-wide association study of body weight in chicken F2 resource population[J].PLoSONE,2011,6(7):e21872.
[7] XIE L,LUO C,ZHANG C,et al.Genome-wide association study identified a narrow chromosome 1 region associated with chicken growth traits[J].PLoSONE,2012,7(2):e30910.
[8] LIU W,LI D,LIU J,et al.A genome-wide SNP scan reveals novel loci for egg production and quality traits in white leghorn and brown-egg dwarf layers[J].PLoSONE,2011,6(12):e28600.
[9] WOLC A,ARANGO J,SETTAR P,et al.Genom-wide association analysis and genetic architecture of egg weight and egg uniformity in layer chickens[J].AnimGenet,2012,43(S1):87-96.
[10] NOORAI R E,FREESE N H,WRIGHT L M,et al.Genome-wide association mapping and identification of candidate genes for the rumpless and ear-tufted traits of the araucana chicken[J].PLoSONE,2012,7(7):e40974.
[11] LIU R,SUN Y,ZHAO G,et al.Genome-wide association study identifies loci and candidate genes for body composition and meat quality traits in Beijing-You chickens[J].PLoSONE,2013,8(4):e61172.
[12] LI D F,LIAN L,QU L J,et al.A genome-wide SNP scan reveals two loci associated with the chicken resistance to Marek’s disease[J].AnimGenet,2013,44(2):217-222.
[13] LUO C,QU H,MA J,et al.Genome-wide association study of antibody response to Newcastle disease virus in chicken[J].BMCGenet,2013,14(1):42.
[14] PURCELL S,NEALE B,TODD-BROWN K,et al.PLINK:a tool set for whole-genome association and population-based linkage analyses[J].AmJHumGenet,2007,81(3):559-575.
[15] WANG D,SUN Y,STANG P,et al.Comparison of methods for correcting population stratification in a genome-wide association study of rheumatoid arthritis:principal- component analysis versus multidimensional scaling[J].BMCProceed,2009,3(Suppl 7):S109.
[16] WALL M E,A RECHTSTEINER,L M ROCHA.Singular value decomposition and principal component analysis.A practical approach to microarray data analysis[M].US:Springer,2003:91-109.
[17] RAMOS A M,CROOIJMANS R P,AFFARA N A,et al.Design of a high density SNP genotyping assay in the pig using SNPs identified and characterized by next generation sequencing technology[J].PLoSONE,2009,4(8):e6524.
[18] JOHNSON R C,NELSON G W,TROYER J L,et al.Accounting for multiple comparisons in a genome-wide association study(GWAS)[J].BMCGenomics,2010,11:724.
[19] EBERLEIN A,TAKASUGA A,SETOGUCHI K,et al.Dissection of genetic factors modulating fetal growth in cattle indicates a substantial role of the non-SMC condensin I complex,subunit G(NCAPG) gene[J].Genetics,2009,183(3):951-964.
[20] GLENSKE K,BRANDT H,ERHARDT G.Association of the Ile-442-Met substitution inNCAPGwith birth weight in German Angus and German Simmental cattle(Brief Report)[J].ArchivfurTierzucht-ArchAnimBreed,2011,54(1):104-106.
[21] PAUSCH H,FLISIKOWSKI K,JUNG S,et al.Genome-wide association study identifies two major loci affecting calving ease and growth-related traits in cattle[J].Genetics,2011,187(1):289-297.
[22] SETOGUCHI K,FURUTA M,HIRANO T,et al.Cross-breed comparisons identified a critical 591-kb region for bovine carcass weight QTL(CW-2) on chromosome 6 and the Ile-442-Met substitution in NCAPG as a positional candidate[J].BMCGenet,2009,10(1):43.
[23] SETOGUCHI K,WATANABE T,WEIKARD R,et al.The SNP c.1326T> G in the non‐SMC condensin I complex,subunit G(NCAPG) gene encoding a p.Ile442Met variant is associated with an increase in body frame size at puberty in cattle[J].AnimGenet,2011,42(6):650-655.
[24] SORANZO N,RIVADENEIRA F,CHINAPPEN-HORSLEY U,et al.Meta-analysis of genome-wide scans for human adult stature identifies novel Loci and associations with measures of skeletal frame size[J].PLoSGenet,2009,5(4):e1000445.
[25] MAYER M L.Crystal structures of theGluR5 andGluR6 ligand binding cores:molecular mechanisms underlying kainate receptor selectivity[J].Neuron,2005,45(4):539-552.
[26] MULLE C,SAILER A,PÉREZ-OTAO I,et al.Altered synaptic physiology and reduced susceptibility to kainate-induced seizures inGluR6-deficient mice[J].Nature,1998,392(6676):601-605.
[27] ROGAWSKI M A,GRYDER D,CASTANEDA D,et al.GluR5 kainate receptors,seizures,and the amygdala[J].AnnalsNewYorkAcadSci,2003,985(1):150-162.
[28] SMOLDERS I,BORTOLOTTO Z A,CLARKE V R,et al.Antagonists ofGLUK5-containing kainate receptors prevent pilocarpine-induced limbic seizures[J].NatNeurosci,2002,5(8):796-804.
[29] DAVIES A,HENDRICH J,VAN MINH A T,et al.Functional biology of the alpha(2)deltasubunits of voltage-gated calcium channels[J].TrendsPharmacolSci,2007,28(5):220-228.
[30] HOU G Y,YUAN Z R,GAO X,et al.Genetic polymorphisms of theCACNA2D1 gene and their association with carcass and meat quality traits in cattle[J].BiochemGenet,2010,48(9-10):751-759.
[31] BIGOS K L,MATTAY V S,CALLICOTT J H,et al.Genetic variation inCACNA1Caffects brain circuitries related to mental illness[J].ArchGeneralPsychiat,2010,67(9):939-945.
[32] GUTIéRREZ-GIL B,WILLIAMS J,HOMER D,et al.Search for quantitative trait loci affecting growth and carcass traits in a cross population of beef and dairy cattle[J].JAnimSci,2009,87(1):24-36.
[33] LI J,YANG M,XU N.Single nucleotide polymorphism in 3UTR of porcineCACNA2D1 gene and its genetic effect on meat quality traits[J].ActaAgriculturaeBoreali-Sinica,2007,22(4):76-79.
[34] JAVERZAT S,FRANCO M,HERBERT J,et al.Correlating global gene regulation to angiogenesis in the developing chick extra-embryonic vascular system[J].PLoSONE,2009,4(11):e7856.
[35] OSTENDORFF H P,TURSUN B,CORNILS K,et al.Dynamic expression of LIM cofactors in the developing mouse neural tube[J].DevDynamics,2006,235(3):786-791.
[36] 吴 丹.北京油鸡体重和屠体性状的全基因组关联研究[D].北京:中国农业科学院,2012. WU D.Genome-wide association study of loci affecting body weight and carcass traits in Beijing-You Chickens[D].Beijing:Chinese Academy of Agricultural Sciences,2012.(in Chinese)
[37] SORANZO N,RIVADENEIRA F,CHINAPPEN-HORSLEY U,et al.Meta-analysis of genome-wide scans for human adult stature identifies novel Loci and associations with measures of skeletal frame size[J].PLoSGenet,2009,5(4):e1000445.
[38] BONNE A,VREEDE L,KUIPER R P,et al.Mapping of constitutional translocation breakpoints in renal cell cancer patients:identification of KCNIP4 as a candidate gene[J].CancerGenetCytogenet,2007,179(1):11-18.
[39] HIMES B E,SHEPPARD K,BERNDT A,et al.Integration of mouse and human genome-wide association data identifies KCNIP4 as an asthma gene[J].PLoSONE,2013,8(2):e56179.
[40] LINDHOLM-PERRY A K,SEXTEN A K,KUEHN L A,et al.Association,effects and validation of polymorphisms within the NCAPG-LCORL locus located on BTA6 with feed intake,gain,meat and carcass traits in beef cattle[J].BMCGenet,2011,12(1):103.
(编辑 郭云雁)
A Genome-wide Association Study on Body Composition of Jinghai Yellow Chicken
ZHANG Tao1,2,FAN Qing-can1,2,ZHANG Xiang-qian1,2,ZHANG Gen-xi1,2, WANG Jin-yu1,2*,GU Yu-ping3
(1.CollegeofAnimalScienceandTechnology,YangzhouUniversity,Yangzhou225009,China; 2.KeyLaboratoryofAnimalGenetics,Breeding,ReproductionandMolecularDesignofJiangsuProvince,Yangzhou225009,China;3.JiangsuJinghaiPoultryGroupCo.,Ltd.,Nantong226103,China)
To scan SNPs and candidate genes affecting body composition traits and provide new methods for further genetic improvement of Jinghai Yellow chicken,a genome-wide association study on 13 body composition traits was carried out using the Illumina chicken 60K SNP Beadchip in Jinghai Yellow chicken in the present study.The result showed that a total of 13 SNPs reached 5% Bonferroni genome-wide significant level(P<1.8E-6) and 130 SNPs reached “suggestive” genome-wide significant level(P<3.59E-5) with body composition traits.The 13 significant SNPs were located nearby or in 12 candidate genes includingGRIK1,NCAPG,KCNIP4,CACNA2D2,and so on,among which 6 SNPs were located in a region approximately 1.6 Mb in length on chicken chromosome 4(74.3-75.9 Mb).Among the 130 “suggestive” significant SNPs,25 SNPs were located in a region 7.4 Mb(71.5-78.9 Mb) in length on chicken chromosome 4.5 650 haplotpyes were established and 14 of them were found to be associated with 6 body composition traits.Nine out of 14 haplotypes were located in the region of 74.3-75.9 Mb on chicken chromosome 4.Five candidate genes ofLCORL,QDPR,KCNIP4,LDB2 andFAM184Bwere located in this region.The present study suggested that genes located in the region of 71.5-78.9 Mb on chicken chromosome 4 andGRIK1,NCAPG,KCNIP4,CACNA2D2,LCORL,QDPR,KCNIP4,LDB2,FAM184Bgenes might play important role in regulation of body composition of Jinghai Yellow chicken.
Jinghai Yellow chicken;GWAS;body composition;haplotype
10.11843/j.issn.0366-6964.2015.09.004
2014-10-13
国家肉鸡产业技术体系(nycytx-42-G1-05);江苏高校优势学科建设工程;江苏省动物遗传繁育与分子设计重点实验室
张 涛(1990-),男,山东临沂人,博士生,主要从事动物遗传育种与繁殖研究,E-mail:zt991279320@126.com
*通信作者:王金玉,教授,博士生导师,E-mail:jywang@yzu.edu.cn
S831.2
A
0366-6964(2015)09-1502-13
