海南与越南缅甸蟒线粒体控制区遗传多样性及差异性分析
2015-03-20刘雅芳侯冠彧于萍赵春萍曹婷张立岭
刘雅芳 侯冠彧 于萍 赵春萍 曹婷 张立岭

摘要:为了更有效地保护、管理和繁育缅甸蟒(Python bivittatus)濒危物种,有必要利用分子技术对地方物种多样性和遗传分化进行研究。试验采用TA克隆双向测序方法对来自海南的37条缅甸蟒和来自越南的28条缅甸蟒部分控制区II序列进行了测定。结果表明,两个地理群体共分为25个单倍型,单倍型多样度(Hd)为0.877±0.025,核苷酸多样性(π)为0.004 76±0.000 41,平均核苷酸差异数(k)为2.676,显示缅甸蟒整体遗传多样性不高;通过比较两群体遗传多样性,发现海南缅甸蟒的单倍型多样度要高于越南缅甸蟒,但核苷酸多样性和平均核苷酸差异数低于越南缅甸蟒;基于控制区构建ML树和Network网络分析显示,两群体均各自形成了明显的两大分支;通过计算得到两群体的固定指数(Fst)为0.201(P<0.001),显示海南缅甸蟒与越南缅甸蟒存在极显著差异。
关键词:缅甸蟒(Python bivittatus);控制区;遗传多样性;差异性
中图分类号:Q959.6;Q78 文献标识码:A 文章编号:0439-8114(2015)02-0398-04
DOI:10.14088/j.cnki.issn0439-8114.2015.02.035
Genetic Diversity and Differentiation of Control Region Sequences in Mitochondrial Genome of Python bivittatus from Hainan and Vietnam
LIU Ya-fang1,HOU Guan-yu2,YU Ping1,ZHAO Chun-ping1,CAO Ting2,ZHANG Li-ling1,3
(1.Department of Animal Sciences, College of Agriculture, Hainan University, Haikou 570228,China;
2.Tropical Crops Genetic Resources Institute, Chinese Academy of Tropical Agricultural Sciences, Danzhou 571737,Hainan,China;
3.Hainan Python Institute, Wenchang 571346, Hainan,China)
Abstract: It was necessary to study genetic diversity and differentiation of local species with molecular techniques for effectively protecting, managing and breeding Burmese python(Python bivittatus). TA bidirectional sequencing was used to obtain part of mtDNA control region II of P.bivittatus from 37 Hainan individuals and 28 Vietnam individuals. A total of 25 haplotypes were obtained. Haplotype diversity(Hd), nucleotide diversity π and the average number of nucleotide difference k were 0.877±0.025, 0.004 76±0.000 41 and 2.676, respectively, indicating that the genetic diversity of P.bivittatus was not very high. By comparing the genetic diversity of the two populations, the diversity of haplotype in P.bivittatus from Hainan was higher than that from Vietnam. The nucleotide diversity π and the average number of nucleotide difference k of the former population was lower. ML trees and network analysis based on mtDNA control region showed that the two populations were divided into 2 clades. The fixation index (Fst) reached 0.201(P<0.001), indicating that there were significant differences between the two populations.
Key words:Python bivittatus; control region; genetic diversity; genetic differentiation
缅甸蟒(Python bivittatus)主要分布于东南亚国家和中国海南、云南、广东、广西、福建、四川等地[1],是国家一级保护动物,被列于《濒危动植物种国际贸易公约》(CITES)附录II。缅甸蟒用途广泛,蟒皮可用来制作民族乐器、高档皮具,据统计,2000-2010年缅甸蟒皮的出口量仅在越南就已达到年平均10万张[2];蟒油具有疏通经脉、消肿抑菌等作用[3];蟒蛇胆具有止咳、祛痰、平喘等作用[4]。此外,缅甸蟒因性情温顺可爱,也是世界流行的宠物。如此大的市场需求,导致野生蟒蛇急剧减少,加上蟒蛇传统栖息地面积的萎缩和数量的减少,使蟒蛇成为濒危物种。有数据显示,中国大陆的野生蟒蛇仅约62 000条,且数量仍在减少[5]。
海南岛位于中国南部,与越南隔海相望。岛屿具有与陆地界限明确、受陆地物种影响小等独有特点,因此是研究物种进化、演替和新物种形成的绝好地理环境。海南岛蟒蛇具有这些特征。目前,关于越南缅甸蟒线粒体基因组全序列和来自海南缅甸蟒线粒体基因组全序列已经公布[6],通过比对发现其在控制区II位置变异最大。此外,人们在饲养过程中,发现同龄海南缅甸蟒与越南缅甸蟒在个体大小和生长速度上有显著性差异,因此有必要利用分子技术探讨这两种缅甸蟒遗传差异。
国内外学者利用线粒体上控制区、CYTB以及微卫星标记对缅甸蟒遗传多样性进行了研究[7-9],取得了一定的研究成果,对于地方种群的来源、结构、种群多样性以及对入侵种群的管理有一定的参考价值,但目前关于海南缅甸蟒种群遗传多样性的研究国内外尚未见报道。为了更有效地保护、管理和繁育缅甸蟒濒危物种,特别是海南缅甸蟒,有必要采用现代分子遗传技术对种群进行群体遗传结构、遗传分化分析。本试验通过测定中国海南岛和越南的缅甸蟒控制区II 3′端序列,研究两地方种群缅甸蟒遗传多样性,探讨海南与越南缅甸蟒遗传结构及遗传差异,以期能为海南和越南缅甸蟒的正确区分提供重要的科学依据。
1 材料与方法
1.1 样本采集
本试验从海南蟒蛇研究所的10 000条蟒蛇中随机抽取海南缅甸蟒37条、越南缅甸蟒28条,采用心脏采血法每条抽取1.5 mL血液,柠檬酸钠抗凝,立即提取总 DNA(参照天根 DP304离心柱型基因组提取试剂盒说明书操作),检测样品浓度和纯度,并稀释至100 ng/μL,-20 ℃分装保存。
1.2 PCR扩增及克隆测序
PCR扩增引物,上游引物序列为:5′-CTCCGAATAAATCCCAATC-3;下游引物序列为:5′–TTGTTGCCGCTTCTGTGG-3′。PCR扩增体系为50 μL:Taq Master Mix混合液25 μL,总DNA模板100 ng,最后补水至50 μL。PCR反应程序为:94 ℃预变性3 min;94 ℃ 变性30 s,50 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸5 min。
PCR产物经1%凝胶电泳检测,回收目的条带。将目的条带进行克隆测序。具体步骤:T4 DNA连接酶作用下16 ℃过夜→连接至PMD20-T载体→转化至大肠杆菌 DH5α→LB培养基培养12 h→挑取阳性克隆菌落→菌落PCR验证→提取质粒→生工生物工程(上海)股份有限公司ABI3730型全自动测序仪双向测序。
1.3 序列分析
所得序列用DNASTAR 7.10中的SeqManII进行序列校对及拼接。用BioEdit7.0软件进行多重比对,DnaSP3.0统计多态位点、简约性位点、单倍型、核苷酸多样性(π)、单元型多样性(h)、平均核苷酸差异数(k)及中性检验。用MEGA 3.1基于Kumar 2-parameter模型进行种群内遗传距离及分子系统分析,进化树中节点的自举置信度水平由自引导值(Bootstrap value)估计,重复次数为1 000。利用Arlequin 3.01软件中分子方差分析(AMOVA)估算两群体间的固定指数(Fst)。用Network进行单倍型网络关系分析。
2 结果与分析
2.1 缅甸蟒控制区遗传多样性
在65条序列中,碱基组成如下:T为23.9%, C为 27.2%,A为37.8%,G为11.0%。A+T明显高于C+G。共发现38个多态位点,包括36个单变异位点和2个简约性位点。转换与颠换比为5.35。所有序列均已提交至GenBank(登录号:KF991017-KF991081)。
除去控制区内串联重复序列,得到长度570 bp左右的65条序列。共定义25个单倍型(表1),其中单倍型Hap3频率最高,为24.6%。其次是单倍型Hap16和Hap17,均为18.5%。65条缅甸蟒单倍型多样度(Hd)、核苷酸多样性(π)和平均核苷酸差异数(k)见表2。基于Kimura 2 parameter模型计算65条缅甸蟒种群内遗传距离在0.002~0.016之间。
2.2 海南与越南缅甸蟒控制区差异
从表2可知,37条海南缅甸蟒控制区序列分为12个单倍型,单倍型比例为32.4%;28条越南缅甸蟒控制区序列分为15个单倍型,单倍型比例为53.6%。海南缅甸蟒的单倍型多样度要高于越南缅甸蟒,但核苷酸多样性和平均核苷酸差异数低于越南缅甸蟒。群体固定指数Fst值在0.00~0.05之间,表明其各亚群间不存在分化;Fst值在0.05~0.15之间,为中度分化;Fst值在0.15~0.25之间,则为高度分化[10]。计算得出海南与越南缅甸蟒两群体间的Fst值为0.201(P<0.001),遗传距离为0.007,核酸歧义度(Dxy)为0.006 56±0.000 90,显示两群体间存在极显著差异。
2.3 缅甸蟒分子进化与单倍型网络关系分析
利用65条缅甸蟒控制区序列构建ML分子系统树(图1)。由图1可知,海南缅甸蟒与越南缅甸蟒分为明显的两大分支(除Y9个体在海南缅甸蟒分支中和H24、H34个体在越南缅甸蟒分支中)。
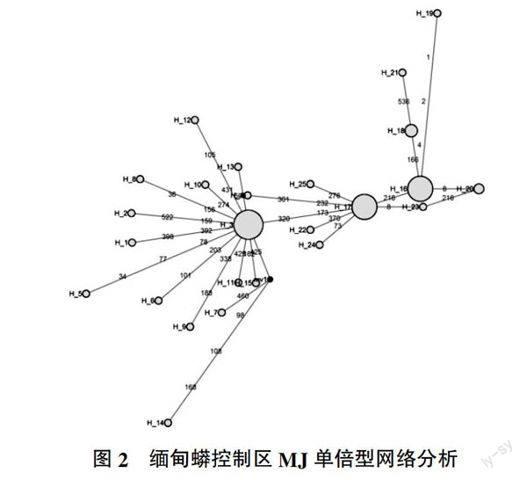
对65条序列进行单倍型网络关系分析(图2),网络以Hap3、Hap16、Hap17为中心。经中性检验,Tajimas D为-2.180 50(P<0.01)、Fu's Fs statistic为-16.963及Mismatch分析图为单峰,表明缅甸蟒曾经历过群体扩张事件。
3 小结与讨论
3.1 缅甸蟒的遗传多样性及保护
从表2中可以看出,缅甸蟒总体上群体遗传具有较高的单倍型多样度,但是表现出较低的核苷酸多样性,显示群体遗传多样性并不高。遗传多样性是一个种群进化和生存的重要基础,遗传多样性的缺失会威胁到种群的生存。加上栖息地的缺失和人类的大量捕杀,缅甸蟒濒危物种数量急剧下降。缅甸蟒的基因多样性匮乏也给缅甸蟒的人工驯养及繁殖带来一定的困难。在人工养殖中,应加强遗传管理,建立清晰详细的谱系关系,防止近亲衰退。同时可以引入遗传多样性高的种群,或将各农户的蟒蛋、幼蟒、种蟒进行交换来提高本地蟒蛇种群基因交流程度,提高本地种群的遗传多样性。
3.2 海南与越南缅甸蟒遗传分化
对比海南与越南缅甸蟒控制区II序列分析可知,相对于越南缅甸蟒来说,海南缅甸蟒的单倍型多样度高,核苷酸多样性和平均核苷酸差异数低,根据Wright的遗传平衡理论,推测海南缅甸蟒可能是由一个小种群迅速增长,长期的地理和时间隔离,导致随机的遗传漂移,引起单倍型多样度增大,但是没有累积核苷酸方面的多态性[11]。构建分子系统树对于鉴别物种来源具有重要的参考价值。图1中分子系统树将65条缅甸蟒分为两大支,其中一支海南缅甸蟒个体占97.2%,另一支越南缅甸蟒占93.1%,地理位置几乎与ML分子系统树完全对应,表明海南与越南缅甸蟒起源于两个不同的母系。进一步利用Median Joining法构建单倍型网络分析图,发现缅甸蟒分为3个主要的单倍型,Hap3、Hap16和Hap17。其中Hap3由87.5%越南缅甸蟒组成,Hap16、Hap17 100%由海南缅甸蟒组成。在单倍型网络分析图中也可看出海南与越南两个明显的区域,说明海南与越南缅甸蟒在线粒体基因组的遗传分化方面确实有明显的差异,反映出这两个群体在进化过程中产生的不同遗传特征。
计算得出海南与越南缅甸蟒Fst为0.201,结果也表明两个地理种群已出现较明显的种群分化。海南与越南同属热带季风气候,地理位置仅有一北部湾之隔,构成了地理上的隔离。日积月累,使缅甸蟒在这两个地方种群产生了极显著的分化。Collins等[7]利用CYTB和控制区基因分析156条美国大沼泽地国家公园缅甸蟒,发现其群体内遗传分化很小,并与越南缅甸蟒群体Fst为0.088,呈现中度分化。You等[9]利用来自台湾金门的26条缅甸蟒、来自福州的2条缅甸蟒和来自越南的5条缅甸蟒的CYTB和COI基因探讨了台湾金门岛缅甸蟒起源,发现金门岛与福州缅甸蟒最为接近,两者都与越南缅甸蟒亲缘关系较远,否认了金门岛上缅甸蟒来自东南亚地区。
对于不同物种,其表观形态差距大,较易区分开。但是对于同一物种不同地域种群来说,很难通过形态特征来加以区分和研究。利用控制区序列来构建分子系统树,可以大致区分本地种和外来种,消除外来物种的基因可能混杂当地种群的潜在风险,也为野生动物保护监管机构及海关检验蟒蛇的来源提供了一个生物学检测方法。这种方法类似于现在DNA条形码概念,不同的是DNA条形码大多数用于种及种以上的分类和鉴定,对于种内来源鉴别很少[12,13]。而控制区是线粒体上进化最快的区域,常用来研究种内遗传分化和多样性[14-16]。总之,通过构建分子系统树、单倍型网络分析图和计算两群体间的固定指数Fst,均已表明海南与越南缅甸蟒在分子水平上已出现明显的分化。
本试验对海南和越南两个地理种群缅甸蟒进行了遗传多样性和遗传差异研究,得出整体缅甸蟒群体遗传多样性并不高,但海南与越南缅甸蟒在分子水平上已出现明显的分化。本研究成果对保护蟒蛇资源、鉴别蟒蛇活体及其产品的来源、人工养殖、种群管理等将具有重要的理论意义和应用价值。
参考文献:
[1] 赵尔宓,黄美华,宗 愉.中国动物志爬行纲[M].北京:科学出版社,1998.34-35.
[2] KASTERINE A, ARBEID R, CAILLABET O, et al. The Trade in South-East Asian Python Skins [M].Geneva:International Trade Centre (ITC), 2012.
[3] 赵 静,陈 琦,张立岭,等.缅甸蟒脂肪酸分析[J].四川动物, 2009(6):899-900.
[4] 管淑玉,曾常青,刘广锋.中药蛇胆的鉴别及质量控制现状[J]. 广东药学院学报,2010,26(5):537-539.
[5] 国家林业局.中国重点陆生野生动物资源调查[M].北京:中国林业出版社,2009.
[6] 刘雅芳,曹 婷,刘建才,等.缅甸蟒线粒体基因组全序列测定与分析[J].四川动物,2014,32(1):24-31.
[7] COLLINS T M, FREEMAN B, SNOW S. Final report: genetic characterization of populations of the nonindigenous Burmese python in Everglades National Park[R]. Miami: Final Report for the South Florida Water Management District,2008.
[8] HUNTER M E, HART K M. Rapid microsatellite marker development using next generation pyrosequencing to inform invasive Burmese Python-Python molurus bivittatus-management [J]. Int J Mol Sci, 2013, 14(3): 4793-4804.
[9] YOU C W, LIN Y P, LAI Y H, et al. Return of the pythons: first formal records, with a special note on recovery of the Burmese python in the demilitarized Kinmen islands [J]. Zoological Studies, 2013, doi:10.1186/1810-522X-52-8.
[10] WRIGHT S. Evolution and the Genetics of Populations. Volume 4. Variability Within and Among Natural Populations [M]. Chicago: University of Chicago Press,1978.
[11] AVISE J C. Phylogeography the History and Formation of Species [M]. Cambridge: Harvard University Press, 2000.
[12] VENCES M, THOMAS M, BONETT R M,et al. Deciphering amphibian diversity through DNA barcoding: chances and challenges[J]. Phil Trans R Soc B, 2005, 360(1462):1859-1868.
[13] WHITTAKER D J, MORALES J C, MELNICK D J. Resolution of the Hylobates phylogeny: Congruence of mitochondrial D-loop sequences with molecular, behavioral, and morphological data sets[J]. Molecular Phylogenetics and Evolution, 2007, 45(2):620-628.
[14] NORMAN J A,MORITZ C, LIMPUS C J.Mitochondrial DNA control region polytmorphisms:genetic markers for ecological studies of marine turtles[J]. Molecular Ecology, 1994, 3(4):363-373.
[15] BARBER P H. Phylogeography of the canyon treefrog, Hyla arenicolor(Cope) based on mitochondrial DNA sequence data [J]. Molecular Ecology, 1999, 8(4):547-562.
[16] 李慧芳,徐文娟,朱文奇,等.福建省家鸭的遗传多样性及其与野鸭的亲缘关系[J].生物多样性,2008,16(6):607-612.
