梅毒螺旋体TprF蛋白B细胞表位的预测与鉴定
2015-03-17曹龙古蔡恒玲赵飞骏欧阳丹明陈苏芳吴移谋曾铁兵
曹龙古,凌 晖,蔡恒玲,赵飞骏,欧阳丹明,陈苏芳,吴移谋,曾铁兵
梅毒螺旋体TprF蛋白B细胞表位的预测与鉴定
曹龙古1,2,凌 晖3,蔡恒玲1,赵飞骏1,欧阳丹明2,陈苏芳2,吴移谋1,曾铁兵1
目的 预测和鉴定梅毒螺旋体(Tp) 膜蛋白TprF氨基端保守区(TprFN)的优势B细胞表位,为深入研究梅毒多价表位疫苗提供依据。方法 从GenBank获取TprFN的氨基酸序列,采用Mobyle、ABCpred和IEDB在线软件综合分析预测TprFN的B细胞表位并人工合成多肽;表达重组蛋白TprFN并经Western blot鉴定后免疫兔,获取血清并测定抗体效价;以TprFN免疫兔血清、梅毒患者血清(设正常人血清和正常兔血清为阴性对照),间接ELISA测定预测的7条人工合成的B细胞表位多肽的免疫反应性和特异性。结果 软件综合预测TprFN的P1 (43-62AA)、P2(57-71AA)、P3(81-88AA)、P4(89-103AA)、P5(125-138AA)、P6(231-251AA)、P7(268-279AA)可能为B细胞表位;表达一可溶性蛋白,WB鉴定为目的蛋白,其免疫抗体效价为1∶12 800以上;ELISA结果显示,预测表位P1、P3与TprFN免疫兔血清及梅毒患者血清均呈阳性反应,而与对照血清均不反应。结论 P1、P3为TprF潜在的优势B细胞表位。
梅毒螺旋体;TpF;B细胞表位
Supported by the National Natural Science Foundation of China (Nos. 81273322 and 81373230), the Scientific Research Fund of Hunan Provincial Education Department (No. 13C876), the Chenzhou Municipal Science and Technology Bureau (No. 2012CJ126), and the Hunan Provincial Key Laboratory for Special Pathogens Prevention and Control Foundation (No. 2014-5) Corresponding author:Zeng Tie-bing,Email:nhdxztb@126.com
梅毒是由梅毒螺旋体(Treponemapallidum, Tp) 引起的一种严重危害成人和新生儿健康的性传播疾病(STD),并可明显促进艾滋病传播[1]。近年我国梅毒发病呈迅速上升趋势[2]。应用疫苗是梅毒防控的根本措施[3-4],但至今未获成功。
目前认为,梅毒疫苗分子主要存在于Tp的外膜蛋白(OMPs)中[3-4]。TprF是近年来属于Tpr家族蛋白第Ⅰ亚家族成员,包含369个氨基酸,软件分析预测为OMP[5]。研究发现,在不同Tp株感染兔的体内存在针对重组TprF的N端保守区域(TprF蛋白全长第21~284位氨基酸,简称TprFN)的强烈的抗体应答和T细胞应答,免疫兔后明显减轻不同Tp株攻击所致损伤和减缓疾病进展[5-6],表明TprFN是颇具希望的候选疫苗分子[3]。
联合多个具有保护作用的抗原分子是研发梅毒高效疫苗的趋势[3-4],但在同一载体上串联多个抗原编码基因技术上存在困难,并可能引起转录干扰[5]。多价表位疫苗能克服该困扰,同时去除了蛋白分子中可能的无关、干扰甚至抑制表位,且可避免免疫耐受,此外还具有保护范围广、针对性强、稳定、高效、制备简单、经济安全等优点,具有广阔的应用前景[7]。本研究对TprF蛋白保守区的B细胞表位进行筛选和鉴定,为发展梅毒多价表位疫苗奠定基础。
1 材料与方法
1.1 实验材料 Tp(Nichols株)为上海皮肤性病医院检验科顾伟鸣馈赠,本研究所传代保存。表达载体pET30a(+)、限制性内切酶、Tp基因组提取试剂盒分别为美国Novagen、Thermo、德国Qiagen公司产品。羊抗兔HRP-IgG、羊抗人HRP-IgG购自北京康为世纪有限公司。96孔高结合力酶标板为美国Corning公司产品。确诊梅毒阳性血清、健康人血清标本分别来自南华大学附属第一医院检验科和健康体检中心。
1.2 方法
1.2.1 TprFN的B细胞表位预测 获得TprF氨基酸序列(http://www.ncbi.nlm.nih.gov/protein/AGF43524.1),将去除信号肽后的保守区氨基酸序列(TprF全长第21~284位)输入在线Mobyle软件(http://mobyle.pasteur.fr/cgi-bin/portal.py?#forms::antigenic)和ABCpred软件(http://www.imtech.res.in/raghava/abcpred/ABC_submission.html),结果按分值高低大小依次显示。再应用在线预测软件IEDB(http://www.immuneepitope.org/tools/bcell/iedb_input)综合分析TprF氨基酸序列表面可及性、亲水性、抗原性、β拐角区域以及易屈性等理化因素。综合以上3种软件预测结果预测TprFN的B细胞抗原表位。
1.2.2 预测表位多肽合成 委托北京泽溪源生物科技股份有限公司人工合成预测的表位多肽,经反相高效液相色谱纯化,质谱分析鉴定。
1.2.3 TprFN的表达与鉴定 从GenBank查取TprF全基因序列,设计针对TprFN的一对特异性引物F:5′ CGCGGATCCTATGCAGGCGTACTCACTCC 3′ (下划线为BamH I酶切位点)、R:5′ CCGCTCGAGGGCAGGGTCCTGGTGTTG 3′ (下划线为Hind Ⅲ酶切位点),以试剂盒提取的Tp DNA为模板,PCR扩增保守区基因片段,构建pET30a(+)/TprFN的原核表达重组体,经测序和双酶切鉴定,30 ℃、以1.0 mmol/L IPTG诱导表达蛋白4 h,以1∶100稀释梅毒患者血清为一抗,1∶2 000稀释标记的山羊抗人HRP-IgG,Western Blot(WB)鉴定纯化蛋白。
1.2.4 TprFN多抗制备 以纯化TprFN加弗氏佐剂免疫5只体重约2.5 kg的新西兰兔(200 μg/只·次),间隔2 w免疫1次,共5次,末次免疫后第10 d收集免疫血清,ELISA测定重组蛋白抗体效价。
1.2.5 TprFN预测B细胞表位鉴定 分别将10 μg/mL合成的B表位多肽和重组TprFN(阳性对照)作为抗原包被在酶标板上(100 μL/孔),以1∶100稀释血清(TprFN免疫兔血清、正常兔血清各5份、梅毒患者血清、正常人血清各10份)为一抗,同时设空白对照,每个样品做3个平行孔,1∶15 000稀释的羊抗兔或羊抗人HRP-IgG进行间接ELISA,酶标仪双波长测定吸光值A630/450nm。实验重复3 次。
1.3 统计学处理 结果以A均值表示,采用SPSS 19.0软件进行统计分析,各组间进行t检验,P<0.05代表有统计学意义。
2 结 果
2.1 TprF蛋白B细胞表位在线软件预测与合成 综合ECBI、ABCpred和Mobyle在线软件,预测出如下7段氨基酸序列有可能为TprFN蛋白的B细胞表位:P1 (43-62AA)、P2(57-71AA)、P3(81-88AA)、P4(89-103AA)、P5(125-138AA)、P6(231-251AA)、P7(268-279AA)。合成多肽纯度均大于90%,质谱分析合成多肽分子量测定值与理论值一致。

表1 预测的TprFN蛋白的B细胞表位
2.2 重组TprFN的表达、鉴定及免疫原性分析 经PCR、双酶切及测序鉴定的目的基因表达了一分子量大小约36 kDa的可溶性蛋白条带(图1-A);Western blot检测显示,重组蛋白仅与梅毒阳性病人血清发生特异性反应(图1-B);ELISA检测其免疫兔多抗效价均在1∶12 800以上,显示良好的免疫原性。
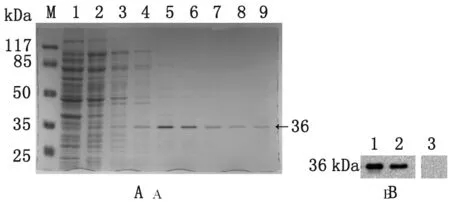
A. M:Protein molecular weight maker; 1:flow-through fluid; 2-4:W1-W3; 5-9:purified recombinant protein.
B. 1,2:syphilitic sera; 3:normal human sera.
图1 纯化重组蛋白TprFN的SDS-PAGE分析与Western Blot鉴定
Fig.1 Analysis and identification of purified recombinant protein TprFNby SDS-PAGE (A) and Western Blot (B)
2.3 TprF蛋白预测B细胞表位的鉴定 间接ELISA结果显示,在P1和P3与TprFN免疫兔血清(图2) 和梅毒血清(图3)反应的吸光度A值与相应对照组差异均有统计学意义(P<0.05),表明P1和P3为TprFN蛋白的潜在优势B细胞表位。
3 讨 论
各种类型疫苗均基于表位(抗原决定簇)发挥作用。B细胞表位是抗原分子中能被BCR/抗体特异性识别的基本单位。在抗Tp感染中,B细胞分泌特异性抗体介导的体液免疫应答发挥着重要作用,主要方式包括阻断Tp粘附、促进巨噬细胞的吞噬(调理作用)、激活补体溶菌作用等[3-4,8]。本研究预测鉴定TprF的B细胞表位,为构建多价表位疫苗奠定基础。
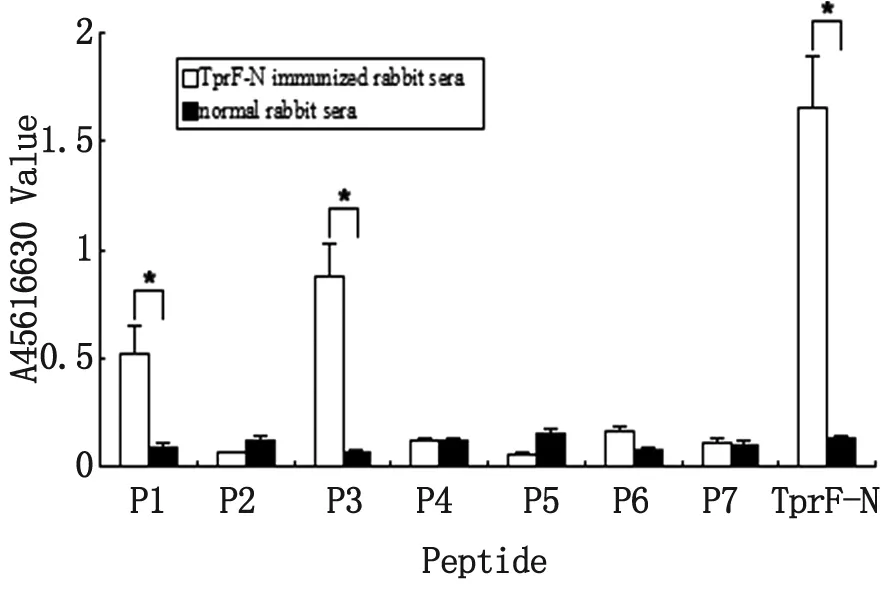
The “*” symbol indicate a significant difference (P<0.05) compared with normal rabbit sera control groups.
图2 ELISA检测TprFN预测表位多肽与兔血清免疫反应性 (A450 /630)
Fig.2 Immunoreactivity of predicted TprFNepitopes with rabbit sera by ELISA
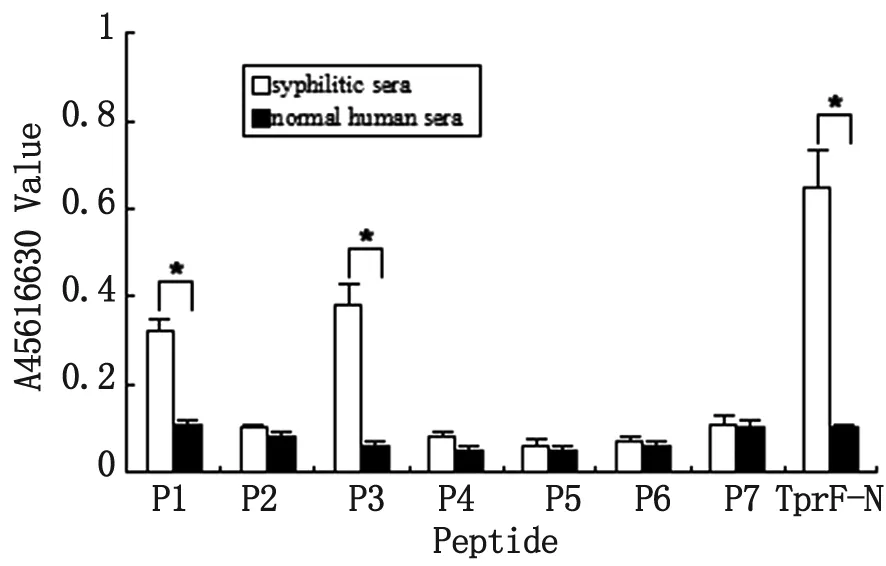
The “*” symbol indicate a significant difference (P<0.05) compared with normal human sera control groups.
图3 ELISA检测TprFN预测表位多肽与人血清免疫反应性 (A450 /630)
Fig.3 Immunoreactivity of predicted TprFNepitope with human sera by ELISA
传统筛选鉴定表位的方法主要有合成重叠肽法[9]、噬菌体肽库展示法[10]、水解法[11]、生物学或化学方法[12]等,其优点是能较全面筛选抗原蛋白可能表位,但工作难度大,耗时长。应用计算机软件分析筛选抗原表位,可减少盲目筛选的庞大工作量,大大提高了发现新表位的效率[13]。目前几乎所有B细胞表位预测软件均以唯象理论为基础,即通过计算蛋白亚序列二级结构或者理化性质,再利用B细胞表位与其相关性来预测。为提高预测的准确性,通常需综合多种方案来分析[14-15],本文运用IEDB在线软件中提供多种方案分析亲水性、抗原性、易屈性、表面可及性与β-转角,再结合ABCpred和Mobyle在线软件总结得出7条TprF抗原最有可能的B细胞表位多肽。由于在线软件均基于现有的表位数据模拟和推测待预测氨基酸序列,存在这一定风险和偏差,需鉴定所预测的表位。
鉴定TprF的B表位首先要确定其免疫原性和免疫反应性。由于至今Tp不能体外人工培养,且其外膜蛋白量少而脆弱,无法直接分离TprF,为此本研究首先通过E.coli表达了可溶性TprFN。可溶性表达无需对纯化蛋白进行变性和复性处理,保持了抗原天然的空间构象,以其免疫能获得针对TprFN的各种B细胞表位的混合抗体。以重组TprFN免疫兔,获得了高滴度的抗体,表明该重组抗原具有良好的免疫原性;免疫印迹法(WB)鉴定重组抗原也表明其有良好的免疫反应性和特异性。WB和ELISA均可用于B细胞表位的鉴定,但人工合成的表位多肽分子量太小,不可采用WB来鉴定。本研究以各合成短肽为抗原,ELISA结果显示,预测的P1和P3能与TprFN免疫兔血清发生特异性强免疫反应,为进一步确认,同时以梅毒感染者血清鉴定,结果与免疫兔血清的反应一致,进一步表明P1和P3为TprFN的优势B细胞表位。
本研究筛选出TprFN的优势B细胞表位,为进一步构建多表位梅毒疫苗奠定了基础,但其是否具有免疫保护性及通过何种方式发挥抗Tp感染作用有待深入研究。
[1]Douglas JM Jr. Penicillin treatment of syphilis:clearing away the shadow on the land[J]. JAMA, 2009, 301(7):769-771. DOI:10.1001/jama.2009.143
[2]Tucker JD, Cohen MS. China’s syphilis epidemic:epidemiology, proximate determinants of spread, and control responses[J]. Curr Opin Infect Dis, 2011, 24(1):50-55. DOI:10.1097/QCO.0b013e32834204bf
[3]Cameron CE, Lukehart SA. Current status of syphilis vaccine development:need, challenges, prospects[J]. Vaccine, 2014, 32(14):1602-1609. DOI:10.1016/j.vaccine.2013.09.053
[4]Cullen PA, Cameron CE. Progress towards an effective syphilis vaccine:the past, present and future[J]. Expert Rev Vaccines, 2006, 5(1):67-80. DOI:10.1586/14760584.5.1.67
[5]Sun ES, Molini BJ, Barrett LK, et al. Subfamily ITreponemapallidumrepeat protein family:sequence variation and immunity[J]. Microbes Infect, 2004, 6:725-737. DOI:10.1016/j.micinf.2004.04.001
[6]Hevner GLK, Centurion-Lara A. Gene organization and transcriptional analysis of the tprJ, tprI, tprG, and tprF loci inTreponemapallidumstrains Nichols and Sea 81-4[J]. J Bacteriol, 2005, 187(17):6084-6093. DOI:10.1128/JB.187.17.6084-6093.2005
[7]Pishraft Sabet L, Taheri T, Memarnejadian A, et al. Immunogenicity of multi-epitope DNA and peptide vaccine candidates based on core, E2, NS3 and NS5B HCV epitopes in BALB/c mice[J]. Hepat Mon, 2014, 14(10):e22215. DOI:10.5812/hepatmon.22215
[8]Lafond RE, Lukeart SA. Biological basis for syphilis[J]. Clin Microbiol Rev, 2006, 19:29-49. DOI:10.1128/CMR.19.1.29-49.2006
[9]Williams KM, Bigley EC 3rd, Raybourne RB. Identification of murine B-cell and T-cell epitopes ofEscherichiacolioutermembrane protein F with synthetic polypeptides[J]. Infect Immun, 2000, 68(5):2535-2545. DOI:10.1128/IAI.68.5.2535-2545.2000
[10]Freund NT, Enshell-Seijffers D, Gershoni JM. Phage display selection, analysis, and prediction of B cell epitopes[M]. Curr Protoc Immunol, 2009, Chapter 9:Unit 9.8. DOI:10.1002/0471142735.im0908s86
[11]Reynolds SR, Dahl CE, Harn DA.T and B epitope determination and analysis of multiple antigenic peptides for theSchistosomamansoniexperimental vaccine triose-phosphate isomerase[J]. J Immuno, 1994, 152(1):193-200.
[12]Morris GE. Epitope mapping by chemical fragment[J]. Methods in Mol Biol, 1996, 66:121-127.
[13]Soria-Guerra RE, Nieto-Gomez R, Govea-Alonso DO, et al. An overview of bioinformatics tools for epitope prediction:implications on vaccine development[J]. J Biomed Inform, 2015, 53:405-414. DOI:10.1016/j.jbi.2014.11.003
[14]Lian Y, Ge M, Pan XM. EPMLR:sequence-based linear B-cell epitope prediction method using multiple linear regression[J]. BMC Bioinformatics, 2014, 15(1):414. DOI:10.1186/s12859-014-0414-y
[15]EL-Manzalawy Y, Honavar V. Building classifier ensembles for B-cell epitope prediction[J]. Methods Mol Biol, 2014, 1184:285-294. DOI:10.1007/978-1-4939-1115-8_15
Prediction and identification of B-cell epitopes ofTreponemapallidumrepeat protein F
CAO Long-gu1,2,LING Hui3,CAI Heng-ling1,ZHAO Fei-jun1,OUYANG Dan-ming2,CHEN Su-fang2,WU Yi-mou1,ZENG Tie-bing1
(1.InstitutionofPathogenicBiology,MedicalCollege,UniversityofSouthChina,Hengyang421001,China;2.DepartmentofPreventiveMedicineandMedicalExamination,XiangnanUniversity,Chenzhou423000,China;3.InstituteofCancerResearch,MedicalSchool,UniversityofSouthChina,Hengyang421001,China)
To predict and identify the dominant B-cell epitopes of conserved region ofTreponemapallidumrepeat protein F (TprFN) and provide the basis for development of polyvalent epitope-based syphilis vaccine, the amino acid sequence of TprFNwas obtained from GenBank and analyzed with comprehensive meta-analysis Mobyle, ABCpred and IEDB online software. The peptides containing predicted epitopes were artificially synthesized. To obtain and measure the titers of antibodies against TprFN, New Zealand rabbits were immunized with recombinant protein TprFNexpressed inE.coliand identified by Western blot (WB) . Sera from TprFN-immunized rabbits, syphilis patients, and normal human and normal rabbits were used to determine the immunoreactivity and specificity of 7 predicted peptides of TpFNby indirect ELISA. Comprehensive meta-analysis of online software showed that P1 (43-62AA), P2(57-71AA), P3(81-88AA), P4(89-103AA), P5(125-138AA), P6(231-251AA) and P7(268-279AA) might be the B-cell epitopes. A protein was expressed in a soluble form and identified as TpFNby WB. The ELISA indicated that P1 and P3 were active with TprFN-immunized rabbit sera and syphilis patient sera but not with negative control sera. These results indicate that P1 and P3 are the potential dominant B-cell epitopes.
Treponemapallidum; TprF; B-cell epitope
国家自然科学基金(No. 81273322,81373230);湖南省教育厅资助科研项目(No.13C876);郴州市科技局资助科研项目(No.2012CJ126);特殊病原体防控湖南省重点实验室资助项目(湘科计字[2014]5号)
曾铁兵,Email:nhdxztb@126.com
1.南华大学医学院病原生物学研究所,衡阳 421001; 2.湘南学院预防医学与医学检验系,郴州 423000; 3.南华大学医学院肿瘤研究所,衡阳 421001
10.3969/j.issn.1002-2694.2015.10.005
R377
A
1002-2694(2015)10-0919-04
2015-05-19;
2015-07-24
