大庆龙凤湿地底泥氨氧化细菌与氨氧化古菌多样性分析
2015-03-13马茹霞晏磊王国兴王长虹贾军邓兵沈婷婷王伟东
马茹霞,晏磊,王国兴,王长虹,贾军,邓兵,沈婷婷,王伟东
(1.黑龙江八一农垦大学生命科学技术学院,大庆 163319;2.上海岩土工程勘察设计研究院有限公司)
硝化作用在地球的氮素循环过程中起重要的作用,其主要包括两个步骤,第一步通过氨氧化菌将氨氧化为亚硝酸盐,第二步通过亚硝酸盐氧化菌将亚硝酸盐氧化为硝酸盐。其中,氨氧化细菌(Ammoniaoxidizing bacteria,AOB)介导的氨氧化作用是其限速步骤,主要由属于变形杆菌门的β-亚纲和γ-亚纲的氨氧化菌来完成[1]。AOB进行的氨氧化作用是由氨单加氧酶(ammoniamonooxygenase,AMO)催化的,编码AMO的基因(amo A,amo B和amo C)分别编码其3个亚基[2]。1997年,Rotthauwe首次使用amo A-PCR方法研究了自养氨氧化细菌群落结构[3],此后,amo A基因被广泛运用于各种生态系统中自养氨氧化细菌的群落结构研究[3]。有研究发现 Nitrosomonas sp.和Nitrosospira sp.为一些堆肥过程中的关键AOB菌群[4],由于Nitrosospira sp.生长速度较慢,目前在活性污泥中很少检测到[5]。研究发现,在海洋和陆地环境的一些嗜温和嗜热古菌中也含有amo A基因的同系物,并且能催化氨氧化反应[6-7],这一发现将参与氮素循环的微生物扩展到了古菌领域。进一步研究表明,海洋中AOA的数量占了整个微生物数量的39%,这些AOA很有可能在海洋环境的硝化作用中起着重要的作用[8-9]。近期,第一株 AOA-Nitrosopumilu smaritimus的成功培养以及古菌amo A基因的mRNA对氨盐添加的响应[6],强有力地证实了AOA在自然界中的真实存在。到目前为止,富集培养AOA已有4种Nitrosopumilu smaritimus[6]、Cenarchaeumsymbiosum[10]、Nitrososphaera gargensis[11]和 Nitrosocaldus yellowstonii[7]。最近,大量的研究发现属于泉古菌门的AOA也能氧化氨,广泛分布于土壤、河口沉积物、牛粪堆肥及污水处理厂等环境中[12],自然界中AOA的多样性和功能研究迅速成为新的研究热点。
龙凤湿地位于黑龙江省大庆市,是全国最大的城市湿地,常年淹水,水来源于自然降水、嫩江引水和城市污水厂净化后的水,是典型的受人工干扰的湿地类型[13]。湿地生态系统对氨态氮有显著的截留和净化功能,而氨态氮是AOB和AOA进行氨氧化反应的底物,氮循环是湿地生态系统中最基本和重要的物质循环之一。实验分别以AOB和AOA的amo A功能基因为目标,构建其amo A基因克隆文库,研究龙凤湿地底泥中AOB和AOA的多样性,并对其底泥理化参数进行分析,以期为人工干扰型湿地氨氧化微生物研究提供基础数据。
1 材料和方法
1.1 样品采集
样品采集于黑龙江省大庆市龙凤湿地的底泥(2013年10月31日),采用多点取样法,垂直取水下10~25 cm深的底泥,每个点取样量相同,混合均匀后装入灭菌的封口聚乙烯袋中迅速带回实验室,其中一部分样品用于理化参数的测定,另一部分存置于-20℃冰箱冻存用于后续DNA的提取。
1.2 实验方法
1.2.1 底泥理化参数的测定
底泥中盐分采用重量法测定,铵态氮采用靛酚蓝比色法测定,硝态氮采用酚二磺酸比色法测定,亚硝态氮采用重氮化偶合分光光度法测定,全氮采用开氏消煮法测定,全磷采用酸溶-抗比色法测定,全钾采用氢氟酸-高氯酸消煮-火焰光度法测定,上述测定方法见参考文献[14]。pH利用数字pH计(日本)测定。
1.2.2 样品总DNA的提取
取0.1 g底泥样品,利用改进的氯化苄法提取总DNA[15],重复3次。为除去腐殖酸等杂质,将提取的DNA混合,纯化试剂盒纯化粗提的总DNA,采用1%琼脂糖凝胶电泳检测。
1.2.3 AOB amo A基因的PCR扩增
利用引物amo A-1F(5′-GGGGTTTCTACTGGTG GT-3′)和amo A-2R(5′-CCCCTCKGSAAAGCCTTCT T-C-3′)对AOB amo A基因进行扩增。PCR的反应体系为25μL体系:2.5μL 10×buffer,0.5μL 10 mM dNTP,0.5μL模板 DNA,1μL Taq DNA聚合酶(5 U),引物各0.25μL,加ddH2O补足至25μL。反应条件:94℃预变性5 min;94℃变性1 min,52℃退火1min,72℃延伸1min,30个循环;72℃最后延伸5min。PCR扩增产物用1%的琼脂糖凝胶电泳检测,EB染色后用凝胶成像系统照相。
1.2.4 AOA amo A基因的PCR扩增
利用引物Arch-amo AF(5′-STAATGGTCTGGCT TAGACG-3′)和Arch-amo AR(5′-GCGGCCATCCAT CTGTATGT-3′)对AOA amo A基因进行扩增。PCR反应体系为 25μL体系:2.5μL 10×buffer,0.5μL 10 mM dNTP,0.5μL模板DNA,1μL Taq DNA聚合酶(5 U),引物各 0.25μL,加ddH2O补足至25μL。反应条件:95℃预变性5 min;95℃变性1 min,55℃退火50 s,72℃延伸1 min,30个循环;72℃最后延伸5min。PCR扩增产物用1%的琼脂糖凝胶电泳检测,EB染色后用凝胶成像系统照相。
1.2.5 PCR产物的纯化与连接
每次PCR重复3次且设置阴性对照,PCR均停止于4℃,产物采用纯化试剂盒回收。
PCR连接反应体系为:PGEM-T Easy载体1μL,10×T4 DNA Ligation Buffer 5μL,PCR回收产物3μL,T4 DNA Ligase 1μL。轻弹离心管使混合均匀,16℃连接过夜。
早在1910年与1937年HARTMANN和JAUDON就提出低血糖是新生儿常见疾病,会造成神经系统损伤与危害[1],其危险因素与低体温、早产儿、低出生体质量儿、喂养不当、孕母糖尿病等有关[2]。研究亦报道新生儿低血糖症发病越早、程度越重、存在时间越长,越易造成新生儿智力低下、脑瘫等中枢神经系统的永久性损害[3]。因此,掌握新生儿血糖检测值在临床工作中尤为重要,对高危儿的血糖干预逐渐成为常规。而照顾新生儿的医护人员对于影响血糖值相关因素的认知,更与临床提供安全有效的高质量护理密切相关。
1.2.6 amo A基因克隆文库的构建
大肠杆菌感受态细胞100μL,置于冰浴中,完全解冻后轻轻将细胞均匀悬浮,加入5μL连接液,轻轻摇匀,冰上放置30 min;42℃水浴热激70 s,冰上放置 2 min,加 LB培养基 400μL,200~250 rpm,37℃震荡培养1 h,室温下4 000 rpm离心5 min,弃去400μL上清液,用剩余的培养基重悬细胞,将细胞涂布在预先用100μL 20mg·mL-1X-gal和20μL 100 mM·L-1IPTG涂布的含有氨苄青霉素LB固体平板上。平板正向放置40min后倒置培养过夜。选择在IPTG-Xgal平板上生长的白色斑点,分别用无菌tip头挑取各转化子至含氨苄青霉素的LB固体平板上,37℃培养过夜。通过蓝白斑选择阳性克隆子,分别得到AOB与AOA的amo A基因克隆文库。
1.2.7 克隆文库的限制性长度多态性分析
利用引物M13-47(5′-CGC CAG GGT TTT CCC AGT CAC GAC-3′)和RV-M(5′-GAG CGG ATA ACA ATT TCA CAC AGG-3′)对克隆文库的插入片段进行 PCR扩增。扩增反应体系为:2.5μL 10× Reaction Buffer,0.5μL 10 mM dNTP,2μL 25 mM MgCl2,引物M13-47和RV-M各0.25μL,菌少许,Taq DNA聚合酶(5 U)0.25μL,补充ddH2O至25μL。PCR反应条件:94℃预变性 5 min;94℃预变性30 s;62℃变性30 s;72℃延伸90 s,30个循环;72℃最后延伸10min。用1.5%的琼脂糖凝胶电泳检测扩增产物,2.0μg·mL-1EB染色10min后检测。
采用限制性内切酶MspⅠ和HinfⅠ对PCR扩增产物进行限制性内切酶酶切分型。酶切反应体系为:MspⅠ0.3μL,HinfⅠ0.3μL,10×M Buffer 1μL,DNA 3μL,ddH2O 5.4μL。37℃恒温水浴10min,2%的琼脂糖凝胶电泳检测RFLP带型。
1.2.8 系统发育树的构建
通过分析RFLP带型图谱,选择克隆子进行测序。文库中将序列之间的相似性≥97%的序列作为一个操作分类单元(operational taxonomic unit,OTU),基因序列Genbank数据库中进行BLAST序列比对(http://www.ncbi.nlm.nih.gov/BLAST/),利用MEGA4.0软件中的邻接法构建系统发育树[16]。
2 结果和分析
2.1 龙凤湿地底泥理化参数分析
表1为大庆龙凤湿地底泥的理化参数,其pH达到9.1,呈弱碱性,且其盐分含量达到1.0%,说明大庆龙凤湿地有盐碱化。全氮的含量为2.61 mg·kg-1(以干样计,下同)。氨态氮与硝态氮的含量分别为3.86 mg·kg-1和16.87mg·kg-1,说明此湿地底泥为低氮环境。

表1 湿地底泥的理化参数Table 1 The physicochemical parameters ofwetland sediment
2.2 湿地底泥AOB的多样性与系统发育分析
根据各时期的OTU数与克隆子数绘制的AOB rarefaction曲线如图1。可以看出,AOB的rarefaction曲线趋于平稳,这说明所选取的克隆均具有很好的代表性,可以代表文库中大多数AOB的类型。

表2 湿地底泥AOB amo A基因序列统计结果Table 2 The statistical results of gene sequences on AOB amo A ofwetland sediment
基于湿地底泥中氨氧化细菌amo A基因构建的系统发育树如图3。所有的序列均属于Nirtosonomonas,与海洋亚硝化球菌(Nitrosococcus oceani)聚于进化树的不同分支。其中,LF-AOB-1在所有的克隆子中所占的比例为47.15%,与Liu等在河水泥沙中获得的序列(KF803085)相似率为99%。LF-AOB-2和LF-AOB-3分别与来自污水处理厂(JQ277647)和湿地沉积物(HQ202453)的序列聚在进化树的同一分支。

图1 湿地底泥中AOB克隆文库的rarefaction曲线Fig.1 Curve of AOB clone library ofwetland sediment rarefaction

图2 湿地底泥中AOA克隆文库的rarefaction曲线Fig.2 Curve of AOA clone library ofwetland sediment rarefaction
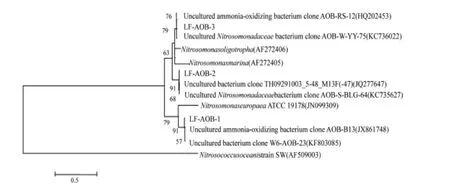
图3 基于amo A基因构建的AOB系统发育树Fig.3 Thephylogenetic treeofAOBbased on amo Agenesequences
2.3 湿地底泥AOA的多样性与系统发育分析
利用Arch-amo AF和Arch-amo AR对龙凤湿地底泥AOA的amo A基因扩增,得到大小约600 bp的条带。构建的amo A基因克隆文库经双酶切测序后得到3个OTU类型(97%相似率)(表3)。所有的序列与来自与河口沉积物等环境中的AOA amo A基因就有较高的相似性。
根据各时期的OTU数与克隆子数绘制的AOA rarefaction曲线如图2。可以看出,AOA的rarefaction曲线基本趋于平稳,这说明所选取的克隆均具有很好的代表性,可以代表文库中大多数AOA的类型。

表3 湿地底泥AOA amo A基因序列统计结果Table 3 The statistical results of gene sequences on AOA amo A ofwetland sediment
从AOA的amo A基因系统发育树来看(图4),所有的序列均属于不可培养的泉古菌门(Crenarchaeota)。其中,LF-AOA-1和LF-AOA-3分别与来自土壤和淡水湿地的序列聚在进化树的同一分支,其克隆子在文库中分别占11.6%和0.47%。LFAOA-2在所有的克隆子中所占的比例最高(83.7%),为底泥中AOA的优势菌属。
3 讨论
从AOB amo A基因克隆文库系统发育看到,本试验只检测到Nitromonas,并没有发现Nitrosospia,菌种类型较少,其与来自江河口沉积物和污水处理厂的序列均有较高的相似率。研究证明,在污水处理厂、人工湿地等pH较高的环境中通常能够检测到Nitrosomonas[17]。贺纪正等在封丘长期定点试验的碱性环境研究中也没有发现Nitrosospira的存在[18],推测Nitrosospira偏爱于pH值较低的环境条件中生存,这与研究的结果相似。
研究发现AOA普遍存在于土壤[12]、河口沉积物、海水沉积物、陆地热泉口等自然环境中,这些研究结果预示着AOA在自然环境中的氮素循环中具有重要作用。本研究在龙凤湿地底泥中,同样证实AOA的存在,所获得的AOA amo A基因序列均属于泉古菌门(Crenarcharota)。有研究认为,与AOB相比,AOA更适合低浓度铵氮的环境条件,并且在此条件下的硝化作用中起主导作用[19]。Hatzenpichler等[11]认为AOA的生长能够受到高浓度铵的抑制。研究中的湿地底泥铵态氮浓度较低,有利于AOA的生长。AOA的发现改变了人们对传统氮素循环中氨氧化过程完全由AOB驱动的认识,而AOA与AOB对湿地底泥中氨氧化作用的相对贡献率还需要进一步深入研究。
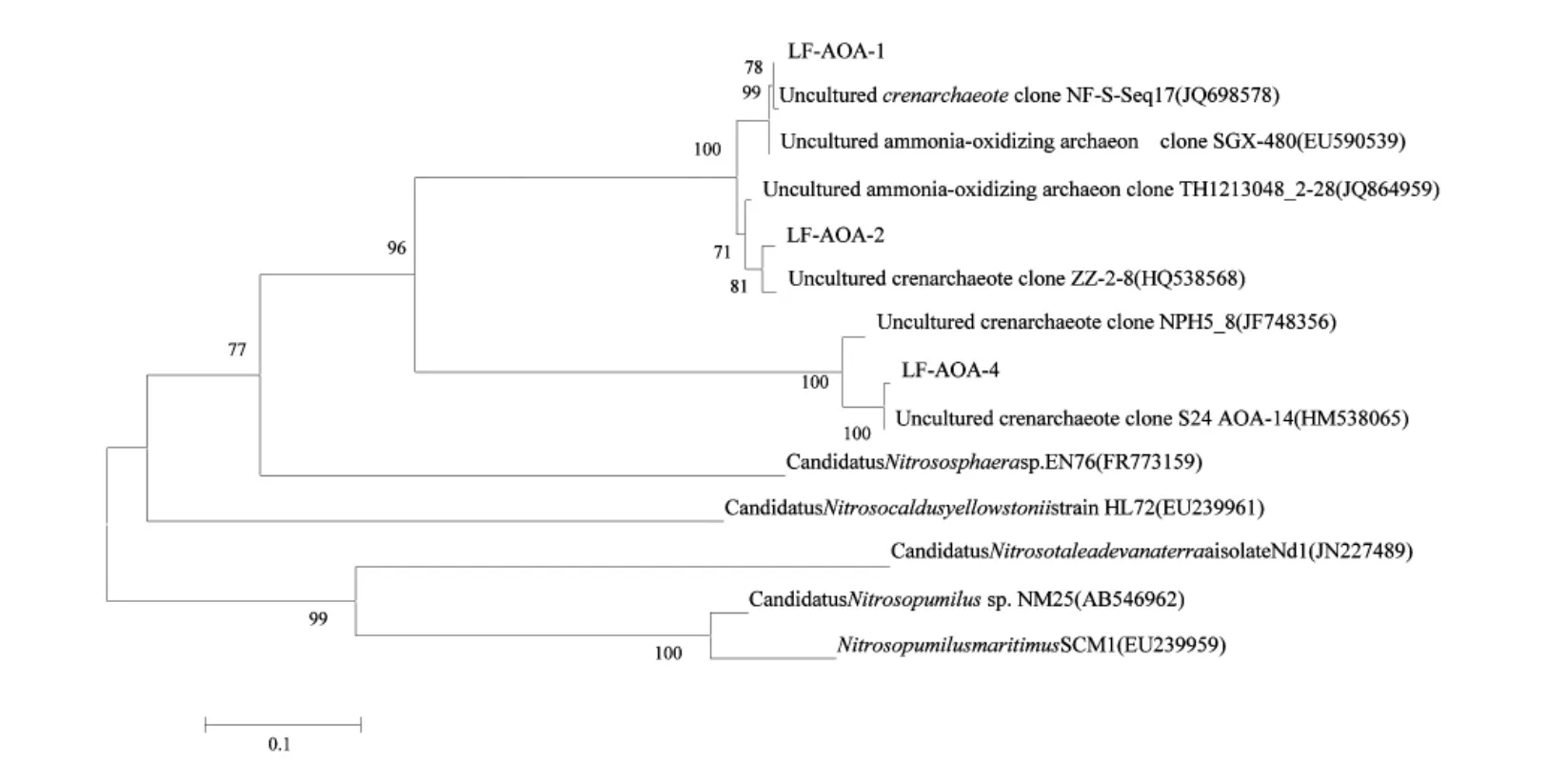
图4 基于amo A基因构建的AOA系统发育树Fig.4 The phylogenetic tree of AOA based on amo A gene sequences
4 结论
(1)龙凤湿地底泥中得到3个AOB的OTU类型,所有的AOB的amo A基因序列均属于Nitromonas,未发现Nitrosospia的存在,这可能与此湿地底泥中较高的pH环境条件有关。
(2)龙凤湿地底泥中得到3个AOA的OTU类型,所有的AOA的amo A基因序列均属于Crenarcharota,与来自河口沉积物等环境中的序列具有较高的相似性,预示着AOA在人工干扰类型湿地生态系统的氮素循环过程中可能起重要的作用。
[1]Holt JG,Krieg N R,Sneath P H A.Ber Gey's Manual of Determinative Bacteriology [M].Baltimore(USA):Williams and Wilkins,1994.
[2]Arp D J,Sayavedra-Soto L A,Hommes NG.Molecular biology and biochemistry of ammonia oxidation by Nitrosomonas europaea[J].Archives of Microbiology,2002,178(4):250-255.
[3]Rotthauwe J H,Witze K P,Liesack W.The Ammonia Monooxygenase structural gene amo A as a functional marker:Molecular fine-scale analysis of natural ammoniaoxidizing populations[J].Applied and Environmental Microbiology,1997,63(2):4704-4712.
[4]Kowalchuk G A,Naoumenko Z S,Derikx P J L,et al.Molecular analysis of ammonia-oxidizing bacteria of theβ subdivision of the class proteobacteria in compost and compostedmaterials[J].Appl Environ Microbiol,1999,65(2):396-403.
[5]Siripong S,Rittmann B.Diversity study of nitrifying bacteria in full-scale municipal wastewater treatment plants[J].Water Res,2007,41(5):1110-1120.
[6]Kǒnneke M,Bernhard A E,Torre JD L,et al.Isolation of an autotrophic ammonia-oxidizing marine archaeon[J].Nature,2005,437(7058):543-546.
[7]Torre JD L,Walker C B,Ingalls A E,et al.Cultivation of a thermophilic ammonia oxidizing archaeon synthesizing crenarchaeol[J].Environmental Microbiology,2008,10(3):810-818.
[8]Agogue'H,Brink M,Dinasquet J,et al.Major gradients in putatively nitrifying and non-nitrifying Archaea in the deep North Atlantic[J].Nature,2008,456(7223):788-791.
[9]Karner MB,DeLong EF,Karl DM.Archaeal dominance in the mesopelagic zone of the Pacific Ocean[J].Nature,2001,409(6819):507-510.
[10]Hallam S J,Konstantinidis K T,Putnam N,etal.Genomic analysis of the uncultivated marine crenarchaeote Cenarchaeum symbiosum[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(48):18296-18301.
[11]Hatzenpichler R,Lebedeva E V,Spieck E,et al.A moderately thermophilic ammonia-oxidizing crenarchaeota from a hot spring[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(6):2134-2139.
[12]Chen X P,Zhu Y G,Xia Y,et al.Ammonia oxidizing archaea:important players in paddy rhizosphere soil[J].Environ Microbiol,2008,10(1111):1978-1987.
[13]孙志远,晏磊,王彦杰,等.牛粪堆肥高温期氨氧化古菌与氨氧化细菌的多样性分[J].黑龙江八一农垦大学学报,2013,25(6):17-22.
[14]鲍士旦,秦怀英,史瑞和,等.土壤农化分析[M].北京:中国农业出版社,2000.
[15]Wang X F,Wang W D,Gao L J,et al.Protocols of applification of denaturing gradient gel electrophoresis(DGGE)in studies of environmentalmicroorganism[J].JournalofChina AgriculturalUniversity,2006,11(5):1-7.
[16]Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24(8):1596-1599.
[17]Otawa K,Asano R,Ohba Y,et al.Molecular analysis of ammonia-oxidizing bacteria community in intermittent aeration sequencing batch reactors used for animal wastewater treatment[J].Environ Microbiol,2006,8(11):1985-1996.
[18]Shen J P,Zhang L M,Zhu Y G,et al.Abundance and composition of ammonia-oxidizing bacteria and ammonia -oxidizing archaea communities of an alkaline sandy loam[J].Environmental Microbiology,2008,10(11):1601-1611.
[19]刘晶静,吴伟祥,丁颖,等.氨氧化古菌及其在氮循环中的重要作用[J].应用生态学报,2010,21(8):2154-2160.
