间充质干细胞与肝样转化细胞的基因谱差异分析
2015-03-05高建鹏张志波王丽昆
姜 华,高建鹏,王 辉,张志波,王丽昆
(昆明医科大学附属延安医院消化内科,云南昆明 650051)
近年来研究发现,与肝移植相比,干细胞移植具有可反复进行,取材广泛,创伤小等优点,并且明显改善肝脏疾病和降低肝衰竭的病死率,可作为替代肝移植的一种新的移植治疗方法[1]。大鼠骨髓间充质干细胞(bone mesenchymal stem cells,BMSCs)为终末肝脏疾病的治疗提供新的途径。但不可否认的是:大鼠BMSCs虽有一定的治疗效果,但远没有达到人们的期望。尤其从大鼠BMSCs分化为肝样细胞的过程中,基因表达谱系的时空变化、起关键作用的信号通路以及相关的表观遗传学调控机制等尚不清楚。本研究先行从全基因组表达芯片分析人手,筛选出分化前后发生显著差异表达的基因,通过基因本体(gene ontology,GO)分析和信号通路(pathway)分析方法初步明确这些差异表达基因的大体功能分类和参与的信号通路,进一步寻找决定分化方向的关键基因及其调控机制。
1 材料与方法
1.1 基因芯片分析
选取大鼠骨髓MSCs经肝细胞提取液诱导分化后肝细胞做基因芯片实验,分析MSCs与分化后肝细胞之间的基因表达谱差异。本实验采用Illumina大鼠全基因组基因表达芯片ratref-12(由上海伯豪生物科技有限公司完成)。芯片结果采用illumina SherlockTM1000分析扫描仪进行扫描,BeadStudio软件读取、处理数据。通过芯片图像分析软件对芯片灰度扫描图进行分析,可以得到芯片上每个基因点的原始信号值,即所有有效重复点的前景信号值减去背景信号值的平均信号值(Avg_Signal),Detection Pval等。根据这些参数值进行后续的数值分析。本实验选择的归一化方法为:Cubic Spline。mRNA使用了limma进行显著性水平的计算,设置参数 P <0.05,fold change≥2[Foldchange=Sig-nal MIA-PaCa2(TIChigh)/Signal BxPc-3(TIClow)]。对差异基因进行功能分析(GO-Analysis)和通路分析(path-way-analysis),根据差异基因间的基因调控网络,从网络当中得到基因间的相互作用关系,及在网络当中处于调控关键点的基因。
1.2 实时定量PCR验证
用筛选出网络当中的5个关键基因及进行验证。依照GenBank序列号获取全长cDNA序列由上海伯豪生物设计并合成上下游引物。在ABIPRSIM7900检测仪上进行SYBRGreen法荧光定量PCR检测。
1.3 统计学分析
2 结果
2.1 基因表达谱GO及Pathway生物信息学分析
通过对12张大鼠的全基因表达谱芯片的差异基因进行注释系统(gene ontology)分析和信号通路(pathway)分析,探讨这种表达现象在生物学上的意义。分析结果显示在GO三大类中,生物学过程(biological process,BP)、分子功能(molecular fuction,MF)及细胞组分(cellular component,CC)总共有1 633个基因表达水平发生了变化。在Pathway分析结果中显示共有45个信号通路发生了显著性变化,具有显著性意义。基于KEGG数据库中的基因间的调控关系,对差异表达基因之间的关系进行整合处理,得到差异基因间的信号通路调控网络(图1)。通过对基因调控网络当中的基因进行网络学分析,按照基因的度(degree)筛选出网络当中的关键基因,如表1所示。
2.2 荧光定量PCR实验对结果进行验证分析结合全基因表达的GO分析及Pathway
分析挑选出可信值高,并可能在大鼠MSCs诱导HLCs相关的基因做进一步的芯片验证实验。以证实芯片的结果的可靠性。我们这里挑选了基因调控网络当中5个关键基因以及 Notch、Mapk、Wnt、JAK/STST途径中的基因进行验证。具代表性的11个差异基因在芯片中的差异倍数和在real-time PCR实验(图2)中的差异倍数进行比较,得到real-time PCR结果和基因芯片表达的结果相符(表2)。

图1 差异基因间的关系网络Fig 1 Network relationship between gene difference

图2 实时定量PCR的结果Fig 2 Real-time PCR results
3 讨论
通过GO分析得到差异基因主要涉及细胞分裂、细胞周期、有丝分裂、DNA复制、四氢叶酸生物合成的过程、血管生成、积极调控转录因子的活动等,提示在大鼠MSCs定向分化为HLCs的过程正如其在胚胎发育的过程中表现的情形类似,开启细胞的相关增殖、分化、代谢等过程,大量合成成熟分化后细胞所行使生物学功能相关的蛋白组分。我们可以看出大鼠MSCs在诱导分化为HLCs的过程中是处于一种高度的合成状态。合成分化后细胞需要的各个部件,同时也在关闭某些基因的表达,这可能与组织的特化相关。在细胞成分GO分类中,我们可以看出染色体着丝粒区域、细胞骨架、蛋白质的细胞外基质等细胞内外上的组分相关基因均变化差异较大。这一系列基因表达的变化对于MSCs分化为HLCs均可能是必要的。差异基因的显著性Pathway分析基于KEGG,对差异基因进行Pathway注释,其主要涉及DNA复制、细胞周期、嘧啶代谢、嘌呤代谢、脂类代谢、错配修复、谷胱甘肽代谢、烟酸和烟酰胺代谢、叶酸生物合成、趋化因子信号通路、癌症通路等。以上可以看出差异基因的显著性Pathway以细胞增殖、分化、各种物质代谢和调控为主。按照基因的度(degree)筛选出信号通路调控网络当中的关键基因与肝细胞的增殖、分化、转移、凋亡,以及肝细胞特有的能量代谢密切相关,在骨髓间充质干细胞向肝系细胞分化过程中具有重要意义。肝脏发育涉及多种信号因子及通路。有报道MSCs可自行表达肝细胞生长因子及肝细胞生长因子受体/C-met,肝细胞生长因子与其受体相结合,启动一跨膜信号传导通路,通过Notch信号通路参与MSCs分化为肝细胞过程,研究结果表明Notch信号通路在MSCs分化成肝细胞过程进行调控是必需的,但是分化必须在信号通路下调的情况下才能进行下去[2],也有提出Notch信号对肝细胞增殖存在正性调控。此外,Heo等通过实验证明,Wnt/β-catenin信号通路对胚肝细胞的增殖分化起作用[3]。我们知道DNA甲基化在定向分化的过程中起着非常重要的作用,某些基因的甲基化和去甲基化,直接决定分化后的生物表型和功能,在生长发育和疾病发生中有指导作用[4]。5-AzaC是一种DNA甲基转移酶抑制剂.其去甲基化作用可促进肝脏相关基因的表达。MSCs向HLCs分化过程中,肝细胞微环境可能与MSCs向肝细胞分化紧密相关[5]。从MSCs定向分化为HLCs,基因表达谱的变化控制蛋白组的变化,进而控制细胞功能和表型,从表达谱中差异基因入手,找到关键基因和通路,可望阐明肝细胞定向分化的调控机制,为临床实践进行理论和方法学探索。

表1 关键基因在网络当中的网络参数Table 1 The network parameters of key genes in the network
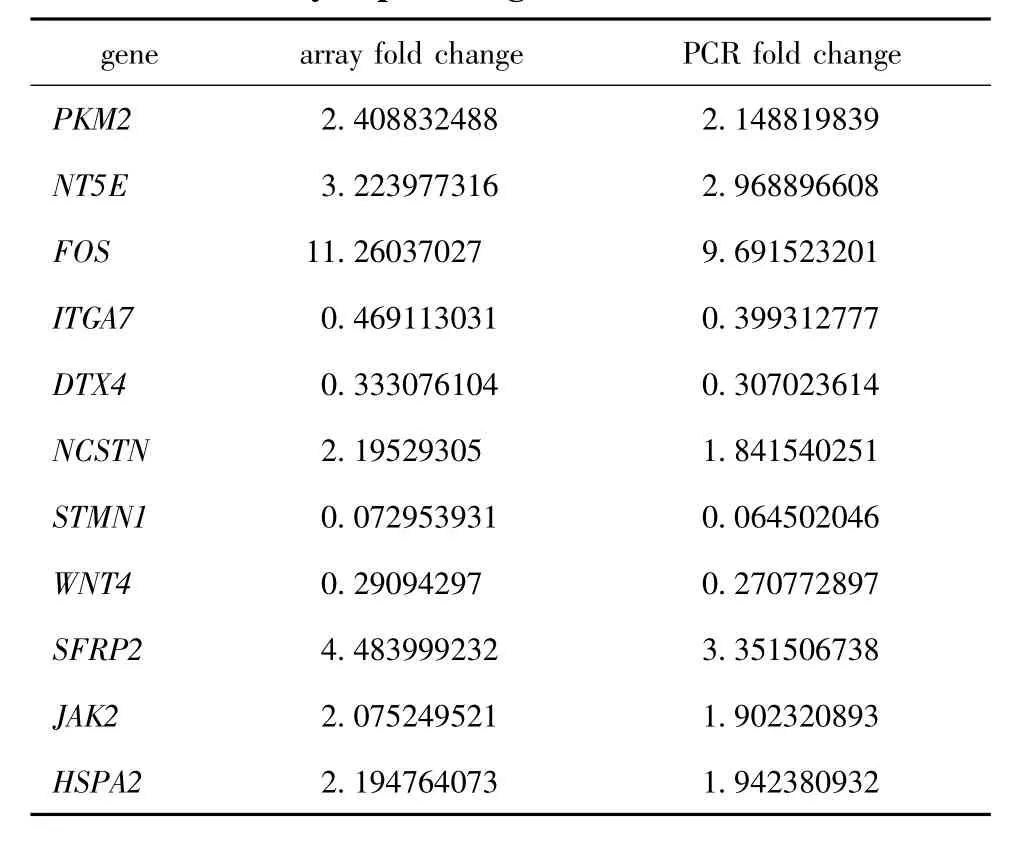
表2 差异表达基因的qRT-PCR验证结果Table 2 Results of qRT-PCR to verify the differentially expressed gene
[1]Ren H,Zhao Q,Cheng T,et al.No contribution of umbilical cord mesenchymal stromal cells to caDillarization and venularization of hepatic sinusoids accompanied by hepatic differentiation in carbon tetrachloride-induced mouse liver fibrosis[J].Cytotherapy,2010,12:371-383.
[2]柳柯.Notch信号通路在骨髓间充质干细胞向肝细胞分化过程中的动态表达特征[J].中国临床解剖学杂志,2013,3:308-313.
[3]Heo J,Ahn EK,Jeong HG,et al.Transcriptional characterization of Wnt pathway during sequential hepatic differentiation of human embryonic stem cells and adipose tissue-derived stem cells[J].Biochem Biophysi Res Commun,2013,434:235-240.
[4]Xenaki D,Martin IB,Yoshida L,et al.Fudey AJ.F3/contactin and TAGl play antagonistic roles in theregulation of sonic hedgehog-induced cerebellar granuleneuron progenitor proliferation[J].Development,2011,138:519-529.
[5]Kaibori M,Adachi Y,Shimo T,et al.Stimulation of liver regeneration after hepatectomy in mice by injection of bone marrow mesenchymal stem cells via the portal vein [J].Transplant Proc,2012,44:1107-1109.
