植物磷酸化蛋白质组学研究进展
2015-03-02刘秋林钟月仙万伟峰林授楷薛李春艾玉芳柯玉琴何华勤
刘秋林,钟月仙,万伟峰,田 甜,林授楷,2,黄 健,薛李春,艾玉芳,柯玉琴,何华勤
(1.福建农林大学生命科学学院,福建 福州350002;2.莆田学院环境与生物工程学院,福建 莆田 351100)
植物磷酸化蛋白质组学研究进展
刘秋林1,钟月仙1,万伟峰1,田 甜1,林授楷1,2,黄 健1,薛李春1,艾玉芳1,柯玉琴1,何华勤1
(1.福建农林大学生命科学学院,福建 福州350002;2.莆田学院环境与生物工程学院,福建 莆田 351100)
首先阐述了植物细胞蛋白质磷酸化的作用机制,并从植物磷酸化蛋白质的分离与鉴定技术、植物磷酸化蛋白质数据库及植物蛋白质磷酸化位点的预测工具,对植物磷酸化蛋白质组的研究进展进行综述.最后分析了植物磷酸化蛋白质组研究中亟需关注的3个问题,即磷酸化蛋白质的分离与鉴定技术的提升、位点预测工具的完善及蛋白质功能的分析.
植物;磷酸化蛋白质组学;进展
蛋白质磷酸化是蛋白质翻译后修饰的一种重要方式,是一种可逆的过程,包括蛋白质磷酸化修饰与去磷酸化修饰[1-5].蛋白质磷酸化修饰是在蛋白激酶的催化下供体磷酸基团转移到受体蛋白质的某个氨基酸残基上的过程,而蛋白质去磷酸化是在磷酸化酶的催化下进行的(图1).在真核生物中,蛋白质磷酸化主要发生在丝氨酸、苏氨酸和酪氨酸上;在原核生物中蛋白质磷酸化也可能发生在天冬氨酸、谷氨酸和组氨酸上[6].研究表明,真核生物中发生磷酸
化修饰的酪氨酸、苏氨酸和丝氨酸的数量比约为1∶200∶1800[7].被磷酸化修饰的蛋白质称为磷酸化蛋白质,一个磷酸化蛋白质可能含有1个或多个磷酸化氨基酸位点[8].
研究[9,10]表明生物体内蛋白激酶的激活、细胞变异与转化、细胞发育等都受到蛋白质磷酸化的调节.而且蛋白质的可逆磷酸化是目前人类所知道的生物体内最主要的信号传递方式[11].真核生物体内随时都有三分之一的蛋白质被磷酸化[6],且真核生物中编码蛋白激酶和磷酸化酶的基因占全基因组的2%-4%[12].但通过生物化学试验从各物种中鉴定获得的磷酸化蛋白质的数量十分有限[13].随着磷酸化蛋白质富集与鉴定技术的不断发展,探索生物体生命活动过程的蛋白质磷酸化现象,特别是分析蛋白质磷酸化修饰对功能蛋白质表达的调控作用已成为一个热点问题[14].
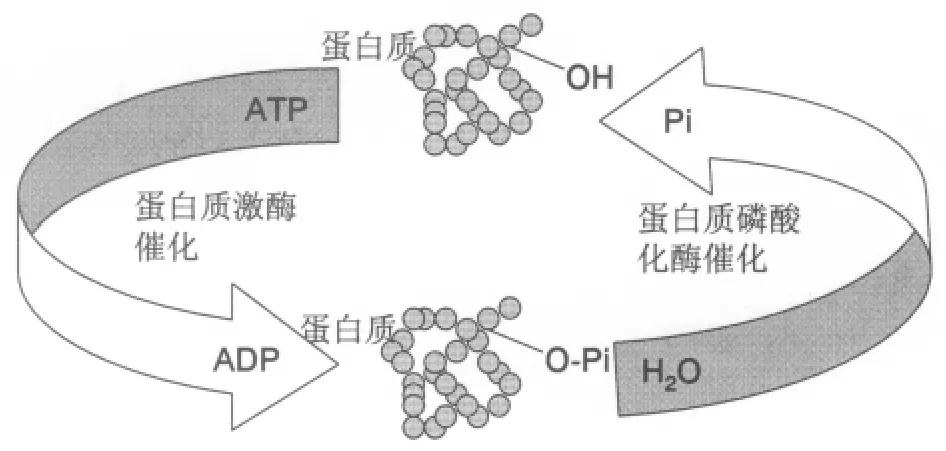
图1 蛋白质磷酸化修饰过程Fig.1 Process of protein phosphorylation and de-phosphorylation
1 植物蛋白质磷酸化修饰的作用机理
植物在适应各种不同生理生态环境(如光、水、营养、病虫害)以及在不同生长发育过程中都伴随着蛋白质磷酸化或去磷酸化修饰现象的发生[15].蛋白质磷酸化修饰的作用主要体现在如下3个方面.一是通过磷酸化修饰改变了受体蛋白质的活性.蛋白质磷酸化或去磷酸化修饰起到开启或关闭蛋白质活性的作用.Chao et al[16]应用质谱技术检测发现拟南芥磷酸丙酮酸羧化酶(PEPCK)的第55位点丝氨酸(Ser55)、58和59位点苏氨酸(Thr58、Thr59)在光诱导下发生磷酸化修饰,尤其是Ser55位点的磷酸化修饰程度与PEPCK的活性成负相关,表明第55位点丝氨酸的磷酸化抑制了光诱导的PEPCK脱羧活性.二是磷酸化蛋白质参与植物体内信号的传导.Medzihradszky et al[17]研究发现拟南芥叶片中光受体——光敏素B(Phytochrome B)中位于第86位点的丝氨酸(Ser86)若被磷酸化修饰,光敏素B的活性就会受到抑制,从而阻碍了植株光诱导的信号传导.三是蛋白质磷酸化影响蛋白质间的互作.由于在氨基酸残基上结合或失去了磷酸基团,从而改变了受体蛋白质的结构,影响了该受体蛋白质与其他蛋白质间的互作.在水稻和拟南芥中,三维结构分析表明α-微管蛋白的Thr349位于与β-微管蛋白互作的交界面上,对于α与β微管蛋白的互作及二聚体蛋白的形成至关重要.而Thr349的磷酸化修饰,影响了α与β微管蛋白质间的互作,导致微管解聚[18].
细胞中蛋白质磷酸化水平是一个动态的变化过程,其细微差异都可能导致细胞代谢水平上的变化[8].因此,蛋白质磷酸化对植物生长发育的影响是全方位的.
2 植物磷酸化蛋白质的富集与鉴定技术
富集和分离出磷酸化蛋白质是后续对磷酸化位点进行鉴定的前提.目前对磷酸化蛋白质的分离富集技术主要有32P放射性标记法、特异抗体的免疫共沉淀法、荧光染料染色和色谱分离法等技术[19].由于细胞中磷酸化蛋白质在植物组织总蛋白质中所占的比例较小,检测时易受其他蛋白质的干扰.因此,各种不同的分离富集方法都是从组织总蛋白质中富集分离出磷酸化蛋白质,再通过下游的质谱技术鉴定磷酸化蛋白质.
2.1 32 P 标记法
该方法利用32P同位素示踪标记蛋白质氨基酸上的磷酸基团,从而确定蛋白质磷酸化修饰的状态.日本学者Khan et al[20]运用[γ32P]标记分析GA处理下水稻叶鞘中的磷酸化蛋白质,共鉴定出44个发生磷酸化修饰蛋白质,其中有13个磷酸化蛋白质参与信号传递.由于32P标记具有放射性,试验操作要求较高,也比较繁琐.因此,近年来,随着新型磷酸化蛋白质分离富集方法的兴起,使用32P标记法来分离磷酸化蛋白质的研究少见报道.
2.2 免疫共沉淀法
该方法首先应用双向电泳技术(2-DE)分离组织细胞的总蛋白质,然后用磷酸化丝氨酸、磷酸化苏氨酸和磷酸化酪氨酸的特异抗体先后进行免疫共沉淀(western-blotting),成像后从对应的2-DE凝胶上切取磷酸化蛋白质点,胶上酶解后进行质谱分析鉴定.Kaufmann et al[21]首次合成磷酸化丝氨酸、磷酸化苏氨酸和磷酸化酪氨酸的特定抗体.应用该方法,He et al[22]、Ke et al[23]鉴定出ABA调控及干旱胁迫下水稻的磷酸化蛋白质.Rush et al[24]富集了4种细胞的600多个酪氨酸磷酸化的肽段,其中磷酸化位点约559个.相对而言,磷酸化丝氨酸和苏氨酸抗体的抗原决定簇较小,免疫共沉淀较为困难,而磷酸化酪氨酸抗体的抗原决定簇适中,更适用于免疫共沉淀分析.
2.3 荧光染料染色(Pro-Q diamond phosphoprotein stain,Pro-Q DPS)
Pro-Q可以高效地与磷酸化的氨基酸残基结合.先采用2-DE技术对组织细胞的总蛋白质进行分离,然后用Pro-Q对SDS-PAGE胶进行染色,只有磷酸化蛋白质才会结合Pro-Q而显色.而且Pro-Q可与银染或考马斯亮蓝一起使用.Agrawa et al[25]发现Pro-Q DPS染色液稀释3倍后同样能保持较高的灵敏度与线性度,使Pro-Q DPS试验成本大幅降低.他们使用改进后的Pro-Q DPS方法,鉴定出在油菜籽发育不同阶段均发生磷酸化修饰的70个蛋白质,并分析其功能,此研究是人类第1次大规模检测植物的磷酸化蛋白质组[26].美国学者Chittetiet al[27]运用SYPro Rubby染色法和Pro-Q染色法分离鉴定盐胁迫下水稻根系的磷酸化蛋白质与差异表达蛋白质,共鉴定出28个磷酸化蛋白质和31个差异表达蛋白质.Chen et al[28]也应用Pro-Q技术分离并鉴定出高温胁迫下苗期水稻叶片的磷酸化蛋白质.但荧光染料染色(Pro-Q DPS)对低丰度磷酸化蛋白点的结合率较低,易造成低丰度磷酸化蛋白质的丢失.
2.4 色谱柱法
该方法是根据磷酸化蛋白质和非磷酸化蛋白质或杂质的不同性质,通过特异性材料进行选择性吸附和洗脱,达到分离磷酸化蛋白质的目的.
2.4.1 固相金属离子亲和色谱(immobilized metal affinity chromatography,IMAC)1975年Porath提出该方法,刚开始只用于分离标记组氨酸,后来逐渐用于分离富集磷酸化蛋白质[29].其原理是磷酸化蛋白质会与IMAC硅脂柱上的固相化金属离子(Ga2+或Fe3+)通过静电作用结合,然后先洗脱非磷酸化的杂质,最后富集和分离出磷酸化蛋白质[30].该方法也存在一些缺点,如比较难洗脱有多个磷酸化位点与IMAC柱紧密结合的蛋白质或多肽;部分磷酸化蛋白质和多肽因未与IMAC柱结合而丢失[31];有些带负电的酸性氨基酸(如天冬氨酸、谷氨酸)可能与柱材料结合而引入杂质.研究[32]发现富集前对含酸性氨基酸的肽段进行甲酯化反应,把羧基转化为羧甲酯,可减弱非特异性吸附.
2.4.2 强阳离子交换色谱(SCX) 大多数肽段经过强阳离子交换与胰蛋白酶处理后带上2个负电荷,而磷酸基团带有1个负电荷.经SCX色谱柱时,带单电荷的磷酸化多肽由于其与柱的结合力弱先流出,带多电荷的肽段由于其与柱的结合力强后流出.因此可以从含有多电荷复杂的非磷酸化肽段中分离出磷酸化肽段[33].研究人员应用该技术从海拉细胞核中分离鉴定出967种蛋白质的2002个磷酸化位点[34],而且发现将SCX和IMAC联用的分离效果更佳[35].
2.4.3 金属氧化物亲和色谱 是一种新兴的磷酸化多肽富集技术,常用的金属氧化物有 TiO2和Al(OH)3等.TiO2柱层析的原理与IMAC类似.TiO2是一种两性电解质,在酸性溶液中Ti带正电,能与磷酸盐结合;在高pH或磷酸缓冲液中又可被洗脱下磷酸化多肽[36].这种方法会受到溶液中酸性氨基酸的影响,若在上样溶液中加入2,5-二羟基苯甲酸(2-hydroxy-5-methoxybenzoicacid,DHB),可提高TiO2富集的特异性[37].Prak et al[38]应用改进后的TiO2柱层析法富集拟南芥细胞膜的磷酸化蛋白质,找到6个新的磷酸化位点.
Al(OH)3富集原理是Al(OH)3中Al3+与磷酸基团具有亲和作用,而非磷酸化多肽及酸性多肽与Al3+的结合率较低.采用Al(OH)3色谱,Wolschin et al[39]鉴定出拟南芥种子中9个蛋白质的16个磷酸化位点.
2.5 磷酸钙沉淀法
该方法的基本原理是在碱性条件下磷酸基团与磷酸钙螯合而沉淀,在酸性条件下又被释放.先用磷酸钙沉淀蛋白质酶解液中的磷酸化肽段,然后再用IMAC或者TiO2法富集磷酸化肽段,可显著提高富集效率.该方法快捷、成本低,可对复杂样品进行初筛.Zhang et al[40]通过将磷酸钙沉淀法与IMAC联用,鉴定了水稻胚细胞中125种蛋白质的242个磷酸化位点.
2.6 化学标记法
化学标记法是利用磷酸化的丝氨酸或苏氨酸经过消除反应后形成双键,再与硫代乙二醇发生加成反应,用巯基取代磷酸根,然后在巯基上连接生物素或加入双重功能的新配基后进行分离富集[41].化学标记技术的最大不足是磷酸化的酪氨酸不能发生消除反应.
2.7 磷酸化蛋白质的鉴定技术
质谱技术是一种常用、可靠的蛋白质或多肽分析方法,但近年来兴起的蛋白质微阵列技术在磷酸化蛋白质的检测上表现出自身的优势.蛋白质微阵列是指将大量蛋白质或配基按一定序列高密度地固定在特殊处理的载玻片上,形成蛋白质探针点阵.蛋白质微阵列技术具有快速、灵敏和易于自动化等多种优点[42],可用于检测低丰度的磷酸化蛋白质[43].可以选择不同的蛋白激酶作探针来检测磷酸化蛋白质.Kersten et al[44]制作了第1张植物(拟南芥)蛋白质微阵列,并采用该技术分析植物中促分裂素原活化蛋白激酶的信号传导途径[45].研究人员在采用多肽阵列技术研究拟南芥根系细胞特异性的磷酸化蛋白质及其激酶时,发现H2O2能诱导MPK3催化而使mRNA的剪接因子SLC30发生磷酸化,而H2O2不影响富含丝氨酸和精氨酸的蛋白质特异性激酶4(SRPK4),这个发现为证明激酶的动态性提供了新的依据[46].
由上述分析可知,不同的磷酸化蛋白质富集分离技术方法各有优势,也都存在不足之处.为了减少鉴定结果的假阳性,可通过多种方法互补进行磷酸化蛋白质的分离与鉴定.
3 植物磷酸化蛋白质数据库
生物学数据的不断积累,促进了相应生物学数据库的发展,使生物学数据库的研究逐渐成为相应学科研究的重要组成部分[47].研究人员应用各种生物化学方法,从不同物种中鉴定出大量的蛋白质磷酸化位点.生物信息学研究人员对这些蛋白质磷酸化位点数据进行整理,开发出具有不同用途的磷酸化蛋白质数据库,如收集来自各种物种磷酸化蛋白质的数据库——Phospho.ELM,还有关于蛋白质翻译后修饰的数据库dbPTM,甚至前人还专门开发出拟南芥蛋白磷酸化位点数据库——PhosPhAt2.2[48-50].下面简要介绍一些常用的植物磷酸化蛋白质数据库.
3.1 Phospho.ELM 数据库
该数据库是通过手动的方式从公开发表的文献资料中收集经试验鉴定来自不同物种的蛋白质磷酸化位点.现共存储有42574个非冗余的丝氨酸、苏氨酸和酪氨酸磷酸化位点.在Phospho.ELM中不仅收录了相应激酶催化的蛋白质磷酸化位点,还附加了相关的信息,包括参考文献、亚细胞分布、参与的信号传导途径等[48].还有一些新的特征也被存储在其中,如结构性乱紊型、规则型以及保守型位点等.最后还链接到分子互作数据库(MINT).
3.2 dbPTM 数据库
dbPTM的数据来源主要是从SWISS-PROT,O-GLYCBASE和Phospho.ELM数据库中整合已经获得试验验证的蛋白质翻译后的修饰位点数据,并不断地更新和加入新的蛋白质翻译后的修饰数据.dbPTM还对一些注释信息较少的数据,尤其是磷酸化、硫酸化和糖基化蛋白质进行预测和注释[49].
3.3 PhosPhAt数据库
PhosPhAt数据库专门收录拟南芥的蛋白质磷酸化位点数据.目前共收录了6300多个蛋白质的14000多个磷酸化位点信息[50].同时,PhosPhAt也是一个专门用于拟南芥的蛋白质磷酸化位点的预测工具.
可以预见,蛋白质磷酸化数据库的开发将往分类精细、信息全面、数据准确、冗余率低的方向发展.
4 植物蛋白质磷酸化位点的预测工具
生物学数据的不断积累使得通过数据训练算法开发出各种预测工具成为可能.2006年报道了150个新的在线预测工具[10].研究人员开发出许多蛋白质磷酸化位点预测工具,实现对蛋白质磷酸化位点预测的高通量、自动化与低成本化[51].由于哺乳动物和人类磷酸化蛋白质数据的积累较快,前期研究人员开发出的蛋白质磷酸化位点预测工具大多更适用于预测动物和人类的蛋白质磷酸化位点[51].近年来,随着植物磷酸化蛋白质组学研究的不断发展,研究人员也开发出适用于植物的蛋白质磷酸化预测工具.
4.1 M usite 预测工具
Musite适用于所有或特定激酶催化的磷酸化位点预测[52].它利用机器学习的方法将磷酸化位点预测模拟为一个失衡的分类问题.该预测工具收集了多种生物体磷酸化蛋白质组的可靠试验数据,用这些数据来训练磷酸化位点的预测模型.Musite工具使用的3种特征提取方法分别为K-Nearest Neighbor(KNN)、Protein Disorder和氨基酸频率.KNN方法能获取由相同激酶或激酶家族所催化的磷酸化位点周围序列的相似性.Disorder分值反映了磷酸化残基在无序区域出现的可能性.
Musite 1.0共训练了6种生物体的蛋白质磷酸化位点预测模型和13个激酶或激酶家族的特定激酶预测模型.这6种生物体分别是Homo sapiens(人类)、Muroidea musculus(老鼠)、Drosophila melanogaster(果蝇)、Caenorhabditis elegans(蠕虫)、Saccharomyces cerevisiae(酵母)和Arabidopsis thaliana(拟南芥),用户可以自由选择一个生物模型进行预测.Musite可满足用户对不同置信度的要求.用户可以根据需要,把预测的阈值类型设置为特异性或分值,并且可以连续地调整其特异性值或分值.
4.2 PlantPhos预测工具
PlantPhos应用最大依赖性分解(MDD)方法[53],将所有磷酸化片段进行聚类,形成具有显著位点特异性的磷酸化片段子集[54];并且把每个MDD聚类产生的序列子集的模序,与Phospho.ELM数据库中已知激酶的作用模序进行对比[48];对MDD聚类产生的每个优化序列子集,运用隐马尔可夫模型(HMM)分别建立相应模型[55].用户在搜索时,HMMER会返回1个HMMER值和期望值(E值).HMMER值是比率的对数值,该比率是查询序列具有显著匹配的可能性与序列是由随机模型产生可能性的比值.E值表示期望值大于或等于返回的HMMER值的序列数目.若某位点的查询结果中,HMMER值大于临界值,则表明该位点被预测为磷酸化位点[54].
4.3 PhosphAt预测工具
以试验鉴定出的拟南芥蛋白质磷酸化位点数据为训练集,使用支持向量机算法(SVM)预测蛋白质的丝氨酸磷酸化位点.如果SVM计算获得的预测值大于0,则认为该位点被磷酸化;如果该预测值小于0,则认为该位点未被磷酸化[56].
4.4 PhosphoRice 预测工具
PhosphoRice是专门用于水稻蛋白质磷酸化位点的预测工具.常见的磷酸化蛋白质预测工具包括Netphos2.0、Kinasephos、Scansite、NetphosK、Disphos、Kinasephos2.0 和 Predphosphos,首先利用它们对训练数据库的蛋白质磷酸化位点数据进行预测分析,获得各工具预测结果的灵敏度Sn、特异性Sp、准确度ACC和马修斯系数MCC等指标.从中选择若干种预测效果较好的工具进行整合.整合方法包括限制性网格搜索法、未加权表决法、减数加权表决法和加权表决法等.通过设定不同的权重和阈值,从中获得最佳的权重分配,最后集合成一个专门针对水稻的蛋白质磷酸化位点预测工具——PhosphoRice[57].
5 展望
近年来,数学、物理、化学和生物科学等学科间的不断融合,促进了植物磷酸化蛋白质组学研究技术与方法的发展,使得大规模磷酸化蛋白质的分离与鉴定成为可能;植物磷酸化蛋白质数据库的功能趋于精细;蛋白质磷酸化位点预测工具的性能日渐成熟.通过加强以下3个方面的研究促进植物磷酸化蛋白质组学研究往纵深发展.
(1)优化蛋白质磷酸化位点的分离与鉴定技术.当前植物磷酸化蛋白质组学研究发展的一个瓶颈是分离与鉴定技术.各种技术普遍存在假阳性高、自动化程度低等弊端,而且由于缺乏类似DNA扩增的技术,低丰度磷酸化蛋白质也不易被检测到.只有磷酸化蛋白质分离与鉴定技术取得突破性进展,才能支撑植物磷酸化蛋白质组学研究的跨越性发展.
(2)开发高通量、精确性高的植物蛋白质磷酸化位点预测工具,引领植物磷酸化蛋白质组学研究从“尝试型”向“验证型”方式的转变.传统的生物学研究策略大都采用“尝试—错误—再尝试—发现”的模式,为发现一个蛋白质磷酸化的位点,往往需要花费大量的时间和经费.如果应用预测工具先筛选出可能的磷酸化位点,再应用生物化学试验进行验证,这种“验证型”研究策略不但提高了试验效率,也节省了开支.因此,未来植物磷酸化蛋白质组学研究的一个方向就是要在磷酸化蛋白质位点数据持续积累的基础上,采用合理有效的特征提取策略,训练优异的算法,开发出更精确、高通量的蛋白质磷酸化位点预测工具,为下游生化试验指明方向[51].
(3)磷酸化蛋白质的功能分析.磷酸化蛋白质的功能分析是后基因组时代的重要研究内容.尤其是在蛋白质磷酸化位点的预测和生物化学鉴定技术不断完善的前提下,蛋白质磷酸化修饰或去磷酸化修饰的功能分析就显得尤为必要.氨基酸点突变[17]、去磷酸化[22]及基因敲除等技术,都可用于蛋白质磷酸化修饰的功能分析研究.
[1]PECK SC.Early phosphorylation events in biotic stress[J].Current Opinion Plant Biology,2003,6(4):334-338.
[2]KHAN M,TAKASAKIH,KOMATSU S.Comprehensive phosphoproteome analysis in rice and identification of phosphoproteins responsive to different hormones/stresses[J].Journal of Proteome Research,2005,4:1592-1599.
[3]CHOU K C,WATENPAUGH K,DAND HEINRIKSON R L.A model of the complex between cyclin-dependent kinase 5(Cdk5)and the activation domain of neuronal Cdk5 activator[J].Biochemical and Biophysical Research Communications,1999,259:420-428.
[4]CHOU K C.Structural bioinformatics and its impact to biomedical science[J].Current Medicinal Chemistry,2004,11:2105-2134.
[5]HUBBARD M,JAND CP.On targetwith a newmechanism for the regulation of protein phosphorylation[J].Trends Biochem Sci,1993,18(5):172-177.
[6]姜铮,王芳,何湘,等.蛋白质磷酸化修饰的研究进展[J].生物技术通讯,2009,20(2):233-237.
[7]HUNTER T.The phosphorylation of proteins on tyrosine:its role in cell growth and disease[J].Phil Trans R Soc LondB,1998,353:583-605.
[8]张倩,杨振,安学丽,等.蛋白质的磷酸化修饰及其研究方法[J].首都师范大学学报:自然科学版,2006,27(6):43-49.
[9]HUNTER T.Signaling-2000 and beyond[J].Cell,2000,100:113-127.
[10]WAN J,KANG S,TANG C,et al.Meta-prediction of phosphorylation sites with weighted voting and restricted grid search parameter selection[J].Nucleic Acids Research,2008,36(4):22.
[11]YANG X H,SLADEK T L.Novel phosphorylated forms of E2F-1 transcription factor bind to the retinoblastoma protein in cells overexpressing an E2F-1 cdna[J].Biochemical and Biophysical Research Communications,1997,2:336-339.
[12]MANN M,ONG SE,GRONBORG M,et al.Analysis of protein phosphorylation usingmass spectrometry:deciphering the phosphoproteome[J].Trends Bioteehnol,2002,20:261-268.
[13]ZHANG B,CHEN Y,ZHENG Y,etal.Reaction of1,4-dien-3-one in ADD with some common protective agents of carbonyl[J].Fudan University Journal of Medical Sciences,2002,5:387-389.
[14]REINDERS J,SICKMANN A.State-of-the-art in phosphoproteomics[J].Proteomics,2005,5:4052-4061.
[15]KRSTEN B,AGRAWALG K,IWAHASHIH,et al.Plant phosphoproteomics:along road ahead[J].Proteomics,2006,6:5517-5528.
[16]CHAOQ,LIU X Y,MEIY C,etal.Light-regulated phosphorylation ofmaize phosphoenolpyruvate carboxykinase plays a vital role in its activity[J].Plant Mol Biol,2014,85(1-2):95-105.
[17]MEDZIHRADSZKY M,BINDICS J,ÁDÁM É,et al.Phosphorylation of phytochrome B inhibits light-induced signaling via accelerated dark reversion in Arabidopsis[J].Plant Cell,2013,25(2):535-44.
[18]BAN Y,KOBAYASHIY,HARA T,et al.α-tubulin is rapidly phosphorylated in response to hyperosmotic stress in rice and Arabidopsis[J].Plant Cell Physiol,2013,54(6):848-58.
[19]魏鑫,李海霞,陈卫卫,等.植物蛋白质磷酸化的研究技术[J].植物生理学通讯,2009,45(9):899-906.
[20]KHAN M,TAKASAKIH,KOMATSU S.Comprehensive phosphoproteome analysis in rice and identification of phosphoproteins responsive to different hormones/stresses[J].JProteome Res,2005,4(5):1592-9.
[21]KAUFMANN H,BAILEY JE,FUSSENEGGER M.Use of antibodies for detection of phosphorylated proteins separated by two-dimensional gel electrophoresis[J].Proteomics,2001,1(2):194-199.
[22]HE H,LIJ.Proteomic analysis of phosphoproteins regulated by abscisic acid in rice leaves[J].Biochemical and Biophysical Research Communications,2008,371:883-888.
[23]KE Y,HAN G,HE H,et al.Differential regulation of proteins and phosphoproteins in rice under drought stress[J].Biochemical and Biophysical Research Communications,2009,379:133-138.
[24]RUSH J,MORITZA,LEEK A,etal.Immunoaffinity profiling of tyrosine phosphorylation in cancer cells[J].Nat Biotechnol,2005,23:94-101.
[25]AGRAWALG K,THELEN JJ.Development of a simplified,economical polyacrylamide gel staining protocol for phosphoproteins[J].Proteomics,2005,5:4684-4688.
[26]AGRAWALG K,THELEN JJ.Large-scale identification and quantitative profiling of phosphoproteins expressed during seed filling in oil seed rape[J].Mol Cell Proteomics,2006,5:2044-2059.
[27]CHITTETIB R,PENG Z.Proteome and phosphoproteome differential expression under salinity stress in rice(Oryza sativa)roots[J].JProteome Res,2007,6(5):1718-1727.
[28]CHEN X,ZHANGW,HE H.Phosphoproteins regulated by heat stress in rice leaves[J].Proteome Science,2011,9:37.
[29]LAUGESEN S,BERGOIN A,ROSSIGNOLM.Deciphering the plant phosphoproteome:tools and strategies for a challenging task[J].Plant Physiol Biochem,2004,42:929-936.
[30]FEUERSTEIN I,MORANDELL S,STECHER G,et al.Phosphoproteomic analysis using immobilized metalion affinity chromatography on the basis of cellulose powder[J].Proteomics,2005,5:46-54.
[31]STENSBALLE A,ANDERSEN S,JENSEN O N.Characterization of phosphop roteins from electrophoretic gels by nanoscale Fe(Ⅲ)affinity chromatog raphy with off-linemass spectrometry analysis[J].Proteomics,2001,1:207-222.
[32]FICARRO SB,MCCLELAND M L,STUKENBERG P T,et al.Phosphoproteome analysis bymass spectrometry and its application toSaccharomyces cerevisiae[J].Nat Biotechnol,2002,20:301-305.
[33]BALLIF B A,VILLEN J,BEAUSOLEIL SA,etal.Phosphoproteomic analysis of the developingmouse brain[J].Mol Cell Proteomics,2004,2:1234-1243.
[34]BEAUSOLEIL SA,JEDRYCHOWSKIM,SCHWARTZD,etal.Large-scale characterization of HeLa cell nuclear phosphoproteins[J].ProcNatl Acad Sci USA,2004,101:12130-12135.
[35]TRINIDAD JC,SPECHTCG,THALHAMMER A,etal.Comprehensive identification of phosphorylation sites in postsynaptic density preparations[J].Mol Cell Proteomics,2006,5:914-922.
[36]PINKSEMW H,UITTOPM,HILHORSTM J,etal.Selective isolation at the femtomole levelof phosphopeptides from proteolytic digests using 2D-NanoLC-ESI-MS/MS and titanium oxide precolumns[J].Anal Chem,2004,76:3935-3943.
[37]LARSEN M R,THINGHOLM T E,JENSEN O N,et al.Highly selective enrichment of phosphorylated peptides from mixtures using titanium dioxidemicrocolumns[J].Molecular & Cellular Proteomics,2005,47:873-886.
[38]PRAK S,HEM S,BOUDET J,et al.Multiple phosphorylations in the C-terminal tail of plant plasmamembrane aquaporins[J].Mol Cell Proteomics,2008,7:1019-1030.
[39]WOLSCHIN F,WECKWERTHW.Combiningmetal oxide affinity chromatography(MOAC)and selectivemass spectrometry for robust identification ofin vivoprotein phosphorylation sites[J].Plant Methods,2005,1:9-14.
[40]ZHANG X,YE JY,JENSEN O N,et al.Highly efficient phosphopeptide enrichment by calcium phosphate precipitation combined with subsequent IMAC enrichment[J].Mol Cell Proteomics,2007,6:2032-2042.
[41]隋少卉,王京兰,蔡耘,等.磷酸化蛋白质组学分析和定量技术的研究进展[J].生物化学与生物物理进展,2007,34(3):240-245.
[42]LOYET K M,STULTS J T,ARNOTT D.Mass spectrometric contributions to the practice of phosphorylation site mapping through 2003:a literature review[J].Mol Cell Proteomics,2005,4:235-245.
[43]KREUTZBERGER J.Protein microarrays:a chance to study microorganisms[J].Appl Microbiol Biotechnol,2006,70:383-390.
[44]KERSTEN B,FEILNER T,KRAMER A,et al.Generation of Arabidopsis protein chips for antibody and serum screening[J].Plant Mol Biol,2003,52:999-1010.
[45]FEILNER T,HULTSCHIG C,LEE J,et al.High throughput identification of potentialArabidopsismitogenactivated protein kinases substrates[J].Mol Cell Proteomics,2005,4:1558-1568.
[46]DE LA FUENTEVAN BENTEM S,ANRATHER D,DOHNAL I,et al.Site-specific phosphorylation profiling ofArabidopsisproteins bymass spectrometry and peptide chip analysis[J].Proteome Res,2008,7:2458-2470.
[47]张晓东,张传富,彭科峰,等.生物信息学数据库研究进展[J].生物信息学,2006,3:143-145.
[48]DIELLA F,CAMERON S,GEMÜND C,et al.Phospho.ELM:a database of experimentally verified phosphorylation sites in eukaryotic proteins[J].BMC Bioinformatics,2004,5:79.
[49]LEE TY,HUANG H D,HUNG JH,etal.DbPTM:an information repository of protein post-translationalmodification[J].Nucleic Acids Research,2006,34:622-627.
[50]HEAZLEWOOD JL,DUREK P,HUMMEL J,et al.PhosPhAt:a database of phosphorylation sites inArabidopsis thalianaand a plant specific phosphorylation site predictor[J].Nucleic Acids Research,2008,36:1015-1021.
[51]QUE S,WANG Y,CHEN P,et al.Evaluation of protein phosphorylation site predictors[J].Protein and Peptide Letters,2010,17:61-68.
[52]GAO J,THELEN J,DUNKER A K,etal.Musite,a tool for global prediction of general and kinase-specific phosphorylation sites[J].Molecular & Cellular Proteomics,2010,9:2586-2600.
[53]LEE T Y,LIN ZQ,HSIEH S J,et al.Exploitingmaximal dependence decomposition to identify conserved motifs from a group of aligned signal sequences[J].Bioinformatics,2011,27(13):1780-1787.
[54]LEE T,BRETANA N,LU C.PlantPhos:using maximal dependence decomposition to identify plant phosphorylation sites with substrate site specificity[J].BMC Bioinformatics,2011,12:261.
[55]EDDY SR.Profile hidden Markov models[J].Bioinformatics,1998,14(9):755-763.
[56]DUREK P,SCHMIDTR,HEAZLEWOOD JL,etal.PhosPhAt:the arabidopsis thaliana phosphorylation site database[J].Nucleic Acids Research,2010,38:828-834.
[57]陈铭.水稻蛋白质磷酸化位点预测工具Meta-Server的构建[D].福州:福建农林大学,2011.
(责任编辑:叶济蓉)
New advance in the research of plant phosphoproteome
LIU Qiu-lin1,ZHONG Yue-xian1,WANWei-feng1,TIAN Tian1,LIN Shou-kai1,2,HUANG Jian1,XUE Li-chun1,AIYu-fang1,KE Yu-qin1,HE Hua-qin1
(1.College of Life Sciences,Fujian Agriculture and Forestry University,Fuzhou,Fujian 350002,China;2.College of Environmental and Biological Engineering,Putian University,Putian,Fujian 351100,China)
The functions of protein phosphorylation were discussed firstly.Then,the advances in the research of plant phosphoproteome were reviewed,including themethods of phosphoproteins extraction and identification,the databases of protein phosphorylated sites and the predictors of protein phosphorylation sites.Finally,the new research areas in plant phosphoproteomewere put forward,including the improvement of the technology of phophosprotein extraction and identification,the enhancement of the performance of predictors on phophorylation sites,and the analysis of the function of phosphorylated protein.
plant;phosphoproteomics;advance
Q51
A
1671-5470(2015)03-0225-07
10.13323/j.cnki.j.fafu(nat.sci.).2015.03.001
2014-11-25
2015-01-05
国家自然科学基金资助项目(31270454);福建省自然科学基金资助项目(2013J01077).
刘秋林(1988-),男,硕士研究生.研究方向:植物蛋白质组学.通讯作者何华勤(1968-),男,教授,博士生导师.研究方向:植物逆境生理与分子生态学.Email:hehq3@fafu.edu.cn.
