应用变性高效液相色谱法检测幽门螺杆菌对克拉霉素的耐药性
2015-02-14吴文冰蔡鹏威方超英
吴文冰,蔡鹏威,方超英,沈 菁,陈 雯
应用变性高效液相色谱法检测幽门螺杆菌对克拉霉素的耐药性
吴文冰1,蔡鹏威2,方超英3,沈 菁1,陈 雯1
目的 建立DHPLC快速检测Hp对克拉霉素耐药性的新方法。方法 抽提56份经纸片琼脂扩散法鉴定为克拉霉素耐药的Hp菌株DNA及其对应的胃粘膜组织标本DNA,通过PCR技术扩增23S rRNA区187 bp基因,运用DHPLC技术对PCR产物进行突变分析,并进行DNA测序。结果 56份克拉霉素耐药的Hp菌株DNA经DHPLC分析,有51份存在异源双链峰,测序证实均存在点突变,5份无异源双链峰,测序证实无点突变,故DHPLC对基因突变耐药菌株的敏感性和特异性为100%,而且56份组织标本DNA检测结果与菌株DNA检测结果一致。结论 DHPLC可应用于Hp对克拉霉素耐药性的快速检测,具有广阔的临床应用前景。
幽门螺杆菌;23S rRNA;克拉霉素耐药;变性高效液相色谱法;基因突变

幽门螺杆菌(Helicobacterpylori,Hp是一种在人类胃粘膜中发现的革兰氏阴性微需氧菌,它与各种消化性疾病,例如消化性溃疡、胃炎及淋巴相关淋巴组织淋巴瘤等关系密切,是胃癌的高危因素之一。克拉霉素是根治Hp感染最重要的抗生素之一,近年来其耐药性呈上升趋势,而Hp对克拉霉素耐药是导致治疗失败的主要原因。变性高效液相色谱法(denaturing high performance liquid chromatography, DHPLC)可自动检测单碱基替代及小片段核苷酸的插入或缺失,已被证实为一种快速、简便的检测基因突变的技术,目前广泛应用于肿瘤和遗传病等的基因研究。本研究应用DHPLC来检测Hp的23S rRNA V区DNA序列突变,探讨23S rRNA V区基因与耐克拉霉素的的相关性及其耐药的分子机制,同时为临床提供更为简便、经济、快速的检测方法。
1 材料与方法
1.1 研究对象 收集本院胃镜室尿素酶快速检测法(福建三强公司)阳性的胃窦部或胃体部粘膜活检标本160份,其中确诊为消化性溃疡62份,胃炎98份,男性104份,女性56份,年龄20~72岁,平均年龄(37.1±13.9)岁,所有研究对象均签署知情同意书。所有活检标本经充分研磨混匀后分成两份,一份用于抽提组织DNA,另一份用于培养及药敏鉴定。
1.2 研究方法
1.1.1Hp的培养及药敏鉴定 培养采用含Hp选择添加剂(OXOID公司)的哥伦比亚琼脂(OXOID公司)培养基,微需氧环境(5%的O2,10%的CO2及850%的N2)培养5 d,所得单个菌落经显微镜镜检形态吻合,且尿素酶、过氧化氢酶及氧化酶均阳性的判定为Hp。药敏鉴定参照文献[1]采用纸片琼脂扩散法,即取纯培养72 h的菌落,用生理盐水调成1.0×108CFU/mL,接种于含5%羊血的水解酪蛋白(Mueller-Hinton,M-H)培养基平板,在培养基平板中央贴15 μg的克拉霉素药敏纸片(OXOID公司),微需氧环境(5%的O2,10%的CO2及85%的N2)培养5 d,采用临床和实验室标准协会(2012)标准,用游标卡尺读取抑菌环直径,当抑菌环直径大于17 mm时为克拉霉素敏感菌株,当抑菌环直径小于13 mm时为克拉霉素耐药菌株。
1.1.2Hp的DNA抽提及PCR扩增 随机抽取50份克拉霉敏感菌株和所有克拉霉素耐药菌株,采用QIAamp DNA Mini Kit和DNeasy Tissue kit(Qiagen公司)分别抽检菌株及其对应的胃粘膜组织DNA,严格按操作说明书进行,所得产物作为模板,根据GenBank中Hp的23S rRNA V 区DNA序列(编号U27270)设计合成PCR扩增引物,上游引物为:5′- CGTAACGAGATGGGAGCTGT -3′,下游引物为:5′- ACTCCATAAGAGCCAAAGCCC -3′(上海铂尚公司合成),PCR条件:95 ℃变性30 s,95 ℃30 s、57 ℃30 s及72 ℃30 s,共35个循环,72 ℃延伸2 min,取5 μL产物进行琼脂糖电泳,紫外灯下观察结果,其余产物保存于-20 ℃冰箱。
1.1.3 DHPLC分析 以幽门螺杆菌标准菌株(编号:NCTC11637,本实验室保存)为DHPLC分析的标准参考模板,将敏感菌和耐药菌的菌株及组织DNA的PCR产物分别和标准菌株DNA的PCR产物等量混和,采用WAVETM2100型分析仪(Transgenomic公司,美国)进行检测,在仪器分析软件推荐的变性温度上下1~2 ℃范围内筛选出最佳变性温度,所得数据采用随机分析软件WAVEMaker®进行分析。
1.1.4 DNA测序 将50份敏感菌和耐药菌的菌株DNA及其对应的胃粘膜组织DNA送上海铂尚公司进行测序。
2 结 果
2.1 克拉霉素耐药率 从160份活检样本中鉴定出56份耐克拉霉素的Hp,耐药率为35%。
2.2 PCR扩增产物电泳结果 50份敏感菌和56份耐药菌的菌株和胃粘膜组织抽提所得的DNA均扩增出特异性条带,扩增片段大小为187 bp,与设计的片段大小相一致,见图1-2。

1: NCTC11637 wild-type; 2:Helicobacterpyloriresistant isolates; 3:Helicobacterpylorisensitivity isolates; M: DL500 DNA Marker (Takara).
图1 幽门螺杆菌DNA扩增产物电泳图
Fig.1 PCR product of clinicalHelicobacterpyloriisolates
2.3 DHPLC分析结果 56份耐药菌的菌株DNA

1-16:Helicobacterpyloriresistant isolates; M: DL500 DNA Marker (Takara).
图2 部分耐药菌菌株DNA扩增产物电泳图
Fig.2 PCR product of someHelicobacterpyloriresistant isolates
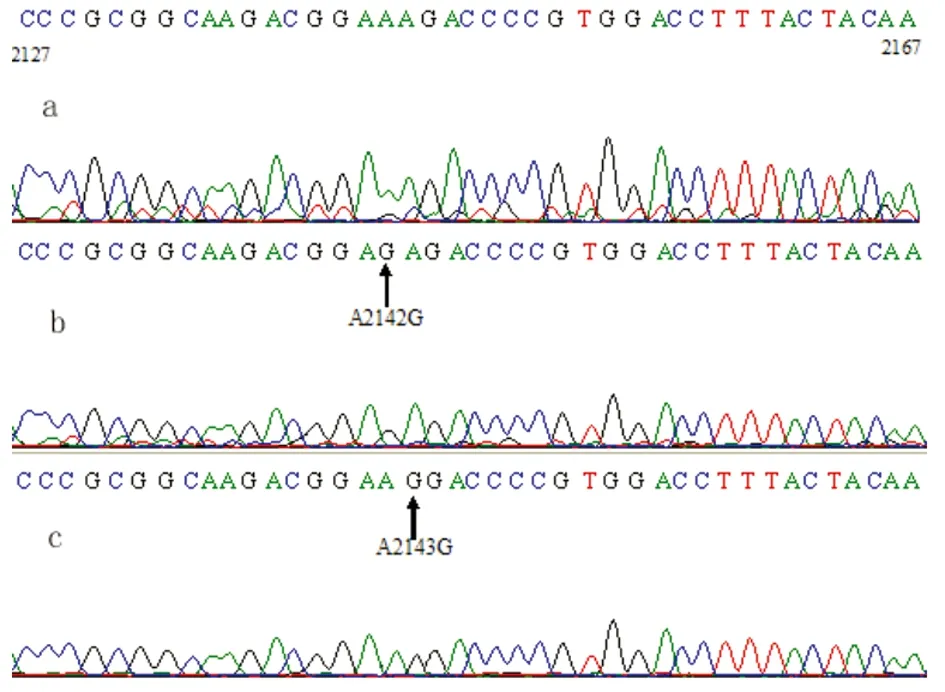
和胃粘膜组织DNA的PCR产物经DHPLC分析,结果完全一致。其中51份存在异源双链峰,异常峰一致的样本为同一基因突变型;5份无异源双链峰,峰型与野生型一致。50份敏感菌菌株DNA和胃粘膜组织DNA的PCR产物经DHPLC分析,结果也完全一致,均无异源双链,峰型与野生型一致。DHPLC分析结果见,图3。

A: NCTC11637 wild-type; b: the point mutations of A2142G; c: the point mutations of A2143G.
图3 DHPLC分析结果
Fig.3 Analysis of the DHPLC
2.4 50份敏感菌的菌株DNA和胃粘膜组织DNA的PCR产物经DNA测序,结果完全一致,与NCTC11637野生株DNA完全一致。 56份耐药菌的菌株DNA和胃粘膜组织DNA的PCR产物经DNA测序,结果也完全一致,其中51份存在异源双链峰,有31份为A2142G,20份为A2143G,测序结果见图4。
3 讨 论
克拉霉素的化学名称是6-甲基红霉素, 是当前应用最广泛的大环内酯类抗生素之一。其抗菌机制是其能进入细胞内并结合在Hp23S rRNA功能区V区的50S大亚基上,抑制肽酰基转移酶,影响核糖体的位移过程,阻止肽链的延长,从而抑制细菌蛋白合成,达到清除Hp的目的[2],此外克拉霉素具有对酸稳定和在胃黏膜中浓度高、口服后生物利用率高和不良反应少等优点,加之临床上含克拉霉素的三联疗法与不含克拉霉素的三联疗法相比,Hp根除率可以提高10%~20%,因此克拉霉素很快成为了根除Hp治疗方案的主要抗菌药物。
A: NCTC11637 wild-type; b: the point mutations of A2142G; c: the point mutations of A2143G.
图4 DNA测序图
Fig.4 Sequencing of the DNA
随着克拉霉素的广泛使用,临床上逐渐开始出现耐克拉霉素的Hp,早期的耐药率基本低于10%,而近年来其耐药率呈明显的上升趋势,超过了20%(如亚洲为21%,美国为29.3%)[3],更为严重的是在儿童中也出现了高耐药的Hp[4],因此阐明克拉霉素的耐药机制迫在眉睫,目前有关克拉霉素耐药机制的研究越来越多,使人们对克拉霉素的耐药有了比较全面深入的研究,最主要的机制可能克拉霉素结合靶位突变有关,包括:23S rRNA V区的肽基转移酶基因编码区域出现点突变和23S rRNA转录后甲基化区域的突变[5-6],前者是目前比较公认的重要耐药机制之一,包括A2143G、A2142G、A2142C、A2115G、G2141A、C2147G、T2190C、C2195T、A2223G和C2694A[7],这些突变表明任何V区基因突变都可能导致Hp对克拉霉素的耐药,其中以A2143G和A2142G最常见,但不同地区,不同种族,不同人群的主要突变类型可不相同,如Vega等研究认为儿童以A2143G基因突变为主,成人以A2142G基因突变为主[8]。其次还与多重耐药流出泵(multidrug resistance efflux pump)基因改变[9-11],大环内酯类药物之间的交叉耐药[12-13],以及rRNA甲基化酶引起大环内酯类药物失活、细胞膜渗透性下降、大环内酯类药物排出增加等[14]有关。
目前检测Hp耐药的方法可以分为两类:一类是表型检测,另一类是基因型检测。表型检测主要是培养加药敏鉴定(包括琼脂糖稀释法和Etest法),但Hp培养条件较为苛刻,且培养周期较长,一般要1~2周才能出报告,而且由于活检标本污染等原因导致其有10%的漏检率,从而限制了其临床应用。随着对Hp耐药研究的深入,越来越多的研究表明Hp23S rRNA的V区基因点突变是导致Hp耐药的主要原因,于是基于PCR的分子生物学方法被引入了Hp耐药的检测,1996年Versalovic等[15]首次通过PCR-限制性片段长度多态性证实了Hp23S rRNA的V区的A2142G和A2143G与Hp耐药有关,此后人们相继应用PCR-寡核苷酸连接分析法、PCR-DNA酶联免疫测定法DNA、PCR逆转录杂交线探针分析法、 PCR选择性同源杂交分析法等检测Hp的基因点突变,这些方法的操作都相对复杂,而且耗时,难以在临床推广。
新近,一种新的高通量筛选DNA序列变异的技术-变性高效液相色谱法开始逐步应用于临床,该技术可在短时间内(10 min左右)自动检测单碱基替代及小片段核苷酸的插入或缺失,其原理是用离子对反向高效液相色谱法分离并检测异源双链。该方法具有自动化、高通量、快速、检出率高(可达95%以上)、检出DNA片段大小范围广等优点,主要用于人类基因多态性和疾病相关性的研究。Patrizia Posteraro等首先尝试将DHPLC应用到Hp23S rRNA的V区基因点突变的检测,其研究表明DHPLC能检测到所有81份耐药菌株和96份胃粘膜组织DNA中点突变,并且每种点突变都伴有特异的洗脱峰型[16]。我们实验结果表明,在56份耐克拉霉素的Hp耐药菌株DNA中检出51份存在峰型异常,经基因测序证实都存在点突变,其中31份为A2142G,20份为A2143G,5份未检测出峰型异常,基因测序也未见突变,50份敏感菌菌株DNA经DHPLC分析均无异源双链峰,基因测序也未见突变,与NCTC11637野生株一致,表明DHPLC对基因突变耐药菌株的敏感性和特异性均达到100%,同时,我们也将这50份敏感菌菌株和56份耐药菌株对应的组织DNA进行DHPLC分析,得到了相同的的实验结果,提示DHPLC不但可以用于菌株DNA,也可以用于组织DNA的检测,这与Patrizia Posteraro等研究相似,这就为临床提供了一个更为快速的检测方法,从而避免了耗时的培养过程,更为临床尽早采取正确的治疗方案提供依据。
[1]Li JZ, Yan QH, Bing PC, et al.Helicobacterpyloriisolates from ethnic minority patients in Guangxi: Resistance rates, mechanisms, and genotype[J]. World J Gastroenterol, 2014, 20(16): 4761-4770.
[2]Garrido L, Toledo H. Novel genotypes inHelicobacterpyloriinvolving domain V of the 23S rRNA gene[J]. Helicobacter, 2007, 12(5): 505-509.
[3]Yang JC, Lu CW, Lin CJ. Treatment ofHelicobacterpyloriinfection: Current status and future concepts[J]. World J Gastroenterol, 2014, 20(18): 5283-5293.
[4]Pacifico L, Osborn JF, Bonci E, et al. Probiotics for the treatment ofHelicobacterpyloriinfection in children[J]. World J Gastroenterol, 2014, 20(3): 673-683.
[5]Gerrits MM, van Vliet AH, Kuipers EJ, et al.Helicobacterpyloriand antimicrobial resistance: molecular mechanisms and clinical implications[J]. Lancet Infect Dis, 2006, 6(11): 699-709.
[6]Megraud F, Lehours P.Helicobacterpyloridetection and antimicrobial susceptibility testing[J]. Clin Microbiol Rev, 2007, 20(2): 280-322.
[7]Zhao LJ, Huang YQ, Chen BP, et al.Helicobacterpyloriisolates from ethnic minority patients in Guangxi: Resistance rates, mechanisms, and genotype[J]. World J Gastroenterol, 2014, 20(16): 4761-4770.
[8]Vega AE, Alarcon T, Domingo D, et al. Detection of resistance to clarithromycin in clinical isolates ofHelicobacterpylorifrom children and adults[J]. Rev Esp Quimioterap, 2003, 16(1): 53-57.
[9]Tsugawa H, Suzuki H, Muraoka H, et al. Enhanced bacterial efflux system is the first step to the development of metronidazole resistance inHelicobacterpylori[J]. Biochem Biophys Res Commun, 2011, 404(2): 656-660.
[10]Hirata K, Suzuki H, Nishizawa T, et al. Contribution of efflux pumps to clarithromycin resistance inHelicobacterpylori[J]. J Gastroenterol Hepatol, 2010, 25(Suppl): S75-S79.
[11]Alvarez-Ortega C, Olivares J, Martinez JL. RND multidrug efflux pumps: what are they good for?[J]. Front Microbiol, 2013, 4: 7.
[12]Agudo S, Alarcon T, Cibrelus L, et al. High percentage of clarithromycin and metronidazole resistance inHelicobacterpyloriclinical isolates obtained from Spanish children[J]. Rev Esp Quimioter, 2009, 22(2): 88-92.
[13]Pacifico L, Osborn JF, Bonci E, et al. Probiotics for the treatment ofHelicobacterpyloriinfection in children[J]. World J Gastroenterol, 2014, 20(3): 673-683.
[14]Agudo S, Perez-Perez G, Alarcon T, et al. High prevalence of clarithromycin-resistantHelicobacterpyloristrains and risk factors associated with resistance in Madrid, Spain[J]. J Clin Microbiol, 2010, 48(10): 3703-3707.
[15]Versalovic J, Osato MS, Spakovsky K, et al. Point mutations in the 23S rRNA gene ofHelicobacterpyloriassociated with different levels of clarithromycin resistance[J]. J Antimicrob Chemother, 1997, 40(2): 283-286.
[16]Posteraro P, Branca G, Sanguinetti M, et al. Rapid detection of clarithromycin resistance inHelicobacterpyloriusinga PCR-based denaturing HPLC assay[J]. J Antimicrobial Chemother, 2006, 57(1): 71-78.
Detection of clarithromycin resistance inHelicobacterpyloriby denaturing high performance liquid chromatography assay
WU Wen-bing1,CAI Peng-wei2,FANG Chao-ying3,SHEN Jing1,CHEN Wen1
(1.DepartmentofLaboratoryMedicine,FujianProvincialHospital,ProvincialClinicalCollegeofFujianMedicalUniversity,Fuzhou350001,China;2.DepartmentofLaboratoryMedicine,FujianProvincialSouthHospital,ProvincialClinicalCollegeofFujianMedicalUniversity,Fuzhou350001,China;3.DepartmentofEndoscopeCenter,FujianProvincialHospital,ProvincialClinicalCollegeofFujianMedicalUniversity,Fuzhou350001,China)
The aim of this study is to evaluate the denaturing high performance liquid chromatography (DHPLC) assay for the rapid detection of clarithromycin resistance inHelicobacterpylori. A 187 bp fragment of the 23S rRNA gene was amplified using DNA from 56 clinicalHelicobacterpyloriisolates, which were shown to be resistant to clarithromycin by agar dilution, and directly from the homologous gastric biopsies. DHPLC and sequencing were used to detect mutations in all PCR products. For the 56 resistant isolates, 51 of the 56 resistant isolates showed heteroduplex peaks, which were easily distinguishable from the homoduplex pesks of the wild-typeHelicobacterpylorireference strain. Sequencing revealed point mutations in all the 51 resistant isolates. Five of the 56 resistant isolates showed homoduplex peaks, sequencing revealed no point mutations in all the 5 resistant isolates. Our results suggested that the DHPLC assay, whose sensitivity and specificity were 100% in detecting point mutations, was a valid tool for rapid assessment of clarithromycin resistance inHelicobacterpyloriand that in the future it could be used directly on biopsy specimens, avoiding the need for culture-based methods.
Helicobacterpylori; 23S rRNA; clarithromycin resistance; denaturing high performance liquid chromatography assay; gene mutations
福建省卫生厅青年科研课题(No.2009-2-3)资助
蔡鹏威,Email:cpweico@163.com
1.福建医科大学省立临床医学院 福建省立医院检验科,福州 350001; 2.福建医科大学省立临床医学院 福建省立金山医院检验科,福州 35000; 3.福建医科大学省立临床医学院 福建省立医院内镜中心,福州 350001
10.3969/j.issn.1002-2694.2015.11.013
R378
A
1002-2694(2015)11-1050-04
2015-07-02;
2015-09-11
