辽宁传统发酵豆酱中乳酸菌及酵母菌分离鉴定
2015-01-30武俊瑞王晓蕊唐筱扬王茜茜乌日娜岳喜庆
武俊瑞,王晓蕊,唐筱扬,王茜茜,乌日娜,2,*,岳喜庆
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2.江南大学食品学院,食品科学与技术国家重点实验室,江苏 无锡 214122)
传统发酵豆酱是以大豆为原料,经过煮制、挤压成块、制曲、发酵而成,在制曲和发酵过程中,利用原料和环境中的微生物自然发酵,微生物利用蛋白质、脂肪、碳水化合物等大分子物质分解成为小分子的风味物质,使豆酱的pH值和各种养分发生变化[1-5]。由于其良好的风味和营养价值广受人们欢迎。传统发酵豆酱中含有异黄酮类、多酚类、大豆皂苷、类黑精、肽类、维生素等大量对人体有益的生理活性物质,具有很好的保健功能[3,5-7]。
豆酱中微生物的种类繁多,包括乳酸菌、酵母菌、霉菌等。乳酸菌是豆酱呈味的主要微生物,可将精氨酸、酪氨酸、组氨酸及天冬氨酸分解,同时对丝氨酸、苏氨酸和苯丙氨酸等进行特异性脱羧基作用,左右着酱的香气[8-10]。酵母菌在豆酱发酵过程中主要起着发酵糖类产酸和产生醇类的作用[11-12]。另外乳酸菌与酵母菌协同作用,乳酸和乙醇生成乳酸乙酯,和鲁氏酵母生成糠醇,从而产生独特的酱香气。同时乳酸菌发酵产生的乳酸使得体系pH值降低,有利于酵母菌的增殖,而酵母菌发酵产生的大量乙醇却会制约着乳酸菌的繁殖[13]。乳酸菌和酵母菌在豆酱发酵中相互协同也相互影响。
本研究以采自辽宁地区的38份传统发酵豆酱样品为实验材料,从豆酱中分离出乳酸菌和酵母菌疑似菌株,再利用16S rDNA序列分析方法和26S rDNA D1/D2序列同源性分析,对其进行鉴定并保藏,为进一步开发利用豆酱中乳酸菌和酵母菌资源提供了基础数据。
1 材料与方法
1.1 材料
从辽宁省地区传统手工制酱农家采集豆酱样品38份,见表1。

表1 样品采集结果Table1 Geographic sources of samples collected for this study
1.2 培养基与试剂
MRS培养基、马铃薯葡萄糖(potato dextrose agar,PDA)培养基、孟加拉红琼脂培养基 本实验室自行配制。
细菌基因组DNA小量制备试剂盒、DNA凝胶回收试剂盒、Taq DNA聚合酶、DNA Marker 上海桑尼生物科技有限公司。
1.3 仪器与设备
JNOEC XS-212-201生物显微镜 日本Olympus公司;5804R型离心机 德国Eppendorf公司;SHP-1500型低温生化培养箱 上海精宏实验设备有限公司;HZQ-F160全温振荡培养箱 哈尔滨东联电子技术开发有限公司;2720型Thermal Cycler PCR仪 美国ABI公司;Tanon 2500凝胶成像系统 中国天能公司;ND-1000型微量紫外分光光度计 美国Nano Drop公司。
1.4 乳酸菌分离鉴定及其多样性分析
1.4.1 乳酸菌的分离纯化与保存
采用倾注培养法,将豆酱样品进行10倍梯度稀释后,取10-4~10-7的样品稀释液100 μL于平板中,倒入含有CaCO3的灭菌MRS琼脂培养基20 mL,混合均匀并凝固后,于30℃培养24~48 h。挑取有钙圈的单菌落,在灭菌后的MRS固体培养基上反复划线分离,置于30℃培养24~48 h,确定为单菌落后置于10倍放大镜下进行菌落形态学观察,并且进行革兰氏染色,于100倍光学显微镜下进行菌体形态学观察,并记录[14-16]。最后用MRS固体培养基进行穿刺保藏。
1.4.2 利用16S rDNA序列分析进行鉴定
1.4.2.1 总DNA的提取及纯度的检测
采用十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB)法提取供试菌株基因组DNA[17-18]。使用ND-1000型微量紫外分光光度计检测基因组DNA的浓度以及OD260nm/OD280nm比值,再用1%的琼脂糖凝胶进行电泳检测。
1.4.2.2 聚合酶链式反应(polymerase chain reaction,PCR)扩增16S rDNA序列
16S rDNA 扩增引物:正向引物为27f:5’-AGAGTTT G A T C C T G G C T C A G-3’;反向引物为1 4 9 5 r:5’-CTACGGCTACCTTGTTACGA-3’。PCR扩增反应体系(25 μL):上下游引物各1 μL(10 pmol/μL),模板DNA(100 ng/μL)1 μL,10×PCR Buffer 2.5 μL,dNTP Mix 2 μL,超纯水17.2 μL,rTaq酶0.3 μL。PCR扩增反应程序为:94℃预变性5 min;94℃变性1 min,58℃退火1 min,72℃延伸2 min,循环30次;72℃延伸10 min,4℃保温[19-20]。
1.4.2.3 检测16S rDNA扩增片段及测序
取3 μL的PCR产物与1 μL的6×Loading Buffer混合,加入1%的琼脂糖凝胶点样孔中,在电压为5 V/cm,电泳液为0.5×TBE(Tris硼酸)中电泳。电泳结束后,用溴化乙锭(ethidium bromide,EB)进行染色,染色30 min,将胶板置于凝胶成像系统中观察。PCR产物检测后,片段长度约为1 500 bp的阳性产物直接送上海桑尼生物科技有限公司进行序列测定[21-22]。
1.4.2.4 乳酸菌同源性分析和系统发育树的构建
将所得序列在GenBank数据库中进行基本局部相似性比对搜索工具(basic local alignment search tool,BLAST)同源性比对分析,运用Mega4.0软件的相邻计算法构建系统发育树,判定目的菌的分类地位或它的系统发育地位[18,21]。
1.5 酵母菌分离鉴定及其多样性分析
1.5.1 酵母菌的分离纯化与保存
将豆酱样品进行梯度稀释,取10-4~10-7的样品稀释液100 μL,均匀涂布在孟加拉红琼脂培养基中,25℃培养48 h。挑取与酵母菌菌落形态相符的单个菌落,接于PDA液体培养基中,25℃培养24 h。传至二代,用平板划线的方法,接种于PDA琼脂培养基上,25℃培养48 h[23-24]。挑取培养好的平板上分离出的单菌落,接于PDA液体培养基中依上述纯化过程再次纯化两代。用接种环挑取纯化过的二代菌种,以划线的方式保藏于PDA固体斜面上,25℃培养48 h。
1.5.2 酵母菌26S rDNA D1/D2序列同源性分析
1.5.2.1 酵母菌总DNA的提取和纯度检测
采用酵母基因组DNA提取试剂盒(离心柱型)对酵母菌总DNA进行提取。使用ND-1000型微量紫外分光光度计检测基因组DNA的浓度和OD260nm/OD280nm比值,用1%的琼脂糖凝胶对基因组DNA进行电泳检测。
1.5.2.2 PCR扩增26S rDNA D1/D2序列
26S rDNA D1/D2扩增引物:正向引物为NL1-FA:5’-GCAGAGTTCTCGGAGTCACGAGCATATCAATA AGCGGAGGAAAAG-3’;反向引物为NL4-RA:5’-AG CGGATCACTTCACACAGGAGGTCCGTGTTTCAAG ACGG-3’。PCR扩增反应体系(50 μL):上下游引物各1 μL(10 pmol/μL),模板 DNA(100 ng/μL)1 μL,10×PCR Buffer 5 μL,4×dNTP Mix 1 μL,超纯水40 μL,rTaq酶1 μL。PCR扩增反应程序为:95℃预变性5 min;94℃变性1 min,52℃退火1 min,72℃延伸1 min 20 s,循环36次;72℃延伸8 min,4℃保温[23-26]。
1.5.2.3 检测26S rDNA D1/D2扩增片段及测序
取2 μL的PCR产物与1 μL的6×Loading Buffer混合,加入1%的琼脂糖凝胶点样孔中,在电压为5 V/cm,电泳液为0.5×TBE中电泳。电泳结束后,用EB进行染色,染色30 min,将胶板置于凝胶成像系统中观察。PCR产物检测后,将片段长度约为600 bp的阳性产物直接送上海桑尼生物科技有限公司进行序列测定。
1.5.2.4 酵母菌同源性分析
将所得序列在GenBank数据库中进行BLAST同源性比对分析[25-26]。
2 结果与分析
2.1 豆酱中乳酸菌的分离与鉴定
2.1.1 乳酸菌的表型特征
本实验共从豆酱样品中分离出62株乳酸菌,其中有28株球菌(45.2%),34株杆菌(54.8%)。在MRS固体培养基中,菌落形态为乳白色或透明且表面光滑的圆形菌落。菌落直径集中在0.5~1.5 mm。革兰氏染色发现62株菌均为革兰氏阳性菌,菌落形态分为球状和杆状,排列方式为单个、成对或短链。
2.1.2 乳酸菌总DNA的提取及16S rDNA扩增结果检测

图1 部分菌株16S rDNA PCR扩增产物电泳图Fig.1 Electrophoresis of 16S rDNA PCR products from lactic acid bacteria
图1所示为部分菌株16S rDNA PCR扩增产物经琼脂糖凝胶电泳检测结果,可以观察到所有泳道的1 500 bp左右位置均出现了一条亮带,并且无明显非特异扩增现象,符合测序要求。将PCR片段长度在1 500 bp左右的阳性产物进行序列测定。
2.1.3 乳酸菌16S rDNA同源性分析
将62株乳酸菌16S rDNA扩增产物测序结果同国家生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库进行BLAST同源性对比分析(http://www.ncbi.nlm.nih.gov/BLAST/)。结果如表2所示。

表2 乳酸菌16S rDNA序列分析结果Table2 Results of 16S rDNA sequence analysis for 62 LAB strains
由表2可知,DD1-2等19株菌与Lactobacillus plantarum KJ026699.1的同源性达到99%或100%,所以将其鉴定为L.plantarum;DD2-1等14株与Enterococcus faecium FJ972173.1的同源性达到99%或100%,所以将其鉴定为E.faecium;DD1-1等12株菌与Tetragenococcus halophilus FJ715470.1的同源性达到99%或100%,所以将其鉴定为T.halophilus;DD2-2等11株菌与Lactobacillus sakei EU626014的同源性达到99%或100%,所以将其鉴定为L.sakei;SY1-1等4株菌与Lactobacillus fermentum EF371900.1的同源性达到99%或100%,所以将其鉴定为L.fermentum;DL1-1与DL3-2与Pediococcus pentosaceus AJ305321的同源性达到99%或100%,所以将其鉴定为P.pentosaceu。
本研究从辽宁省14个地区采集到的38份样品中共分离出了62株乳酸菌,通过16S rDNA序列分析后共鉴定出6个种,L.plantarum19株,是样品中分离数量最多的菌种,除锦州和鞍山外均分离到该菌株,占总数的30.65%;E.faecium有14株,除盘锦、锦州和辽阳外均分离到该菌株,占总数的22.58%;T.halophilus有12株,占总数的19.35%;分离到L.sakei 11株,占总数的17.74%。另外,还分离到4株L.fermentum和2株P.pentosaceus,这2株P.pentosaceus均分离自大连样品。14个地区中,有85.71%的地区都分离到了L.plantarum,78.57%的地区分离到了E.faecium,有71.43%的地区分离到了T.halophilus,可初步推测这3种菌为辽宁省地区自然发酵豆酱中存在的优势菌。
2.1.4 豆酱样品中乳酸菌系统发育树的构建
根据同源序列的搜索结果,下载相关模式株的序列,利用本地软件Mega4.0将测得序列与GenBank中的模式菌株16S rDNA序列进行分析,构建系统发育树(图2),获得目的菌的分类地位和它的系统发育地位。
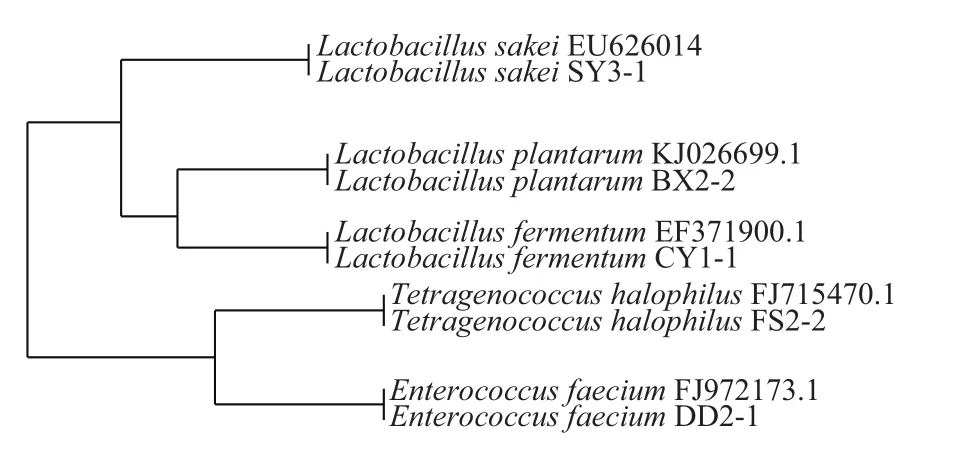
图2 部分乳酸菌16S rDNA序列系统发育树Fig.2 Phylogenetic tree of 16S rDNA sequences of lactic acid bacteria
从系统发育树(图2)可以看出,Lactobacillus sakei SY3-1形成了第一群,Lactobacillus plantarum BX2-2和Lactobacillus fermentum CY1-1的16S rDNA序列在进化关系上亲缘关系较近,形成了第二类群;Tetragenococcus halophilus FS2-2和Enterococcus faecium DD2-1形成了第三类群,所有分离株与相对应的参考菌株同源性都达到了99%以上,并与其聚在一起,证明乳酸菌16S rDNA区域序列分析结果的准确性。
2.1.5 豆酱样品乳酸菌生物多样性分析
本研究从辽宁省14个地区38份样品中共分离到6个种62株乳酸菌,其中乳球菌为3个种28株(45.16%),乳杆菌3个种34株(54.84%),表3汇总了每个种的乳酸菌的地区分布情况。
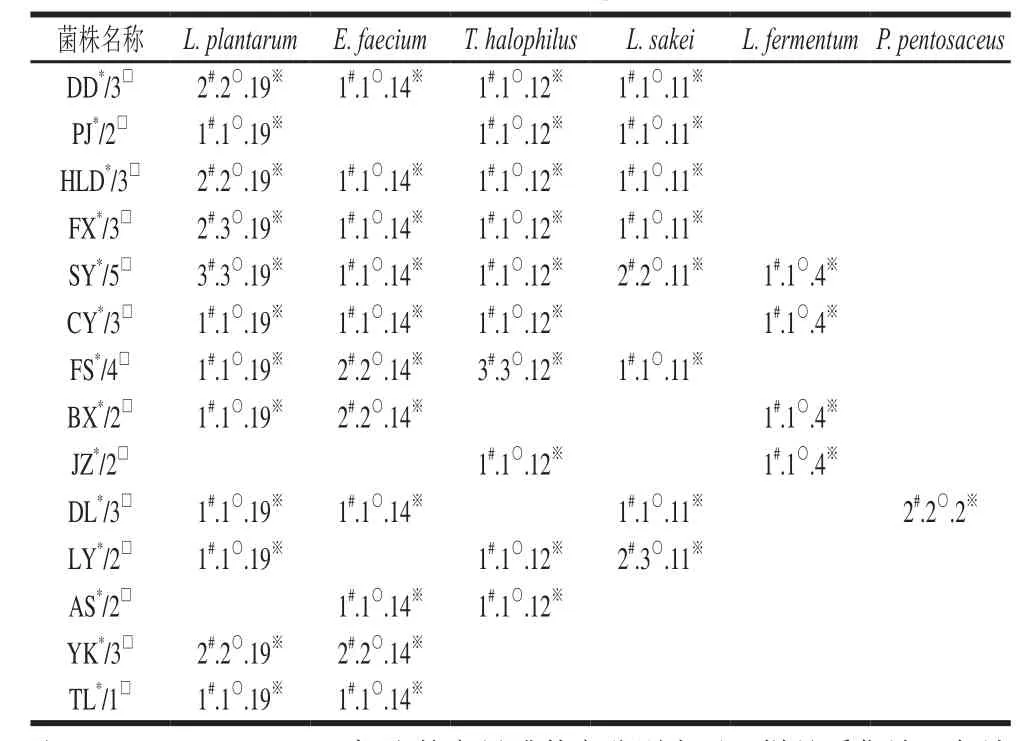
表3 各地区豆酱样品中乳酸菌分离鉴定情况Table3 Distribution of lactic acid bacteria in fermented soybean paste from different regions
由表3可知,从丹东市采集的3个样品中共分离到4个种(占种数的66.67%)5株乳酸菌(占总菌数的8.06%);盘锦市采集的2个样品中分离出乳酸菌3个种(占种数的50%)3株(占总菌数的4.83%);葫芦岛市的3个样品中分离出乳酸菌4个种(占种数的66.67%)5株(占总菌数的8.06%);来自阜新市的3个样品中分离出4个种(占种数的66.67%)乳酸菌6株(占总菌数的9.68%);沈阳市的5份样品中分离出乳酸菌5个种(占种数的83.33%)8株(占总菌数的12.9%);朝阳市的3份样品中分离出乳酸菌4个种(占种数的66.67%)乳酸菌4株(占总菌数的6.45%);抚顺市的4份样品中分离出乳酸菌4个种(占种数的66.67%)乳酸菌7株(占总菌数的11.29%);本溪市的2份样品中分离出乳酸菌3个种(占种数的50%)乳酸菌4株(占总菌数的6.45%);来自锦州市的2个样品中分离到乳酸菌2个种(占种数的33.33%)2株乳酸菌(占总菌数的3.23%);大连市采集的3个样品中分离出4个种(占种数的66.67%)乳酸菌5株(占总菌数的8.06%);辽阳市采集的2个样品中分离出3个种(占种数的50%)乳酸菌5株(占总菌数的8.06%);鞍山市采集的2个样品中分离出乳酸菌2个种(占种数的33.33%)2株(占总菌数的3.23%);营口市采集的3个样品中分离出乳酸菌2个种(占种数的33.33%)4株(占总菌数的6.45%);铁岭市采集的1个样品中分离出乳酸菌2个种(占种数的33.33%)2株(占总菌数的3.23%)。
2.2 酵母菌分离鉴定及多样性分析
2.2.1 酵母菌的表型特征
实验共从豆酱样品中分离出56株酵母菌。在PDA固体培养基中,菌落颜色为乳白色或奶油色,表面光滑,呈圆形或卵圆形。菌落大而厚,直径大于3 mm。
2.2.2 酵母菌总DNA的提取及26S rDNA D1/D2扩增结果检测
图3为部分菌株26S rDNA D1/D2扩增产物经琼脂糖凝胶电泳检测结果,所有泳道的600 bp左右位置均出现了一条亮带,并且无明显非特异扩增现象,符合测序要求。将PCR片段长度在600 bp左右的阳性产物进行序列测定。
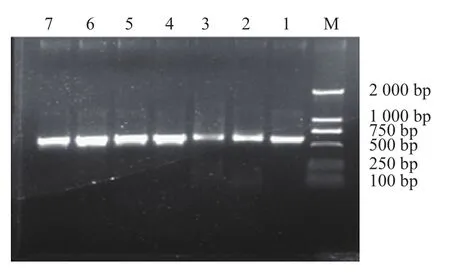
图3 部分菌株26S rDNA D1/D2 PCR扩增产物电泳图Fig.3 Electrophoresis of 26S rDNA D1/D2 PCR products from yeast
2.2.3 酵母菌26S rDNA D1/D2同源性分析
将56株酵母菌26S rDNA D1/D2扩增产物测序结果同NCBI数据库进行BLAST同源性对比分析(http://www.ncbi.nlm.nih.gov/BLAST/)。结果如表4所示。

表4 酵母菌26S rDNA D1/D2序列分析结果Table4 Results of 26S rDNA D1/D2 sequence analysis for yeast
通过在NCBI数据库进行BLAST同源性对比分析(表4),SY2等18株菌被鉴定为Zygosaccharomyces;SY1-B等13株被鉴定为Candida;SY1-A等14株菌被鉴定为C.parapsilosis;SY3-A等7株菌被鉴定为D.hansenii;BX1-B等4株菌被鉴定为Debaryomyces。
本研究从辽宁省14个地区采集到的38份样品中共分离出了56株酵母菌,通过26S rDNA D1/D2序列分析后共鉴定出5个种,Zygosaccharomyces18株,是样品中分离数量最多的菌种,占总数的32.14%;Candida 13株,占总数的23.21%;C.parapsilosis 14株,占总数的25%;分离到D.hansenii 7株,占总数的12.5%。还分离到4株Debaryomyces,分别来自本溪、抚顺、辽阳、营口。14个地区中,有34.21%的地区都分离到了Zygosaccharomyces和C.parapsilosis,26.32%的地区分离到了Candida,可初步推测这3种菌为辽宁省自然发酵豆酱中存在的优势菌。
2.2.4 豆酱样品酵母菌生物多样性分析
本研究从辽宁省14个地区38份样品中共分离到5个种56株酵母菌,表5汇总了每个种的酵母菌的地区分布情况。

表5 各地区豆酱样品中酵母菌分离鉴定情况Table5 Distribution of yeast in fermented soybean paste from different regions
由表5可知,从阜新市采集的3个样品中共分离到4个种(占种数的80%)6株酵母菌(占总菌数的10.71%);沈阳市采集的5个样品中分离出酵母菌4个种(占种数的80%)7株(占总菌数的12.5%);大连市的3个样品中分离出酵母菌4个种(占种数的80%)4株(占总菌数的7.14%);来自鞍山市的2个样品中分离出4个种(占种数的80%)乳酸菌4株(占总菌数的7.14%);营口市的3份样品中分离出酵母菌4个种(占种数的80%)4株(占总菌数的7.14%)。由此可见,辽宁省阜新市和沈阳市自然发酵豆酱中酵母菌多样性较好。
3 结 论
本研究从辽宁省14个地区38份样品中共分离到乳酸菌62株,并用乳酸菌16S rDNA序列分析法对分离到的乳酸菌进行鉴定,鉴定结果为L.plantarum 19株、E.faecium 14株、T.halophilus 12株、L.sakei 11株、L.fermentum 4株、P.pentosaceus 2株。分离到酵母菌56株,经过26S rDNA D1/D2序列分析方法鉴定结果为Zygosaccharomyces 18株、Candida 13株、C.parapsilosis 14株、D.hansenii 7株、Debaryomyces 4株。
14个地区中,有85.71%的地区都分离到了L.plantarum;78.57%的地区分离到了E.faecium;有71.43%的地区分离到了T.halophilus,可初步推测这3种菌为辽宁省自然发酵豆酱中存在的优势菌。14个地区中,有34.21%的地区都分离到了Zygosaccharomyces和C.parapsilosis,26.32%的地区分离到了Candida,可初步推测这3种菌为辽宁省自然发酵豆酱中存在的优势菌。
丹东市、沈阳市、阜新市等地区的样品中乳酸菌的多样性较好,而营口市和铁岭市均发现2种乳酸菌,多样性较差。辽宁省阜新市和沈阳市自然发酵豆酱中酵母菌多样性较好。
[1]包启安.酱及酱油的起源及其生产技术(一)[J].中国调味品, 1992,17(9): 1-4.
[2]汤慧娟, 韩翠萍, 刘洋, 等.传统发酵豆酱的养分变化分析[J].食品与发酵工业, 2013, 39(4): 64-67.
[3]SHUKLA S, PARK H K, KIM J K, et al.Determination of biogenic amines in Korean traditional fermented soybean paste[J].Food and Chemical Toxicology, 2010, 48(5): 1191-1195.
[4]谢韩, 杨中梁.酱和酱油的传承[J].中国酿造, 2013, 32(3): 169-173.
[5]黄胜君.辽东地区传统大豆酱制作工艺[J].中国酿造, 2008, 27(19):75; 81.
[6]高秀芝, 王小芬, 李献梅, 等.传统豆酱和商品豆酱发酵过程中营养及理化指标动态[J].食品科学, 2009, 30(3): 222-226.
[7]KIM H G, HONG J H, SONG C K, et al.Sensory characteristics and consumer acceptability of fermented soybean paste[J].Journal of Food Science, 2010, 75(Suppl 7): 375-383.doi: 10.1111/j.1750-3841.2010.01771.x.
[8]武俊瑞, 张苗, 岳喜庆, 等.黑龙江传统发酵豆酱中乳酸菌的分离鉴定[J].食品与发酵工业, 2014, 40(3): 83-86.
[9]赵建新, 王森, 毛丙永, 等.接种乳酸菌与酵母菌改善豆酱品质[J].食品与发酵工业, 2011, 37(11): 39-45.
[10]梁恒宇, 马莺, 程建军, 等.自然发酵黄豆酱中嗜盐乳酸菌的分离、鉴定与筛选[J].中国酿造, 2006, 25(8): 24-27.
[11]柴洋洋, 葛菁萍, 宋刚, 等.传统发酵豆酱中酵母菌的分离、筛选及功能酵母的鉴定[J].中国食品学报, 2013, 13(3): 183-188.
[12]黄永.黑龙江地区自然发酵黄豆酱中酵母菌的分离鉴定及筛选[D].哈尔滨: 东北农业大学, 2006: 8-10.
[13]李志明, 郭洁, 崔希勇, 等.黄豆酱类食品的微生物状况分析[J].中国调味品, 2008, 33(12): 75-76; 87.
[14]武俊瑞, 李欣, 李晓忱, 等.自然发酵酸菜汁中乳杆菌的分离鉴定[J].食品科学, 2012, 33(15): 191-194.
[15]凌代文, 东秀珠.乳酸细菌分类鉴定及试验方法[M].北京: 中国轻工业出版社, 1999: 84-113.
[16]杨洁彬, 郭兴华, 张篪, 等.乳酸菌: 生物学基础及应用[M].北京: 中国轻工业出版社, 1996: 69-83.
[17]YU J, WANG W H, MENGHE B L G, et al.Diversity of lactic acid bacteria associated with traditional fermented dairy products in Mongolia[J].Journal of Dairy Science, 2011, 94(7): 3229-3241.
[18]乌日娜.内蒙古传统酸马奶中乳杆菌的分离鉴定及16S rDNA序列同源性分析[D].呼和浩特: 内蒙古农业大学, 2005: 13-14.
[19]热娜·米吉提, 古丽斯玛依·艾拜都拉, 苏豫梅, 等.新疆饲料乳酸菌的多样性与系统进化分析[J].安徽农业科学, 2012, 40(6): 3253-3257.
[20]WU R, WANG L P, MENGHE B, et al.Isolation and preliminary probiotic selection of lactobacilli from koumiss in Inner Mongolia[J].Journal of Basic Microbiology, 2009, 49(6): 1-9.
[21]乌日娜, 张和平, 孟和毕力格.酸马奶中乳杆菌Lb.casei Zhang和ZL12-1的16S rDNA基因序列及聚类分析[J].中国乳品工业, 2005,33(6): 4-9.
[22]李欣, 武俊瑞, 田甜, 等.大庆自然发酵酸菜中乳酸菌的分离鉴定及耐酸菌株初步筛选[J].食品科学, 2014, 35(1): 150-154.doi:10.7506/spkx1002-6630-201401029.
[23]XIE Jianping, JIANG Hongchen, LIU Xinxing, et al.16S rDNA based microbial diversity analysis of eleven acid mine drainages obtained from three Chinese copper mines[J].Journal of Central South University of Technology, 2011, 18(6): 1930-1939.
[24]陈晓前, 赵萍, 王家利, 等.贮藏苹果中酵母菌的分离初步鉴定及其抑制研究[J].中国酿造, 2009, 28(10): 51-62.
[25]吴斯日古冷.内蒙古西部地区酸面团中酵母菌和乳酸菌的分离鉴定及其生物多样性研究[D].呼和浩特: 内蒙古农业大学, 2011:13-14; 19-22.
[26]唐玲, 刘平, 黄瑛, 等.酵母的分子生物学鉴定[J].生物技术通报,2008(5): 84-87.
