微生物对偶氮染料脱色降解的研究进展
2015-01-07解井坤马宏瑞彭香玉
花 莉,解井坤,朱 超,马宏瑞,彭香玉
(陕西科技大学 资源与环境学院, 陕西 西安 710021)
微生物对偶氮染料脱色降解的研究进展
花 莉,解井坤,朱 超,马宏瑞,彭香玉
(陕西科技大学 资源与环境学院, 陕西 西安 710021)
近年来,随着印染与染料工业的发展,染料的数量和品种不断增多,由染料废水造成的污染呈增加的趋势,开发环境友好、高效、快速、低成本的染料废水处理方法是当前研究的热点。目前利用微生物处理偶氮染料废水的应用和研究居于首位,许多研究者致力于高效脱色偶氮染料微生物的筛选、分离和驯化。文章详细介绍了微生物对偶氮染料的脱色机制,并对目前研究所涉及的细菌、真菌、藻类以及混合菌群脱色偶氮染料的现状进行了分析,对今后的研究方向和重点内容进行了展望,为偶氮染料废水微生物处理技术的应用提供参考。
偶氮染料;脱色降解;细菌;真菌;混合菌群
目前,国内外常用的偶氮染料废水处理的方法可以分为物理法、化学法和生物法3类,其中物理法主要包括吸附法、沉淀法、过滤法、气浮法、膜分离法、超声波气振法和高能物理法等,化学法主要有化学混凝法、化学氧化法、光催化氧化法、电化学法和高温深度氧化法等,因物理及化学法存在着比较严重的二次污染,处理成本也比较高,所以很难在实际中得到广泛应用。生物法的本质是利用微生物酶系实现有机污染物的降解,在处理含偶氮染料废水方面具有经济高效、环境友好、产泥量少、末端产物无毒害和耗水量小的优势,是国内外处理印染废水的主要方法。提高微生物脱色降解效率的关键在于高活性和高适应性菌株的驯化和分离,本研究主要介绍了目前发现的能够高效脱色和降解偶氮染料的微生物菌株和混合菌群,以及其各自的作用机制和影响因素,以期为偶氮染料废水微生物处理技术的研究与应用提供参考。
1 微生物对偶氮染料的脱色机制
1.1 吸附脱色
微生物对偶氮染料的脱色机制主要有吸附脱色、酶降解以及两者的共同作用,目前用于偶氮染料吸附脱色的微生物主要有藻类、酵母菌、丝状真菌以及细菌等。微生物的吸附性能主要取决于菌体表面所含的蛋白质、脂质、糖类等大分子物质,以及分子上所含的多种功能基团,如氨基、羧基、羟基、磷酸盐及其他带电荷基团,这些基团通过极性作用、氢键、静电作用以及成键作用将偶氮染料吸附于细胞壁上[4]。适当的预处理能够优化微生物的吸附性能,如对微生物进行灭菌[5],添加酸、甲醛、NaOH、NaHCO3或CaCl2等物质[6],能够改变细胞的表面性能,增加吸附性能的结合位点。另外,以灭活微生物作为脱色偶氮染料的吸附剂具有明显优势,因为死细胞不需要营养物质,能够长期保存使用,而且可通过有机溶剂和表面活性剂再生。微生物的吸附效率受多种因素的影响,如pH、温度、离子强度、接触时间、吸附剂和染料的浓度、染料的结构以及微生物的种类等。
1.2 有氧条件下偶氮的降解
偶氮染料的微生物降解可分为好氧和厌氧2种机制。对于好氧条件下的偶氮还原,基本认为是由特异性酶催化完成的,这些酶是非黄素依赖的偶氮还原酶。最早纯化的偶氮还原酶是由羧基橙降解菌株K22产生的Orange Ⅰ偶氮还原酶和菌株KF46产生的Orange Ⅱ偶氮还原酶,这2个酶都是单体非黄素依赖还原酶[7]。目前在有氧条件下作用的偶氮还原酶有6种已被纯化,这些酶都有1个NAD(P)H结合位点,即都以NAD(P)H作为辅酶。一些好氧微生物在偶氮还原酶和辅助因子存在的条件下,能够还原偶氮化合物产生芳香胺类物质,然后芳香胺类物质在好氧条件下进一步被微生物降解。Yan等[8]将类球红细菌的偶氮还原酶基因在大肠杆菌中进行表达纯化,发现在纯化酶还原偶氮染料生成芳香胺的过程中有羟基化偶氮中间体存在,为偶氮染料还原必须通过一个非完全的还原过渡阶段提供了依据。另外,单加氧和双加氧酶能与还原偶氮染料形成的芳香胺类物质的芳香环结合,使芳香环开裂继而进一步使偶氮染料降解矿化。
许多具有偶氮染料还原能力的外酶,如木质素过氧化物酶(LiP)、锰过氧化物酶(MnP)以及漆酶也是从微生物中分离出来的[9]。LiP和MnP均为血红素蛋白酶,具有相似的降解反应机制:以H2O2为最初氧化底物,先将染料氧化形成高度活性的自由基中间体,继而发生链反应产生许多自由基促使底物氧化。此类降解反应在细胞外进行,从而使得菌体对降解底物的毒性有较高耐受能力并表现出广谱性。漆酶是一类含铜的多酚氧化酶,其利用O2作为电子受体,催化多酚类物质经4次单电子传递形成醌及自由基,不需要H2O2参与。这种自由基反应是高度非特异性的和无立体选择性的,因此在降解有机物时呈现广谱有效的特性。
按照国家标准[12-13],将降噪后的试验数据在数值分析软件中进行加权计算.主要为座椅处测点的x,y,z轴加速度值进行加权,其加权系数分别是1.4,1.4和1.0.得出座椅处的加权均方根值,如表2所示.
1.3 厌氧条件下偶氮染料的降解
厌氧条件下偶氮染料的降解是一种非特异性的还原过程,其中包括直接酶催化和依赖氧化还原中间体的偶氮还原过程。直接酶催化是指微生物体内存在非特异性的偶氮还原酶可以直接催化偶氮染料接受电子被还原。例如在利用肠道菌Clostridiumperfringens厌氧脱色偶氮染料的过程中,发现菌体培养基的上清液仍然有偶氮还原活性,可能是菌体分泌了一种胞外的偶氮还原酶,且其活性与黄素无关[10],但对于这种酶的存在目前仍没有确凿的证据,所以依赖辅助因子以及氧化还原中间体降解还原反应可能是厌氧条件下去除偶氮染料的主要机理。目前,主要的氧化还原中间体有醌、2-萘醌硫磺酸酯(2-anthraquinonesulfonate AQS)、2-羟基-1,4-萘醌(law sone)、FAD、NAD(P)H等,其可以接受来自偶氮还原酶的电子而被还原,然后又将电子转移给偶氮染料使偶氮键(-N=N-)断裂,产生芳香胺类物质,从而使偶氮染料脱色。在偶氮染料与氧化还原中间体之间进行的是自发非特异性的还原反应,主要决定于两者氧化还原电势的大小。氧化还原电势为-320~-50 mV的醌类氧化还原中间体通常具有厌氧还原偶氮染料的能力,这是由氧化还原辅助因子NAD(P)H等的氧化还原电势和偶氮复合物的氧化还原电势共同决定的[11]。
目前已经分离出了多种偶氮还原酶,其作用方式各有不同。其中一种酶是位于细胞质中的黄素氧化还原酶,其对偶氮染料的降解速率取决于细胞对染料的透性,而且对于不同的染料具有选择透过性,分子体积大的较难进入细胞,如酸性染料的摩尔体积小于直接染料,从而比较容易进入细胞中而被胞质黄素氧化还原酶所还原,但染料分子的透性并不仅仅取决于分子大小,同时还取决于分子的极性[12]。由于细胞膜含有大量的脂质及部分蛋白质和糖类,所以极性亲水的偶氮染料不易进入细胞,而且染料分子中的极性基团能和细胞膜上的某些官能团形成氢键从而更加不利于染料分子进入细胞[13]。另外一种酶是位于细胞膜上与呼吸链有关的泛醌类氧化还原酶,其不依赖于偶氮染料进入细胞内部,对偶氮染料的还原集中于细胞膜上。泛醌类偶氮还原酶接受来自呼吸链的电子并将其用于氧化还原中间体的还原,然后氧化还原中间体将电子转移给偶氮染料从而使其还原。由于不依赖染料分子的透过性,因此泛醌类氧化还原酶的还原速率大大提高,而且向反应体系中加入氧化还原中间体也将有利于偶氮染料的还原[14]。另外,在利用大肠杆菌厌氧降解偶氮染料的过程中,前人在细胞质中发现了一种NADH依赖的law sone还原酶,而且发现law sone还原酶的活性几乎完全存在于细胞质部分,向反应体系添加law sone能显著提高还原速率,可能是law sone将还原力从胞内带到了胞外。law sone作为一个合适的氧化还原中间体,不仅仅因为其具有氧化还原电势,而且因为其具有良好的脂溶性,可通过菌体细胞膜扩散,取代了偶氮染料的跨膜扩散,从而使还原速率增大[15]。
2 微生物对偶氮染料的脱色降解
目前,能够脱色降解偶氮染料的微生物呈现出广泛的多样性,其中包括细菌、真菌、放线菌、藻类等[16]。不同种类微生物生长环境和代谢机制的差异会影响偶氮染料的脱色效果,因此深入了解不同微生物的脱色降解机制,可以为微生物创造适宜的生存环境,以促进偶氮染料的脱色降解。
2.1 细菌对偶氮染料的脱色降解
细菌由于易于培养,在好氧和厌氧条件下有较快的生长速率,能产生多种氧化还原酶,并且适应性强,在高盐度和高温等环境中生长良好[17],因此在染料废水的生物处理中应用最多。对于细菌的脱色作用,最早的报道见于1977年,Horitsu[18]首次分离出可以降解偶氮染料的细菌BacillossubrilisIF03002。Wong等[19]于1996年分离得到1株可以降解甲基红的细菌KlebsiellapseumonineRS-12,研究发现,该菌无论在静止条件还是在摇动条件下均对甲基红有非常好的降解效果,加入葡萄糖或(NH4)2SO4可以促进降解,但加入乙醇无促进效果。Nachiyar等[20]研究发现,以葡萄糖为碳源时Pseudomonasaeruginosa在有氧条件下对制革染料有很好的脱色效果,同时还对其他多种染料都有脱色效果。国内对降解偶氮染料的菌株也进行了大量研究。徐文东等[21]从处理毛纺厂染料废水的活性污泥中分离得到1株高效降解菌,经鉴定该菌为嗜热鞘氨醇杆菌(Sphingobacteriumthalpopilum),5 d内其对派拉丁蓝RRN的脱色率为34%。许玫英等[22]分离到1株具有染料降解能力的希瓦氏菌新菌种,命名为脱色希瓦氏菌ShewanelladecolorationisS12T,将该菌株在偶氮染料质量浓度为50 mg/L的培养基中培养4 h后,染料脱色率达96%,其对偶氮染料最高脱色质量浓度可以达到2 000 mg/L,此菌另外一个重要的特点就是具有很强的Fe(Ⅲ)还原活性。Han等[23]从台湾海峡分离出2种能够脱色偶氮染料废水的新细菌菌种DX2b和SH7b,其PCR扩增的16S rRNA序列分析表明,所得菌株的核酸序列与Rahnellaaquatilis、Axinetobacterguillouiae、Microvirgulaaerodenitrificans有99%的相似性。
细菌对偶氮染料的还原主要是在偶氮还原酶作用下完成的,在厌氧条件下偶氮还原反应是在非特异还原酶作用下的电子传递过程,偶氮染料作为最终电子受体被氧化还原中间体传来的电子还原,但好氧条件下氧会抑制细菌对偶氮染料的还原。而Olukanni等[24]研究发现,Micrococcussp.在厌氧条件下脱色活性染料需24 h,而在好氧条件下只需6 h。Oturkar等[25]利用Bacilluslentus对活性红120进行脱色,发现在自然静置和供氧条件下的脱色率分别为98%和70%,而在无氧条件下不能脱色,说明B.lentus的脱色过程需要一定量的氧参与。一些细菌脱色偶氮染料的相关研究结果如表1所示。
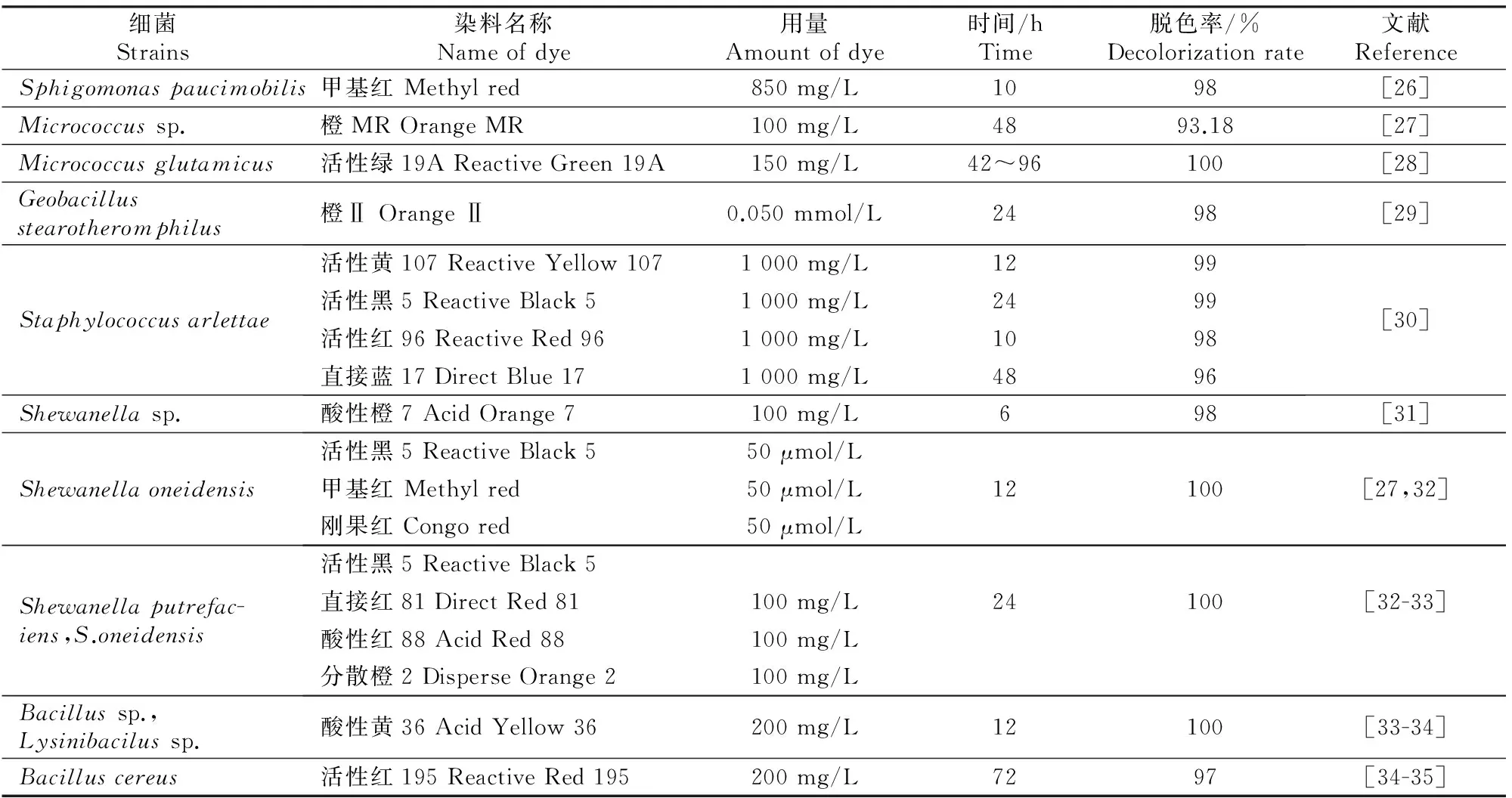
表1 细菌对不同偶氮染料的脱色率Table 1 Decolorization rates of different azo dyes by bacteria
2.2 真菌对偶氮染料的脱色降解
真菌对染料的脱色主要依靠吸附和酶降解,灭活的Cunninghamellaelegans、冻干的Trametesversicolor对染料的吸附能力均优于活性炭,在pH 2~3时以上真菌的吸附性能较高,可能与染料分子表面的电荷与细胞表面电荷的吸引力有关[36]。真菌的吸附能力随温度的升高而增加,这是因为温度的升高增大了细胞的表面活性和染料的动能[37],然而温度过高脱色率会有所下降,可能是因为高温会导致真菌吸附表面失活或者是活性部位被破坏。
在真菌中,酵母菌由于生长快、对染料和重金属(如PbⅡ和CdⅡ)的累积量较高、比丝状真菌脱色速率快、适应不良环境能力较强而广泛用于染料废水的生物处理。与微藻类相似,酵母菌的脱色机制包括吸附、酶降解以及两者的共同作用。酵母菌在较低pH下有较高的吸附能力。例如Candidaalbicans在pH 2.5时对直接紫51吸附量最大,Candidatropicalis对直接紫3吸附量最大时的pH为4.0。同种酵母菌对不同染料会表现出不同的吸附性能,不同酵母菌对同种染料的吸附能力也不同。如C.tropicalis对雷马素蓝和活性红的吸附率分别为94%和44%,然而Trichosporonakiyoshidainum对活性蓝和活性红的吸附率分别为63%和90%[38]。偶氮染料的降解脱色与酵母菌生长和初级代谢密切相关,偶氮染料能够诱导酵母菌产生多种氧化还原酶,如MnP、Tyr 以及NADH-DCIP,这些酶对偶氮染料都有不同程度的降解能力[39]。酵母菌对偶氮染料的酶降解机制与丝状真菌类似,其在厌氧和好氧条件下都有降解偶氮染料的特性。有研究表明,利用GalactomycesGeotrichum处理雷马素红、金黄HER、红玉GFL、大红RR、甲基红、棕3REL以及亮蓝组成的混合染料废水,其在好氧条件下的脱色率及COD、TOC去除率均明显优于兼氧或厌氧环境[40]。而Candidarugopeeliculosa则在静置条件下对活性蓝有较好的脱色效果[41]。
真菌酶解偶氮染料的过程中受环境状况和营养水平的影响较大,尤其是碳源、氮源、时间、pH、温度、添加剂、盐度以及氧含量[42]。真菌在好氧条件下的脱色能力优于厌氧条件,而且搅拌能促进氧的传递亦能提高脱色效果。如用Trametesvesicorsanguineus脱色蓝49、橙12、橙13、黑5和雷马素艳蓝R[43],Aspergillusniger脱色刚果红[44],Aspergillusflavus、Alternatiasp.、Penicilliumsp.脱色酸性红151和橙Ⅱ[45],当增加氧含量或提高搅拌速度时都能使脱色效果有明显提升。Grinhut等[46]在研究Trametessp.脱色降解橙Ⅱ和艳蓝R250的过程中发现,当C∶N为53∶1时能促进脱色,而当C∶N为10∶1时脱色受阻。Phanerochaetechrysosporium[47]和Bjerkanderasp.[48]在氮限制培养条件下能取得最大脱色率,说明碳源和氮源对脱色有很大影响。向培养基中加入添加剂亦能提高脱色效果,如琥珀酸能促进P.chrysosporium产生MnP[49];藜芦醇能提升LiP的产量,从而促进真菌对活性黑5和吐温80的脱色[50];Mn2+能提高MnP的活性,从而提高脱色率[51]。由于成本低且具有将染料完全降解的可能性,关于真菌脱色偶氮染料的研究也取得了丰硕的成果,具体如表2所示。
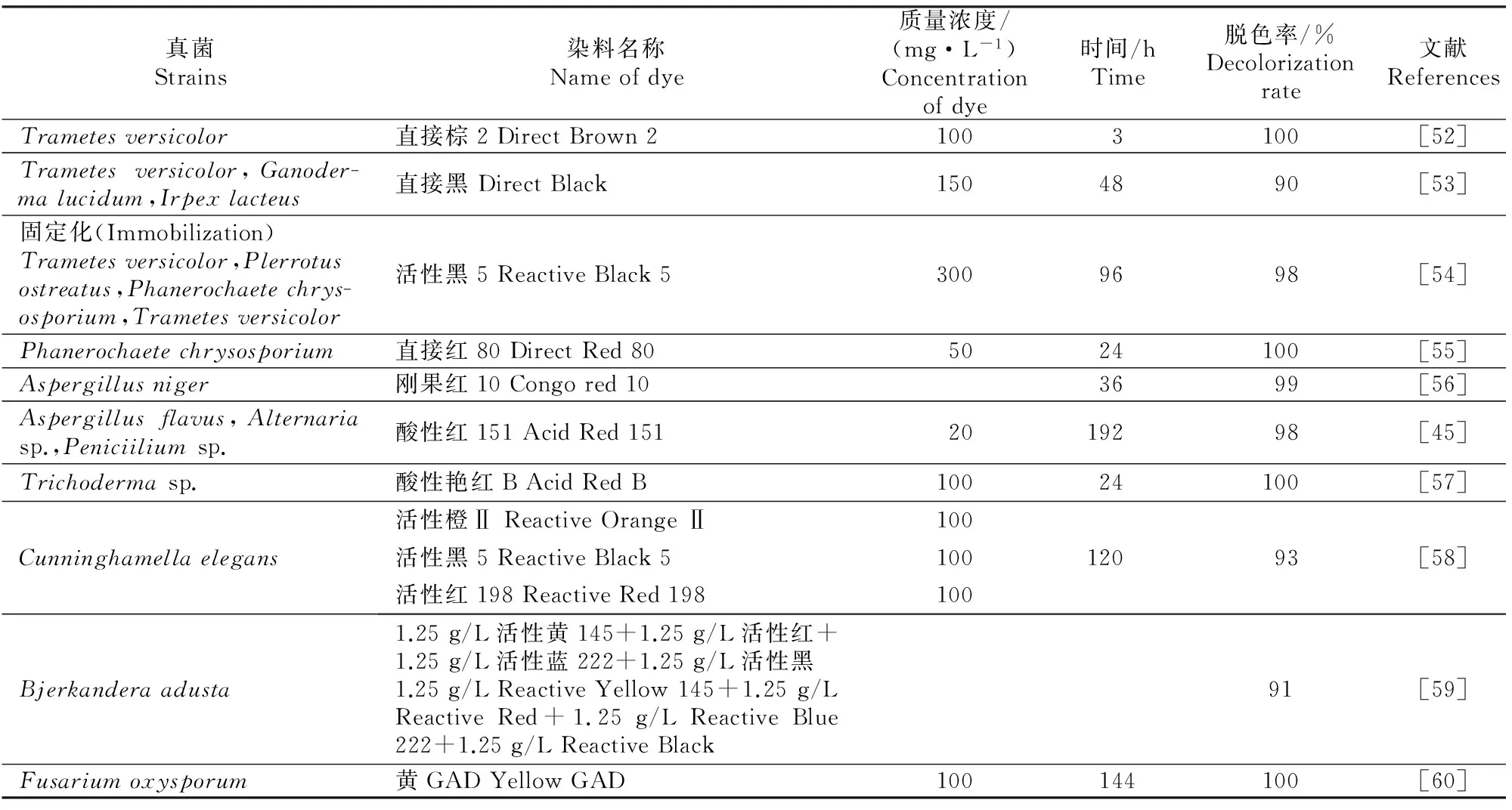
表2 真菌对偶氮染料的脱色率Table 2 Decolorization rates of different azo dyes by fungi
2.3 藻类对偶氮染料的脱色
许多偶氮染料对水体中的生物表现出高毒性,但对藻类的生长影响却不明显。因此,微藻类是染料废水生物处理的一个很好的选择。与细菌和真菌不同,藻类能够从大气和太阳光中获得碳源和能源,因此大量培养藻类的费用很低。藻类脱色机制包括酶降解、吸附以及两者的共同作用。同细菌相似,藻类也能通过诱导偶氮还原酶使偶氮染料的偶氮键断裂产生芳香胺类物质,从而实现对偶氮染料的脱色,此脱色过程也包含氧化酶的作用[61]。
藻类的吸附性能受染料的结构、藻类种类、环境pH影响较大。在较低的pH值下藻类对偶氮染料有较高的吸附量,可能是由于带负电荷的染料阴离子和藻类带正电的表面活性细胞之间的静电吸引所致。如在pH=2时,利用Scenedesmusquadricauda[62]、Chlorellavulgaris[63]、Spirogyrasp.[63]处理偶氮染料废水可以取得较好的脱色效果。藻类对偶氮染料的酶解一部分用于自身的同化,一部分用于偶氮染料的矿化。许多藻类能降解偶氮染料产生芳香胺类物质,进而将芳香胺类物质降解为简单有机物或矿化为CO2[64]。藻类一般先将偶氮染料吸附于表面然后通过诱导产生的偶氮还原酶对其进行还原降解,所以固定化微藻是一个具有一定应用价值的脱色方法。目前关于微藻类脱色偶氮染料的相关研究如表3所示。
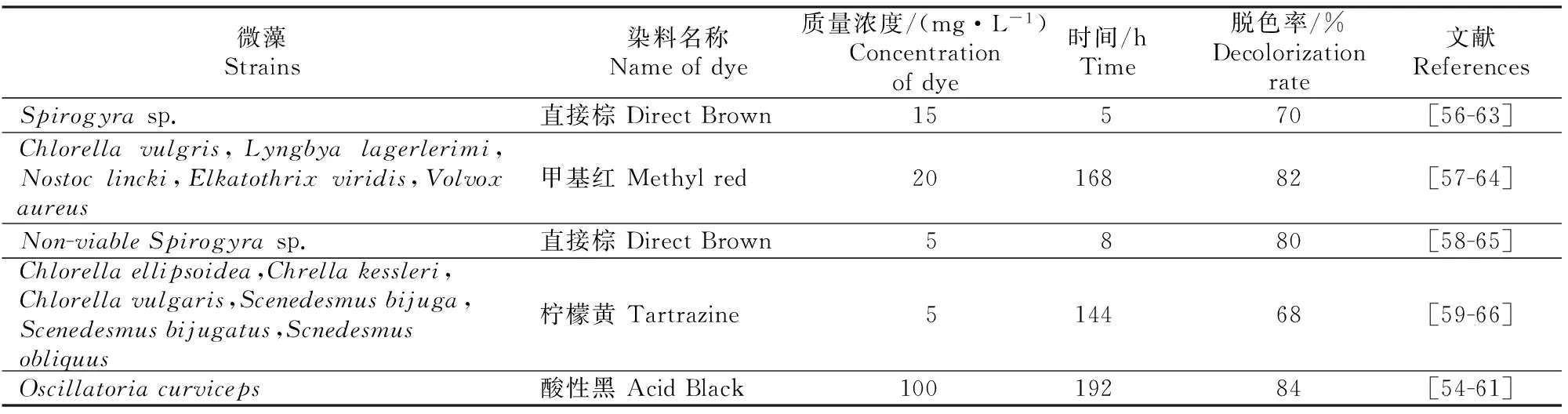
表3 微藻类对不同偶氮染料的脱色率Table 3 Decolorization rates of different azo dyes by algae
2.4 混合菌群对偶氮染料的脱色降解
对于纯培养的单一菌株而言,一般只是将发色基团打开,对中间产物如苯胺类致癌物质很难进一步降解。微生物通常只对特定的染料有降解性能,很难适应成分复杂的实际染料废水,而且染料的完全降解需要多种酶的催化作用,这就确定了利用微生物混合菌群处理染料废水的优越性。混合菌群中的不同菌株会作用于染料分子的不同部分和基团,有些菌株与其他菌株可以构成共代谢机制,并能对其他菌株的代谢产物有降解作用。
Jadhav等[67]的研究表明,假单胞菌SUK1(Pseudomonassp.SUK1)能够脱色染料,假单胞菌LBC2(Pseudomonassp.LBC2)和LBC3(Pseudomonassp.LBC3)则不能,但当3种菌株混合时却能得到良好的染料脱色效果。Enterobactercloacae和Enterococcuscasseliflavus培养15 min对橙Ⅱ的脱色率分别为10%和23%,但混合时能将橙Ⅱ完全脱色[68]。Yang 等[69]考察了真菌和细菌组成的混合菌群对偶氮染料的降解能力,其中真菌和细菌的比例为6.8∶1~51.8∶1时能取得良好脱色效果,36S rRNA序列分析表明,其中70%的真菌为念母菌属的酵母菌。夏枯草(Prunellavulgaris)和Micrococcusglutamicus对猩红R的完全脱色时间分别为14和20 h,而两者混合脱色时仅需3 h[70]。Phugare SS混合菌群包括普罗维登藻(Providenciasp.)和绿脓杆菌(Pseudomonasaeruginosa),二者培养1 h能将红HE3B完全脱色,并能使97%的雷马素黑脱色,而其各自单独使用时的脱色时间为5 和48 h[71]。在培养72 h时,假单胞菌(Pseudomonassp.SUK1)和赭曲霉(Aspergillusochraceus)对活性藏青HE2R的脱色率分别为78%和61%,而两者混合处理时对活性藏青HE2R的脱色率可达92%[72]。鉴于混合菌群在偶氮脱色处理中的优越性,现已开展了许多相关研究,具体如表4所示。

表4 微生物群落对不同偶氮染料的脱色率Table 4 Decolorization rate of different azo dyes by consortia
3 微生物脱色的影响因素
3.1 碳源和氮源
在微生物脱色偶氮染料过程中,以染料作为碳源和氮源往往不能满足微生物生长的需要,有些染料甚至对微生物会产生毒副作用,因此需要外源碳源和氮源通过共代谢机制实现染料的脱色或降解[83]。各种外源碳源在微生物的代谢过程中被氧化,产生的电子通过电子传递链传递给染料,使其偶氮键断裂,从而促进染料的脱色。但也有研究表明,外源碳源有时不能很好地促进染料脱色,首先由于外源碳源的易降解性和微生物对碳源利用的偏好性导致其对染料的脱色率降低;其次在微生物代谢过程中产生的电子可能会传递给外源碳源或其中间代谢产物,从而使染料的降解受到限制[71]。然而外源氮源如蛋白胨、尿素等能够促进NADH的再生,加速偶氮染料的还原[84]。近年来,由于存在来源广泛、经济合理等优点,木质素作为外源物质在促进微生物脱色偶氮染料的应用中倍受关注。例如木质素能诱导Comamonassp. UVS产生木质素酶类从而促进直接蓝GLL的脱色[85]。Saratale等[28]利用MicrococcusglutamicusNCIM-2168处理活性绿19A以及用细菌菌落GR处理猩红R时,分别以甘蔗渣粉末、稻草、稻壳为外源物质考察其对偶氮染料的脱色效果,结果表明以稻壳为外源物质时其对脱色表现出较好的促进效果。
3.2 温度和pH
温度对于生物化学反应和酶活性来说非常重要,其是影响微生物脱色偶氮染料的一个极为关键的因素。偶氮染料的脱色率在低温度范围内随着温度的升高而升高,在最适温度达到最大,之后随着温度的继续升高脱色率下降,这可能是由于高温下细胞活力的减弱以及酶活的降低所致。然而有些微生物制剂则具有一定的耐高温能力,当温度上升到60 ℃ 时仍表现出一定脱色能力。另外,更高温度能导致菌体细胞破裂,使细胞表面积增大,反而有助于偶氮染料的吸附脱色[86]。
pH值能影响微生物细胞膜对染料分子的透过性,因此pH也是影响微生物脱色的一个重要因素。实验室条件下微生物脱色的最适pH为6~10。但实际染料废水由于其所含成分极为复杂,pH大都呈酸性或碱性而不利于微生物的生存,因此分离筛选极端适酸和极端适碱的脱色微生物具有重要的实际意义。如分离株Citrobactersp.CK3能够在强酸(pH=4)和强碱(pH=12)条件下使活性红190有效脱色[87]。
3.3 染料分子结构
芳香环上相对于偶氮键的取代基分为两大类,即斥电子基和吸电子基,其中斥电子基主要有羟基、氨基等,而吸电子基主要包括磺酸基、羧基、硝基、羰基、卤素原子等。偶氮键附近的电子云密度越高,偶氮键就越牢固,也就越难断裂;相反,电子云密度越低,染料就越易被降解。目前,有关芳香环上取代基相对于偶氮键的取代位置对染料生物脱色影响的研究还较少,仅有的研究中以磺酸基和羟基取代位置对脱色影响作用的研究较多。较之对位取代而言,-SO3H 和-COOH的邻位取代会在偶氮键附近产生更大的空间位阻,使得偶氮键较难断裂;而-SO3H 的邻位取代较其间位取代更易使偶氮键断裂,这是由于间位取代产生的诱导效应较弱,取代基吸附电子的能力降低,-SO3H取代位置的影响作用大小表现为对位>邻位>间位[88]。另外,如果苯环上相对-OH的邻位上有斥电子基取代,则可提高染料的生物降解率,若是换成具有负诱导效应的吸电子基(如-F、-Cl、-NO3等),则使染料难以被生物降解。
3.4 电子供体
自然界存在各种各样的电子供体和电子受体,研究其在微生物还原脱色偶氮染料过程中的作用机制显得尤为重要。在Bacillussp.脱色偶氮染料 C.I.Reactive Orange 16的过程中,添加醋酸钠、甲酸钠、琥珀酸钠、柠檬酸钠以及丙酮酸钠等电子供体都能增强脱色效果[89]。Hong等[90]在用ShewanelladecolorationisS12 处理多种偶氮染料的过程中,添加电子供体也取得了类似的增强脱色效果。在微生物的厌氧偶氮还原过程中,首先是电子供体被氧化,产生的电子通过一个复杂的电子传递链传给电子受体(偶氮染料),进而完成对偶氮染料的降解。在这个过程中,脱氢酶、细胞色素和甲基萘醌都是极为重要的中间体,其能加速电子的传递。而有些电子供体的存在反而会阻碍偶氮的还原过程,这可能是由于电子供体之间的竞争抑制了电子传递过程。由于不同电子供体在偶氮还原过程中的诱导还原机制以及酶促能力不同,因此针对不同染料废水应通过深入研究寻找其最适的电子供体。
4 展 望
伴随着人们对时尚的追求,越来越多的染料被合成,人们在创造出绚丽世界的同时,也给环境造成了巨大污染。目前广为应用的生物处理方法已不能满足染料废水的处理要求,急需对此展开更为深入、广泛的研究,以提高染料废水的处理效率。许多研究者已开始将生物法与其他方法联用,如生物法和高级氧化技术联用处理高盐度染料废水,现已取得了良好的效果[91]。同时筛选、分离高效脱色降解偶氮染料菌株也尤为重要,近年来,许多人更是将研究重点放在混合菌株对偶氮染料废水的处理上,并证实其处理效果优于单一菌种。另外,微生物固定化技术也是实现高效低耗、简化运行管理的有效措施,其将微生物通过一定的技术手段镶嵌或包埋于一定机械强度的载体内,使微生物保持在反应器中,以提高染料废水的脱色降解效果。构建基因工程菌治理环境污染是环境微生物工程高新技术中的前沿课题,其能定向有效地利用环境微生物细胞中的降解污染物基因去执行净化环境的功能。但是具有降解污染物基因的土著微生物菌株有时难以适应处理环境,而且繁殖速度较慢,清除污染物的速度和效果达不到治理环境的要求。因此,有必要将降解偶氮染料的基因转入繁殖能力强、适应性能佳的受体菌中,构建治理染料废水污染的高效菌株。最近,微生物燃料电池(Microbial fuel cells,MFCs)作为一种发展迅速的新型能源技术开始应用于偶氮染料废水的处理,其通过微生物脱色降解偶氮染料分子过程中的电子传递机制将有机物中的化学能转化为电能,是极具应用前景的污水资源化处理技术,而该技术的核心便是微生物工程菌株的研究和开发[92]。
[1] Babu B R,Parande A K,Raghu S,et al.Cotton textile processing:Waste generation and effluent treatment [J].J Cotton Sci,2007,11:141-153.
[2] Zonllinger H.Colour chemistry-synthesis,properties and applications of organicdyes and pigments [M].5th ed.Weinheim,Germany:VCH Publishers,1991:187.
[3] Estevam V S,Jorge M V.Liquid-phase hydrogenation of benzene to cyclohexene catalyzed by Ru/SiO2in the presence of water-organic mixtures [J].Catalysis Communications,2003,4:91-96.
[4] Charumathi D,Das N.Packed bed column studies for the removal of synthetic dyes from textile wastewater using immobilised deadC.tropicalis[J].Desalination,2012,285:22-33.
[5] Lim S L,Chu W L,Phang S M.Use ofChlorrllavulgarisfor bioremediation of textile wastewater [J].Bioresour Technol,2010,101:7314-7322.
[6] Vijayaraghavan K,Yun Y S.Utilization of fermentation waste (Corynebacteriumglutamicum) for biosorption of reactive black 5 from aqueous solution [J].J Hazard Mater,2007,141:45-52.
[7] Zimmermann T,Gasser F,Kulla H G,et al.Comparison of two azoreductases acquired during adaption to grwth on azo dyes [J].Arch Microbiol,1984,138:37-43.
[8] Yan B,Zhou J T,Wang J,et al.Expression and characteristics of the gene encoding azoreductase fromRhodobatersphaeroidesAS1.1737 [J].FEMS Microbiol Lett,2004,236:129-136.
[9] Liers C,Bobeth C,Pecyna M,et al.Dyp-like peroxidases of the jelly fungus Auricularia auricula-judae oxidize nonphenolic lignin model compounds and high-redox potential dyes [J].Appl Microbiol Biotechnol,2010,85:1869-1879.
[10] Rafii F,Franklin W,Cerniglia C E.Azoreductase activity of anaerobic bacteria isolated from human intestinal microflora [J].Appl Environ Microbiol,1990,56:2146-2151.
[11] Rau J,Knackmuss H J,Stolz A.Effects of differernt quinoideredox mediators on the anaerobic reduction of azo dyes by bacteria [J].Environ Sci Technol,2002,36:1497-1504.
[12] 曹孟德,王孔星.染料微生物降解的研究概况 [J].环境科学与技术,1989(4):5-10.
Cao M D,Wang K X.Research survey of biodegradation of dye [J].Environ Sci Technol,1989(4):5-10.(in Chinese)
[13] 刘金齐,刘厚田.偶氮染料微生物降解的研究进展 [J].环境科学研究,1988,1(6):40-46.
Liu J Q,Liu H T.The development of research on azo-dye degradation by microorgnisms [J].Research of Environ Sci,1988,1(6):40-46.(in Chinese)
[14] Russ R,Rau J,Stolz A.The function of cytoplasmatic flavin reductases in the bacterial reduction of azo dyes [J].Appl Environ Microbiol,2000,66:1429-1434.
[15] 洪义国,许玫英,郭 俊,等.细菌偶氮还原研究进展 [J].应用与环境生物学报,2005,11(5):642-647.
Hong Y G,Xu M Y,Guo J,et al.Advance in bacterial azoreduction research [J].Chi J Appl Environ Biol,2005,11(5):642-647.(in Chinese)
[16] Anjaneyulu Y,Chary N S,Raj D S S.Decolourization of industrial effluents available methods and emerging technologies:A review [J].Rev Environ Sci Biotechnol,2005,4:245-273.
[17] 刘志培,杨惠芳.偶氮染料的微生物降解 [J].环境污染与防治,1987(2):2-5.
Liu Z P,Yang H F.Microbial degradation of azo dyes [J].Environmental Pollution and Control,1987(2):2-5.(in Chinese)
[18] Horitsu H.Degradation of p2 aminozaobenzene byBacillussu-btilis[J].Eur J Appl Microbial,1977,14:217-224.
[19] Wong P K,Yuen P Y.Decolorization and biodegration of met-hyl red byKlebsiellapneumoniaRS-13 [J].Wat Res,1996,30(7):1736-1744.
[20] Nachiyar C V,Rajkumar G S.Degradation of tannery and textile dye,navian fast blue S5R byPseudomonasaerugenosa[J].Enzyme Microb Technol,2005,36:503-508.
[21] 徐文东,文湘华,付莉燕.偶氮染料派拉丁蓝RRN脱色细菌的选育与研究 [J].环境科学学报,2001(S1):127-132.
Xu W D,Wen X H,Fu L Y.Isolation and characterization of bacteria for decoloring of zao dye acid complec blue RRN [J].Journal of Environmental Sciences,2001(S1):127-132.(in Chinese)
[22] 许玫英,钟小燕,曹 渭,等.脱色希瓦氏菌(Shewanelladecolorationis)S12T的脱色特性 [J].微生物学通报,2005,32(1):9-13.
Xu M Y,Zhong X Y,Cao W,et al.The decolorizing characterization ofShewanelladecolorationisS12T[J].Microbiology,2005,32(1):9-13.(in Chinese)
[23] Han J L,Wang Y N.Exploring new strains of dye-decolorizing bacteria [J].Journal of Bioscience and Bioengineering,2012,4:508-514.
[24] Olukanni O D,Osuntoki A A,Gbenle G O.Decolorization of azo dyes by a strain of micrococcus isolated from a refuse dump soil [J].Biotechnol,2009,8(4):442-448.
[25] Oturkar C C,Nemade H N,Mulik P K,et al.Mechanistic investigation of decolorization and degradation of reactive red 120 byBacilluslentusB1377 [J].Bioresour Technol,2011,102(28):83-89.
[26] Ayed L,Mahdhi A,Cheref A,et al.Decolorization and degradation of azo dye Methyl Red by an isolatedSphingomonaspaucimobilis:Biotoxicity and metabolites characterization [J].Desalination,2011,274(1):272-277.
[27] Rajee O, Patterson J. Decolorization of azo dye (Orange MR) by an autochthonous bacteriumMicrococcussp.DBS2 [J].Indian J Microbiol,2011,51(2):159-163.
[28] Saratale R G,Saratale G D,Chang J S,et al.Ecofriendly decolorization and degradation of reactive green 19A usingMicrococcusglutamicusNCIM-2168 [J].Bioresour Technol,2009,110:3897-3905.
[29] Evangelista-Barreto N S,Albuquerque C D,Vieira R H S F,et al.Cometabolic decolorization of the reactive azo dye Orange Ⅱ byGeobacillusstearothermophilusUCP986 [J].Text Res J,2009,79:1266-1273.
[30] Elisangela F,Rea Z,Fabio D G,et al.Biogegradation of textile azo dyes by a facultativeStaphylococcusarlettaestrain VN-11 using a sequential microaerophilic/aerobic process [J].Int Biodeter Biodegr,2009,63:280-288.
[31] Wang J,Liu G F,Lu H,et al.Biodegradation of Acid Orange 7 and its auto-oxidative decolorization product in membrane-aerated biofilmreaction [J].Int Biodeter Biodegr,2012,67:73-77.
[32] Wu J,Kim K S,Sung N C,et al.Isolation and characterization ofShewanellaoneidensisWL-7 capable of decolorizing azo dye Reactive Black 5 [J].J Gen Apll Microbiol,2009,55:51-55.
[33] Khalid A,Arshad M,Crowley D.Decolorization of azo dyes byShewanellasp.under saline conditions [J].Appl Microbiol Biotechnol,2008,79:1053-1059.
[34] Anjaneya O,Souche S Y,Santoshkumar M,et al.Decolorization of sulfonated azo dye Metanil Yellow by newly isolated bacterial strains:Bacillussp.strain AK1 andLysinibacillussp.Strain AK2 [J].J Hazard Mater,2011,190(35):1-8.
[35] Modi H A,Rajput G,Ambasana C.Decolorization of water soluble azo dyes by bacterial cultures,isolated from dye house effluent [J].Bioresour Technol,2010,101:6580-6583.
[36] Balamurugan B,Thirumarimurugan M,Kannadasan T.Anaerobic degradation of textile dye bath effluent usingHalomonassp. [J].Bioresour Technol,2011,102(10):6365-6369.
[37] Erden E,Kaymaz Y,Pazarlioglu N K.Biosorption kinetics of a direct azo dye Sirius Blue K-CFN byTrametesversicolor[J].Electron J Biotechnol,2011,14:1-10.
[38] Pajot H F,Figrueroa L I C,Farina J I.Dye-decolorizing activity in isolated yeasts from the ecoregion of Las Yungas(Tucuman Argntina) [J].Enzyme Microbiol Technol,2007,40:1503-1511.
[39] Martorell M M,Pajot H F,Figueroa L I C.Dye-decolorizing yeasts isolated from Las Yungas rainforest dye assimilation and removal used as selection criteria [J].Int Biodeter Biodegr,2012,66:25-32.
[40] Waghmode T R,Kurade M B,Govindwar S P.Time dependent degradation of mixture of stucturally different azo and non azo dyes by usingGalactomycesgeotrichumMTCC 1360 [J].Int Biodeter Biodegr,2011,65(4):79-86.
[41] Viror V,Corso C R.Decolorization of textile dye byCandidaalbicansisolated from industrial effluents [J].J Ind Microbiol Biotechnol,2008,35(11):1353-1357.
[42] Kaushik P,Malik A.Fungal dye decolourization:Recent advances and future potential review article [J].Environ Int,2009,35(1):27-41.
[43] Pilatin S,Kunduhoglu B.Decolorization of textile dyes by ne-wly isolatedTrametesversicolorstrain [J].Life Sci Biotechnol,2011,1(1):25-35.
[44] Ali N,Hameed A,Siddiqui M F,et al.Application ofAspergillusnigerSA1 for the enhanced bioremoval of azo dyes in simulated textile effluent [J].Afr J Biotechnol,2009,8:38-45.
[45] Ali N,Hameed A,Ahmed S.Role of brown-rot fungi in the bioremoval of azo dyes under different donditions [J].Braz J Microbiol,2010,41:907-915.
[46] Grinhut T,Salame T M,Chen Y,et al.Involvement of ligninolytic enzymes and Fenton-like reaction in humic acid degradation byTrametessp [J].Appl Microbiol Biotechnol,2011,91(11):31-40.
[47] Karimi A,Vahabzadeh F,Bonakdarpour B.Use ofPhanerochaetechrysosporiumimmobilized on kissiris for synthetic dye decolourization:Involvement of manganese peroxidase [J].World J Microb Biot,2006,22:1251-1257.
[48] Axelsson J,Nilsson U,Terrazas E,et al.Decolorization of the textile dyes Reactive Red 2 and Reactive Blue 4 usingBjerkanderasp.strain BOL13 in a continuous rotating biological contactor reactor [J].Enzyme Microbiol Technol,2006,39:32-37.
[49] Ghasemi F,Tabandeh F,Bambai B,et al.Decolorization of different azo dyes byPhanerochaetechrysosporiumRP78 under optimal condition [J].Int J Environ Sci Technol,2010,7:457-464.
[50] Enayatizamir N,Tabandeh F,Rodriiguez C S,et al.Biodegradation pathway and detoxification of the diazo dye reactive black 5 byPhanetochaetechrysosporium[J].Bioresour Technol,2011,102:10359-10362.
[51] Urek R O,Pazarlioglu N K.Production and stimulation of ma-nganese peroxidase by immobilizedPhanerocaetechrysosporium[J].Process Biochem,2005,40:83-87.
[52] Cano M,Solis M,Solis A,et al.Decoloration de CD 2(Cafe director 2) or enzimas intracelulares y extracelulares deTrametesversiolor[J].Interciencia,2012,37:294-298.
[53] Baccar R,Blanquez P,Bouzid J,et al.Decolorization of a tannery dye:From fungal screening to bioreactor application [J].Biochem Eng J,2011,56(3):184-189.
[54] Fernandez J A,Henao L M,Pedroza-Rodriguez A M,et al.Im-mobilising lignilolytic fungus for removing Reactive Black 5 dye [J].Rev Colomb Biotecnol,2009,11(1):59-72.
[55] Singh S,Pakshirajan K,Daverey A.Enhanced decolorization of Direct Red-80 dye by the white rot fungusPhanerochaetechrysosporiumemploying sequential design of experiments [J].Biodegradation,2010,21:501-511.
[56] Karthikeyan K,Nanthakumar K,Shanthi K,et al.Response su-rface methodology for optimization of culture conditions for dye decolorization by a fungusAspergillusniger HM11 isolated from dye affected soil [J].Iran J Microbiol,2010,2:213-222.
[57] Xin B,Xia Y,Zhang Y,et al.A feasible method for growing fungal pellets in a column reactor inoculated with mycelium fragments and their application for dye bioaccumulation from aqueous solution [J].Bioresour Technol,2012,105:100-105.
[58] Ambrosio S T,Vilar Junior J C,Da Silva C A A,et al.A biosorption isotherm model for the removal of reactive azo dyes by inactivated mycelia ofCumninghamellaelegansUCP542 [J].Molecules,2012,17:452-462.
[59] Anastasi A,Parato B,Spina F,et al.Decolorisation and detoxification in the fungal treatment of textile wastewaters from dyeing processes [J].New Biotechnol,2011,29:38-45.
[60] Porri A,Baroncelli R,Guglielminetti L,et al.Fusariumoxysporumdegradation and detoxification of a new textileglycoconjugate azo dye (GAD) [J].Fungal Biol,2011,115:30-37.
[61] Priya B,Uma L,Ahamed A K,et al.Ability to use the diazo dye C.I.Acid Black 1 as a nitrogen source by the marine cyanobacteriumOscillatoriacurvicepsBDU92191 [J].Bioresour Technol,2011,102:7218-7223.
[62] Ergene A,Ada K,Tan S,et al.Removal of Remazol Brilliant Blue R dye from aqueous solutions by adsorption onto immobilizedScenedesmusquadricauda:Equilibrium and kinetic modeling studies [J].Desalination,2009,249:1308-1314.
[63] Sivarajasekar N,Baskar R,Balakrishnan V.Biosorption of an azo dye from aqueous solutions ontoSpirogyra[J].J Univ Chem Technol Metall,2009,44:157-164.
[64] Ei-Sheekh M M,Gharieb M M,Abou-EI-Souod G W.Biodegradation of dyes by some green algae and cyanobacteria [J].Int Biodeter Biodegr,2009,63:699-704.
[65] Mohan S V,Ramanaiah S V,Sarma P N.Biosorption of direct azo dye from aqueous phase ontoSpiogyrasp.I02:Evaluation of kinetics and mechanistic aspects [J].Biochem Eng J,2008,3861-3869.
[66] Omar H H.Algal decolorization and degradation of monoazo and diazo dyes [J].Pak J Biol Sci,2008,11:1310-1316.
[67] Jadhav J P,Kalyani D C,Telke A A,et al.Evaluation of the efficacy of a bacterial consortium for the removal of color,reduction of heavy metals and toxicity from textile dye effluent [J].Bioresour Technol,2010,101:165-173.
[68] Chan G F,Rashid N A A,Koay L L,et al.Identification and optimization of novel NAR-1 bacterial consortium for the biodegradation of Orange Ⅱ [J].Insight Biotechnol,2011,1:7-16.
[69] Yang Q X,Li C M,Li H J,et al.Degradation of synthetic reactive azo dyes and treatment of textile wastewater by a fungi consortium reactor [J].Biochemical Engineering Journal,2006,43:225-230.
[70] Saratale R G,Saratle G D,Kalyani D C,et al.Enhanced decolorization and biodegradation of textile azo dye Scarlet R by using developed microbial consortium-GR [J].Bioresour Technol,2009,100:2493-2500.
[71] Phugare S S,Kalyani D C,Patil A V,et al.Textile dye degradation by bacterial consortium and subsequent toxicological analysis of dye and dye metabolites using cytotoxicity,genotoxicity and oxidative stress studies [J].J Hazard Mater,2011,186:713-723.
[72] Kadam A A,Telke A A,Jagtap S S,et al.Decolorization of adsorbed textile dyes by developed consortium ofPseudomonassp.SUK1 andPspergillusochraceusNCIM-1146 under solid state fermentation [J].J Hazard Mater,2011,189:486-494.
[73] Ruiz-Arias A,Juarez-Ramirez C,Cobos-Vasconcelos D,et al.Aerobic biodegradation of a sulfonated phenylazonaphthol dye by a bacterial community immobilized in a multistage packed-bed BAC reactor [J].Appl Biochem Biotechnol,2010,162(6):1689-1707.
[74] Kumar K,Dastidara M G,Sreekrishnan T R.Effect of process parameters on aerobic decolourization of reactive azo dye using mixed culture [J].World Acad Sci Eng Technol,2009,58:962-965.
[75] Ayed L,Achour S,Kehelifi E,et al.Use of active consortia of constructed ternary bacterial cultures via mixture design for Congo Red decolorization enhancement [J].Chem Eng J,2010,162:495-502.
[76] Ayed L,Khelifi E,Jannet H B,et al.Response surface methodology enzymes and metabolites characterization [J].Chem Eng J,2010,165:200-208.
[77] Kolekar Y M,Nemade H N,Markad V L,et al.Decolorization and biodegradation of azo dye Reactive Blue 59 by aerobic granules [J].Bioresour Technol,2012,104:818-822.
[78] Waghmode T R,Kurade M B,Khandare R V,et al.A sequential aerobic/microaerophilic decolorization of sulfonated mono azo dye Golden Yellow HER by microbial consortium GG-BL [J].Int Biodeter Biodegr,2011,65(7):1024-1034.
[79] Yemashova N A,Kotova I B,Netrusov A I,et al.Special traits of decomposition of azo dyes by anaerobic microbial communities [J].Appl Biochem Microbiol,2009,45:176-181.
[80] Qu Y,Shi S,Ma F,et al.Decolorization of Reactive Dark Blue K-R by the synergism of fungus and bacterium using response surface methodology [J].Bioresour Technol,2010,101:8016-8023.
[81] Saratale R G,Saratale G D,Chang J S,et al.Decolorization and biodegradation of reactive dyes and dye wastewater by a developed bacterial consortium [J].Biodegradation,2010,21:999-1015.
[82] Dafale N,Rao N N,Meshram S U,et al.Decolorization of azo dyes and simulated dye bath wastewater using acclimatized microbial consortium:Biostimulation and halo tolerance [J].Bioresour Technol,2008,99(7):2552-2558.
[83] Levin I,Melignani E,Ramos A M.Effect of nitrogen sources and vitamins on ligninolytic enzyme production by some white-rot fungi,dye decolorization by selected culture filtrates [J].Bioresour Techno,2010,101:4554-4563.
[84] Chang J S,Lin Y C.Fed-Batch bioreactor strategies for microbial decolorization of azo dye using aPseudomonasluteolastrain [J].Biotechnol Prog,2000,16(6):979-985.
[85] Jadhav U U,Dawkar V V,Ghodake G S,et al.Biodegradation of Direct Red 5B,a textile dye by newly isolatedComamonassp.UVS [J].J Hazard Mater,2008,158(2/3):507-516.
[86] Pearce C I,Lloyd J R,Guthriea J T.The removal of colour from textile wastewater using whole bacterial cells:A review dyes pigments [J].2003,58(3):179-196.
[87] Wang H J,Su Q,Zheng X W,et al.Bacterial decolorization and degradation of the reactive dye reactive red 180 byCitrobactersp.CK3 [J].Int Biodeter Biodeg,2009,63(4):395-399.
[88] Myrna S,Aida S,Hermminial I P,et al.Microbial decolouration of azo dyes:A review [J].Process Biochemistry,2012,47:1723-1748.
[89] Telke A A,Kalyani D C,Dawkar V V,et al.Influence of orangnic and inorganic compounds on oxidoreductive decolorization of sulfonated azo dye C.I.Reactive Orange 16 [J].Hazard Mater,2009,172(1):298-309.
[90] Hong Y,Chen X,Guo J,et al.Effects of electron donors and acceptors on anaerobic reduction of azo dyes byShewanelladecolorationisS12 [J].Appl Microbiol Biotechnol,2007,74(1):23-28.
[91] Saratale R G,Saratale G D,Chang J S,et al.Bacterial decolorization and degradation of azo dyes:A review [J].Journal of the Taiwan Institute of Chemical Engineers,2011,42:138-157.
[92] Komal S,Sindhu S,Suddhasatwa B.Microbial fuel cells for azo dye treatment with electricity generation:A review [J].Bioresour Technol,2013,131:564-571.
Advance in microbial decolorization and degradation of azo dyes
HUA Li,XIE Jing-kun,ZHU Chao,MA Hong-rui,PENG Xiang-yu
(CellegeofResourcesandEnvironment,ShaanxiUniversityofScience&Technology,Xi’an,Shaanxi710021,China)
With the development of textile printing and dying industry in recent years,synthetic dyestuffs are increasing in both number and variety and their improper discharges in aqueous ecosystems lead to severe water pollution.Developing eco-friendly,efficient,fast,and low-cost treatment methods is a hot research spot at present.Currently,the application and research of microorganisms for azo dyes wastewater treatment attract the most attention and many researches focus on the selection, isolation and domestication of potent species.This review introduces the mechanisms involving in the decolorization and degradation of azo dyes and related bacteria,fungi,algae and their consortium.This review also prospects the future application and research highlights to provide reference for the treatment of azo dye-containing wastewaters.
azo dyes;decolorization and degradation;bacteria;fungi;microbial consortium
时间:2015-09-09 15:41DOI:10.13207/j.cnki.jnwafu.2015.10.014
2014-03-20
陕西省自然科学基金项目(2015JM4127);陕西省教育厅专项科研计划项目(2013JK072);陕西科技大学博士科研启动基金项目(BJ12-29);陕西省科技统筹创新项目(省科技厅)(2013KTCL14)
花 莉(1978-),女,贵州贵阳人,副教授,博士,硕士生导师,主要从事环境生物技术研究。 E-mail:106910730@qq.com
X172;X791
A
1671-9387(2015)10-0103-11
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20150909.1541.028.html
