经济树种全基因组测序成果要报
2015-01-05郭起荣
冉 洪,张 莹,胡 陶,冯 云,廉 超,郭起荣
(国际竹藤中心 国家林业局竹藤科学与技术重点开放实验室,北京 100102)
经济树种全基因组测序成果要报
冉 洪,张 莹,胡 陶,冯 云,廉 超,郭起荣
(国际竹藤中心 国家林业局竹藤科学与技术重点开放实验室,北京 100102)
经济树种通常基因组较大,测序的组装、注释等存在较大困难,有必要对这方面的研究进展及存在的问题进行分析比较,以提高经济林木全基因组的研究效率。对已经完成全基因组测序的28种经济树种的全基因组测序成果进行了概述,比较了所采用的测序策略、技术与方法及所利用的测序材料,分析了各物种全基因组的大小、基因数量、基因密度、基因均长、平均内含子长度、GC含量等结构特点,汇集了各树种系统发育中的基因组复制等重要分子事件,探讨了其纤维素、木质素、糖与淀粉、油脂、抗性、生殖等重要生物经济性状的基因组学特征,展望了中国在基因组科学领域中的重要影响。
经济树种;全基因组测序;基因家族;基因;系统发育;生物经济性状
森林占陆地生态系统生物量的90%以上,林业和木材加工业对全球经济的贡献重大。经济林木不仅为人类提供了大量的建筑、造纸等原材料以及其他的可再生能源,而且在保水防沙和提高空气质量等方面起着重要的作用[1]。
基因(Gene)是生命遗传的基本单位,储存着生命的种族、孕育、生长、凋亡等过程的全部信息,解码这张生命的“说明书”是人们长期的梦想。
由于生命的复杂性,突破瓶颈,从基因组而不是从个别基因、基因片段、碱基序列的层面,破译整个基因组的基因序列、基因结构进化分析,通过组建数据平台,利用分子生物学技术手段,可以解析、注释、预测、关联重要经济性状的分子来源。
全基因组测序是对一种生物的基因组中的全部基因进行的测序,即测定其DNA的碱基序列[2]。全基因组测序(Whole-genome sequencing,WGS)不仅能够了解林木的基因组结构及其功能,而且能够了解林木的起源与进化[3]。
1 全基因组测序的技术选择及材料选用
测定生命体的腺嘌呤(A)、胸腺嘧啶(T)、鸟嘌呤的(G)、胞嘧啶(C)碱基的排列方式,在不同的技术、经济时代具有不同的策略、技术与方法,选用理想的测序材料也是全基因组测序的关键。
自从第一个全基因组测序的生命体——流感嗜血杆菌的测序工作完成的1995年以来,第一个真核生物是酿酒酵母(1996年),第一个植物当推拟南芥(2000年,并成为模式植物),水稻(2002)及其近缘的二穗短柄草(2010)、小立碗藓(2008)、烟草(2011)等随着基因组学研究的深入与完善,在一定程度上也相继成为某些模式。
1.1 采用的测序方法
2006年,毛果杨Populus trichocarpa全基因组草图发布在《Science》上[4],这开启了经济树种全基因组时代。统计至2014年底,已经有28种[4-38]经济树木的全基因组发布,对其测序所用的技术、方法和材料见表1,其中,完全采用第一代测序技术的有5种。随着2005年第二代测序技术的出现,已在开展的对苹果等7种经济树种的全基因组测序工作立即联用了二代技术,这使测序的进度、效率和准确率得以提高。对其余16种经济树种纯粹使用第二代测序技术便完成了其全基因组测序,且完美展现了二代测序技术的低成本、高速率、通量大、高覆盖度等特点。第三代单分子测序技术 (SMRT DNA-Sequencing)已经正式进入了实战阶段,人的全基因组测序随之降至的“一千美元”时代,期望着早日在经济树木中现光溢彩。
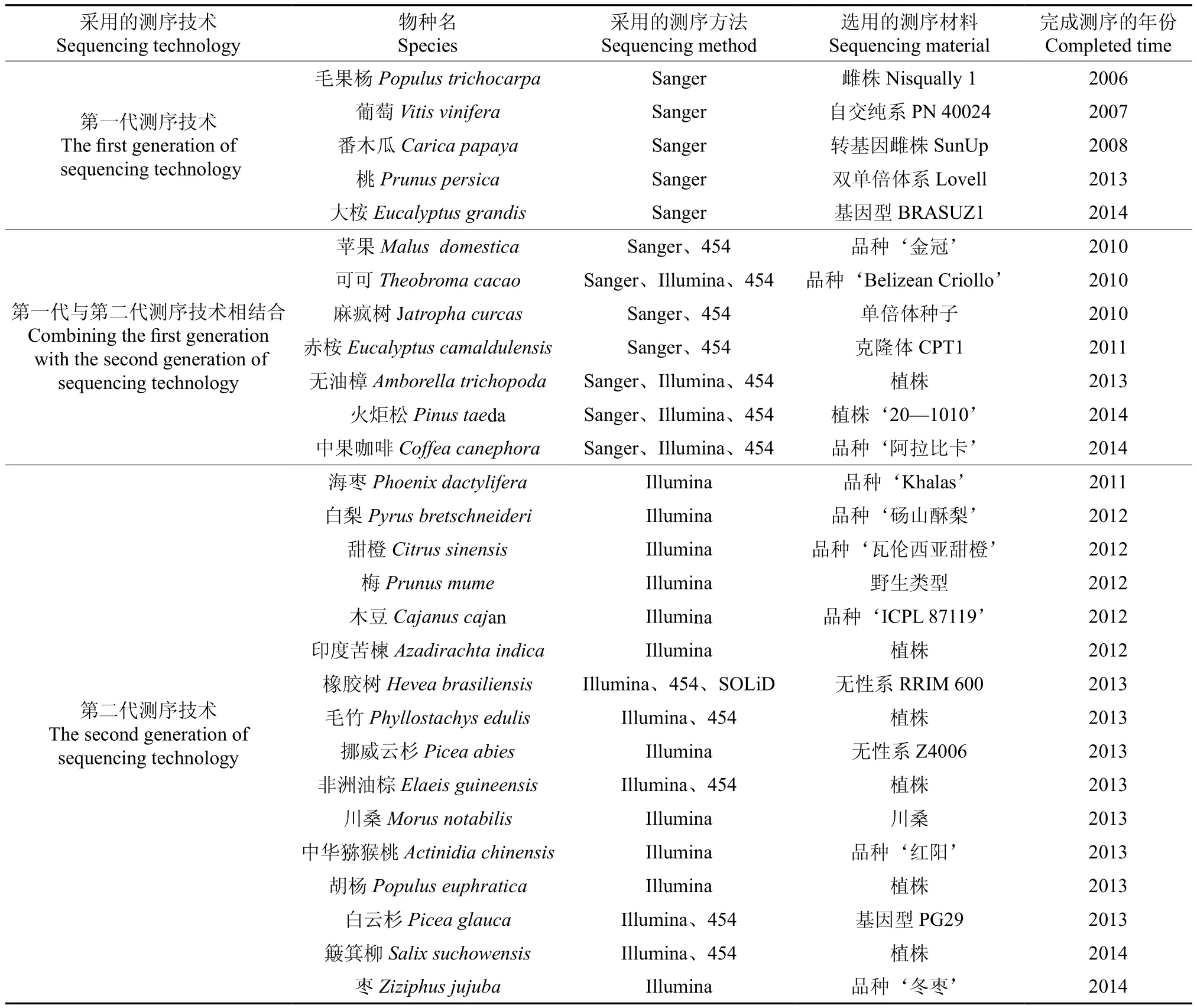
表 1 已完成全基因组测序的28种经济树木Table 1 28 economic tree species completed whole genome sequencing
最原始的DNA测序方法为Maxam-Gilbert法,在对经济树种的测序中未见使用。对28种经济林木的测序最普遍采用的是Sanger测序法,通过合成与单链DNA互补的多核苷酸链来读取待测DNA分子的序列,合成的互补单链可在不同位置随机终止反应。
第二代高通量测序中的主流技术是Roche公司的454焦磷酸测序、Illumina/Solexa聚合酶合成测序和ABI公司的SOLiD连接酶测序技术。这3种二代测序技术的原理各不相同,其数据量产出、数据质量和单Run运行的成本也不一样,华大基因岳桂东等人对其进行了很好的陈述[39]。
1.2 选用的测序材料
在进行全基因组测序时,纯合度越高的材料对于测序完成后的组装拼接越便捷。树木基因组具有高杂合度的固有特点,使用育种方法获取越纯的材料越有利于测序的成功。对葡萄、番木瓜、桃、大桉、麻疯树、赤桉、橡胶树、挪威云杉、白云杉等物种的测序采用转基因、克隆技术等人工方法得到较为理想的测序材料,对毛果杨、苹果、可可、非洲海枣、白梨、甜橙、木豆、中华猕猴桃、中果咖啡、枣等物种的测序选用的材料都是低杂合度的品种。
随着测序技术的进步,对测序材料杂合度进行估算就成为基因组调查的一个重要内容。从已经发表测序的文章中可得知:枣测序材料的杂合度为1.9%,白梨1.02%,中华猕猴桃0.54%,毛果杨0.26%,梅0.03%。

表 2 28种经济树木的全基因组测序数据的比较†Table 2 Comparison of whole genome sequencing data of 28 economic tree species
2 全基因组的组成结构特点
所有细胞形态的生物都以DNA为遗传信息载体,生命多样性的基础在于基因组DNA含量与组成的差异[2]。一个基因包含编码蛋白质肽链或RNA的核酸序列,还包含为保证转录所必需的调控序列:5′非翻译序列、内含子以及3′非翻译序列。表2直观比较了28种经济树木的基因组大小等结构特点。
2.1 基因组大小
基因组的大小就是指一个基因组中拥有的DNA含量的多少。常用于测定基因组大小的方法主要有流式细胞分析(Flow cytometry)法和孚耳根微显影(Feulgen microdensitometry)法等种[40]。
这28种经济树木的全基因组大小为265~23 200 Mb,裸子植物的基因组普遍大于被子植物。比如桃(265)与火炬松(23 200)的基因组大小就相差近88倍,这反映出C值悖理现象,基因组中DNA含量差异很大,它们的差异性与生物的复杂性程度并不完全相关[41]。在物种进化过程中,基因组多倍化、转座子积累是导致基因组增大的主要原因,而同源不平等重组和非正规重组则是基因组DNA丢失的潜在动力,制约着基因组无限制的增大[42-43]。
2.2 基因数量
DNA测序组装完成后,常采用ab inition等生物信息学软件预测物种的基因数量。在已经完成测序的28种经济树种中,赤桉的全基因组预测的基因数量最多(77 121个),白云杉的最少(13 100个),这28种经济树种的基因数量平均为36 600个。
2.3 基因均长
基因是控制生物体性状的基本单元,其长度可用碱基对的多少来度量,常用碱基对(base-pairs,bp) 来表示。基因均长关系到基因组的复杂程度。在已经测序的经济树种中,仅见白云杉的基因均长为5 151 bp,其余树种的都在1~1 000 bp之间。
2.4 内含子平均长度
内含子(intron)是真核生物细胞DNA中的间插序列,是编码区的重要组成部分。内含子增加了基因的长度,提高了基因间的重组频率,且经常含有调控元件,其差别剪接可使一种基因指导合成多种蛋白。真核生物基因组中内含子的长度在基因组内和物种间的差异都比较大[2]。在已经完成测序的28种经济树种中,火炬松的平均内含子长度(2 700 bp)最长,而葡萄的平均内含子长度(213 bp)最短。
2.5 基因密度
基 因 密 度(gene density,genomic density) 指DNA单位长度上的基因数量,常用“基因数/10 kb”来表示。从现有的经济树木全基因组数据来看,裸子植物平均580 bp有一个基因;被子植物平均19 bp有一个基因。从表2中还可看出,裸子植物的基因密度普遍低于被子植物,其基因密度是被子植物的数十分之一甚至数百分之一,反映了其进化关系。
2.6 GC含量
实验过程中,常采用分光光度法或以流式细胞仪测量DNA双螺旋结构的熔化温度,以测定GC的含量。在全基因组测序过程中,基因组的GC含量直接影响着测序的难度。当物种的GC含量高,PCR扩增便困难,测序深度则低,同时也提高了扩增时的错误率。高等植物毛果杨(33.30%)、麻疯树(34.30%)、赤桉(33.6%)、木豆(32.80%)与枣(33.41%)的GC含量均低于35%,故以提高测序深度来保证全基因组测序的成功完成,枣、木豆的测序深度分别到达了68、163.4 倍。
此外,全基因组DNA序列里面包含了许多重复序列(repeated sequence),苹果的重复序列达到了67%,而甜橙的只有20%,这些高度重复的序列主要存在于非编码区,在生物进化过程中发生了扩张,这一方面导致了基因组大小在进化过程中的突发式的快速扩张,另一方面,其中的一部分重复序列因可能携带有可编码序列,从而致使该物种的基因总数也相应急剧增加。此外,扩张后的基因组,其所包含的表达基因或暂时未表达的基因更为丰富,成为该物种应对可能的更为多样性生存环境的后备基因资源,其生存适应性提高了。
3 系统发育分析
在前基因组时期,人们也利用碱基对、基因片段、基因来进行生物品种鉴定、系谱分析及进化关系的研究,并取得了重要成果。一些保守性高且在物种中广泛存在的基因被当作构建进化树的分子标准[44-45],但每个基因有着不同的进化历程,存在横向转移等原因[46-48],基于不同的分子标准得到的结果可能不尽相同,基于全基因组序列的生物系统发育分析(phylogenetic analysis)当然更具准确率和科学性。
根据各物种全基因组测序结果,利用Interactive Tree Of Life(ITOL)在线工具,构建了如图1的28种经济树种的分类树,并评估了部分物种的分化时间、全基因组复制(Whole Genome Duplication,WGD)事件。
由图1可知,被子植物部分与根据基因亲缘关系分类的APG III[49](Angiosperm Phylogeny Group III)分类法构建的种系发生树一致。
全基因组复制事件是系统发育的重要资源[50],几乎所有的被子植物基因组都有WGD证据,多倍体化是被子植物进化的重要推动因素[51]。属于单子叶的毛竹、海枣与如中华猕猴桃、拟南芥、木豆等双子叶植物一样,根据全基因组测序结果可明确推断其发生过WGD,并且在单子叶植物与双子叶植物分化后,共同发生过一次WGD[52],这便构筑了毛竹、海枣基因组的复杂性。双子叶植物与单子叶植物分化后,共同发生的一次WGD,就是古六倍体化事件,对葡萄WRKY转录因子家族全基因组分析[53]也证明了古六倍体化事件。
在28种已经完成全基因组测序的经济树种中,仅有现存被子植物中已知最早与其它被子植物分开演化而倍受植物学家关注的孑遗植物——无油樟和3种裸子植物没有提及WGD分子事件。
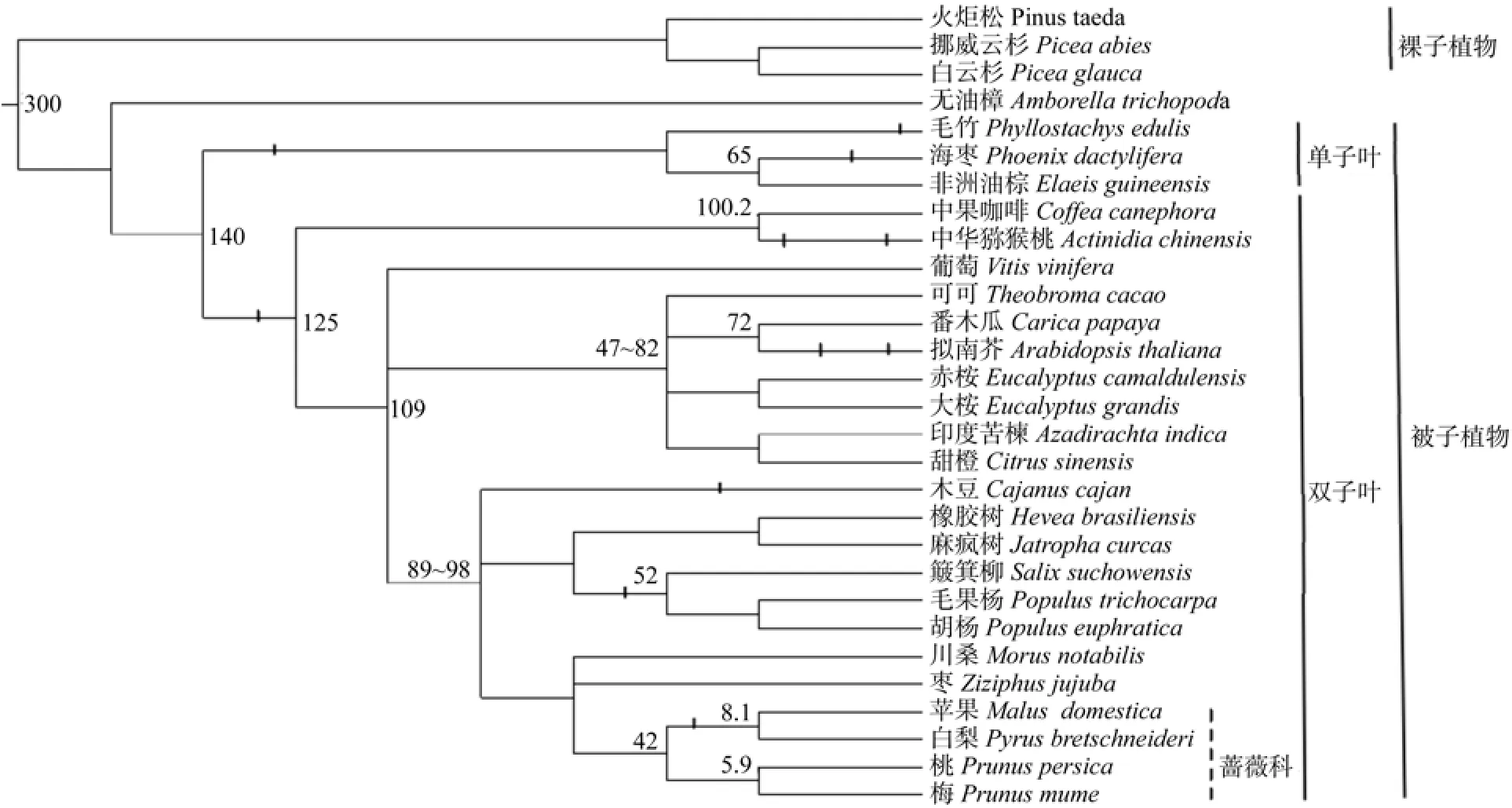
图 1 经济树种系统发育树及全基因组复制事件Fig. 1 Phylogenetic tree and the whole genome duplication events of economic tree species
4 生物性状的基因组学解析
至2014年底,已经完成全基因组测序的28种经济树种中,包含了毛果杨、火炬松、挪威云杉等用材树种6种,苹果、枣等经济树种18种,胡杨、簸箕柳、梅等生态经济树种,“非草非木”的毛竹及极具进化意义的无油樟等树种。毛果杨在很多时候被作为木本植物的模式,用于对控制经济树种重要生物经济性状的基因家族(gene family)、基因(gene)的分子遗传扫描中。
4.1 纤维素
以用材为主的树种其纤维素合成基因是基因组学遗传解析的重点之一。纤维素(cellulose)是植物细胞里的主要成分,其基本单位是吡喃式D-葡萄糖,以糖苷键行使相连,其葡萄糖残基约有2 000~2 500 个,主要以小微纤丝的形式存在,一根微纤丝由36 根β-1,4葡糖苷链结晶而成。人们对纤维素的生物合成已有较清晰的认识[54]。
纤维素合成酶基因(CesA)家族调控着次生壁形成、木质部形成、木材张力的形成等。毛果杨的CesA家族有18个基因,占毛果杨纤维素合成相关基因的1/5,此外,还有纤维素酶基因(KOR)、蔗糖合成酶基因(SuSY)、纤维素定向基因(COBRA)和FRA2。火炬松CesA家族有6个基因、赤桉CesA家族有11个基因,毛竹CesA家族有19个,还有38个类纤维素合酶基因(Csl)。
水果中的纤维素影响其食用品质。在番木瓜中发现有11个CesA、8个COBRA基因与纤维素合成有关,同时还发现了β-葡萄糖转移酶(GT1)基因、α-1,2-岩藻糖酰转移酶(GT37)基因、β-葡糖醛酸转移酶(GT43)基因等相关基因。
4.2 木质素
木质素(lignin)沉积在木质部导管和厚壁组织及韧皮部纤维中,在植物体机械支持、水分运输和病虫害防御中具有重要作用[55-56]。木质素单体在细胞壁中脱氢聚合成木质[57-58]。
有关木质素在植物体内生物合成过程的研究已取得明显的进步[59],如桉树是造纸的重要原材料,通过调控OMT、CCR基因的表达能控制其木质素含量[60-61]。在毛果杨、毛竹中发现,羟基肉桂酰基转移酶(HCT)基因、肉桂酰CoA还原酶(CCR)基因数量占据主导地位;番木瓜中,苯丙氨酸解氨酶(PAL)基因、肉桂酸4-羟基化酶(C4H)基因、4-香豆酰-CoA连接酶(4CL)基因、HCT基因家族数目处于中等水平,而咖啡酸-O-甲基转移酶(COMT)基因、香豆酸-3-羟基化酶(C3H)基因数目分别只有1个和2个。与其它物种相比,番木瓜的CCR基因家族只有1个基因,阿魏酸-5-羟基化酶(F5H)基因家族有4个基因,肉桂醇脱氢酶(CAD)基因家族有18 个基因,它们都在木质素合成的后期发挥作用。
大桉中的PAL、C4H、4CL、HCT、C3H、咖啡酰辅酶A.O.甲基转移酶(CCoAOMT)基因、阿魏酸-5-羟基化酶(F5H)基因、COMT、CCR、CAD这10 个基因家族中,只有4CL、F5H家族含有1个基因,而其余8个家族却至少包含有2个基因,最多的是PAL基因家族,拥有7 个基因。
木质素同时也是石细胞的主要组成成分,其生物合成直接影响着石细胞的含量[62]。石细胞广泛存在于白梨等果实的果肉之中,严重影响着果实品质。在白梨果实发育早期,HCT、p-香豆酰-莽草酸/奎尼酸3'-羟化酶(C3'H)基因、咖啡酰-辅酶AO-甲基转移酶(CCOMT)基因高水平表达,促使p-香豆酰-辅酶A(PCC)向咖啡酰-辅酶A(CFC)和阿魏酰-辅酶A(FC)基因高水平转化,最终导致G型、S型木质素的积累。
4.3 糖类与淀粉
糖类与淀粉是经济林的主要目的性状。蔗糖是光合作用的主要产物,是多数植物体内长距离运输碳水化合物的主要形式,也是某些植物储藏的主要化合物。淀粉则几乎存在于所有的植物之中,是相对稳定的高分子碳水化合物。
有393个基因涉及枣的蔗糖与淀粉的代谢途径,其中的6个主要基因家族分别为木聚糖内糖基转移酶(XET)基因家族、β-葡萄糖苷酶(BGluc)基因家族、UDE-葡萄糖4-表异构酶(GALE)基因家族、果糖二磷酸醛缩酶(FBA)基因家族、棉子糖合酶(RFS)基因家族、MFS转运酶(MFS)基因家族。
在番木瓜中,主要有蔗糖合酶/蔗糖磷酸合酶(GT4)基因、中性转化酶(GH32)基因、己糖转运酶基因等。由糖类合成淀粉的过程中,主要有淀粉合酶(GT5)基因、淀粉分支基因、异淀粉酶(GH13)基因、β-淀粉酶(GH14)基因参与调控合成。
在蔷薇科植物中,光合作用形成碳水化合物主要以山梨糖醇形式转运,而非蔗糖。在苹果、梨、桃等树种中,调控山梨糖醇的基因主要是山梨醇6-磷酸脱氢酶(S6PDH)基因家族、山梨糖醇脱氢酶(SDH)基因家族、山梨糖醇转运酶(SOT)基因家族。
在甜橙全基因组测序中发现了半乳糖醛酸酯途径存在基因的高表达现象,其中的D-半乳醛糖酸还原酶(GalUR)基因、PG基因、PME基因起着关键性作用。
4.4 油 脂
油脂中最主要的成分是脂肪酸和三酰甘油(TAGs)。蔗糖是脂肪酸的主要碳源,通过糖酵解途径生成丙酮酸,再合成脂肪酸的碳供体,从而形成脂肪酸。甘油与脂肪酸进行酯化作用合成三酰甘油[63]。已进行了全基因组测序的油脂树种有典型的麻疯树、可可、油棕3种,材、脂兼用的火炬松未见其全基因组松脂松香方面的研究报道。
可可的油脂存在于种子中,三酰甘油是其重要组成部分,酰基-ACP硫酯酶(FATB)基因、酮脂酰-ACP合酶基因是合成三酰甘油的2个关键基因。
麻疯树种子中的三酰甘油含量很高。其中,乙酰辅酶A羧化酶(ACC)、杂聚肽ACCase生物素羧基载体蛋白亚基(BCCP)、酰辅酶A(LACS)、磷酸乙酰转移酶(GPAT)、溶血磷脂酸酰基转移酶(LTAT)、磷脂酸磷酸酯酶(LPP)、二酰基甘油酰基转移酶(DGAT)、二酰基甘油酰基转移酶(PDAT)、3-酮酰基-ACP合酶(KAS)、脂肪酸去饱和酶(FAD)、酰基载体蛋白(ACP)这11个基因家族调控甘油、脂肪酸的生成以及TAGs的合成。具有抗菌作用的异佛波酯在麻疯树中的产量也很高,其基焦磷酸合成酶(GGPPS)、蓖麻烯合成酶(CS)、萜烯羟化酶、酰基转移酶4个基因起着主要调控作用。
在非洲油棕的中果皮中,WRI1基因控制着油脂的积累,且其油籽中的LEAFY COTY-LEDON1(LEC1)、LEAFY COTYLEDON2(LEC2)、ABSCISIC ACID INSENSITIVE3 (ABI3)、FUSCA这4个基因通过WRI1起着正调控作用,促进油脂的积累。
4.5 维生素C
通过全基因组测序,重点关注生物性状的关键作用基因及其表达途径,分子解析植物体内重要的抗氧化剂及许多酶的辅助因子维生素C(Vc)的生物合成。中华猕猴桃因其高Vc含量而被誉为“水果之王”,其主要通过L-半乳糖途径合成Vc,其中的GDP-D-甘露糖3’,5’-表异构酶(GME)基因、GDP-L-半乳糖磷酸酶 (GGP)基因、D-苏阿醛糖1-脱氢酶(TAD)基因、L-半乳糖酸-1,4-内酯脱氢酶(Ga1LDH)基因起着决定性作用;还发现了碱性磷酸酶(Alase)、L-抗坏血酸过氧化物酶(APX)、肌醇加氧酶(MIOX)、单脱氢抗坏血酸还原酶(MDHAR)这4个涉及Vc合成的基因家族存在基因扩张现象。
4.6 其它次生代谢产物
酚类、萜类及含氮化合物等植物次生代谢产物具有重要的经济用途。
黄酮在植物的生长发育过程中具有重要作用,其形成的聚合物原花青素(类黄烷醇单体及其聚合体的多酚化合物)对人体很有益。在可可中发现,花青素合成酶(ANS)、花青素还原酶(ANR)、无色花色素还原酶(LAR)、黄烷酮醇-4-还原酶(DFR)这4个基因是合成黄烷酮的关键基因。毛果杨的类黄酮生物合成过程受查尔酮异构酶、类黄酮3-羟化酶、黄烷酮3-羟化酶、五色花色素还原酶、类萜合酶这5个基因控制。
可可的萜类物质单萜(C10)、倍半萜(C15)分别占34%、31%,烯萜合酶(TPS)基因是合成这两种萜类物质的最主要基因,可可中的棉子酚具有抵御虫害的作用,其关键基因是杜松烯合酶基因。印度苦楝中的TPS21、lytB/ispH、4-二磷酸胞苷-2-C-甲基赤藓糖激酶(ispE)、牻牛儿牻牛儿基焦磷酸合成酶(GGPS)、FDPS、FDFT1、SQLE是涉及其倍半萜烯、三萜的生物合成的基因。
橡胶树产生的乳胶其生物合成涉及到100个基因,这些基因分别存在于β-1,3-葡聚糖酶、卵磷脂酶基因烯醇酶、超氧化物歧化酶(Mn)、甲壳质酶、脂质转运前体、甘油松香酯合成、几丁质酶等14个基因家族中。
咖啡因是一种生物碱,在中果咖啡中发现了CcXMT、CcMTL、CcNMT3咖啡因合成过程中的最关键的3个基因。
梅香主要成分是苯丙酸类/苯环型化合物、肉桂酸和萜类物质,苄醇乙酰转移酶(BEAT)基因家族34个基因主要调控苯甲基的产生。白梨中的脂氧化酶(LOX)基因、乙醇脱氢酶(ADH)基因涉及α-亚麻酸代谢合成,与梨的挥发物芳香气味有关。
4.7 开花与生殖
磷脂酰乙醇胺结合蛋白(PEBP)FLOWERING LOCUS T(FT)的同源物是被子植物开花过程的重要激活剂,曾有报道记载,在裸子植物中缺乏FT基因的直系同源物,取而代之的是一个FT/TFL1类基因,也许其作用是抑制开花过程[64-65]。有关研究者确认了挪威云杉缺乏FT类基因,在其全基因组组装1.0版本中,首次确认了4个可能的FT/TFL1类基因。
MADS-box基因家族决定花组织的最终发育程度,与果实发育密切相关,可能调节梨果的发育。MADS-box包含12个亚家族,苹果的StMADS11亚家族存在着明显的扩张现象,其基因数达到了15个。不同于呼吸跃变型,甜橙属于非呼吸跃变型果实,其MADS-RIN基因可能是果实成熟的一种关键调节因子。在梅的MADS-box家族中发现了6个串联排列的转录因子家族(DAM),与休眠的诱导和解除有关,控制着梅能在低于0 ℃的温度条件下开花,而苹果中没有发现此类串联。
植物的PAS-FBOX-KELCH基因控制着光信号和开花时间。在番木瓜中发现,只有ZTL基因与PAS-FBOX-KELCH基因直系同源,而ZTL基因还缺乏明显的KELCH域;番木瓜全基因组中仅有少量的KELCH域(37个),由光敏形态建成1(COP1)基因家族的3个旁系同源基因抵抗紫外线。
毛竹成花过程转录因子涉及到花分生组织建成(FMI)、MADS-box、zf-Dof、YABBY这4个基因家族,在促进开花和光周期调节途径中起到关键作用的CONSTANS(CO)基因存在大量重复序列。
麻疯树为雌雄同株植物,涉及其花性别分化的3个基因是APETALA 2(AP2)、APETALA 3(AP3)、PISTILLATA ,与其开花有关的几个基因是CO、FLOWERING LOCUS D(FLD)、FLOWERING LOCUS F(FLF)、LEAFY(LFY)、SUPPRESSOR OF OVEREXPRESSION OF CONSTANS 1(SOC1)。
4.8 抗 性
经济树种的抗逆性是栽培与利用的限制性因子,在全基因组层面探测了其抗病、虫、旱、寒、盐碱等方面的分子信息。
植物的抗病基因R基因及其6个类型的基因数量是各经济树种全基因组重点论述的内容。
毛果杨PAD4、NPR1基因调控着水杨酸,在抗病(旱、寒)方面起作用,其中控制水杨酸累积上调的PAD4基因有2个拷贝,下调的NPR1有5个拷贝。在中果咖啡中发现其SH3基因对于咖啡叶锈病有很强的抵抗作用。川桑进化出能产生更多蛋白酶抑制剂(PIs)机制来抵御虫害,发现有79个PIs基因,其中最主要的是C1半胱氨酸肽酶抑制基因家族(22个基因)、A1/C1丝氨酸肽酶抑制基因家族(19个基因)。
在火炬松中发现了可诱导ABA的同源lp3基因家族的4个成员,其在干旱应答机制中起作用。
在胡杨中发现,其高亲和K+转运蛋白1(HKT1)基因家族有4个基因,抗氧化酶基因家族、热休克蛋白基因家族存在基因扩张现象,甜菜碱醛脱氢酶(BADH)基因、肌醇半糖苷合成酶4(GolS4)基因有更多的拷贝,以调节液泡浓度。
毛竹的全基因测序图示了多个器官材料,共有117个基因,分属21个抗逆基因家族的基因表达量,其全基因组测序文章的附件汇集了各抗性家族的同源基因、蛋白序列归类域。
5 展 望
随着基因组学的迅速发展,越来越多的物种基因组被解码,基因组学正在改变着整个世界——每一个基因组,多一次改变。
确定兴趣树种,评估合适的DNA测序材料,经过测序、组装、建库、提交、注释、进化分析等步骤,获得框架草图,成果发布,这已成为全基因组测序的基本范式。
相关资讯显示,我国杜仲、油茶、油桐、泡桐、白桦、紫竹、桂竹等物种也都已进入全基因组测序时代。买麻藤因其特殊的种系发生学位置,其全基因组研究成果将作为2017年在我国深圳举办的第19届世界植物学大会(XIX IBC)上重磅推出的成果之一;华大基因研究院推动的千种动植物基因组计划……这表明,中国已进入打造全球基因组大国、强国的行列,为生物经济、环境安全和人类健康服务。
[1]Jansson S, Douglas C J. Populus:a model system for plant biology[J]. Annu Rev Plant Biol, 2007,58:435-458.
[2]杨金水.基因组学[M].北京:高等教育出版社,2013:1-131.[3]施季森,王占军,陈金慧.木本植物全基因组测序研究进展[J].遗传, 2012,34(2):145-156.
[4]Tuskan G A, Difazio S, Jansson S,et al.The genome of black cottonwood,Populus trichocarpa(Torr & Gray)[J].Science,2006, 313(5793):1596-1604.
[5]Jaillon O, Aury J M, Noel B,et al.The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla[J]. nature, 2007,449(7161):463-467.
[6]Velasco R, Zharkikh A, Troggio M,et al.A high quality draft consensus sequence of the genome of a heterozygous grapevine variety[J]. PloS one,2007,2(12):e1326.
[7]Ming R, Hou S, Feng Y,et al. The draft genome of the transgenic tropical fruit tree papaya (Carica papayaLinnaeus)[J].Nature,2008,452(7190):991-996.
[8]Verde I, Abbott A G, Scalabrin S,et al.The high-quality draft genome of peach (Prunus persica) identi fi es unique patterns of genetic diversity, domestication and genome evolution[J].Nature genetics,2013,45(5):487-494.
[9]Myburg A A, Grattapaglia D, Tuskan G A,et al. The genome ofEucalyptus grandis[J].Nature, 2014,510(7505):356-362.
[10]Velasco R, Zharkikh A, Affourtit J,et al.The genome of the domesticated apple (Malus domesticaBorkh)[J].Nature genetics,2010, 42(10):833-839.
[11]Argout X, Salse J, Aury J M,et al.The genome ofTheobroma cacao[J].Nature genetics, 2011,43(2):101-108.
[12]Sato S, Hirakawa H, Isobe S,et al.Sequence analysis of the genome of an oil-bearing tree,Jatropha curcasL[J].DNA research, 2011, 18(1): 65-76.
[13]Hirakawa H, Nakamura Y, Kaneko T,et al.Survey of the genetic information carried in the genome ofEucalyptus camaldulensis[J].Plant Biotechnology,2011,28(5):471-480.
[14]Chamala S, Chanderbali A S, Der J P,et al. Assembly and validation of the genome of the nonmodel basal angiospermAmborella[J].Science,2013,342(6165):1516-1517.
[15]Rice D W, Alverson A J, Richardson A O,et al.Horizontal transfer of entire genomes via mitochondrial fusion in the angiospermAmborella[J].Science,2013,342(6165):1468-1473.[16]Albert V A, Barbazuk W B, Der J P,et al.TheAmborellagenome and the evolution of fl owering plants[J].Science, 2013,342(6165): 1241089.
[17]Neale D B, Wegrzyn J L, Stevens K A,et al. Decoding the massive genome of loblolly pine using haploid DNA and novel assembly strategies[J].Genome biology,2014 15(3): R59.
[18]Zimin A, Stevens K A, Crepeau M W,et al. Sequencing and assembly of the 22-Gb loblolly pine genome[J].Genetics, 2014,196(3): 875-890.
[19]Kovach A, Wegrzyn J L, Parra G,et al.ThePinus taedagenome is characterized by diverse and highly diverged repetitive sequences[J].BMC genomics,2010,11(1):420.
[20]Denoeud F, Carretero-Paulet L, Dereeper A,et al.The coffee genome provides insight into the convergent evolution of caffeine biosynthesis[J].science, 2014,345(6201):1181-1184.
[21]Al-Mssallem I S, Hu S, Zhang X,et al. Genome sequence of the date palmPhoenix dactyliferaL[J].Nature communications,2013, 4:2274.
[22]Al-Dous E K, George B, Al-Mahmoud M E,et al.De novo genome sequencing and comparative genomics of date palm(Phoenix dactylifera)[J]. Nature biotechnology, 2011, 29(6):521-527.
[23]Wu J, Wang Z, Shi Z,et al. The genome of the pear (Pyrus bretschneideriRehd)[J]. Genome research, 2013, 23(2):396-408.
[24]Xu Q, Chen L L, Ruan X,et al. The draft genome of sweet orange (Citrus sinensis)[J].Nature genetics,2013,45(1):59-66.[25]Zhang Q, Chen W, Sun L,et al.The genome ofPrunus mume[J].Nature communications, 2012,3:1318.
[26]Varshney R K, Chen W, Li Y,et al.Draft genome sequence of pigeonpea (Cajanus cajan), an orphan legume crop of resourcepoor farmers[J].Nature biotechnology,2012,30(1):83-89.
[27]Krishnan N M, Pattnaik S, Jain P,et al.A draft of the genome and four transcriptomes of a medicinal and pesticidal angiospermAzadirachta indica[J].BMC genomics,2012,13(1): 464.
[28]Krishnan N M, Pattnaik S, Deepak S A,et al. De novo sequencing and assembly ofAzadirachta indicafruit transcriptome [J]. Curr Sci,2011,101:1553-1561.
[29]Rahman A Y A, Usharraj A O, Misra B B,et al. Draft genome sequence of the rubber treeHevea brasiliensis[J]. BMC genomics, 2013, 14(1):75.
[30]Peng Z, Lu Y, Li L,et al. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla) [J]. Nature genetics,2013,45:456-461.
[31]Nystedt B, Street N R, Wetterbom A,et al.The Norway spruce genome sequence and conifer genome evolution[J].Nature, 2013,497(7451): 579-584.
[32]Singh R, Ong-Abdullah M, Low E T L,et al.Oil palm genome sequence reveals divergence of interfertile species in Old and New worlds[J].Nature,2013,500(7462):335-339.
[33]He N, Zhang C, Qi X,et al. Draft genome sequence of the mulberry treeMorus notabilis[J]. Nature communications, 2013,4: 2445.
[34]Huang S, Ding J, Deng D,et al. Draft genome of the kiwifruitActinidia chinensis[J].Nature communications,2013,4:2640.
[35]Ma T, Wang J, Zhou G,et al. Genomic insights into salt adaptation in a desert poplar[J]. Nature communications, 2013, 4: 2797.
[36]Birol I, Raymond A, Jackman S D,et al.Assembling the 20 Gb white spruce (Picea glauca) genome from whole-genome shotgun sequencing data[J].Bioinformatics,2013,29(12):1492-1497.
[37]Dai X, Hu Q, Cai Q,et al. The willow genome and divergent evolution from poplar after the common genome duplication[J].Cell research,2014,24(10):1274-1277.
[38]Liu M J, Zhao J, Cai Q L,et al.The complex jujube genome provides insights into fruit tree biology[J].Nature communications, 2014, 5: 5315.
[39]岳桂东,高 强,罗龙海,等.高通量测序技术在动植物研究领域中的应用[J].中国科学, 2012,42(2):107-124.
[40]陈建军,王 瑛.植物基因组大小进化的研究进展[J].遗传,2009,31(5):464-470.
[41]Thomas Jr C A. The genetic organization of chromosomes[J].Annual review of genetics, 1971,5(1):237-256.
[42]Hawkins J S, Grover C E, Wendel J F. Repeated big bangs and the expanding universe: Directionality in plant genome size evolution[J].Plant Science,2008,174(6):557-562.
[43]Hawkins J S, Hu G, Rapp R A,et al.Phylogenetic determination of the pace of transposable element proliferation in plants: copia and LINE-like elements inGossypium[J].Genome, 2007, 51(1):11-18.
[44]Woese C R, Kandler O, Wheelis M L. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya[J].Proceedings of the National Academy of Sciences,1990, 87(12): 4576-4579.
[45]Doolittle R F, Handy J. Evolutionary anomalies among the aminoacyl-tRNA synthetases[J]. Current opinion in genetics &development,1998,8(6):630-636.
[46]Heinemann J A, Kurenbach B. Eecyclopedia of Microbiology[M].Oxford: S Moselio, 2009, 597-606.
[47]Kalia V C, Lal S, Cheema S. Insight in to the phylogeny of polyhydroxyalkanoate biosynthesis: horizontal gene transfer[J].Gene, 2007, 389(1):19-26.
[48]Philippe H, Douady C J. Horizontal gene transfer and phylogenetics[J]. Current opinion in microbiology, 2003, 6(5):498-505.
[49]Bremer B, Bremer K, Chase M,et al.An update of the Angiosperm Phylogeny Group classi fi cation for the orders and families of fl owering plants: APG III[J].Botanical Journal of the Linnean Society, 2009, 161:105-121.
[50]Jiao Y, Wickett N J, Ayyampalayam S,et al. Ancestral polyploidy in seed plants and angiosperms[J]. Nature, 2011, 473(7345):97-100.
[51]Soltis D E, Bell C D, Kim S,et al.Origin and early evolution of angiosperms[J].Annals of the New York Academy of Sciences,2008,1133(1):3-25.
[52]Tang H, Bowers J E, Wang X,et al. Angiosperm genome comparisons reveal early polyploidy in the monocot lineage[J].Proceedings of the National Academy of Sciences,2010,107(1):472-477.
[53]李成慧,蔡 斌.葡萄WRKY转录因子家族全基因组分析[J].经济林研究,2013,31(4):127-131.
[54]李春秀,齐力旺,王建华,等.植物纤维素合成酶基因和纤维素的生物合成[J].生物技术通报,2005,(4):5-11.
[55]Boerjan W, Ralph J, Baucher M. Lignin biosynthesis[J].Annual review of plant biology,2003, 54(1):519-546.
[56]Baucher M, Halpin C, Petit-Conil M,et al. Lignin: genetic engineering and impact on pulping[J].Critical Reviews in Biochemistry and Molecular Biology,2003,38(4):305-350.
[57]Grima-Pettenati J, Goffner D. Lignin genetic engineering revisited[J]. Plant Science,1999, 145(2):51-65.
[58]Rogers L A, Campbell M M. The genetic control of lignin deposition during plant growth and development[J].New phytologist, 2004,164(1):17-30.
[59]章霄云,郭安平,贺立卡,等.木质素生物合成及其基因调控的研究进展[J].分子植物育种, 2006,4(3):431-437.
[60]谷振军,章怀云,张党权,等.赤桉木质素合成途径OMT基因家族的原核表达与纯化研究[J].中南林业科技大学学报,2014, 34(6):24-27, 60.
[61]陈博雯,盖 颖,蒋湘宁.尾叶桉GLU4肉桂酰-辅酶A还原酶基因克隆及原核表达[J].中南林业科技大学学报,2014,34(11): 71-76, 97.
[62]Tao S, Khanizadeh S, Zhang H,et al.Anatomy, ultrastructure and lignin distribution of stone cells in twoPyrusspecies[J].Plant Science, 2009,176(3):413-419.
[63]周 丹,赵江哲,柏 杨,等.植物油脂合成代谢及调控的研究进展[J].南京农业大学学报,2012, 35(5):77-86.
[64]Karlgren A, Gyllenstrand N, Källman T,et al.Evolution of the PEBP gene family in plants: functional diversification in seed plant evolution[J].Plant physiology, 2011, 156(4): 1967-1977.[65]Klintenäs M, Pin P A, Benlloch R,et al.Analysis of conifer FLOWERING LOCUS T/TERMINAL FLOWER1-like genes provides evidence for dramatic biochemical evolution in the angiosperm FT lineage[J]. New Phytologist, 2012, 196(4):1260-1273.
An essential report on the achievements of whole genome sequencing in economic trees
RAN Hong, ZHANG Ying, HU Tao, FENG Yun, LIAN Chao, GUO Qi-Rong
(SFA Key Laboratory of Bamboo and Rattan Science and Technology, International Center for Bamboo and Rattan,Beijing 100102, China)
There exist some dif fi culties in assembly and annotation of genome sequencing of economic trees owning to its enormous genome. In order to enhance research ef fi ciency on whole genome in economic trees, it is necessary to analyze and compare the research progress at the aspect and the existing problems. The achievements of whole genome sequencing of the 28 economic tree species were summarized. The sequencing strategies, technology, methods,and the sequencing materials were compared. The whole genome size, gene numbers, gene density, average length of genes, average length of introns, GC content and other structure characteristics were analyzed. Moreover, the important molecular events in the evolution process of the species were collected, and the important biological economic traits were dissected, including cellulose, lignin, sugar and starch, grease, resistance, and so on. In addition, the important role of China in the fi eld of genome sciences was forecasted.
economic trees; whole genome sequencing; gene family; gene; phylogenesis; biological economic traits
S602.4
A
1003—8981(2015)02—0149—09
2015-03-02
国家自然科学基金项目(31370631)。
冉 洪,硕士研究生。
郭起荣,教授,博士研究生导师。E-mail:QRGUO@icbr.ac.cn
冉 洪,张 莹, 胡 陶,等.经济树种全基因组测序成果要报[J].经济林研究,2015,33(2):149-157.
10.14067/j.cnki.1003-8981.2015.02.026
http: //qks.csuft.edu.cn
[本文编校:伍敏涛]
