番茄品种SSR标记鉴定技术研究
2015-01-04
(北京市种子管理站,北京100088)
番茄品种SSR标记鉴定技术研究
赵海艳 吴明生*宋 歌 牛 茜 田慧芳
(北京市种子管理站,北京100088)
以16个番茄品种为材料,从200对番茄SSR引物中筛选出20对适合用于番茄品种鉴定的引物,用该20对引物对66个番茄品种进行分析,共检测出52个等位基因变异,等位基因变异范围为2~5个,平均每个位点产生2.6个等位基因变异。通过遗传聚类方法对引物组合的品种鉴别能力进行分析,结果显示仅用9对引物构成的引物组合就可以对66个番茄品种进行有效鉴别,该9对引物组合构建的SSR-DNA指纹图谱可作为品种的遗传标签用于品种真伪鉴定。
番茄;SSR;品种鉴定;指纹图谱
番茄(Solanum lycopersicum L.)是我国的主要栽培蔬菜之一,但我国番茄种质资源相对匮乏,加上消费者对番茄风味的特殊要求,导致育种单位所采用的育种材料集中在少数种质资源上,育成的品种遗传差异小。由于亲缘关系相近的品种很难利用外观形态加以区别,容易出现一品多名、一名多品,或以劣充好的违法行为。因此,亟须建立一套准确、高效的番茄品种鉴定技术。
与传统的品种鉴定方法相比,SSR分子标记具有多态性高、重复性和稳定性好等优点,广泛用于玉米、水稻等主要农作物的品种鉴定和指纹图谱构建(宋建 等,2006),在品种打假维权方面取得很好效果。在番茄方面,Suliman等(2002)利用AFLP、SSR、SNP等技术对番茄品种进行多态性分析,认为SSR标记较适合于种质资源多样性分析。王日升等(2006)比较了SSR 标记和形态标记在番茄栽培品种遗传多样性的分析结果上具有高度一致性,表明SSR标记可用于番茄品种鉴别。本试验对200对番茄SSR引物进行筛选获得适合番茄品种鉴定的候选引物,通过遗传相似聚类分析方法确定适合66个番茄品种鉴定的SSR引物组合,并构建了番茄品种的SSR-DNA指纹,以期为番茄品种分子鉴定和品种权保护提供技术支撑。
1 材料与方法
1.1 试验材料
试验于2013~2014年在北京市种子管理站种子检测室进行。供试番茄材料由本站收集,共计66个品种,包括樱桃番茄和普通番茄,其品种名称和主要性状见表1。
1.2 试验方法
1.2.1 DNA提取 采用新型植物基因组DNA提取试剂盒(TIANGEN,北京)提取番茄的基因组DNA,于-20 ℃保存备用。
1.2.2 SSR引物 200对SSR引物来自Benor等(2008)和番茄基因数据库(http://www.sgn.cornell. edu),普通引物由北京鼎国昌盛生物技术有限责任公司合成,荧光标记引物由上海英骏生物技术有限公司合成。
1.2.3 PCR 扩增 PCR反应体系总体积为12 μL。包括30 ng模板DNA,MgCl22.5 mol·L-1,dNTPs 0.15 mol·L-1,引物0.25 μmol·L-1,Taq 聚合酶0.1 U,1×Taq Buffer。
PCR反应程序:94 ℃预变性5 min;94 ℃变性40 s,58 ℃退火35 s,72 ℃延伸45 s,共36个循环;72 ℃延伸10 min,扩增产物4 ℃保存。
1.2.4 PCR产物检测 普通引物的PCR产物和荧光标记引物的PCR产物分别通过生物大分子分析仪(LabChip GX)和毛细管电泳仪(ABI 3500XL)进行分析。
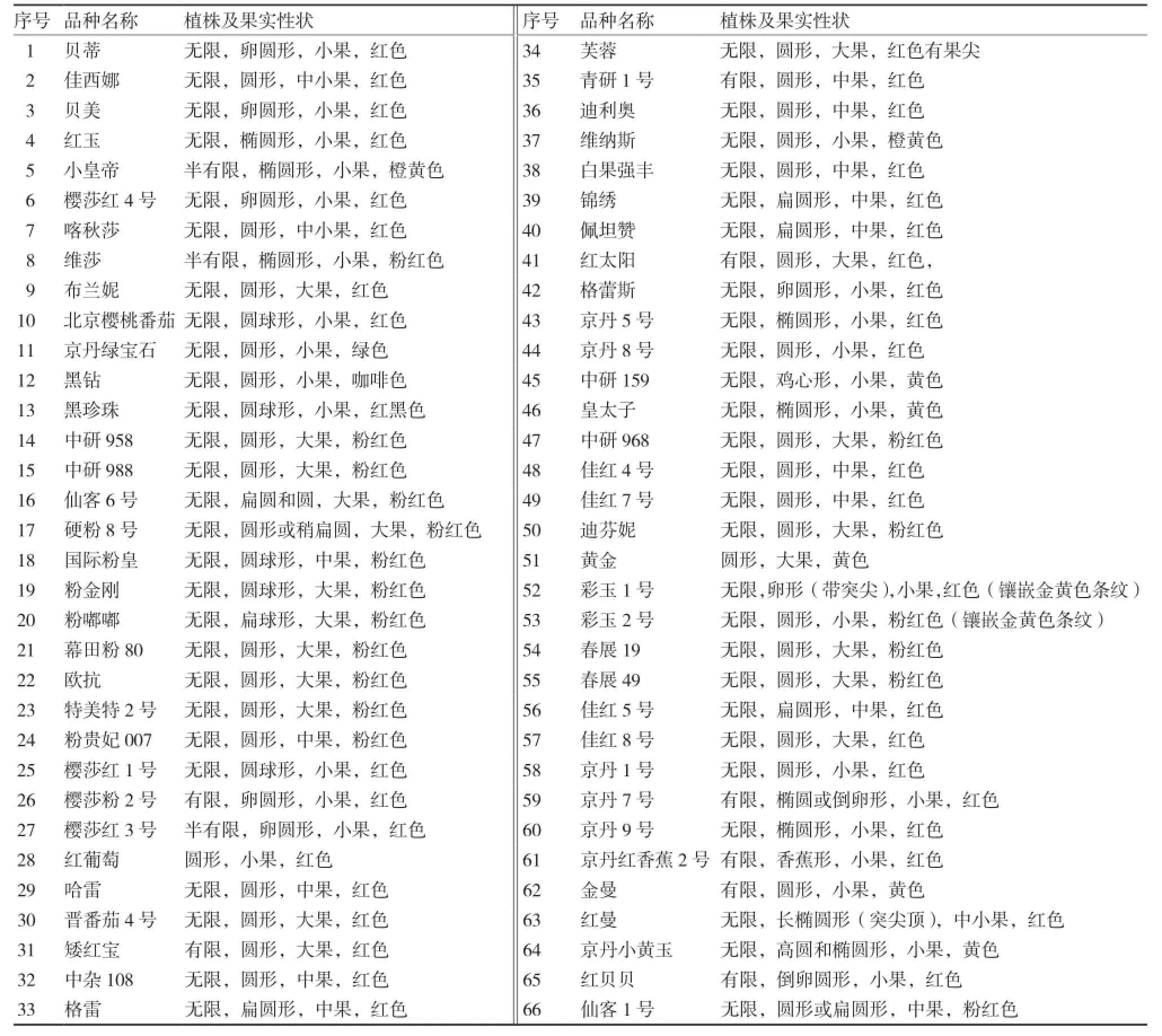
表1 66个番茄品种的名称及主要性状
1.2.5 数据统计与分析 SSR扩增分离结果采用“0,1”统计方法,在同一分子量上,有带记为“1”,无带记为“0”。多态性信息含量PIC=1-∑Pi2,Pi表示在i位点上的等位基因频率。通过NTSYS-pc Version 2.0软件按照非加权配对算数平均法(UPGMA)完成遗传聚类。
2 结果与分析
2.1 引物筛选
基于生物大分子分析仪电泳平台,将不同果形、果色和生长类型的16个番茄品种作为引物筛选材料,对200对SSR引物质量进行评估,从中筛选出等位基因大于2、扩增谱带清晰且重复性好的20对SSR引物作为番茄品种鉴定的候选引物(表2)。基于毛细管电泳平台,用66个番茄品种对筛选的20对引物多态性进行分析,结果显示(表2)20个SSR位点共检测出52个等位基因变异,等位基因变异范围为2~5个,平均每个位点等位基因变异数为2.6个;20个位点共检测出88个基因型(图1),变幅为3~11个,平均为4.4个;20个位点多态性信息量(PIC)变幅为0.678~0.860,平均为0.756,均为高度多态性信息引物,表明筛选出的引物能较好地反映66份番茄品种的基因型多样性水平,可用于番茄品种种质分析。
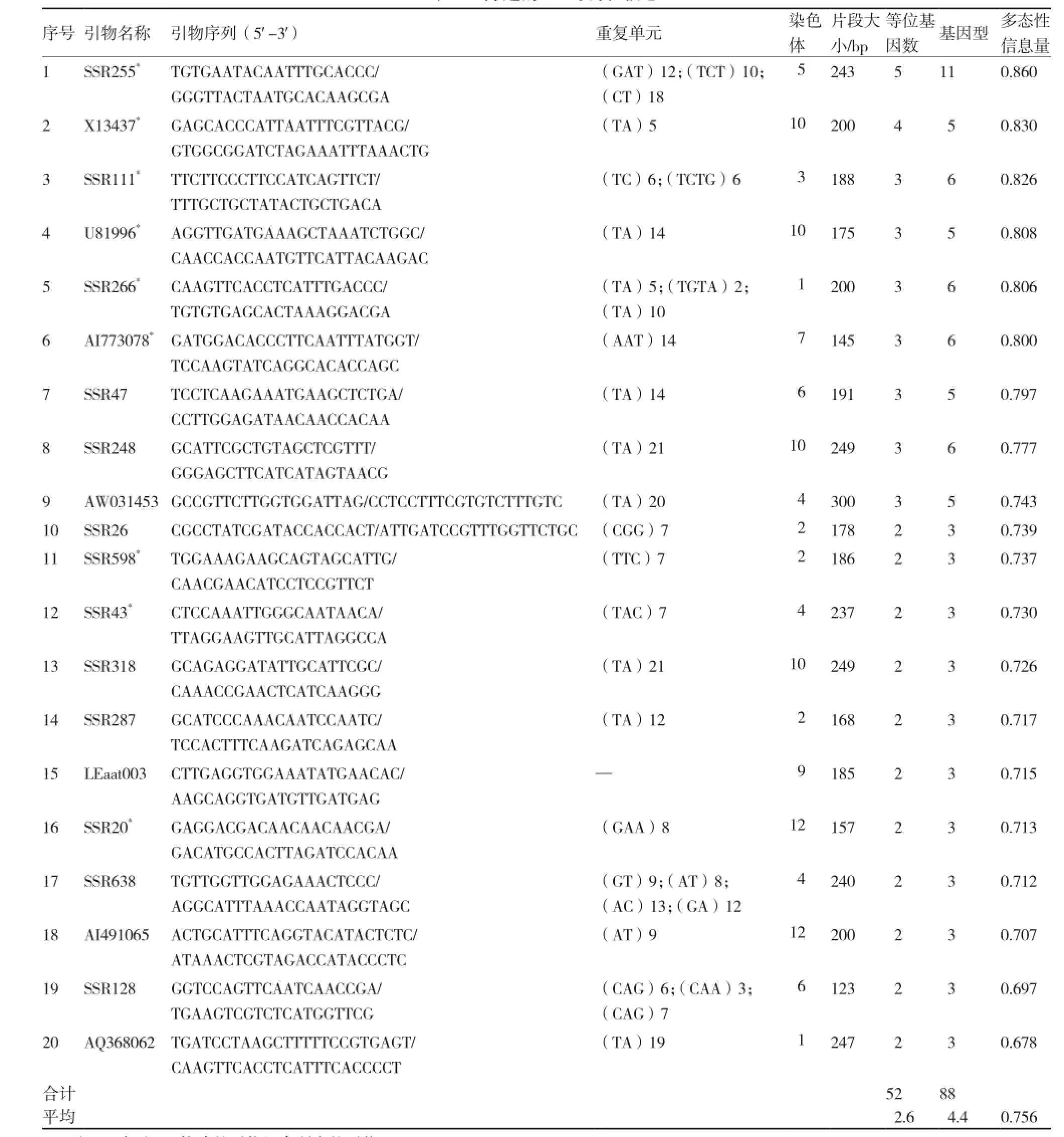
表2 筛选的20对引物信息
2.2 引物组合与指纹图谱构建
为构建66个番茄品种适宜的鉴定引物组合,将筛选的引物按照其多态性信息量高低(表2)排序,分别采用前1对、前2对、前3对等一直到前20对引物对66个番茄品种进行遗传相似性聚类分析。结果显示,当引物组合数量达到16对时才可以将所有品种区别开(图2),品种间遗传相似系数为0.182~0.977,品种间至少存在1个位点的差异。由于前10对引物和前6对引物以及前15对引物和前12对引物在鉴别品种数量上没有差异(图2),引物的增加对整个引物组合鉴别能力没有明显影响,所以将引物7、8、9、10、13、14和15剔除,最终的组合只需9对引物,即引物SSR255、X13437、SSR111、U81996、SSR266、AI773078、SSR598、SSR43和SSR20。用该9对引物组合对66个品种进行遗传相似性聚类分析(图3),品种间遗传相似系数为0.148~0.963,平均遗传相似系数为0.601,与用前16对引物获得平均遗传相似系数0.605差异不是很明显。中研988和硬粉8号之间遗传相似系数最大为0.963,在LEaat003和SSR111位点上有差异。
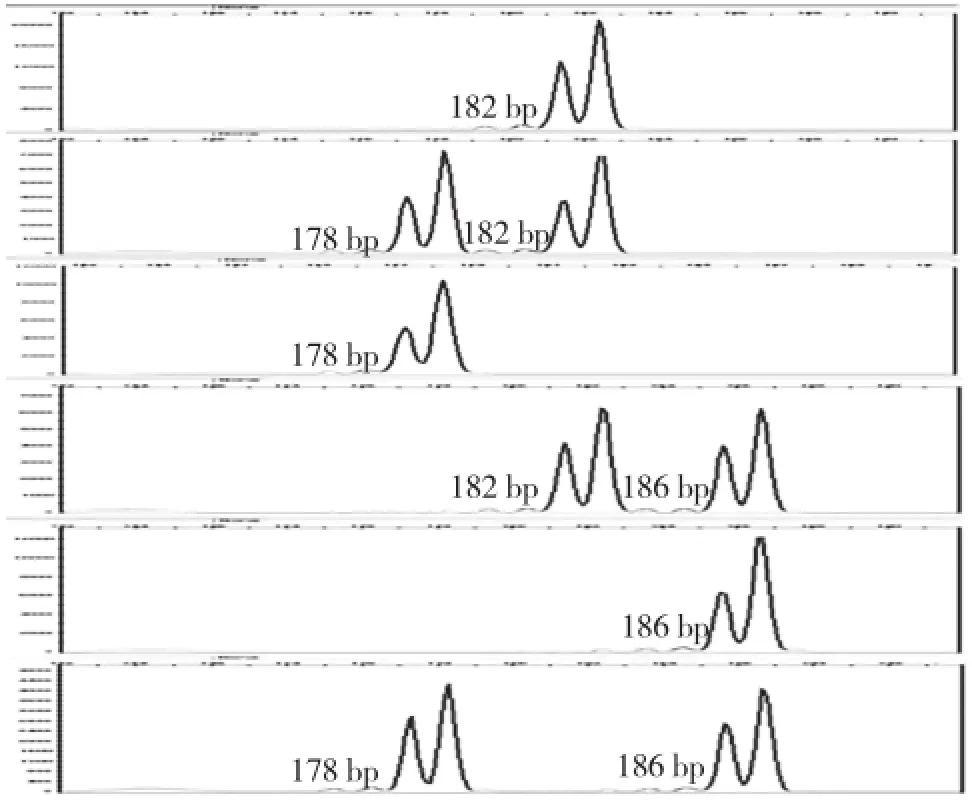
图1 番茄品种在SSR111位点上表现的基因型

图2 引物数量与鉴别品种数量的关系
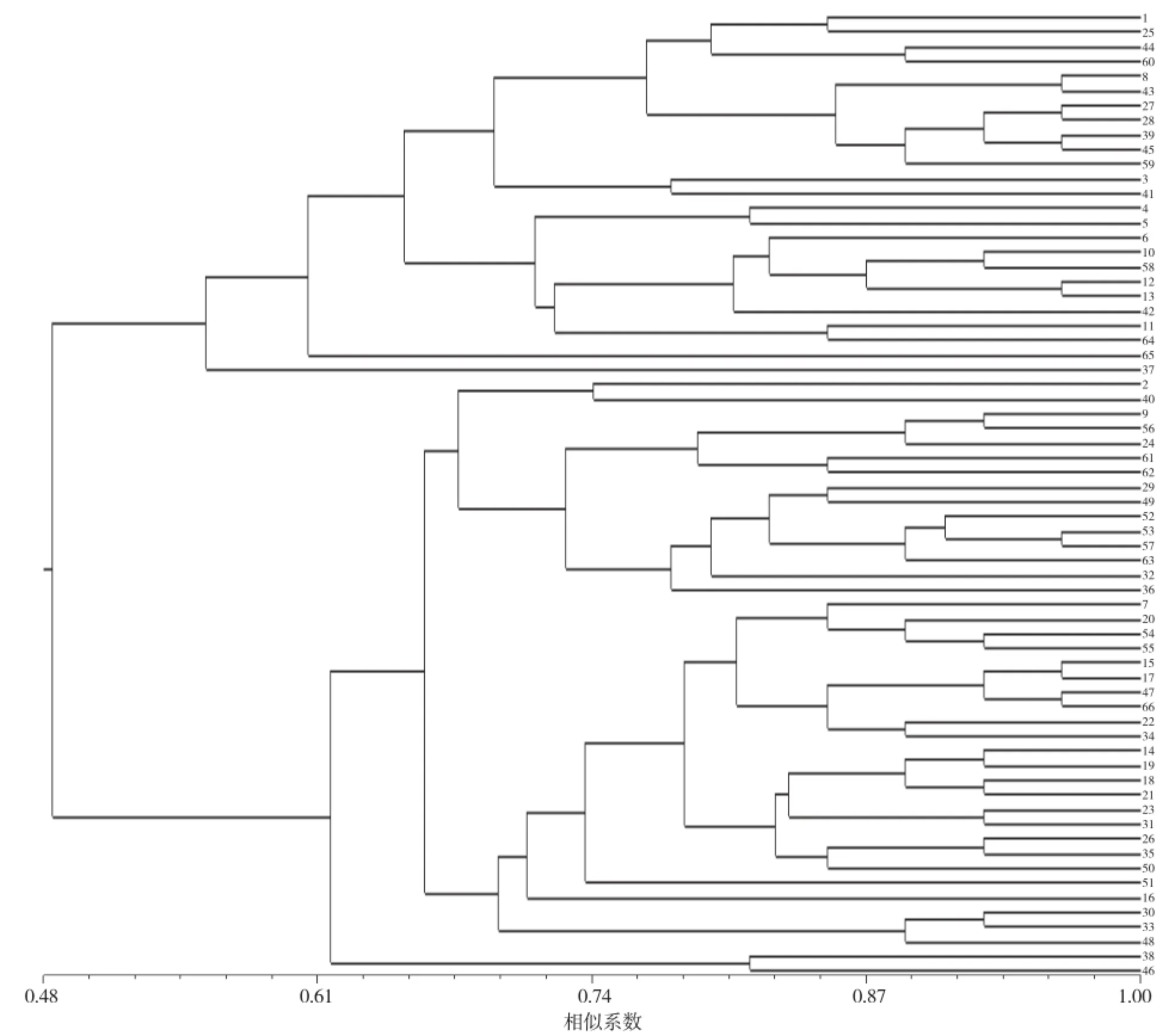
图3 9对引物组合的聚类结果
将9个SSR位点的等位基因按其扩增片段长度从小到大排列,采用“0,1”统计方法,即在同一等位基因上,有带记为“1”,无带记为“0”,基于毛细管电泳结果构建了66个番茄品种的DNA指纹图谱(表3),可作为番茄品种的遗传标签,用于品种的真伪鉴定。

表3 66个番茄品种的DNA指纹
3 结论与讨论
虽然多种DNA标记技术可用于番茄品种的鉴定研究(朱海山 等,2003;苏永涛 等,2010;刘淑君 等,2011),但是要在实际检测中得到广泛应用,选用的标记技术必须具备多态性丰富、结果稳定、重复性好、电泳谱带容易识别、简便易行且使用成本低等特点。相比较RAPD标记的稳定性差和AFLP技术的繁杂,SSR标记则更适合用于品种的实际检测,因此也被国际植物新品种保护联盟(UPOV)列为DNA指纹建库方法之一。目前SSR标记用于番茄品种鉴定的研究相对较少,主要还是用于品种遗传多样性分析(王日升 等,2006;申璐 等,2011),而且结果分析基本上还是基于普通PAGE胶电泳技术,形成的指纹数据也难以整合(王柏柯 等,2010)。本试验在广泛筛选引物的基础上,通过毛细管电泳技术对筛选的引物特征进行分析,筛选获得的20对引物特异性好、峰型简单易辨认、分型结果稳定重演性强,利用其中的9对引物构成的组合就可以对66个番茄品种进行有效鉴别,同时还构建了66个番茄品种SSR-DNA指纹,为番茄品种快速鉴定奠定了基础。
驯化和育种选择使得栽培番茄的遗传变异急剧变窄(Bai & Lindhout,2007),Archak等(2002)的研究显示印度新近育成的番茄品种遗传基础比早期的窄,申璐等(2011)研究发现我国番茄品种也存在这一问题,而且遗传基础比国外的更窄。本试验中66份供试材料所产生的平均遗传相似系数为0.601,最大的遗传相似系数为0.963,也表现出供试品种的遗传基础接近的特点,这不仅制约着我国番茄新品种的培育,而且加大了当下番茄品种的鉴别工作难度。因此要把9对SSR引物组合应用到番茄品种真实性的实际检测中,在更大范围内进行品种甄别,需要补充新的引物到组合中去,这也是下一步研究的重点。
刘淑君,杨悦俭,叶青静.2011.AFLP技术在番茄品种鉴定中的应用.浙江农业科学,(4):749-753.
申璐,沈火林,柴敏.2011.采用InDel和SSR标记分析番茄品种基因组DNA多态性.中国农业大学学报,16(2):34-42.
宋建,陈杰,陈火英.2006.利用SSR 分子标记分析番茄的遗传多样性.上海交通大学学报:农业科学版,24(6):524-528.
苏永涛,刘杨,庄天明.2010.栽培番茄品种指纹图谱的AFLP分析.中国蔬菜,(18):34-39.
王柏柯,余庆辉,杨生保.2010.4个加工番茄新品种SSR指纹图谱的构建与品种鉴定.新疆农业科学,47(7):1474-1478.
王日升,李杨瑞,杨丽涛.2006.番茄栽培品种SSR 标记和形态标记的遗传多样性分析.热带亚热带植物学报,14(2):120-125.
朱海山,张宏,毛昆明.2003.RAPD标记鉴定番茄杂交种子纯度的研究.云南农业大学学报,18(3):270-272.
Archak S,Karihaloo J L,Jain A.2002.RAPD markers reveal narrowing genetic base of Indian tomato cultivars.Current Science,82:1139-1143.
Bai Y L,Lindhout P.2007.Domestication and breeding of tomatoes:what have we gained and what can we gain in the future? Annals of Botany,100(5):1085-1094.
Benor S,Zhang M Y,Wang Z F,Zhang H S.2008.Assessment of genetic variation in tomato(Solanum lycopersicum L.)inbred lines using SSR molecular markers.Genet Genomics,35(6):373-379.
Suliman P S,Kashkush K,Shas H.2002.Generation and mapping of AFLP,SSRs and SNPs in Lycopersicon esculentum.Cell Mol Biol Lett,7(2A):593-597.
Studies on Tomato Variety Identification Technology Using SSR Marker
ZHAO Hai-yan,WU Ming-sheng*,SONG-Ge,NIU-Qian,TIAN Hui-fang
(Beijing Seed Administration Station,Beijing 100088,China)
Taking 16 tomato hybrid varieties as material, 20 pairs of primer suited for tomato variety identification were screened from 200 pairs of SSR primer, and 66 tomato varieties were analyzed. 52 allelic gene variations were identified among these varieties. The allelic gene variation was in the range of 2-5, with an average of 2.6 allelic gene variation each locus. The cultivar identification ability of primer combination was analyzed by genetic clustering method. The results showed that the 66 tomato varieties could be efficiently identified by only 9 pairs of SSR primer. The SSR-DNA fingerprinting constructed with these 9 pairs of primer combination could be used as genetic tag for variety authenticity identification.
Tomato;SSR;Variety identification; Fingerprinting
赵海艳,女,硕士,农艺师,专业方向:种子质量检测,E-mail:zhaoh_yan@163.com
*通讯作者(Corresponding author):吴明生,男,高级农艺师,专业方向:种子质量检测,E-mail:chpwcx2010@sina.com
2014-06-06;接受日期:2014-11-15
北京市科技计划项目(D131100000413001)
