太平洋牡蛎酪氨酸酶基因家族的系统发生分析
2014-11-17于雪于红孔令锋李琪
于雪,于红,孔令锋,李琪
中国海洋大学海水养殖教育部重点实验室,青岛 266003
酪氨酸酶是含铜金属酶,广泛分布于微生物、动物、植物及人体中,是黑色素生物合成的关键酶,可以催化两个反应:催化L-酪氨酸羟基化转变为L-多巴和氧化 L-多巴形成多巴醌,多巴醌经过一系列反应后形成黑色素[1,2]。黑色素合成在生物体中具有重要的生理功能,人类遗传病如白化病、白癜风、耳聋都与黑色素合成中的一个或多个相关基因的突变有关[3]。酪氨酸酶在无脊椎动物先天免疫防御中也起着重要的作用[4,5]。酪氨酸酶还参与昆虫蜕皮后角质的硬化以及软体动物卵囊、足丝、角质层的形成等多种生理反应[6~10]。
目前,软体动物中关于酪氨酸酶的研究主要集中在基因克隆、分离和酶活性分析等方面[8,9,12~15],关于酪氨酸酶进化方面的分析较少。通过对后生动物酪氨酸酶的进化分析,根据系统进化树拓扑结构,可将酪氨酸酶分为刺胞动物和原肢类酪氨酸酶、头索和半索动物酪氨酸酶类似蛋白、脊索动物酪氨酸酶家族和酪氨酸酶相关蛋白[2]。大部分后生动物中,不同物种的酪氨酸酶和酪氨酸酶类似蛋白存在独立的基因扩张现象[2]。Felipe等[16]根据组氨酸排列顺序将酪氨酸酶分为a亚型、b亚型、g亚型,并认为a亚型是最原始类型。然后经过两次复制:第一次复制,早于单纤毛真核生物的分化,分化出了b亚型即胞内型; 第二次复制,早于动物的分化,分化出g亚型即跨膜结构域型。分化出的多细胞真核生物中有酪氨酸酶亚型缺失现象,即一种或两种亚型缺失,留下的亚型进行线性扩张[16]。以上均是针对不同物种酪氨酸酶基因家族的系统进化研究,但是对某个物种中线性扩张的酪氨酸酶基因家族的研究还未见报道。例如,合浦珠母贝(PinctadafucataGould)中已发现并克隆了3个酪氨酸酶基因[8,9],但这些酪氨酸酶的类型以及与其他物种酪氨酸酶的进化关系等问题仍然有待探索。
太平洋牡蛎(Crassostrea gigasThunberg)亦称长牡蛎,隶属软体动物门双壳纲(Bivalvia),是世界上养殖范围最广、养殖产量最大的经济贝类。根据FAO统计,2011年太平洋牡蛎世界总产量达到63.7万吨(http://www.fao.org/fishery/species/3514/en)。2012 年,太平洋牡蛎基因组测序完成,共发现26个酪氨酸酶基因,存在基因扩张现象[11]。
本研究对相关数据库中的太平洋牡蛎和其他一些物种的酪氨酸酶序列进行收集整理、同源比对以及聚类分析,探讨了太平洋牡蛎酪氨酸酶基因家族的分类、演化,旨在为太平洋牡蛎酪氨酸酶基因家族的理论研究和实际应用提供基础资料,为软体动物酪氨酸酶家族的深入研究奠定基础。
1 材料和方法
1.1 酪氨酸酶氨基酸序列的获取
太平洋牡蛎酪氨酸酶 tyr19 基因序列已由本实验室克隆获得(于雪,未发表),其余酪氨酸酶基因序列从太平洋牡蛎基因组数据库(http://www.oysterdb.cn/)下载获得。合浦珠母贝酪氨酸酶基因序列从其基因组数据库(http://marinegenomics.oist.jp/genomes/)下载获得,其他分别隶属于脊索动物门、软体动物门、环节动物门、扁形动物门、线虫动物门和腔肠动物门的17个物种的酪氨酸酶从NCBI下载获得,相关信息见表1。

表1 序列来源及登录号
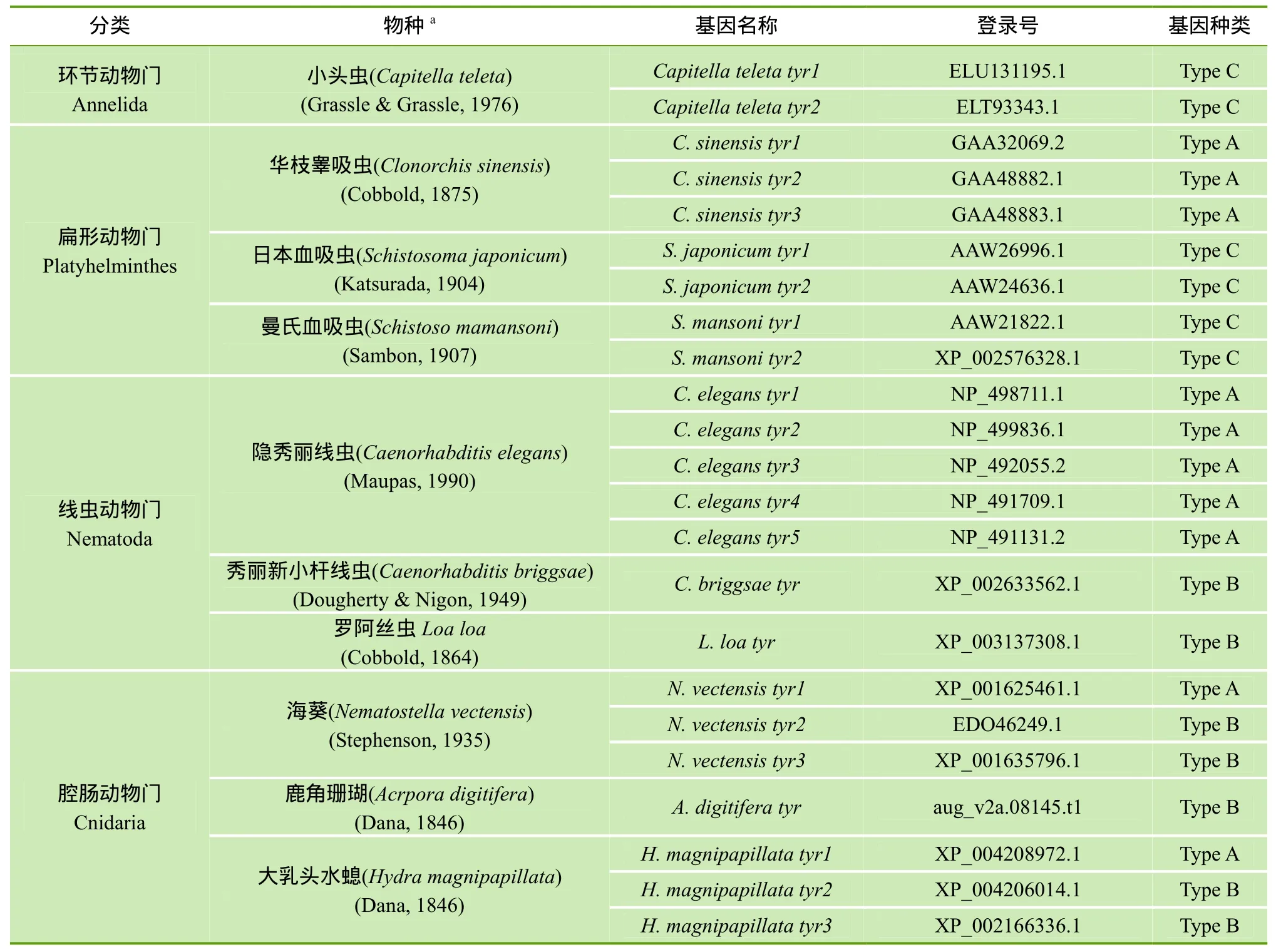
续表1
1.2 太平洋牡蛎酪氨酸酶基因结构分析
太平洋牡蛎酪氨酸酶所在Scaffold、基因组位置等信息从太平洋牡蛎基因组序列数据库(http://www.oysterdb.cn/)获得。
1.3 太平洋牡蛎酪氨酸酶基因氨基酸序列分析
使用 SMART软件(http://smart.embl-heidelberg.de/)分析并选取酪氨酸酶的保守结构域; 用 SignalP 4.0 和 Cholorop 1.1[17,18]对酪氨酸酶信号肽进行预测; 用TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测酪氨酸酶跨膜结构域; 用 ClustalX 1.83软件[19]对太平洋牡蛎酪氨酸酶的保守结构域进行多重比对分析。
1.4 太平洋牡蛎酪氨酸酶基因的进化分析
太平洋牡蛎酪氨酸酶的保守结构域的多重比对在 ClustalX1.83[19]中进行,利用 MEGA5.0[20]采用邻接法(Neighbor-joining method,NJ)并自举(Bootstrap)1000次计算置信值。不同物种酪氨酸酶的保守结构域序列的多重比对也在 ClustalX1.83 中进行。用MrBayes 3.2[21]构建贝叶斯系统发生树。
2 结果与分析
2.1 太平洋牡蛎酪氨酸酶基因所在Scaffold分析
通过太平洋牡蛎基因组数据分析,获得具有较完整酪氨酸酶结构域的酪氨酸酶基因21个,分布于14个Scaffold上(图1)。其中,Scaffold 867上分布的酪氨酸酶基因最多,为4个; Scaffold43702有3个;Scaffold552和Scaffold337各有2个; Scaffold248、Scaffold203、Scaffold483、Scaffold552、Scaffold557、Scaffold612、Scaffold1155、Scaffold1220、Scaffold1719、Scaffold1891和 Scaffold4314均含有一个酪氨酸酶基因。

图1 太平洋牡蛎酪氨酸酸酶基因家族所在Scaffold分析(数字代表两个基因间的距离)
2.2 太平洋牡蛎酪氨酸酶的分类与保守区域分析
所有物种的酪氨酸酶都具有酪氨酸酶保守结构域,酪氨酸酶保守结构域都包含两个保守铜离子结构域Cu(A) 和 Cu(B)。太平洋牡蛎酪氨酸酶基因家族中,包括3种类型的酪氨酸酶。tyr5、tyr11、tyr12、tyr17、tyr18为分泌型(Type A); tyr1、tyr2、tyr3、tyr4、tyr9、tyr13、tyr14、tyr15、tyr16、tyr20、tyr21 为胞内型(Type B)。tyr6、tyr7、tyr8、tyr10、tyr19 为具跨膜结构域型酪氨酸酶(Type C)。太平洋牡蛎酪氨酸酶保守结构域序列比对分析表明,除 tyr9外其他酪氨酸酶都包含6个保守的组氨酸(图2)。
2.3 太平洋牡蛎酪氨酸酶的系统进化
太平洋牡蛎酪氨酸酶的系统进化树分析见图 3,其酪氨酸酶基因为多拷贝基因,21个酪氨酸酶基因中有9对同源基因存在基因重复现象(tyr1和tyr21,tyr2和tyr9,tyr3和tyr13,tyr4和tyr5,tyr7和tyr10,tyr11和tyr14,tyr12和tyr17,tyr19和tyr20),其中4对同源基因(tyr12和tyr17,tyr11和tyr14,tyr4和tyr5,tyr19和tyr20)属于基因内复制,位于相同的Scaffold,其余5对同源基因(tyr1和tyr21,tyr2和tyr9,tyr3和tyr13,tyr7和 tyr10,tyr15和 tyr16)均位于不同的Scaffold,可能属于基因间的重复。

图2 太平洋牡蛎酪氨酸酶Cu(A)和Cu(B)结合域的比对

太平洋牡蛎分泌型酪氨酸酶的系统进化树分析见图 4。太平洋牡蛎所有分泌型酪氨酸酶首先与头足类分泌型酪氨酸酶聚在一起,然后与隐秀丽线虫分泌型酪氨酸酶聚在一起。海葵和水螅分泌型酪氨酸酶单独聚为一支,与太平洋牡蛎、头足类、隐秀丽线虫分泌型酪氨酸酶分化较大。
太平洋牡蛎胞内型酪氨酸酶 tyr1、tyr2、tyr9、tyr15、tyr16与合浦珠母贝胞内型酪氨酸酶聚为一支,与鹿角珊瑚、舌囊虫、大乳头水螅和海葵胞内型酪氨酸酶分化较大(图 5A)。太平洋牡蛎胞内型酪氨酸酶tyr3、tyr11、tyr13和tyr14与罗阿丝虫、秀丽新小杆线虫胞内型酪氨酸酶聚为一支,与华枝睾吸虫、文昌鱼、海葵和大乳头水螅的胞内型酪氨酸酶分化较大(图5B)。
太平洋牡蛎具跨膜结构域型酪氨酸酶 tyr6、tyr7、tyr8、tyr10、tyr19与合浦珠母贝具跨膜结构域型酪氨酸酶聚在一起,与其他具有跨膜结构域型酪氨酸酶种类分化较大(图6)。

图3 太平洋牡蛎酪氨酸酶基因家族的系统进化树

图4 分泌型酪氨酸酶系统进化树

3 讨 论
Felipe等[17]按照组氨酸排列将酪氨酸酶分成 α亚型、β亚型、γ亚型,并认为α亚型全部含有信号肽,为分泌型。通过研究太平洋牡蛎酪氨酸酶基因家族发现,虽然太平洋牡蛎酪氨酸酶在组氨酸排列上属于 α亚型,但类型多样,不仅包括具信号肽的分泌型酪氨酸酶,还包括胞内型酪氨酸酶和具跨膜结构域型酪氨酸酶,说明 α亚型酪氨酸酶结构的复杂性。Sugumaran[22]认为无脊椎动物中的酪氨酸酶缺乏跨膜结构域。但是通过分析发现环节动物门,扁形动物门和软体动物门的某些酪氨酸酶均含有跨膜结构域。本研究说明无脊椎动物酪氨酸酶的多样性及复杂性。太平洋牡蛎和合浦珠母贝具跨膜结构域型酪氨酸酶保守结构域的所在位置不同于脊索动物门、环节动物门和扁形动物门具跨膜结构域型酪氨酸酶。这些物种的跨膜结构域分布在 C末端,而太平洋牡蛎及合浦珠母贝的跨膜结构域分布在N末端与信号肽重叠,而且有些类型只具有跨膜结构域而没有信号肽。这可能导致了太平洋牡蛎以及合浦珠母贝具跨膜结构域类型与脊索动物门,环节动物门和扁形动物门具跨膜结构域型酪氨酸酶的分化较大,使得太平洋牡蛎和合浦珠母贝具跨膜结构域酪氨酸酶在进化中构成一独立分支。
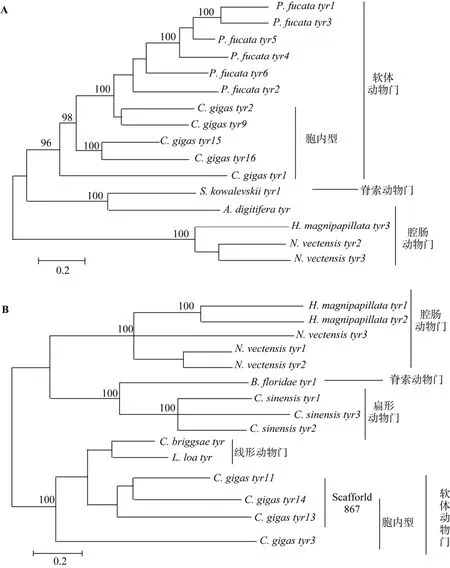
图5 胞内型酪氨酸酶系统进化树

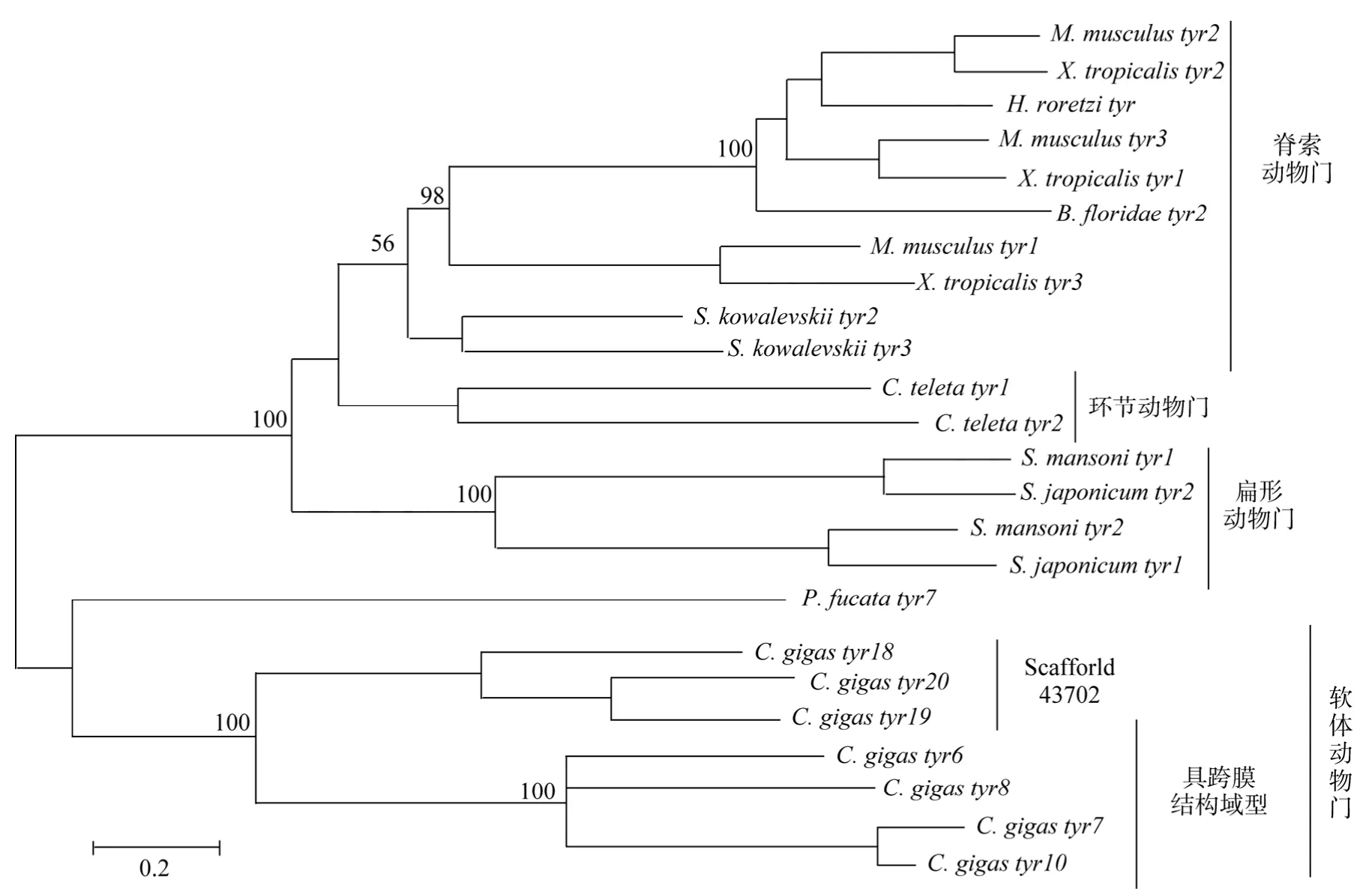
图6 具跨膜结构域型酪氨酸酶系统进化树
通过系统发生分析发现,太平洋牡蛎酪氨酸酶进化树上的分布不仅受保守结构域影响还受基因所在位置影响,如tyr4、tyr21并非分泌型酪氨酸酶,但tyr4与tyr5同位于Scaffold 552,tyr21与tyr17同位于Scaffold 337,仍与太平洋牡蛎分泌型酪氨酸酶聚在一起; tyr11并非太平洋牡蛎胞内型酪氨酸酸酶,但与 tyr13、tyr14同位于scaffold 867,在进化树上与太平洋牡蛎胞内型酪氨酸酶聚在一起; 太平洋牡蛎 tyr18、tyr20 并非太平洋牡蛎具跨膜结构域型酪氨酸酸酶,但与tyr19同位于scaffold 43702,在进化树上与太平洋牡蛎具跨膜结构域型酪氨酸酶聚在一起。以上结果说明酪氨酸酸酶基因所在位置在一定程度上影响了酪氨酸酶保守结构域的分化,也说明了太平洋牡蛎酪氨酸酶基因分化的复杂性,存在基因内和基因间复制。
基因重复是基因家族数量增加的主要方式,太平洋牡蛎酪氨酸酶基因家族中发现9对基因重复现象,说明基因重复是太平洋牡蛎酪氨酸酶基因扩增的一个因素,同时也有力证实了基因重复是基因组合遗传多样化的重要来源[23]。太平洋牡蛎酪氨酸酶的基因重复伴随着结构分化,支持了结构分化在基因复制中的普遍性[24]。根据太平洋牡蛎酪氨酸酶进化树分析,发现Type A酪氨酸中,tyr18与其他Type A酪氨酸酶分化较大,可能是较早分化出来的 Type A酪氨酸酶。Type B酪氨酸酶中,tyr2、tyr9为较早分化出的Type B酪氨酸酶。Type C型酪氨酸中tyr8较早分化出来,而且 Type C型酪氨酸酶与 Type A型、Type B型酪氨酸酶分化较大,沿着较独立的方向进行基因扩张。其中最早分化的酪氨酸酶 tyr2、tyr8、tyr9、tyr18可能参与比较基础的代谢,如软体动物先天性免疫、黑色素形成。随着基因的再次分化、扩张,预示着这些基因可能具有特化的新功能,比如参与软体动物卵囊、足丝、角质层的形成等多种特化的生理反应[8~10]。太平洋牡蛎酪氨酸酶基因家族的扩张可能与酪氨酸酶在太平洋牡蛎功能的分化密切联系。
[1]Ito S,Wakamatsu K,Ozeki H.Chemical analysis of melanins and its application to the study of the regulation of melanogenesis.Pigm Cell Res,2000,13(Suppl 8):103–109.
[2]Esposito R,D’Aniello S,Squarzoni P,Pezzotti MR,Ristoratore F,Spagnuolo A.New insights into the evolution of Metazoan tyrosinase gene family.PLoS ONE,2012,7(4):E35731.
[3]Goding GR.Melanocytes:the new black.Int J Biochem Cell Biol,2007,39(2):275–279.
[4]Gillespie JP,Kanost MR,Trenczek T.Biological mediators ofinsect immunity.Ann Rev Entool,1997,42:611–643.
[5]Luna-Acosta A,Thomas-Guyon H,Amari M,Rosenfeld E,Bustamante P,Fruitier-Arnaudin I.Differential tissue distribution and specificity of phenoloxidases from the Pacific oysterCrassostrea gigas.CompBiochemPhysi,2011,159B:220–226.
[6]Sugumaran M.Unified mechanism for sclerotization of insect cuticle.Adv Insect Physiol,1998,27:229–334.
[7]Sugumaran M.Molecular mechanisms for mammalian melanogenesis comparison with insect cuticular sclerotization(minireview).FEBS Lett,1991,293(1-2):4–10.
[8]Kouhei N,Masato Y,Koichi M,Hiroshi M.Tyrosinase localization in mollusc shells.Comp Biochem Physio B Biochem Mol Biol,2007,146:207–214.
[9]Zhang C,Xie LP,Huang J,Chen L,Zhang RQ.A novel putative tyrosinase involved in periostracum formation from the pearl oyster(Pinctada fucata).BiochemBiopyhs Res Commun,2006,342(2):632–639.
[10]Zhou Z,Ni DJ,Wang MQ,Wang LL,Shi XW,Yue F,Liu R,Song LS.The phenoloxidase activity and antibacterial function of a tyrosinase from scallopChlamys farreri.Fish Shellfish Immunology,2012,33(2):375–381.
[11]Zhang G,Fang X,Guo X,Li L,Luo R,Xu F,Yang P,Zhang L,Wang X,Qi H,Xiong Z,Que H,Xie Y,Holland PW,Paps J,Zhu Y,Wu F,Chen Y,Wang J,Peng C,Meng J,Yang L,Liu J,Wen B,Zhang N,Huang Z,Zhu Q,Feng Y,Mount A,Hedgecock D,Xu Z,Liu Y,Domazet-Loso T,Du Y,Sun X,Zhang S,Liu B,Cheng P,Jiang X,Li J,Fan D,Wang W,Fu W,Wang T,Wang B,Zhang J,Peng Z,Li Y,Li N,Wang J,Chen M,He Y,Tan F,Song X,Zheng Q,Huang R,Yang H,Du X,Chen L,Yang M,Gaffney PM,Wang S,Luo L,She Z,Ming Y,Huang W,Zhang S,Huang B,Zhang Y,Qu T,Ni P,Miao G,Wang J,Wang Q,Steinberg CE,Wang H,Li N,Qian L,Zhang G,Li Y,Yang H,Liu X,Wang J,Yin Y,Wang J.The oyster genome reveals stress adaptation and complexity of shell formation.Nature,2012,490:49–54.
[12]Cong R,Sun W,Liu G,Fan T,Meng X,Yang L,Zhu L.Purification and characterization of phenoloxidase from clamRuditapes philippinarum.Fish Shellfish Immunol,2005,18(1):61–70.
[13]Jiang JW,Xing J,Sheng XZ,Zhan WB.Characterization of phenoloxidase from the bay scallopArgopecten irradians.J Shellfish Res,2011,30(2):273–277.
[14]Aladaileh S,Rodney P,Nair SV,Raftos DA.Characterization of phenoloxidase activity in Sydney rock oysters(Saccostrea glomerata).CompBiochemPhysiol,2007,148(4):470–480.
[15]Luna-Gonza´leza A,Maeda-Martíneza AN,Vargas-Alboresb F,Ascencio-Vallea F,Robles-Mungaraya M.Phenoloxidase activity in larval and juvenile homogenates and adult plasma and haemocytes of bivalve molluscs.Fish Shellfish Immunol,2003,15(4):275–282.
[16]Aguilera F,McDougall C,Degnan MB.Origin,evolution and classification of type-3 copper proteins:lineage-specific gene expansions and losses across the Metazoa.BMC Evol Biol,2013,13:96.
[17]Petersen TN,Brunak S,Von Heijne G,Nielsen H.SignalP 4.0:discriminating signal peptides from transmembrane regions.Nat Meth,2011,8(10):785–786.
[18]Emanuelsson O,Nielsen H,von Heijne G.ChloroP.A neural network-based method for predicting chloroplast transit peptides and their cleavage sites.Protein Sci,1999,8(5):978–984.
[19]Larkin MA,Blackshields G,Brown NP,Chenna R,McGettigan PA,McWilliam H,Valentin F,Wallace IM,Wilm A,Lopez R,Thomps JD,Gibson TJ,Higgins DG.Clustal W and Clustal X version 2.0.Bioinformatics,2007,23(21):2794–2948.
[20]Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance and maximum parsimony methods.Mol Biol Evol,2011,28(10):2731–2739.
[21]Ronquist F,Teslenko M,van der Mark P,Ayres DL,Darling A,Hohna S,Larget B,LiuL,Suchard MA,Huelsenbeck JP.MrBayes 3.2:efficient bayesian phylogenetic inference and model choice across a large model space.Syst Biol,2012,61(3):539–542.
[22]Sugumaran M.Comparative biochemistry of eumelanogenesis and the protective roles of phenoloxidase and melanin in insects.Pigment Cell Res,2002,15(1):2–9.
[23]Lespinet O,Wolf IY,Koonin EV,Aravind L.The role of lineage-specific gene family expansion in the evolution of eukaryotes.Genome Res,2002,12:1048–1059.
[24]Xu G,Guo C,Shan H,Kong H.Divergence of duplicate genes in exon-intron structure.Proc Natl Acad Sci USA,2012,109(4):1187–1192.
