裂殖酵母SAGA各亚基亚细胞荧光定位分析
2014-11-17周幸周楠余垚吕红
周幸,周楠,余垚,吕红
复旦大学生命科学学院,遗传工程国家重点实验室,上海 200433
SAGA(Spt-Ada-Gcn5 Acetyltransferase complex)是一个多亚基的复合物,在裂殖酵母(Schizosaccharomyces pombe)、酿酒酵母(Saccharomy cescerevisiae)、果蝇(Drosophila melanogaster)、小鼠(Mus musculus)以及人(Homo sapiens)等真核生物中都保守,主要负责体内10%的基因转录[1,2]。在裂殖酵母里,SAGA由19个亚基组成,可分为4个功能模块:乙酰化功能模块(Gcn5、Ada2、Ngg1、Sgf29),负责组蛋白的乙酰化; 去泛素化功能模块(Ubp8、Sgf73、Sgf11、Sus1),负责组蛋白的去泛素化; SPT模块(Spt3、Spt7、Spt8、Spt20、Hfi1、Tra1)以及 TAF模块(Taf5、Taf6、Taf9、Taf10、Taf12),这两个模块的亚基是 TATA BOX的相关蛋白,负责染色质重塑和募集其他转录因子到启动子区域,参与基因的转录调控[3,4]。
SAGA可以通过组蛋白乙酰化、组蛋白去泛素化、染色质重塑等方式参与基因的调控,在细胞的不同生理阶段发挥功能。已有研究表明,SAGA中spt8+的缺失影响裂殖酵母隔膜降解途径中关键基因ace2+的转录,从而造成胞质分裂缺陷[5]; Gcn5、Spt8拮抗作用于酵母有性生殖基因ste11+的转录,调控酵母的繁殖方式[6]; hGcn5与TRRAP形成二聚体,激活哺乳动物细胞c-Myc的转录,而Mad抑制其转录,该平衡若被打破会导致过度激活 c-Myc,进而引发癌症[7]; 去泛素化模块的亚基Sus1具有不依赖转录的功能,它直接参与了酿酒酵母mRNA的出核调控[8]。在裂殖酵母里,对于复合物亚基的研究,荧光定位分析是阐述各个蛋白生物学功能直观又有效的手段,因此获得完整的 SAGA各亚基整合荧光定位信息对进一步研究SAGA的功能具有重要意义。此前,Hayashi等[9]构建了1 058个裂殖酵母基因的GFP整合荧光菌株,包括部分SAGA成员,但缺少其中9个亚基的定位信息; Matsuyama等[10]利用过量表达荧光质粒的方式检测裂殖酵母所有蛋白各自的亚细胞定位信息,但仍缺少SAGA中3个亚基的定位信息,且过量表达荧光质粒的定位信息可能与内源性表达蛋白的真实定位存在一定的差异。因此,本研究利用同源重组的方法,在文献[9]的基础上补充了 9个SAGA亚基的整合荧光菌株(Ada2、Sgf29、Sgf73、Sus1、Sgf11、Hfi1、Spt8、Tra1、Taf10),全面分析了内源性表达的19个SAGA亚基的整合荧光定位。
Sgf73是 SAGA的核心亚基,在不同物种中都同源保守[11]。Sgf73负责将去泛素化核心亚基Ubp8连接到SAGA核心模块(SPT模块、TAF模块),且自身与SAGA亚基Ubp8、Sgf11、Sus1形成一个去泛素化亚复合物[12]。已有研究表明,缺失sgf73+会造成多种影响:Ubp8失去了去泛素化功能[12]; Sus1、TREX-II复合物调控 mRNA出核的功能受到影响,并且Sus1有出核的异常现象[12]; PIC复合物(Preinitiation complex)的组装不稳定使基因的复制起始受影响[13]。不仅如此,在对人的研究中,Sgf73的同源蛋白 ATAXIN-7的变异造成了神经退行性疾病,并影响视网膜的发育[14]。这些研究都表明sgf73+的保守性和重要性。缺失sgf73+对 Ubp8、Sus1之外的SAGA亚基是否也有影响还不清楚,因此,本研究在所有 SAGA亚基的整合荧光菌株中分别敲除了sgf73+,通过检测荧光定位的方式首次系统分析了sgf73+的缺失对SAGA各亚基的定位影响。
在分析荧光定位时,本研究发现sgf73+的缺失还造成了胞质分裂的缺陷,表现为隔膜降解受影响。为了进一步分析造成这种缺陷的原因,本研究通过功能回补实验,分析了胞质分裂过程中与膈膜降解相关基因ace2+、mid2+在过量表达的条件下回补△sgf73缺陷的能力,提示Sgf73可能参与了多条影响胞质分裂的途径。
1 材料和方法
1.1 材料
1.1.1 菌株与质粒
本研究使用的原始质粒 pCHGL-GFP、pFAMLRFP、pREP42-URA4、pLEPF-LEU2,大肠杆菌(Escherichia coli)DH5α菌株、Top10菌株均为本实验室长期保存,所用到的裂殖酵母菌株见表1。
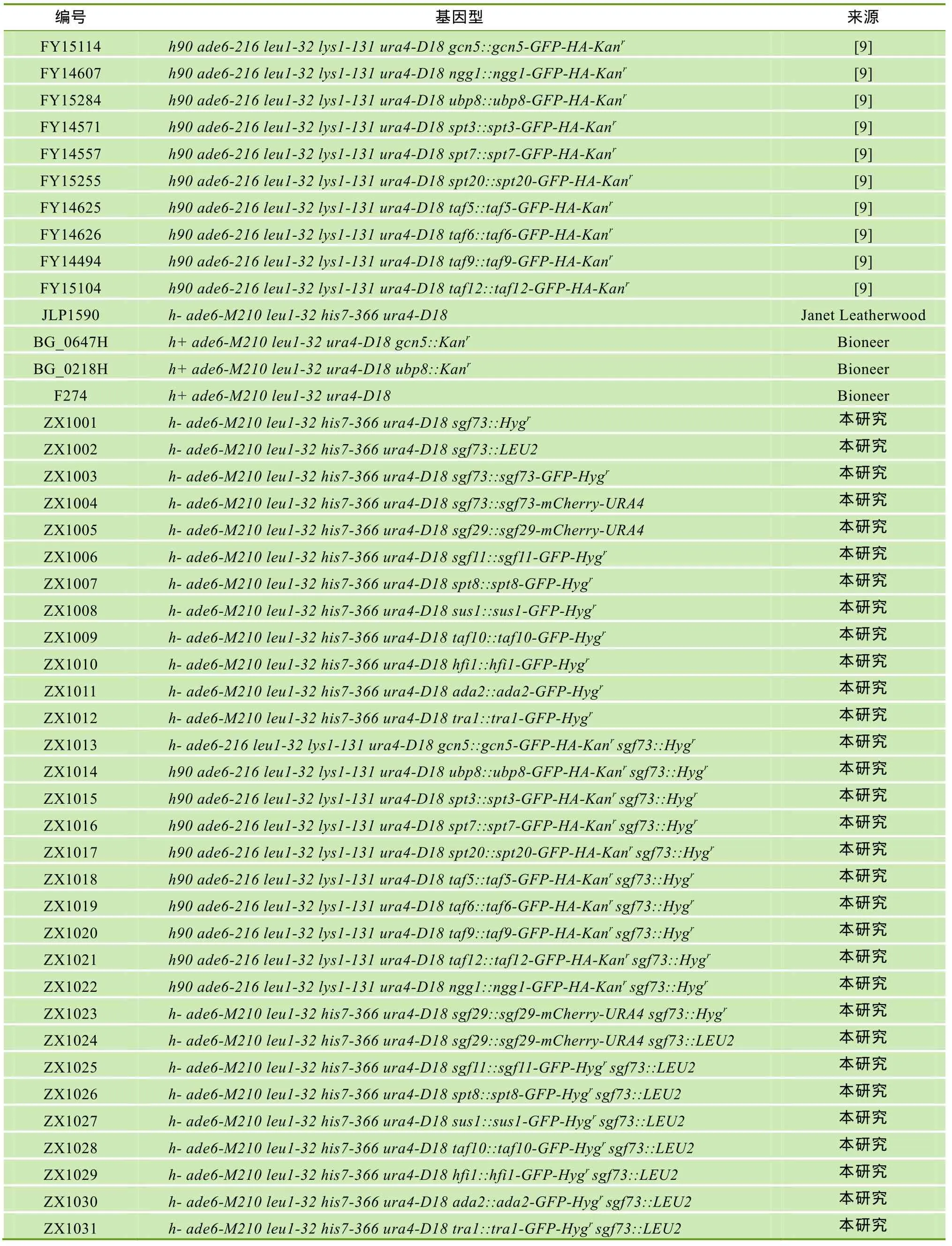
表1 本研究使用的裂殖酵母菌株列表
1.2 方法
1.2.1 裂殖酵母荧光菌株、缺失突变体菌株的构建与验证
裂殖酵母荧光菌株、缺失突变体菌株的构建都采用同源重组的方法[15],菌株构建均以sgf73+为例。在荧光菌株构建过程中(图1A),分别选取大于300 bp的终止密码子上游(Up)和下游(Down)片段,设计带有酶切位点的上下游引物 U-F/U-R、D-F/D-R(上游HindⅢ/BglⅡ,下游SacⅠ/SpeⅠ)。当上下游片段都连接到质粒载体上后,通过HindⅢ/SpeⅠ双酶切获得线性质粒片段,利用酵母转化使片段与宿主菌的基因组产生同源重组。挑取转化后的单克隆菌落,抽取基因组 DNA(gDNA)利用验证引物JD-U-F/ JD-U-R、JD-D-F/JD-D-R进行PCR验证,若两对验证引物能扩增出条带,则菌株构建成功。在缺失突变菌株的构建过程中(图1B),首先分别选取大于300 bp的ORF(Open reading frame)上下游片段,建步骤与荧光菌株一致,仅在验证时多用一对 ORF内部引物ORF-F/ORF-R进行扩增,若该引物对不能扩增出条带,而JD-U-F/JD-U-R、JD-D-F/JD-D-R引物对能扩增出条带,则菌株构建成功。菌株构建引物序列见表2。
1.2.2 酵母转化
制备感受态:收集处于对数期(OD600值为 1~2)的酵母菌体,总OD600值大于 10,用 10 mL LiAC/TE/PEG4000/ddH2O 混合液(1:1:2:6)重悬,离心后再用550 μL混合液重悬,100 μL每管分装待用。LiAC浓度为1 mol/L,pH值为7.5; TE是0.1 mol/L Tris碱与 0.01 mol/L EDTA混合液; PEG4000的浓度为50%。
酵母转化:加入 10 μL待转化的质粒或线性DNA片段,3 μL煮沸的单链DNA于感受态细胞中,充分打匀后加入 260 μL LiAC/TE/PEG4000(1:1:8),漩涡震荡混匀后30℃水浴45 min,42℃热激15 min,低速(小于 5 000 r/min)离心收集并弃去上清液,加入1 mL 培养液于30℃培养4~6 h,涂布于固体选择培养基上,30℃培养3~4 d。
1.2.3 裂殖酵母基因组DNA提取
收集菌体,加入300 μL苯酚、氯仿、异戊醇混合液(25:24:1),300 μL DNA裂解液,300 μL玻璃珠,涡旋震荡1 min,再加入300 μL 1×TE,上下颠倒数次再涡旋震荡1 min。13 000 r/min离心10 min,将上清转入等体积的氯仿、异戊醇混合液(24:1)中,上下颠倒数次,13 000 r/min离心10 min,将上清转入2倍体积的无水乙醇中,-20℃放置 30 min。13 000 r/min离心5 min,弃去无水乙醇,用800 μL 75%的乙醇洗一次,13 000 r/min离心5 min后弃乙醇并烘干,加入 1×TE溶解基因组 DNA,涡旋震荡1 min后待用。

图1 裂殖酵母荧光菌株、缺失突变体菌株的构建与验证(本研究质粒构建示意图均以sgf73+为例)

表2 构建sgf73+荧光菌株和缺失突变体菌株所需引物
1.2.4 显微检测
收集处于对数期(OD600=1左右)的菌体,用ddH2O重悬并离心,用1×的Hoechst(Sigma)重悬染核,避光静置15 min以上,离心收集菌体,取2 μL镜检。荧光显微镜(型号为奥林巴斯X51)蓝色通道激发绿色整合荧光(曝光时间为 300~500 ms); 黄色通道激发红色整合荧光(曝光时间为 100~200 ms); 多功能通道激发 Hoechst染核荧光(曝光时间为 30~50 ms),DIC(微分干涉)为白光(曝光时间为30 ms)。检测时,若检测到特异性荧光,则背景噪音会很少;若特异性荧光很少或者没有特异性荧光,则会有大量背景噪音。
2 结果与分析
2.1 裂殖酵母SAGA全亚基整合荧光定位分析
本研究对裂殖酵母SAGA的乙酰化功能模块、去泛素化功能模块、SPT模块及TAF模块中各亚基的内源表达荧光定位进行系统分析。
荧光分析乙酰化功能模块(Gcn5、Ada2、Ngg1、Sgf29)各亚基的定位发现,Ada2、Ngg1、Sgf29完全定位于核内(图2A),提示它们仅在核内发挥功能。Gcn5是SAGA中重要的酶学功能亚基之一,具有组蛋白乙酰化修饰功能。结果表明,它在核内和胞浆中都有定位(图2A),胞浆的定位提示Gcn5可能在胞浆中也有乙酰化作用。

图2 乙酰化模块和去泛素化模块中各亚基荧光定位
本研究也分析了去泛素化功能模块(Sus1、Sgf11、Sgf73以及 Ubp8)各亚基的荧光定位。在裂殖酵母里,Sus1和Sgf11的荧光定位信息首次被检测到。其中Sus1不仅定位于核内,在核膜上也能检测到明显的点状定位(图2B)。裂殖酵母Sus1的定位与酿酒酵母一致[8],提示在裂殖酵母里 Sus1可能也参与了mRNA的运输。Sgf11、Sgf73、Ubp8的定位方式一致,大部分定位于核内,同时在胞浆中也有少量荧光定位(图2B)。去泛素化也是SAGA的重要酶学功能,其中 3个亚基有少量胞浆的荧光定位提示SAGA可能在胞浆中也有去泛素化作用。
SPT模块是一类与TATA BOX相关的蛋白,裂殖酵母里由 Spt3、Spt7、Spt8、Spt20、Hfi1以及 Tra1组成。结果表明,Spt3、Spt8、Tra1主要定位于胞浆内,核内分布较少(图 3A),提示 Spt3、Spt8、Tra1可能主要在胞浆中发挥功能。Tra1是SAGA最大的亚基,蛋白大小为422.3 kDa,本研究是首次检测到其荧光定位信息。在检测Spt3-GFP菌株的染核荧光时,发现它呈多核表型(单个细胞内超过2个细胞核,图 3A),而其他亚基的基因在融合 GFP后表型正常(图2,图3)。我们推测这可能是由于GFP序列的插入影响了Spt3的某些功能,使细胞分裂出现缺陷而不能及时分开,但复制过程正常进行。Spt7、Spt20大部分定位于核内,胞浆中有少量定位(图 3A),提示它在胞浆中也可能发挥作用。Hfi1在核内与胞浆中较均匀地分布(图 3A),提示它可能在核内和胞浆中能发挥同等功能。
TAF 模块由 Taf5、Taf6、Taf9、Taf10、Taf12组成,且这5个亚基同时也存在于TFIID复合物[16]。本研究发现,Taf5和 Taf6大部分定位于核内,少量定位于胞浆中(图 3B),提示它们在胞浆中也可能发挥作用。Taf9和Taf10仅定位于核内,说明它们仅在核内发挥作用(图2D)。Taf12类似于Hfi1,在核内与胞浆中较均匀地分布,提示它也可能在核内和胞浆中均等地发挥功能(图3B)。

图3 SPT模块,TAF模块中各亚基荧光定位
本研究将获得的整合荧光定位结果与Matsuyama等[10]利用过量表达荧光质粒的方法所获得的荧光定位进行了对比分析。结果发现,只有Ada2、Spt20、Taf5、Taf6的定位情况与本研究的结果一致,其余均有差异(表3)。这种差异可能是过表达质粒无法准确控制蛋白的表达量,或因为是异位表达,导致某些蛋白的定位不准确。
另外,酿酒酵母 SAGA各亚基整合荧光的定位与本研究获得的裂殖酵母的整合荧光液有明显差异[17]。酿酒酵母除Sus1与裂殖酵母一致外,其余所有亚基都完全定位于核内(表3)。尽管酿酒酵母与裂殖酵母同属子囊菌纲,但两者存在 3~4亿年的进化差异,裂殖酵母更接近于哺乳动物细胞[18],裂殖酵母SAGA出现多种定位情况提示它可能具有特异于酿酒酵母的功能。正如Spt8影响了裂殖酵母的膈膜降解,而酿酒酵母中还没有隔膜的形成。
2.2 缺失sgf73+对SAGA各亚基荧光定位的影响
为了分析sgf73+的缺失对SAGA各亚基的荧光定位的影响,本研究在 SAGA各个亚基的整合荧光菌株中分别敲除了sgf73+。如图4A所示,sgf73+的缺失直接影响并减少了乙酰化功能模块中 Gcn5、Ngg1、Sgf29在核内的荧光定位,提示Sgf73可能参与维持了 SAGA乙酰化功能亚基的稳定性。另外,Gcn5和Sgf29核内荧光减弱甚至消失的现象与哺乳动物细胞中观察到的现象相吻合[14],这进一步支持了我们的结论。但是sgf73+的缺失并没有影响Ada2的核内定位,这说明Sgf73对4个乙酰化亚基的作用不一致。
已有研究表明,去泛素化功能模块缺失sgf73+后,Sus1、Sgf11、Ubp8脱离 SAGA 核心[12],并且Ubp8失去了去泛素化的功能,这与本研究的结果相符。荧光数据表明,Sgf11、Ubp8的核内荧光定位几乎完全消失,Sus1的核内定位也有明减少,尽管Sus1在核孔处定位正常(图 4B)。因此,在裂殖酵母里缺失 Sgf73后不仅影响了 Ubp8、Sgf11、Sus1与SAGA的结合,同时还影响了它们在核内的定位,这说明SAGA去泛素化模块的稳定性依赖于Sgf73。Sus1在核孔处定位正常是否影响了TREX-II复合物的功能还需要进一步研究。

表3 分析比较裂殖酵母与酿酒酵母SAGA所有亚基的荧光定位
SPT模块中Hfi1、Spt7、Spt20是SAGA的核心结构亚基[19],它们受到影响则会极大程度影响SAGA的稳定性。缺失sgf73+后,Spt3、Spt8、Spt20、Hfi1、Tra1的定位没有变化,然而Spt7的核内荧光几乎完全消失(图5A)。这说明SPT模块中只有Spt7的稳定性依赖于 Sgf73,而其他亚基的正常定位并不需要它来维持。由于缺失sgf73+影响了Spt7,提示sgf73+的缺失可能通过影响 Spt7的稳定性从而间接造成了SAGA整体的不稳定。
TAF模块的5个亚基是SAGA中仅有的必需基因,这5个亚基缺失sgf73+后的荧光定位(图5B)与缺失前(图 3B)完全一致,说明缺失sgf73+并不会影响到TAF家族蛋白的定位,对它们的稳定性没有影响。
综上所述,在缺失sgf73+后有 7个 SAGA亚基的荧光定位受到影响,分别是Gcn5、Sgf29、Ngg1、Ubp8、Sgf11、Sus1、Spt7,其余的亚基在缺失前后并没有明显的定位变化(表 4)。这说明尽管这 19个亚基属于同一个复合物,但各亚基与 Sgf73的联系并不一致。
2.3 sgf73+缺失对裂殖酵母细胞形态的影响
在分析缺失sgf73+对各亚基荧光定位的影响时,本研究发现SAGA所有亚基荧光菌株都呈多核表型(细胞核数大于2,图4~5),同时,DIC数据也显示在缺失sgf73+后出现了异常表型。在图6中,缺失sgf73+前为Control组,缺失sgf73+后为Dsgf73组。Control组除文章前面提到Spt3-GFP有异常现象以外,其余都与WT(野生型)表型一致(图6,A~E)。Dsgf73组都呈现多膈膜表型(隔膜数≥2)。尽管本研究发现缺失sgf73+影响了Gcn5和Ubp8的荧光定位(图4,A~B),但gcn5+和ubp8+各自的缺失突变体并没有表现出胞质分裂缺陷,表型与WT一致(结果未发表)。这说明造成sgf73的分裂缺陷与 SAGA酶学活性亚基Gcn5和Ubp8都无关。
2.4 隔膜降解相关基因ace2+、mid2+过表达回补分析
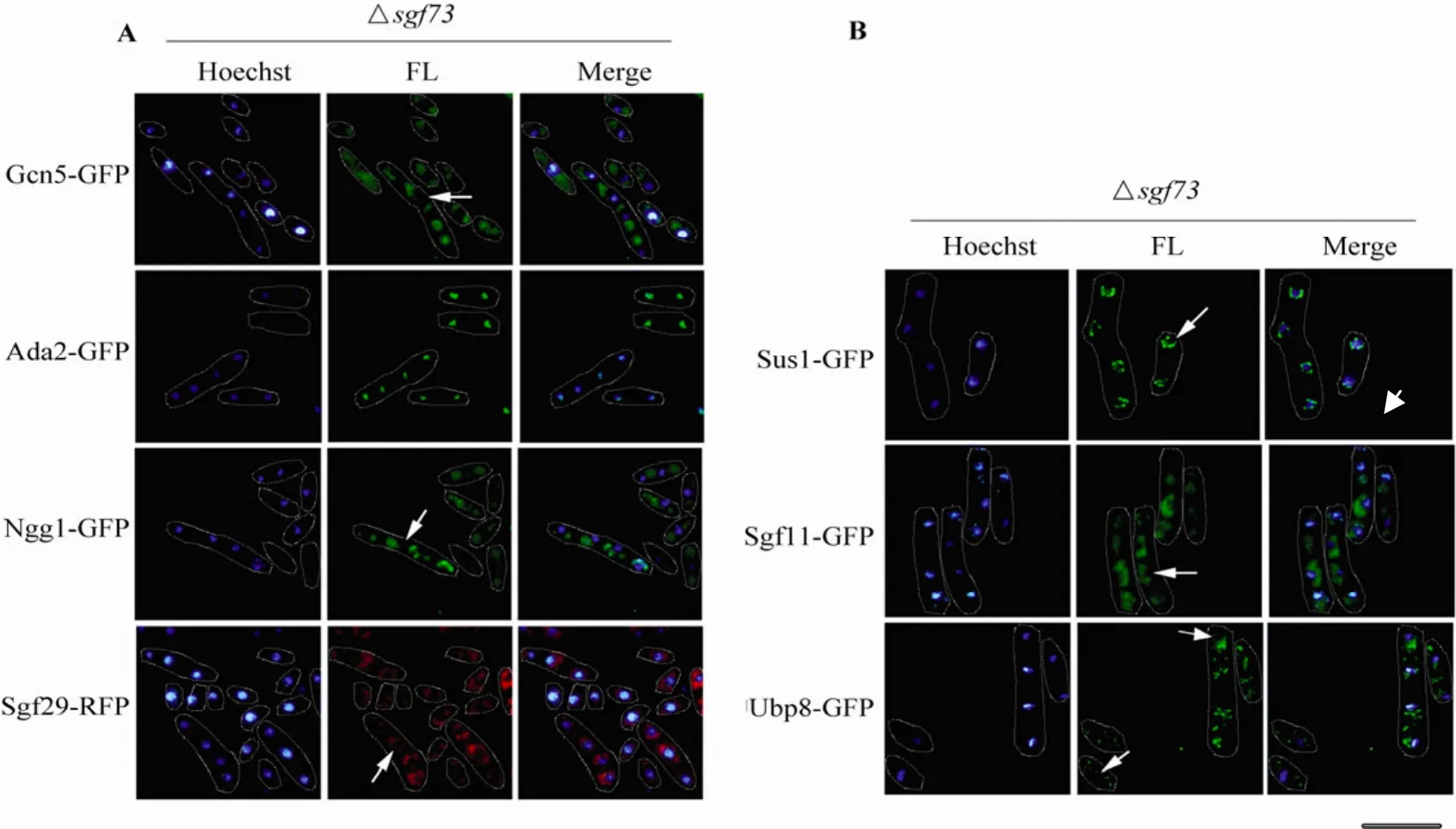
图4 sgf73+的缺失对乙酰化模块和去泛素化模块各亚基荧光定位的影响
为了进一步研究缺失sgf73+引起的多隔膜现象,本研究在sgf73菌株中分别转入过表达质粒pRep42-ace2+、pRep42-mid2+、pRep42-sgf73+,检测在过量表达ace2+、mid2+、sgf73+的情况下能否回补sgf73的胞质分裂缺陷。
去除培养基中的Thaimine后,sgf73+几乎完全回补自身的缺陷,ace2+则完全不能回补,而mid2+能够部分回补该表型(图7)。Helmlinger等[20]的芯片数据显示,△sgf73与△spt8都造成ace2+和mid2+的mRNA 水平下调,而过表达ace2+、mid2+出现回补差异。因此,在△sgf73中可能是Ace2调控Mid2出现异常,导致过量表达ace2+也无法使mid2+处于正常水平。由于ace2+能够完全回补△spt8的缺陷而无法回补△sgf73的缺陷,表明缺失sgf73+和spt8+所造成的胞质分裂缺陷不一致。另外,mid2+也只能部分回补△sgf73的多膈膜表型,说明sgf73+的缺失不仅通过影响mid2+从而造成胞质分裂缺陷,可能还通过其他途径影响了胞质分裂。
3 讨 论
本研究以 Hayashi等[9]和 Matsuyama等[10]的工作为基础,完整地获得了裂殖酵母 SAGA所有亚基的整合荧光定位,其中首次成功地在裂殖酵母里检测到了Sus1、Sgf11以及 Tra1的荧光定位,分析总结了SAGA各亚基的荧光定位类型,并归类为4类:第一类,荧光完全定位于核内,提示这些亚基仅在核内发挥作用,包括 Ada2、Ngg1、Sgf29、Taf9、Taf10;第二类,荧光大部分定位于核内,仅少量定位于胞浆中,包括 Gcn5、Sgf11,Sgf73、Ubp8、Spt7、Spt20、Taf5、Taf6,提示这些亚基可能在胞浆中也发挥功能,但至今尚未有相关报道; 第三类,核内与胞浆中都有分布,且比较均匀,如 Hfi1、Taf12,它们可能在核内与胞浆中有等同的作用,但也无相关报道; 第四类,这些亚基有较特殊的定位,如 Sus1、Tra1、Spt3、Spt8(表3)。这些亚基不同的定位方式体现了SAGA的复杂性与多功能性,但 SAGA的功能还未完全揭开,仅少数亚基有单独的研究,新功能还有待发现。本研究的整合荧光定位信息对今后的功能研究起到一定的提示和参考作用。
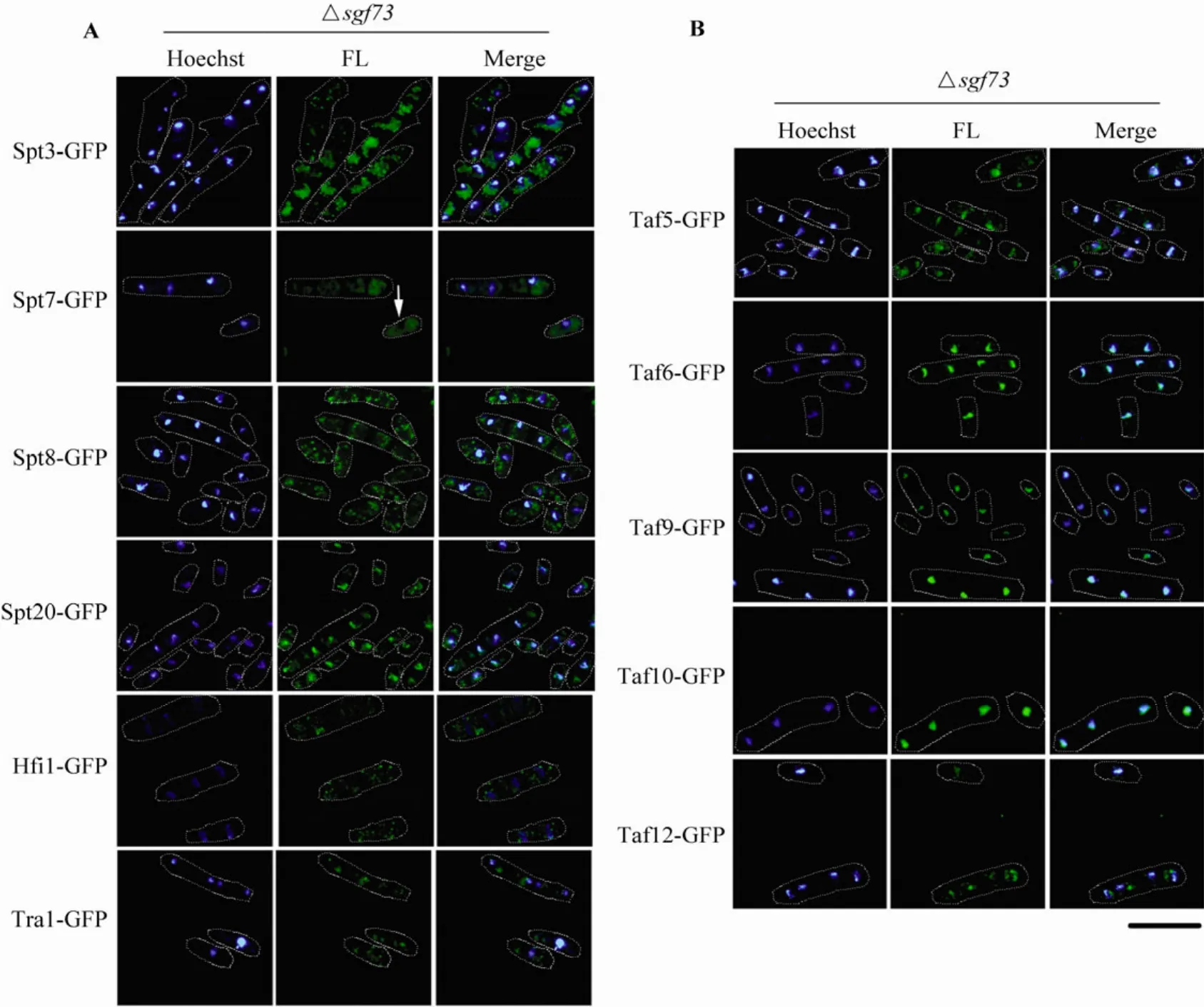
图5 sgf73+的缺失对SPT模块、TAF模块各亚基荧光定位的影响
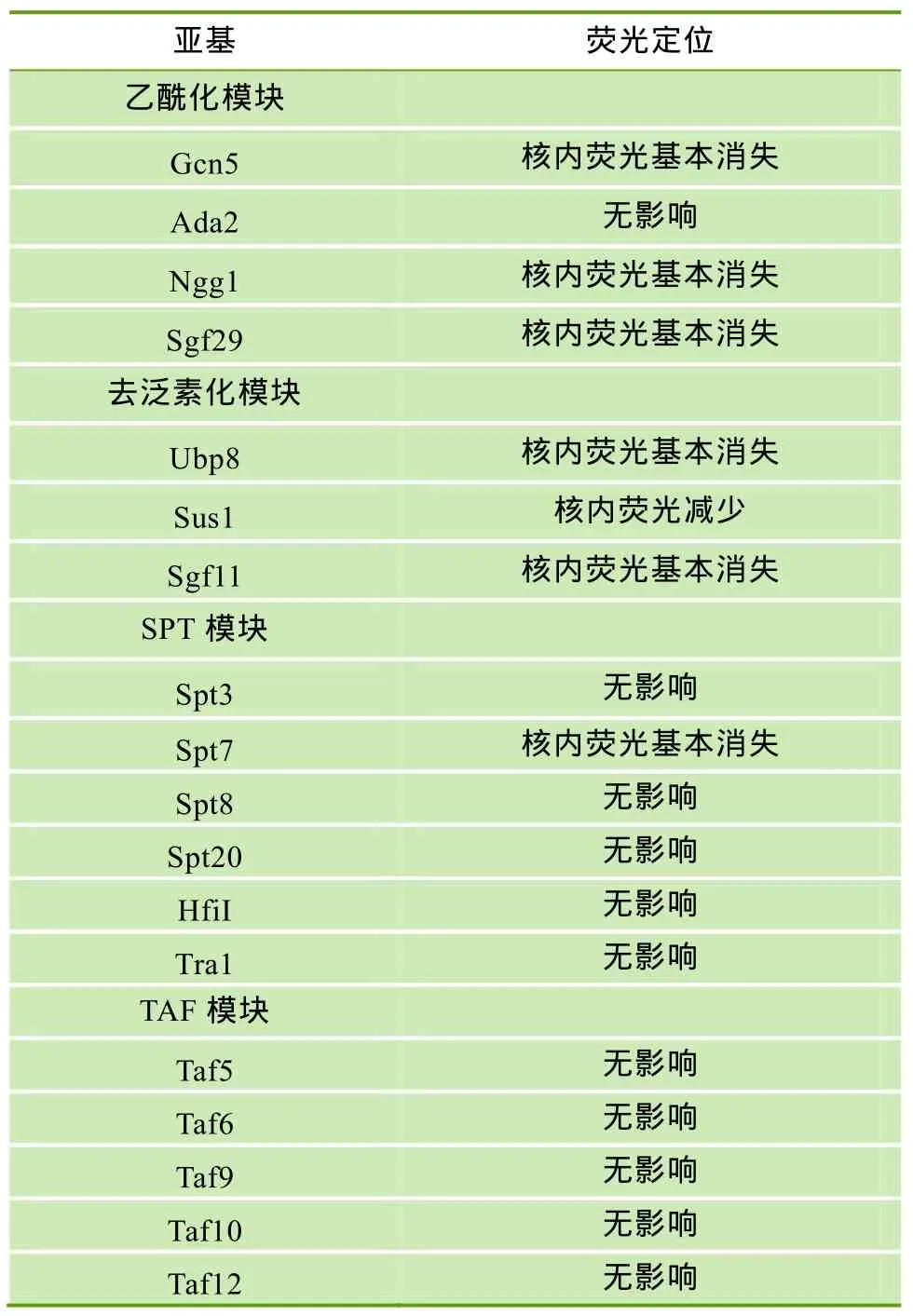
表4 SAGA各亚基缺失sgf73+前后荧光对比
裂殖酵母整合荧光与过表达荧光出现的差异可能是文献[10]所述的方法精确度不高,难以保证加入Thaimine的量和处理的时间恰好使Pnmt1启动子与整合荧光的内源启动子处于同一启动水平。这种方法是将基因的ORF以及YFP(Yellow fluorescence protein)构建在含有Pnmt1启动子的过表达质粒上,通过与裂殖酵母2号染色体上的基因Leu1同源获得荧光菌株,并通过加入Thaimine来控制荧光质粒的表达量; 另外,由于所有需要表达荧光的基因都是与裂殖酵母2号染色体上基因Leu1同源,而并非该基因原位,因此,在定位上与整合荧光可能会有部分差异(表3)。裂殖酵母与酿酒酵母定位的差异说明SAGA尽管保守,但由于裂殖酵母在进化程度上高于酿酒酵母,且已经出现有丝分裂,那些在胞浆中有定位的亚基可能以某种方式参与到了有丝分裂过程或其他酿酒酵母不具备的生理过程中。
本研究还利用敲除技术系统分析了缺失sgf73+对其他SAGA亚基的蛋白定位影响。如表4所示:Gcn5、Sgf29、Ngg1、Ubp8、Sgf11、Sus1、Spt7这 7个亚基在缺失sgf73+后核内荧光定位明显减少甚至消失,而其余亚基的荧光定位在缺失前后并没有明显变化。这说明在整个 SAGA里,部分亚基的稳定性或功能依赖于 Sgf73,而其余亚基的稳定性或功能并不需要Sgf73来维持。
本研究还发现△sgf73菌株呈现多隔膜表型(图6),并同时有两个或两个以上的细胞核(图4,图5)。这说明缺失sgf73+影响了细胞的胞质分裂,但对DNA复制没有造成影响,且这种影响不依赖于酶学活性亚基Gcn5和Ubp8。裂殖酵母的膈膜分为两层,外层由Agn1负责降解[21],内层由Eng1负责降解[22],这两个降解酶都受到Ace2的调控。在Ace2转录途径中,Mid2受 Ace2的调控,而 Mid2本身调控Septin(胞裂蛋白环)的形成,因此它也参与了胞质分裂过程。为了验证△sgf73的分裂缺陷是否由ace2+、mid2+的转录水平下降引起的,本研究在△sgf73中过量表达了sgf73+、ace2+、mid2+,回补结果显示ace2+完全不能回补△sgf73的多隔膜表型,而mid2+也仅能部分回补,提示缺失sgf73+还影响了其他调控胞质分裂的途径。另外,虽然△sgf73与△spt8所造成的多隔膜表型相似,但由于过量表达ace2+并不能回补△sgf73的表型缺陷,说明造成这两者胞质分裂缺陷的机制并不一致。
胞质分裂是控制形成子细胞的重要生理过程,在高等生物中它的异常会导致细胞不正常分裂甚至诱发癌变。SAGA各亚基中除△spt8和△sgf73出现多膈膜表型外,最新的研究还发现△spt7、△spt20、△hfi1同样呈现多隔膜表型[23]。针对这几个 SAGA亚基的缺失突变体菌株的分裂缺陷至今还未有深入的机制研究。SAGA是否可以直接与调控细胞周期或胞质分裂的关键因子相互作用仍然不清楚。因此,阐明SAGA是仅仅通过转录的方式还是有其他途径参与调控细胞周期或胞质分裂是一个值得研究的问题,从而进一步揭示SAGA的多功能性。
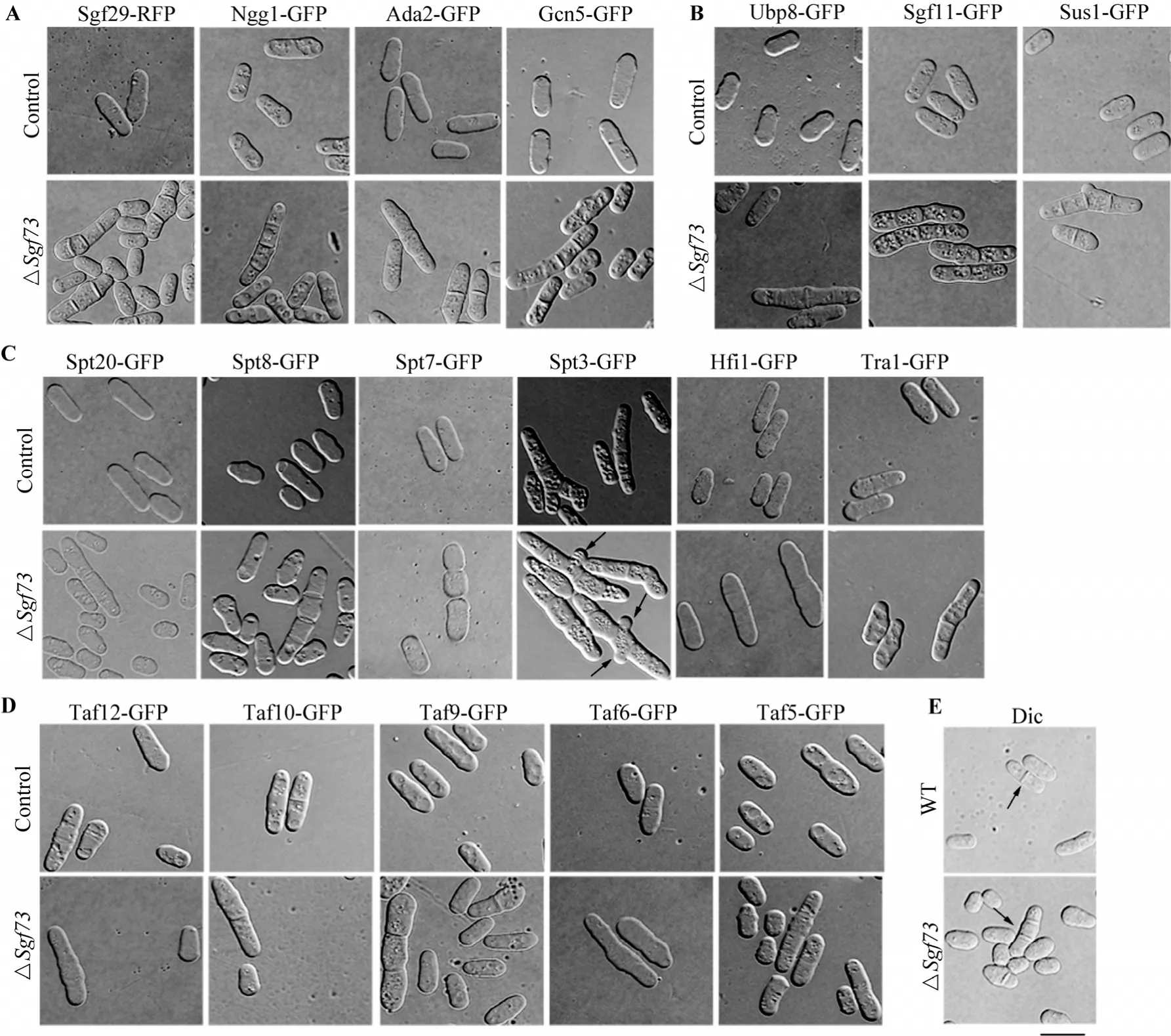
图6 SAGA各亚基荧光菌株中缺失sgf73+后的细胞表型

图7 sgf73突变体中过表达ace2+、mid2+、sgf73+后多隔膜细胞比例
[1]Huisinga KL,Pugh BF.A Genome-wide housekeeping role for TFIID and a highly regulated stress-related role for SAGA inSaccharomyces cerevisiae.Mol Cell,2004,13(4):573–585.
[2]Zhang XY,Varthi M,Sykes SM,Phillips C,Warzecha C,Zhu WT,Wyce A,Thorne AW,Berger SL,Mcmahon SB.The putative cancer stem cell marker USP22 is a subunit of the human SAGA complex required for activated transcription and cell-cycle progression.Mol Cell,2008,29(1):102–111.
[3]Koutelou E,Hirsch CL,Dent SY.Multiple faces of the SAGA complex.Curr Opin Cell Biol,2010,22(3):374–382.
[4]Rodriguez-Navarro S.Insights into SAGA function during gene expression.EMBO Rep,2009,10(8):843–850.
[5]Batta G,Szilagyi Z,Laczik M,Sipiczki M.The involvement of theSchizosaccharomyces pombesep9/spt8gene in the regulation of septum cleavage.FEMS Yeast Res,2009,9(5):757–767.
[6]Helmlinger D,Marguerat S,Villén J,Gygi SP,Bähler J,Winston F.TheS.pombeSAGA complex controls the switch from proliferation to sexual differentiation through the opposing roles of its subunits Gcn5 and Spt8.Genes Dev,2008,22(22):3184–3195.
[7]Mcmahon SB,Wood MA,Cole MD.The essential cofactor TRRAP recruits the histone acetyltransferase hGCN5 to c-Myc.Mol Cell Biol,2000,20(2):556–562.
[8]Rodńguez-Navarro S,Fischer T,Luo MJ,Antúnez O,Brettschneider S,Lechner J,Pérez-Ortín JE,Reed R,Hurt E.Sus1,a functional component of the SAGA histone acetylase complex and thes nuclear pore-associated mRNA export machinery.Cell,2004,116(1):75–86.
[9]Hayashi A,Ding DQ,Tsutsumi C,Chikashige Y,Masuda H,Haraguchi T,Hiraoka Y.Localization of gene products using a chromosomally tagged GFP-fusion library in the fission yeastSchizosaccharomyces pombe.Genes Cells,2009,14(2):217–225.
[10]Matsuyama A,Arai R,Yashiroda Y,Shirai A,Kamata A,Sekido S,Kobayashi Y,Hashimoto A,Hamamoto M,Hiraoka Y,Horinouchi S,Yoshida M.ORFeome cloning and global analysis of protein localization in the fission yeastSchizosaccharomyces pombe.Nat Biotechnol,2006,24(7):841–847.
[11]Helmlinger D,Hardy S,Sasorith S,Klein F,Robert F,Weber C,Miguet L,Potier N,Van-Dorsselaer A,Wurtz JM,Mandel JL,Tora L,Devys D.Ataxin-7 is a subunit of GCN5 histone acetyltransferase-containing complexes.Human Mol Genet,2004,13(12):1257–1265.
[12]Köhler A,Schneider M,Cabal GG,Nehrbass U,Hurt E.Yeast Ataxin-7 links histone deubiquitination with gene gating and mRNA export.Nat Cell Biol,2008,10(6):707–715.
[13]Shukla A,Bajwa P,Bhaumik SR.SAGA-associated Sgf73p facilitates formation of the preinitiation complex assembly at the promoters either in a HAT-dependent or independent mannerin vivo.Nucleic Acids Res,2006,34(21):6225–6232.
[14]Palhan VB,Chen S,Peng GH,Tjernberg A,Gamper AM,Fan Y,Chait BT,La Spada AR,Roeder RG.Polyglutamine-expanded ataxin-7 inhibits STAGA histone acetyltransferase activity to produce retinal degeneration.Proc Natl Acad Sci USA,2005,102(24):8472–8477.
[15]Gregan J,Rabitsch PK,Rumpf C,Novatchkova M,Schleiffer A,Nasmyth K.High-throughput knockout screen in fission yeast.Nat Protoc,2006,1(5):2457–2464.
[16]Grant PA,Schieltz D,Pray-Grant MG,Steger DJ,Reese JC,Yates JR,Workman JL.A subset of TAFIIs are integral components of the SAGA complex required for nucleosome acetylation and transcriptional stimulation.Cell,1998,94(1):45–53.
[17]Huh WK,Falvo JV,Gerke LC,Carroll AS,Howson RW,Weissman JS,O'Shea EK.Global analysis of protein localization in budding yeast.Nature,2003,425(6959):686–691.
[18]Sipiczki M.Where does fission yeast sit on the tree of life?Genome Biol,2000,1(2):S1011.
[19]Grant PA,Duggan L,Côté J,Roberts SM,Brownell JE,Candau R,Workman JL.Yeast Gcn5 functions in two multisubunit complexes to acetylate nucleosomal histones:characterization of an Ada complex and the SAGA(Spt/ Ada) complex.Genes Dev,1997,11(13):1640–1650.
[20]Helmlinger D,Marguerat S,Villén J,Swaney DL,Gygi SP,Bähler J,Winston F.Tra1 has specific regulatory roles,rather than global functions,within the SAGA co-activator complex.EMBO J,2011,30(14):2843–2852.
[21]Garcia I,Jimenez D,Martin V,Duran A,Sanchez Y.The alpha-glucanase Agn1p is required for cell separation inSchizosaccharomyces pombe.Biol Cell,2005,97(7):569–576.
[22]Martin-Cuadrado AB,Dueñas E,Sipiczki M,Vázquez DAC,Del RF.The endo-beta-1,3-glucanase eng1p is required for dissolution of the primary septum during cell separation inSchizosaccharomyces pombe.J Cell Sci,2003,116(Pt 9):1689–1698.
[23]Hayles J,Wood V,Jeffery L,Hoe KL,Kim DU,Park HO,Salas-Pino S,Heichinger C,Nurse P.A genome-wide resource of cell cycle and cell shape genes of fission yeast.Open Biol,2013,3(5):130053–130067.
