番茄中与致病密切相关miRNA的挖掘及特性分析
2014-11-17孙广鑫栾雨时崔娟娟
孙广鑫,栾雨时,崔娟娟
大连理工大学生命科学与技术学院,大连 116024
MicroRNA(miRNA)普遍存在于生物体内,是由内源基因编码的长度为22nt左右的单链非编码小分子 RNA[1],可与 RNA诱导沉默复合体(RNA-induced silencing complex,RISC) 结合识别其靶mRNA。植物中miRNA序列高度保守并呈现出独特的表达时序性和组织特异性。在植物细胞核中,内源miRNA基因先形成前体转录本pri-miRNA,然后被加工形成较为稳定的初级茎环结构,再进一步形成具有发夹结构的 pre-miRNA,最后经 DCL1酶剪切形成 miRNA/miRNA*双链复合体,从细胞核进入细胞质形成成熟的 miRNA[2],与 RISC结合后作用于靶基因,发挥其翻译阻遏或者降解作用[3]。miRNA参与植物生长发育的多种生理生化过程以及各种生物与非生物胁迫反应[4]等。
番茄作为重要的园艺作物,不仅具有很高的经济价值,而且在生命科学研究中也占有举足轻重的地位。由于番茄具有易杂交、繁殖系数高、遗传资源丰富、突变体库较多、遗传图谱广泛、遗传转化体系高效稳定等特性,使其成为作物研究中的模式植物之一。在植物与病原物互作的研究方面,由于番茄基因组复杂且病原种类丰富,陆续得到了众多的理论信息[5]。2009年,Feng等[6]率先检测了与病毒相关的番茄 miRNA,其后的研究表明 miRNA在黄瓜花叶病毒(Cucumber mosaic virus,CMV)、番茄不孕病毒(Tomato aspermy virus,TAV)、番茄卷叶新德里病毒(Tomato leaf curl new delhi virus,ToLCNDV)、番茄灰霉病等病害的防御和发生中均发挥着重要的调控作用。因此,对miRNA的抗逆功能研究可为番茄种质资源改良提供一定的理论依据。
目前对 miRNA的挖掘主要有直接克隆(Direct cloning)、正向遗传学(Forward genetics)筛选和生物信息学(Bioinformatics)分析等方法。直接克隆法比较简单,第一条植物miRNA就是用该方法发现的,很多实验室已经利用这种方法从不同生物中发现了众多miRNA[7~9],该方法更倾向于发现中、高量表达的miRNA,但很难发现一些表达量低、组织特异性强或受诱导表达的miRNA。近年来,高通量测序技术的发展极大地完善了该方法,并使得大规模鉴定miRNA成为可能[10,11],很多植物的miRNA都是通过高通量测序获得的[12~14]。正向遗传学筛选是在获得突变个体后,将突变基因分离出来,从而得到一个新的产物。最初的lin-4和let-7就是利用正向遗传学筛选发现的[15,16],该方法可以清楚地了解miRNA的功能,但效率很低,只能鉴定到少量的miRNA,因此,正向遗传学筛选方法无法成为发现miRNA的主要手段,只是在 miRNA研究早期使用较多。生物信息学分析方法可筛选出经各种测序方法获得的大量数据中的miRNA,它能够弥补直接克隆法的不足,发现一些丰度低、组织特异性强或受诱导表达的miRNA。随着一些物种基因组序列、大量EST(Expressed sequence tag)和GSS(Genome survey sequences)等序列的公布,也可以不通过测序,而是根据miRNA的保守性和独有的特征,利用生物信息学方法在基因组、EST和 GSS中预测 miRNA前体,进而找到保守的 miRNA,但通过该方法获得的miRNA还需经过实验验证以确定其真实性[17,18]。生物信息学分析方法快速、简单、高效,但是很难发现与现有miRNA无同源性的新的miRNA。
当番茄受病原物侵染时,体内的多种miRNA及相关靶基因的表达量都会发生变化,说明miRNA在番茄响应生物胁迫中发挥着关键的调控作用[19,20]。为了筛选出番茄体内与致病密切相关的miRNA,本研究采用生物信息学手段,从Sanger miRNA数据库——miRBase(Release 20:June 2013,http://www.mirbase.org/)和已报道的文献中找到34个miRNA的成熟序列,预测了它们的靶基因,利用Cytoscope软件构建这些 miRNA之间的调控网络,从中筛选出miR169、miR482、miR5300、miR6024、miR6026和 miR6027,进行了进一步的分析及实时定量 PCR验证,为深入研究miRNAs的作用机制奠定基础。
1 材料和方法
1.1 材料
野生醋栗番茄(Solanum pimpinellifolium)材料L3708为本实验室保藏; 致病疫霉菌种P12103为西北农林科技大学单卫星教授惠赠。通过NCBI、Web of Knowledge及文献检索等途径,统计出番茄中与致病相关的 miRNA共 34个(表1),其中 miR156、miR159、miR160、miR162、miR166、miR167、miR169、miR171、miR172、miR319、miR395、miR397、miR399、miR482、miR5300、miR6022、miR6023、miR6024、miR6026和 miR6027是在miRBase中已经登录的。利用Primer5软件分析设计其中 5个 miRNA的上游引物序列及内参基因Actin(GenBank:U60478.1)的上、下游引物序列(表 2)。
1.2 方法
1.2.1 致病相关miRNA序列的获得及靶基因预测
从 Sanger miRNA数据库——miRBase(http://www.mirbase.org/search.shtml)(release 20)和已报道的文献及其附件中收集到的34个miRNA全部家族成员的成熟序列。
将上述 miRNA利用软件 psRNATarget(http://plantgrn.noble.org/psRNATarget) 进行靶基因预测。在Solanum Lycopersicon(tomato)、transcript、cDNA library version2.3数据库中预测番茄miRNA的靶基因。预测参数除Maximum expectation设为5.0外,其余均使用程序默认参数。
1.2.2 致病密切相关miRNA的筛选
利用Cytoscope软件构建病原诱导的miRNA及其靶基因的调控网络。根据各靶基因的个数确定对应圆点的大小,从中筛选出与致病密切相关的miRNA作为主要的研究和分析对象。
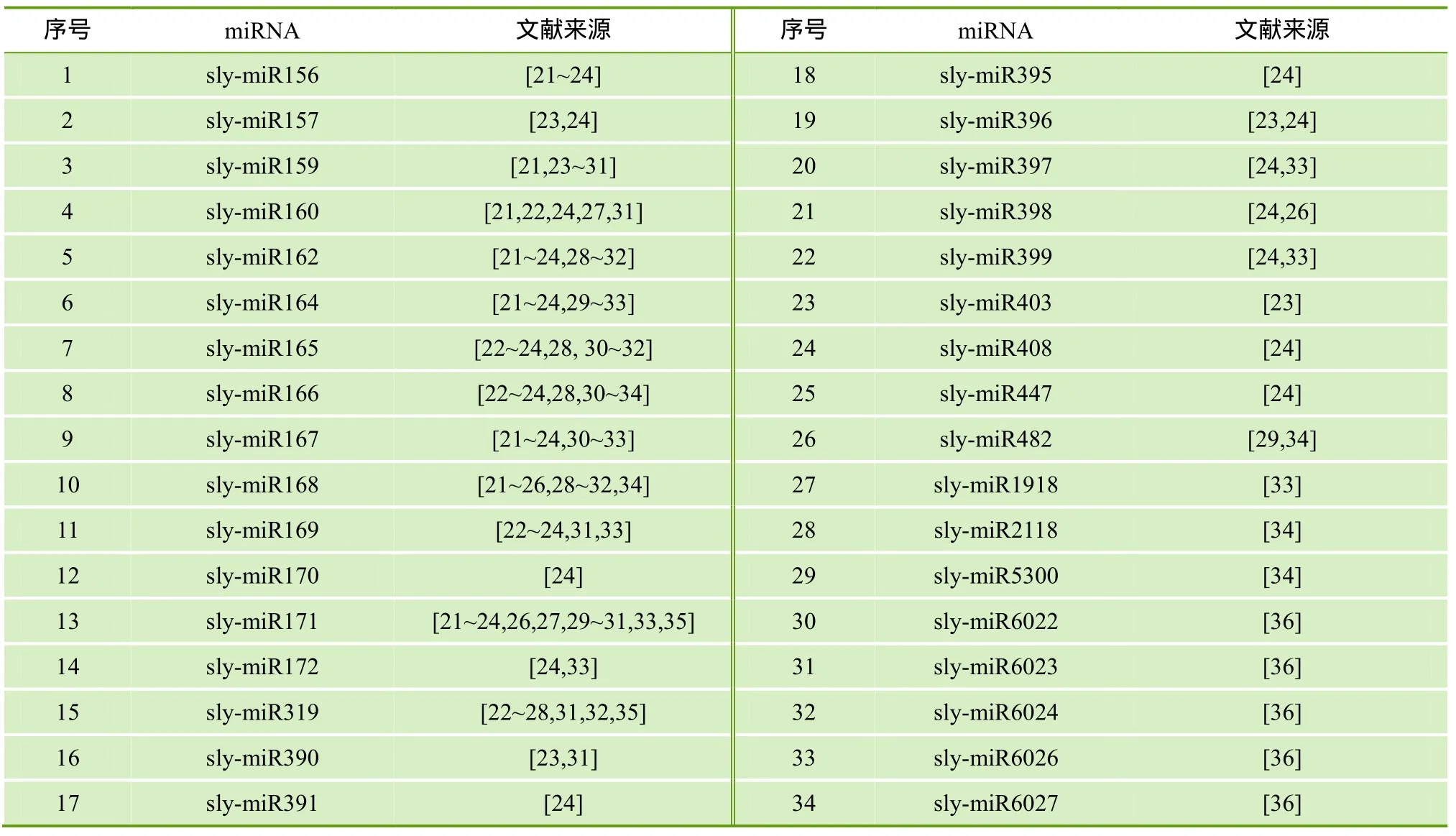
表1 番茄中与致病相关的34个miRNA
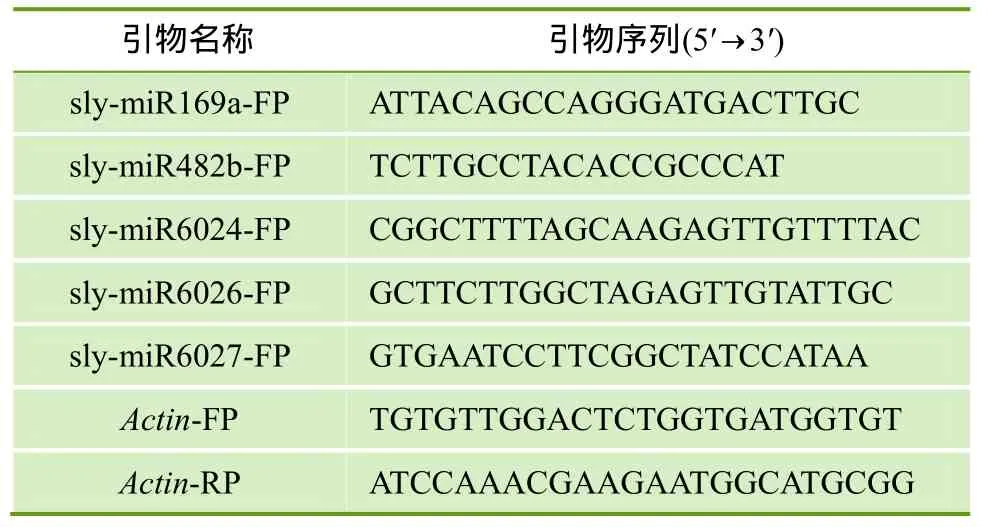
表2 设计的5个miRNA的上游引物及Actin的上、下游引物序列
1.2.3 致病密切相关miRNA的靶基因功能分析
根据上述靶基因预测结果,针对筛选出的若干miRNA的靶基因名称和 GO注释号在 Uni-protKB(http://www.uniprot.org/help/uniprotkb)蛋白数据库中分析各靶基因的功能。统计出与病害相关的靶基因。
1.2.4 致病密切相关miRNA的启动子分析
针对筛选出的若干 miRNA 的基因 5′端上游1 500 bp的启动子片段,利用 PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在线程序进行上游启动子预测及分析。找出与病害相关的启动子元件。
1.2.5 致病密切相关miRNA的实时定量PCR验证
选取长势相同的番茄五叶期幼苗为试材,以106个孢子/mL的致病疫霉孢子悬浮液喷施叶片,分别于处理后 0 h、3h、6 h、12 h、24 h、48 h、72 h、96 h时取样,用RNAiso plus(TaKaRa,Japan)试剂盒提取总RNA,再用miRNA反转录试剂盒One Step PrimeScript miRNA cDNA Synthesis Kit(TaKaRa,Japan)对样品中的 miRNA进行加尾,加上一个长为60 bp左右的接头,通过实时定量PCR进行验证。本研究使用的各miRNA的上游引物见表2,下游引物为试剂盒中提供的通用引物Uni-miR qPCR Primer。反应体系为 25 μL,包括:2 × SYBR®Premix ExTaqTM12.5 μL,cDNA 模板 2 μL,正反向引物各 1 μL,ddH2O 8.5 μL。反应条件:95℃预变性10 s; 95℃变性5 s; 60℃复性20 s,40个循环。用番茄Actin作为内参基因,荧光数据在每个循环的复性末期采集。对得到的结果用 2-DDCt法分析绘出柱状图,并对实时定量PCR产物进行4%琼脂糖凝胶电泳检测。
2 结果与分析
2.1 致病密切相关miRNA的靶基因预测及筛选
整理分析psRNATarget在线预测出的miRNA及其靶基因。先构建miRNA与靶基因间的Cytoscope调控网络。发现在整个网络中有多个miRNA携带它的靶基因单独存在,还有一部分miRNA是通过它们的靶基因与少数几个miRNA相关联,而只有一部分miRNA(miR156、miR157、miR159、miR167、miR169、miR171、miR319、miR403、miR482、miR5300、miR6024、miR6026和 miR6027)是通过它们的靶基因与多个 miRNA相关联形成了致密网络。再以这13个分布较集中的 miRNA为研究对象构建得到它们相互间的Cytoscope调控网络(图1)。从图1可知miR169、miR482、miR5300、miR6024、miR6026和miR6027的靶基因个数较多且与其他miRNA的关联性较大(至少有4个相关联的miRNA)。
2.2 致病密切相关miRNA的靶基因功能分析
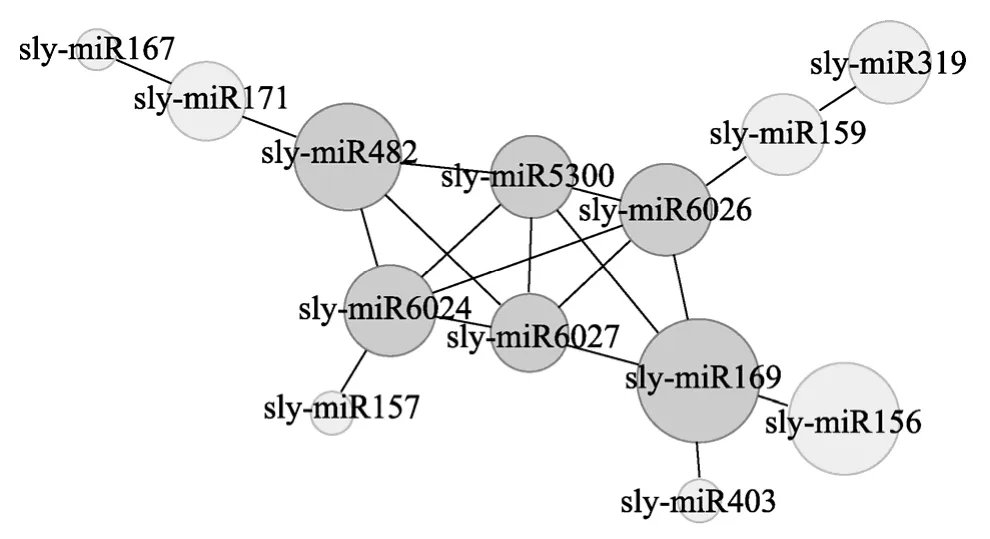
图1 致病密切相关各 miRNA之间的调控网络(圆的大小与靶基因数目呈正相关)
用psRNATarget在线软件预测miR169、miR482、miR5300、miR6024、miR6026和 miR6027的靶基因,经筛选后各获得多条靶基因。根据各靶基因的名称和GO注释号在Uni-protKB蛋白数据库中查询每条靶基因的功能,可将它们分为 5类:(1)编码与胁迫响应相关的蛋白质;(2)编码与代谢相关的酶蛋白;(3)编码与跨膜运输相关的蛋白质;(4)编码转录因子;(5)未知蛋白。统计发现 miR169、miR482、miR5300、miR6024、miR6026和 miR6027编码与胁迫响应相关蛋白质的靶基因分别占各自总靶基因的22.45%、80.30%、71.43%、95.45%、59.09%和 100.00%。可见,预测到的靶基因大部分与番茄的胁迫响应相关,其中有很多参与致病过程(表3)。
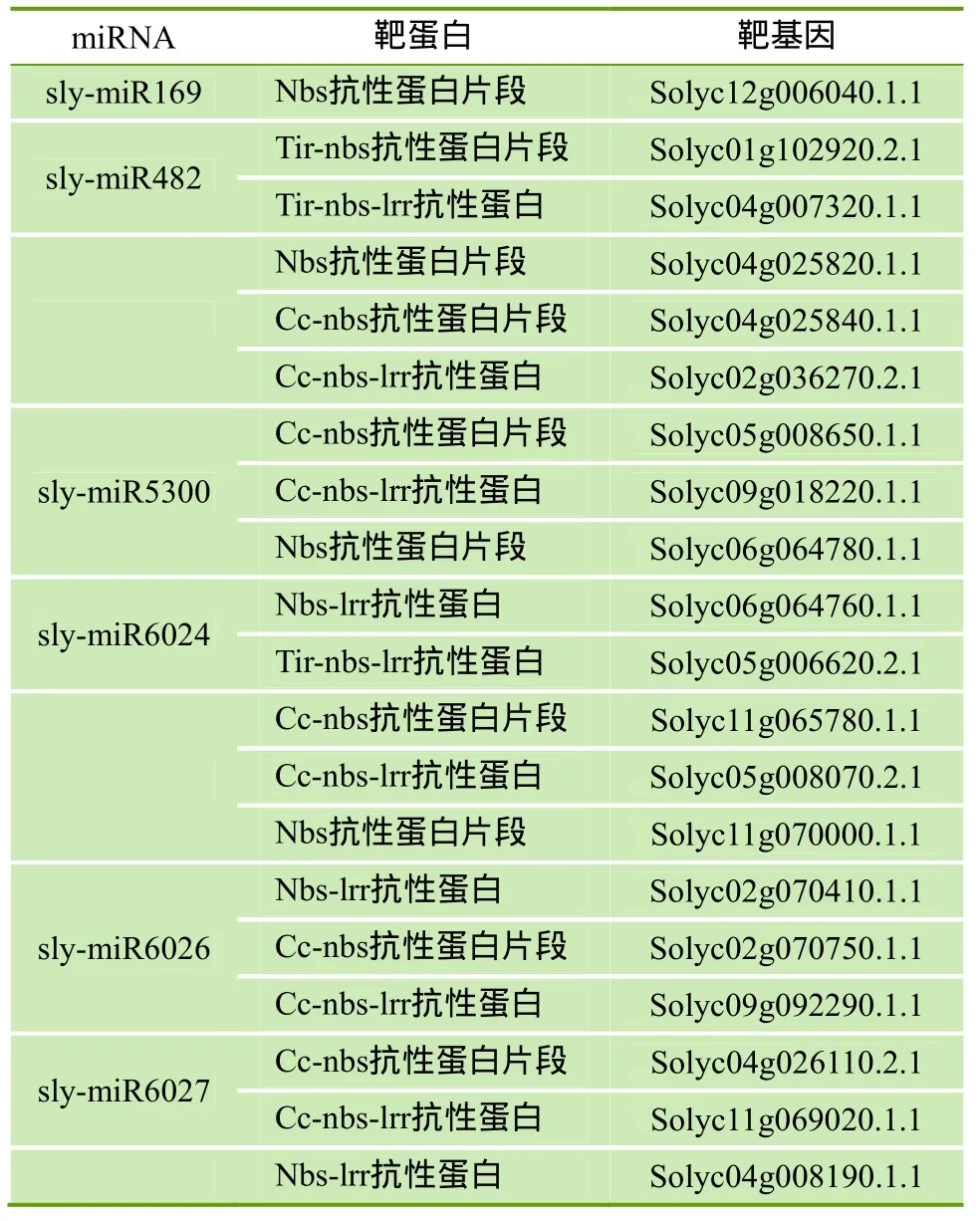
表3 与致病密切相关的各miRNA的靶基因
2.3 致病密切相关miRNA的启动子分析
应用PlantCARE在线程序对miR169、miR482、miR5300、miR6024、miR6026和miR6027前体基因5′端上游1 500 bp的序列进行启动子预测分析。结果表明,除 miR6027外,其他各成员的启动子区域不仅存在典型的具有转录起始功能的 TATA-box和CAAT-box,还存在多种胁迫相关的顺式作用元件(包括激素响应、光响应、生物胁迫响应、厌氧响应、干旱响应和温度响应元件等)以及与生物胁迫相关的启动子元件 TC-rich repeats; 同时 miR169、miR482、miR5300和miR6026中还存在与生物胁迫相关的启动子元件Box-W1。由于不能确定miR6027在染色体上的具体位置,所以未对其进行启动子分析。
2.4 致病密切相关miRNA的实时定量PCR验证
5个miRNA的实时定量PCR检测结果见图2。致病疫霉处理后,番茄叶片中的miR169、miR482、miR6024、miR6026和 miR6027的表达量均在 3 h内迅速降低,在24 h时普遍有所回升,在48 h时又降到最低点。以上结果说明这5个miRNA都能对致病疫霉处理做出响应。且通过琼脂糖凝胶电泳检测(图 3)表明,miR169、miR482、miR6024、miR6026和miR6027的扩增产物都在80 bp左右,说明符合预期长度。
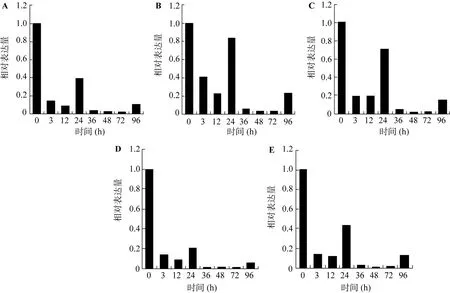
图2 致病疫霉处理后5个miRNA的表达特性
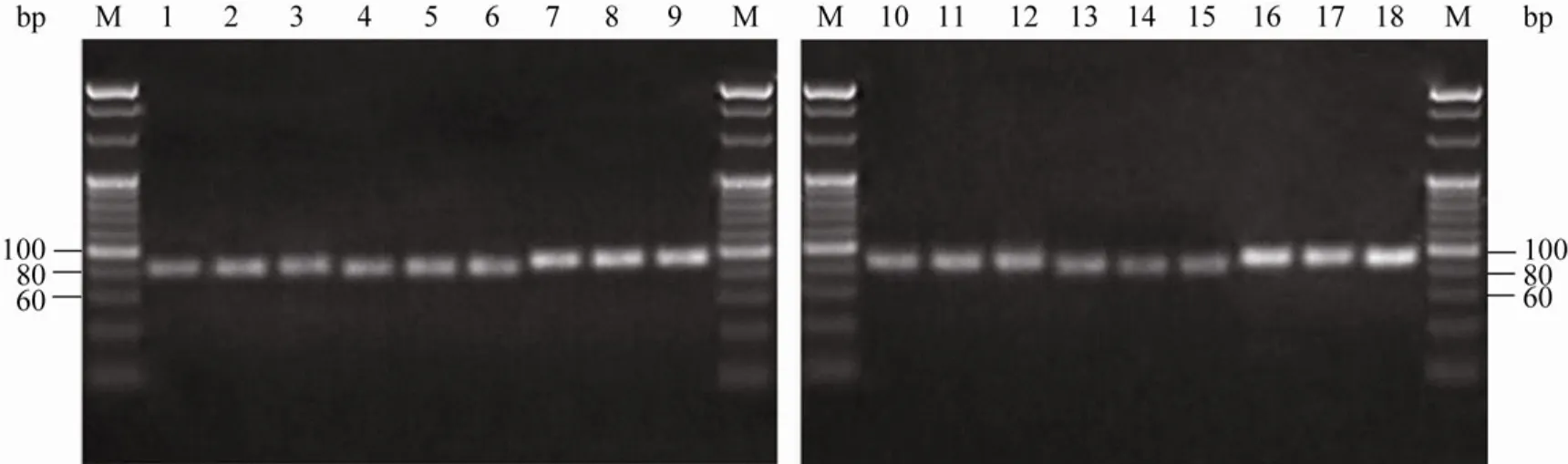
图3 实时定量PCR产物的琼脂糖凝胶电泳检测
3 讨 论
近年来,miRNA作为基因调控因子备受瞩目,已迅速成为生命科学领域的研究热点。众所周知miRNA是植物对胁迫响应过程中不可或缺的调控因子,它通过互补作用靶向mRNA抑制或降解相应基因的表达,参与调控生物体内的各项生命活动。本研究采用生物信息学分析方法构建 Cytoscape网络图,欲筛选与致病密切相关的 miRNA,对选出的miR169、miR482、miR5300、miR6024、miR6026和miR6027进行靶基因功能分析和启动子分析。结果发现,这6个miRNA的靶基因和启动子基本都与生物胁迫相关,表明它们极可能参与了番茄对胁迫响应的调控过程。为了探究各miRNA在致病疫霉处理下的表达情况,本文又对其中5个miRNA进行了表达特性分析。发现这5个miRNA的表达量都有显著变化,且趋势基本一致,均在48 h时降到最低水平,说明这5个miRNA都能对致病疫霉的接种做出响应,且它们之间可能存在某些相似的通路。由于miR5300的引物特异性不理想,所以本文未对其进行实验分析。究竟在胁迫下各miRNA与其靶基因存在怎样的相互关系,后者又是如何表达的,这些问题还有待于进一步的实验验证。
番茄作为重要的栽培植物,在受到真菌、细菌、病毒以及线虫和昆虫的侵害时常造成大量减产[37],甚至绝收,给农业生产带来严重影响。研究发现,植物抗病(R)基因能够识别病原物并引起过敏性反应,它作为防卫系统的一员,在没有病原物侵染时表达量很低,但当病原物侵染后,能够识别激发子,诱导抗病基因的过量表达,并激活抗病信号传导途径,产生防卫反应[38],从而提高作物对病害的抗性。所以,克隆抗性基因将对研究植物和病原物互作、提高植物抗病性具有重要意义。近年来已从不同植物中克隆出了各种R基因。多数R基因具有NBS-LRR结构域,其中NBS(Nucleotide binding site)是抗病基因核苷酸结合位点。而NBS-LRR编码基因是抗病基因中的最大家族,大约 3/4的抗病基因都来自于这一家族。根据N端信号肽的不同,又将NBS-LRR基因分为 TIR-NBS-LRR和CC-NBS-LRR两类[39,40]。这些抗病基因大量存在于植物基因组中,它的作用对象囊括了所有病原物[41]。经靶基因功能分析发现筛选出的6个miRNA的靶基因中均含有NBS-LRR抗性蛋白,推测植物在受到病原物侵染时,这 6个miRNA的表达量也会发生相应的变化,从而说明了我们筛选的miRNA的确是与致病密切相关的。并且已有实验证明,当番茄植株受到CMV、TAV、Tolcv和灰霉病菌侵染时,用基因芯片技术检测到miR169表达量逐渐上调,而其靶基因的表达量下调,说明miR169参与了病原物的胁迫响应。miR482、miR6024、miR6026和 miR6027也被预测到参与植物的免疫应答过程,靶向植物的免疫受体[24]。
由于启动子控制着基因表达的起始时间和表达程度,而某些miRNA的表达又具有时空特异性和组织特异性。所以,对启动子的研究将成为miRNA表达特性分析的重要工具[42]。研究发现,大部分miRNA都含有独立的启动子,是 RNA聚合酶Ⅱ的产物[43],经过转录而形成。所以,它的表达很有可能受到精确的调控。为进一步剖析miRNA在胁迫下的表达模式,本研究利用 PlantCARE[44]对番茄miR169、miR482、miR5300、miR6024 和 miR6026基因上游启动子区域进行分析,发现在这些区域中存在共同的植物病原物侵染和损伤响应顺式作用元件TC-rich repeats和Box-W1。表明这些生物胁迫相关miRNA可能参与了相应的逆境胁迫过程,也暗示植物在响应生物胁迫时可能存在某些共同的通路。研究发现Box-W1存在于如芪合成酶(STS)和病程相关蛋白PR-1、Ypr-10中[45,46],位于多病原体侵染和非生物胁迫响应基因 VvALDH2B8的启动子区域,能特异的结合WRKY转录因子[47],参与胁迫响应基因的表达调控。所以,这些胁迫响应顺式元件的存在为 miRNA参与植物抗逆反应提供了证据。此外,番茄作为一种模式植物,挖掘和分析其与病害相关的 miRNA还有助于揭示植物与病原物互作的分子机制。
[1]Carrington JC,Ambros V.Role of microRNAs in plant and animal development.Science,2003,301(5631):336–338.
[2]Jones-Rhoades MW,Bartel DP,Bartel B.MicroRNAs and their regulatory roles in plants.Annu Rev Plant Biol,2006,57:19–53.
[3]金龙国,王川,刘进元.植物 MicroRNA.中国生物化学与分子生物学报,2006,22(8):609–614.
[4]Kulcheski FR,De Oliveira LFV,Molina LG,Almerão MP,Rodrigues FA,Marcolino J,Barbosa JF,Stolf-Moreira R,Nepomuceno AL,Marcelino-Guimarães FC,Abdelnoor RV,Nascimento LC,Carazzolle MF,Pereira GAG,Margis R.Identification of novel soybean microRNAs involved in abiotic and biotic stresses.BMC Genomics,2011,12(1):307.
[5]Arie T,Teraoka T,Takahashi H,Kodama M.Tomato as a model plant for plant-pathogen interactions.Plant Biotechnology,2007,24(1):135–147.
[6]Feng JL,Wang K,Liu X,Chen SN,Chen JS.The quantification of tomato microRNAs response to viral infection by stem-loop real-time RT-PCR.Gene,2009,437(1–2):14–21.
[7]Sunkar R,Girke T,Jain PK,Zhu JK.Cloning and characterization of microRNAs from rice.Plant Cell,2005,17(5):1397–1411.
[8]Tang GL,Reinhart BJ,Bartel DP,Zamore PD.A biochemical framework for RNA silencing in plants.Genes Dev,2003,17(1):49–63.
[9]Llave C,Xie ZX,Kasschau KD,Carrington JC.Cleavage ofScarecrow-likemRNA targets directed by a class ofArabidopsismiRNA.Science,2002,297(5589):2053–2056.
[10]Lu C,Tej SS,Luo SJ,Haudenschild CD,Meyers BC,Green PJ.Elucidation of the small RNA component of the transcriptome.Science,2005,309(5740):1567–1569.
[11]Nakano M,Nobuta K,Vemaraju K,Tej SS,Skogen JW,Meyers BC.Plant MPSS databases:signature-based transcriptional resources for analyses of mRNA and small RNA.Nucleic Acids Res,2006,34(S1):D731–D735.
[12]Zhao CZ,Xia H,Frazier TP,Yao YY,Bi YP,Li AQ,Li MJ,Li CS,Zhang BH,Wang XJ.Deep sequencing identifies novel and conserved microRNAs in peanuts(Arachis hypogaeaL.).BMC Plant Biol,2010,10(1):3.
[13]Li BS,Qin YR,Duan H,Yin WL,Xia XL.Genome-wide characterization of new and drought stress responsive microRNAs inPopulus euphratica.J Exp Bot,2011,62(11):3765–3779.
[14]Zhou ZS,Zeng HQ,Liu ZP,Yang ZM.Genome-wide identification ofMedicago truncatulamicroRNAs and their targets reveals their differential regulation by heavy metal.Plant Cell Environ,2012,35(1):86–89.
[15]Lee RC,Feinbaum RL,Ambros V.The C.elegans heterochronic genelin-4encodes small RNAs with antisense complementarity tolin-14.Cell,1993,75(5):843– 854.
[16]Reinhart BJ,Slack FJ,Basson M,Pasquinelli AE,Bettinger JC,Rougvie AE,Horvitz HR,Ruvkun G.The 21 nucleotidelet-7RNA regulates developmental timing inCaenorhabditis elegans.Nature,2000,403(6772):901–906.
[17]张志明,宋锐,彭华,罗茂,沈亚欧,刘丽,赵茂俊,潘光堂.用生物信息学挖掘玉米中的 microRNAs及其靶基因.作物学报,2010,36(8):1324–1335.
[18]Zhang JG,Zeng R,Chen JS,Liu X,Liao QS.Identification of conserved microRNAs and their targets fromSolanum lycopersicumMill.Gene,2008,423(1):1–7.
[19]Feng JL,Lai LY,Lin RH,Jin CZ,Chen JS.Differential effects ofcucumber mosaic virussatellite rnas in the perturbation of microrna-regulated gene expression in tomato.Mol Biol Rep,2012,39(1):775–784.
[20]Diermann N,Matousek J,Junge M,Riesner D,Steger G.Characterization of plant mirnas and small rnas derived from potato spindle tuber viroid(pstvd) in infected tomato.Biol Chem,2010,391(12):1379–1390.
[21]Feng JL,Lin RH,Chen JS.Alteration of tomato micro-RNAs expression during fruit development uponCucumber mosaic virusandTomato aspermy virusinfection.Mol Biol Rep,2013,40(5):3713-3722.
[22]Feng JL,Lai LY,Lin RH,Jin CZ,Chen JS.Differential effects ofCucumber mosaic virussatellite RNAs in the perturbation of microRNA-regulated gene expression in tomato.Mol Biol Rep,2012,39(1):775–784.
[23]Feng JL,Liu X,Lai LY,Chen JS.Spatio-temporal expression of miRNAs in tomato tissues uponCucumber mosaic virusandTomato aspermy virusinfections.Acta Biochim Biophys Sin,2011,43(4):258–266.
[24]Naqvi AR,Haq QMR,Mukherjee SK.MicroRNA profiling ofTomato leaf curl new delhi virus(tolcndv) infected tomato leaves indicates that deregulation of mir159/319 and mir172 might be linked with leaf curl disease.Virol J,2010,7(1):281.
[25]Vu TV,Choudhury NR,Mukherjee SK.Transgenic tomato plants expressing artificial microRNAs for silencing the pre-coat and coat proteins of a begomovirus,Tomato leaf curl New Delhi virus,show tolerance to virus infection.Virus Res,2013,172(1-2):35–45.
[26]Várallyay E,Válóczi A,Ágyi A,Burgyán J,Havelda Z.Plant virus-mediated induction of miR168 is associated with repression of ARGONAUTE1 accumulation.EMBO J,2010,29(20):3507–3519.
[27]Stav R,Hendelman A,Buxdorf K,Arazi T.Transgenic expression ofTomato bushy stunt virussilencing suppressor P19 via the pOp/LhG4 transactivation system induces viral-like symptoms in tomato.Virus Genes,2010,40(1):119–129.
[28]Martín R,Arenas C,Daròs JA,Covarrubias A,Reyes JL,Chua NH.Characterization of small RNAs derived fromCitrus exocortis viroid(CEVd) in infected tomato plants.Virology,2007,367(1):135–146.
[29]Pilcher RLR,Moxon S,Pakseresht N,Moulton V,Manning K,Seymour G,Dalmay T.Identification of novel small RNAs in tomato(Solanum lycopersicum).Planta,2007,226(3):709–717.
[30]Feng JL,Wang K,Liu X,Chen SN,Chen JS.The quantification of tomato microRNAs response to viral infection by stem-loop real-time RT-PCR.Gene,2009,437(1-2):14–21.
[31]Feng JL,Wang YW,Lin RH,Chen JS.Altered expression of microRNAs and target mRNAs in tomato root and stem tissues upon different viral infection.J Phytopathol,2013,161(2):107–119.
[32]Cillo F,Mascia T,Pasciuto MM,Gallitelli D.Differential effects of mild and severeCucumber mosaic virusstrains in the perturbation of microRNA-regulated gene expression in tomato map to the 3′ sequence of RNA 2.Mol Plant Microbe Interact,2009,22(10):1239–1249.
[33]Naqvi AR,Choudhury NR,Mukherjee SK,Haq QMR.In silicoanalysis reveals that several tomato micro-RNA/microRNA* sequences exhibit propensity to bind toTomato leaf curl virus(ToLCV) associated genomes and most of their encoded open reading frames(ORFs).Plant Physiol Biochem,2011,49(1):13–17.
[34]Shivaprasad PV,Chen HM,Patel K,Bond DM,Santos BACM,Baulcombe DC.A microRNA superfamily regulates nucleotide binding site–leucine-rich repeats and other mRNAs.Plant Cell,2012,24(3):859–874.
[35]Goswami S,Sahana N,Pandey V,Doblas P,Jain RK,Palukaitis P,Canto T,Praveen S.Interference in plant defense and development by non-structural protein NSs ofGroundnut bud necrosis virus.Virus Res,2012,163(1):368–373.
[36]Li F,Pignatta D,Bendix C,Brunkard JO,Cohn MM,Tung J,Sun H,Kumar P,Baker B.MicroRNA regulation of plant innate immune receptors.Proc Natl Acad Sci USA,2012,109(5):1790–1795.
[37]van Kan JAL.Licensed to kill:the lifestyle of a necrotrophic plant pathogen.Trends Plant Sci,2006,11(5):247–253.
[38]Thurau T,Kifle S,Jung C,Cai DG.The promoter of the nematode resistance gene Hs1pro-1activates a nematode-responsive and feeding site-specific gene expression in sugar beet(Beta vulgarisL.) andArabidopsis thaliana.Plant Mol Biol,2003,52(3):643–660.
[39]Dangl JL,Jones JDG.Plant pathogens and integrated defence responses to infection.Nature,2001,411(6839):826–833.
[40]van Der Biezen EA,Jones JDG.The NB-ARC domain:a novel signalling motif shared by plant resistance gene products and regulators of cell death in animals.Curr Biol,1998,8(7):R226–R227.
[41]He LM,Du CG,Covaleda L,Xu ZY,Robinson AF,Yu JZ,Kohel RJ,Zhang HB.Cloning,characterization,and evolution of the NBS-LRR-encoding resistance gene analogue family in polyploid cotton(Gossypium hirsutumL.).Mol Plant Microbe Interact,2004,17(11):1234–1241.
[42]Sunkar R,Kapoor A,Zhu JK.Posttranscriptional induction of two Cu/Zn superoxide dismutase genes inArabidopsisis mediated by downregulation of miR398 and important for oxidative stress tolerance.Plant Cell,2006,18(8):2051–2065.
[43]Johnston RJ Jr,Hobert O.A microRNA controlling left/right neuronal asymmetry inCaenorhabditis elegans.Nature,2003,426(6968):845–849.
[44]Lescot M,Dehais P,Thijs G,Marchal K,Moreau Y,van de Peer Y,Rouz P,Rombauts S.PlantCARE,a database of plantcis-acting regulatory elements and a portal to tools forin silicoanalysis of promoter sequences.Nucleic Acids Res,2002,30(1):325–327.
[45]Pühringer H,Moll D,Hoffmann-Sommergruber K,Watillon B,Katinger H,Machado MLD.The promoter of an appleYpr10gene,encoding the major allergen Mal d 1,is stress-and pathogen-inducible.Plant Sci,2000,152(1):35–50.
[46]Pape S,Thurow C,Gatz C.The ArabidopsisPR-1promoter contains multiple integration sites for the coactivator NPR1 and the repressor SNI1.Plant Physiol,2010,154(4):1805–1818.
[47]Rushton PJ,Torres JT,Parniske M,Wernert P,Hahlbrock K,Somssich IE.Interaction of elicitor-induced DNA-binding proteins with elicitor response elements in the promoters of parsleyPR1genes.EMBO J,1996,15(20):5690–5700.
