阿氏浮丝藻mcyT基因序列多样性研究
2014-11-05潘倩倩朱梦灵李仁辉
潘倩倩 朱梦灵 刘 洋 林 燊 李仁辉
(1. 中国科学院水生生物研究所, 武汉 430072; 2. 中国科学院大学, 北京 100049)
近年来, 水体富营养化现象日益严重, 国内外许多湖泊相继发生蓝藻水华现象。许多水华蓝藻种类还能产生蓝藻毒素和释放异味物质严重破坏水环境和生态[1—3]。资料显示, 世界各地不同水域发生的蓝藻水华中有 25%—70%可以检测到蓝藻毒素,并且水华蓝藻种类中约有 60%为产毒蓝藻[4]。其中微囊藻毒素(Microcystins, 简称 MCs)是最常见的肝毒素, 主要由微囊藻(Microcystis)、鱼腥藻(Anabaena)、项圈藻(Anabaenopsis)、浮丝藻(Planktothrix)和念珠藻(Nostoc)等蓝藻种类产生[5]。
浮丝藻作为常见的产微囊藻毒素种类而被广泛关注, 其在国内外特别是欧洲国家水体中分布广泛[6—9]。Rantala, et al.研究发现, 浮丝藻是除微囊藻之外最常见的潜在产微囊藻毒素的种类[10]。除微囊藻毒素之外, 浮丝藻还能够产生鱼腥藻毒素-a(Anatoxin-a)、麻痹性贝类毒素(Paralytic shellfish poisoning toxins, 简称PSTs)等多种蓝藻毒素[11,12]。目前阿氏浮丝藻与粉红浮丝藻(Planktothrix rubescens)的微囊藻毒素合成基因簇已全部测序并注释完成[13,14]。阿氏浮丝藻P. agardhii CYA 126/8的微囊藻毒素合成基因簇测序全长约55.6 kb, 包括9个功能基因, 其中8个基因(mcyA, -B, -C, -D, -E, -G, -H和-J)与微囊藻的微囊藻毒素合成基因相似。但是被命名为 mcyT的基因是浮丝藻所特有的编码硫酯酶的基因, 位于浮丝藻微囊藻毒素合成基因簇的 5′末端, 并且其转录方向与其他基因均相反[13]。mcyT基因编码产物硫酯酶与 mcyC基因编码产物硫酯酶结构域相互辅助共同参与微囊藻毒素的合成过程。Christiansen, et al.推测可能由于浮丝藻含有mcyT基因可以编码除 McyC之外的第二种硫酯酶, 而在微囊藻中缺失该基因, 从而导致浮丝藻 Planktothrix CYA 126的微囊藻毒素含量高于微囊藻 M. aeruginosa PCC 7806 和 K139[13]。
在我国, 浮丝藻也是常见水华蓝藻种属, 常与其他水华蓝藻如微囊藻、鱼腥藻等共存, 并且也有浮丝藻占优势的蓝藻水华现象[13,15]。胡鸿钧等报道过阿氏浮丝藻(Planktothrix agardhii)等3个种[16], 林燊等基于分子系统学研究报道过阿氏浮丝藻、假阿氏浮丝藻(Planktothrix pseudagardhii)和蒙氏浮丝藻(Planktothrix mougeotii)等种类[17]。目前我国对产毒微囊藻研究较多, 其他潜在产微囊藻毒素的蓝藻研究较少, 而浮丝藻作为分布广泛的潜在产毒水华蓝藻种类需要引起关注。本文通过PCR扩增方法检测分离自我国不同省份水体的阿氏浮丝藻的微囊藻毒素合成酶基因来初步探讨我国浮丝藻的产毒情况。选取微囊藻毒素合成相关的多肽合成酶基因mcyA、聚酮合成酶基因 mcyE以及硫酯酶基因 mcyT进行PCR检测。鉴于欧洲水体中, mcyT基因广泛存在于产毒浮丝藻与不产毒浮丝藻中, 并且其侧翼区存在一定的多样性[18], 所以我们对浮丝藻的 mcyT基因进行序列扩增测序, 并通过构建分子系统树分析我国浮丝藻 mcyT基因的序列多样性, 为后续进一步研究我国浮丝藻和其他水华蓝藻种类的产毒情况提供参考, 也为有害蓝藻的监测和治理提供一定的理论依据。
1 材料与方法
1.1 藻种及培养
实验所用的13株阿氏浮丝藻分别来自北京、上海、浙江、湖北、云南和新疆等地, 这些藻株均为单种藻株, 保存于中国科学院水生生物研究所有害藻类学科组藻种库(CHAB), 藻株的编号及来源见表1。实验所用藻株均由CT完全培养基[19]静置培养,每日早晚手动摇匀使之不贴壁生长, 光照强度为25 μmol/(m2·s), 光暗周期为 12h︰12h, 温度为(25±1)℃。
1.2 藻株基因组DNA提取
参考 Xanthogenate-SDS(XS)DNA抽提方法[20],并作适当改变用于提取藻株的基因组DNA。方法如下: 取约10 mL对数生长期的浮丝藻藻液(根据藻液密度具体情况而定)于12000 r/min离心5min, 弃上清, 将藻泥转至2 mL离心管, 每管依次加入TER缓冲液(10 mmol/L Tris-HCl, pH 7.4; 1 mmol/L EDTA,pH 8; 100 mg/mL RNase A)100 μL, 置于生物样品均质仪(MasterPrep, China)中细胞破碎6.5 m/s 60s重复2次, 液氮冻融1次。然后每管再依次加入XS buffer[1% potassium ethyl xanthogenate (Fluka, Buchs,Switzerland); 100 mmol/L Tris-HCl, pH 7.4; 20 mmol/L EDTA, pH 8; 1% sodium dodecylsulfate; 800 mmol/L ammonium acetate] 750 μL, 充分混匀, 70℃水浴2h, 其间震荡混匀数次, 冰浴30min。13000 r/min 4℃冷冻离心20min, 取上清于新的2 mL离心管, 加入等体积的异丙醇, 混匀, 室温放置 10min, 然后13000 r/min室温离心 20min, 缓慢倒掉上清, 再加入 1 mL 70%冰乙醇清洗沉淀, 12000 r/min离心15min, 缓慢倒掉乙醇, 50℃真空干燥 30min, 最后加入50 μL无菌双蒸水溶解, 置于–20℃待用。
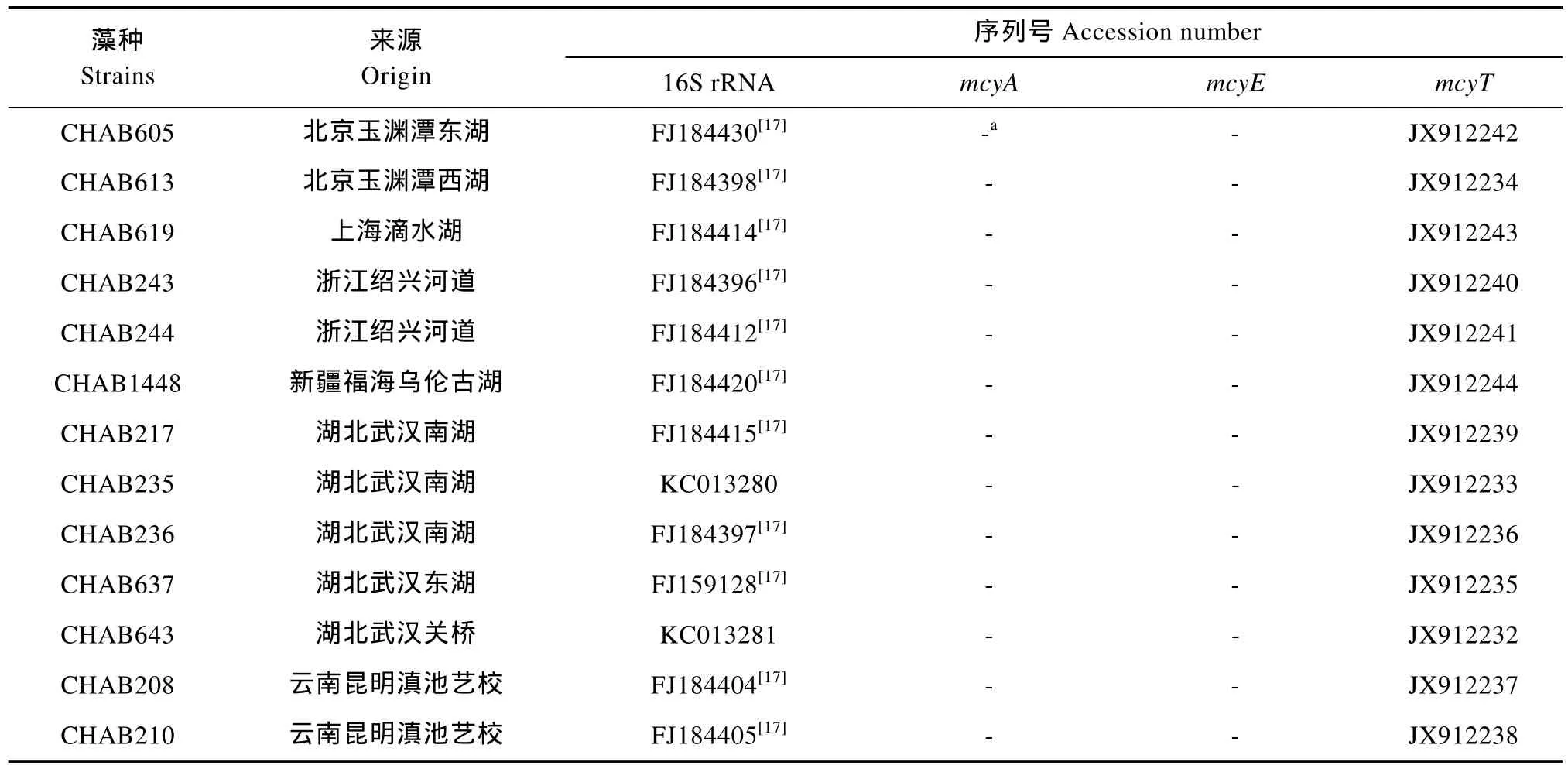
表1 13株阿氏浮丝藻的藻株信息Tab. 1 13 strains of Planktothrix agardhii investigated in this srudy
1.3 浮丝藻微囊藻毒素合成酶基因的 PCR检测、测序及分子系统分析
对本研究的13株阿氏浮丝藻藻株进行PCR检测, 选用微囊藻毒素合成相关的多肽合成酶基因mcyA、聚酮合成酶基因mcyE以及硫酯酶基因mcyT,扩增引物均由上海英骏生物技术有限公司合成, 详细信息见表2。PCR反应体系为50 μL, 包含200 mmol/L dNTP、1.5 mmol/L MgCl2、1×buffer PCR 缓冲液、10 pmol引物、1 U Taq DNA聚合酶(Takara, Japan)。PCR反应条件为: 94℃预变性 5min; 94℃变性 30s,55℃ (F1/R4)、56℃ (mcyE-F2/mcyE-R4)、59℃ (mcyACd 1F/mcyA-Cd 1R)、60℃ (mcyTA-Z+/mcyTA-Z–)退火30s, 72℃延伸1min, 35个循环; 72℃延伸5min。PCR扩增产物用 1%的琼脂糖凝胶电泳检测, 目的片段产物通过胶回收试剂盒回收(BioFlux)。将回收产物连接到载体 pMD18-T (Takara, Japan)上, 然后克隆载体转化至感受态细胞 E. coli DH5α (Takara,Japan)中, 挑取单克隆菌液送北京华大基因科技有限公司进行测序。
测序得到的序列通过 NCBI BLAST进行比对,并将本研究获得的序列和从GenBank数据库中下载的相关基因序列用Clustal X1.83软件进行对位排列整理。然后运用分子进化遗传分析软件 MEGA 5.0基于 Kimura two-parameter模型构建邻接法分子系统关系树(Neighbor-Joining tree, 简称NJ tree), 步展值(bootstrap value)设置为1000, 空位或缺失位点均当作配对删除(Pairwise deletion)处理。
2 结果
2.1 浮丝藻的微囊藻毒素合成酶基因检测
13株浮丝藻的微囊藻毒素合成酶基因检测结果见表1, 从表中可以发现, mcyA和mcyE基因均没有检测到目的产物, 只有 mcyT基因序列有目的扩增片段。mcyT基因序列PCR扩增电泳图见图1。其中编号 CYA126/8代表作为阳性对照的产毒浮丝藻 P.agardhii CYA126/8[16]的mcyT基因序列扩增片段。
2.2 浮丝藻16S rRNA基因及mcyT基因序列分子系统分析
PCR扩增得到浮丝藻CHAB 235及CHAB 643的16S rRNA基因序列全长1452 bp (序列号见表1),其余藻株的序列参考林 燊等的研究结果[17], 将这13株浮丝藻的16S rRNA基因序列与GenBank中相似序列进行序列比对整理, 截取最大可使用的序列长度约 1371 bp, 以 Planktothricoides raciborskiiNSLA3为外类群构建NJ树, 得到结果如图2所示。该分子系统树分为两大分支, 其中第 I簇主要为阿氏浮丝藻, 第 II簇主要为粉红浮丝藻藻株序列, 本实验所用13株浮丝藻均位于第I簇。

表2 实验所用引物信息列表Tab. 2 Primers used in this study
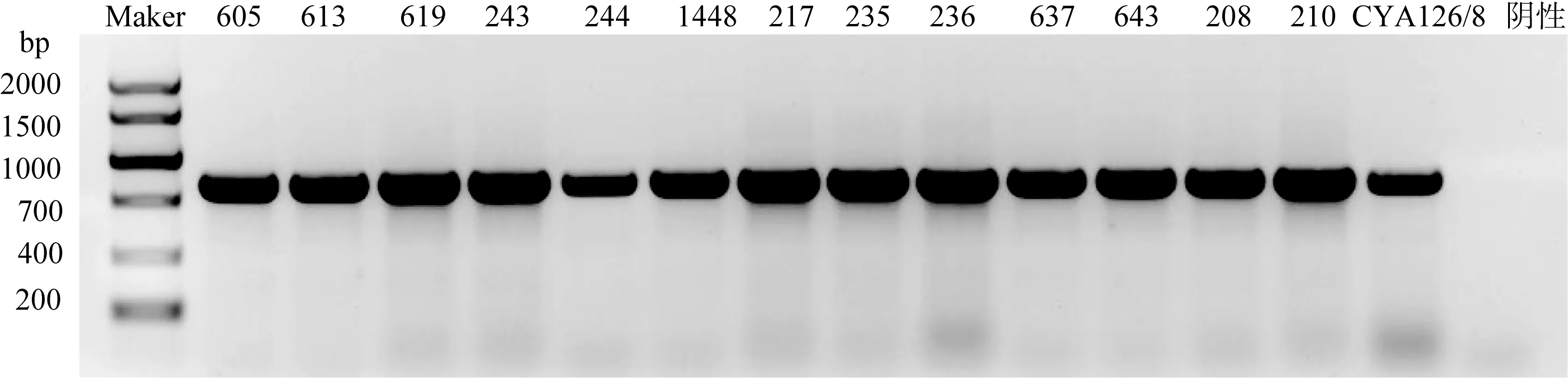
图1 浮丝藻mcyT 基因PCR扩增电泳图Fig.1 Electrophoresis result for testing mcyT gene of planktothrix

图2 基于16S rRNA基因序列的NJ分子系统树(节点处的数字代表1000重复Bootstrap值的百分数, 低于50%的未显示)Fig. 2 Phylogenetic tree (NJ) based on 16S rRNA sequences (The numbers at each node represent the percentage of bootstrap setting as 1000, only showing those above 50%)
PCR扩增得到的13株浮丝藻mcyT序列长度为703 bp(序列已上传至GenBank, 序列号见表1)。通过NCBI BLAST比对, 结果显示均与GenBank中的阿氏浮丝藻的mcyT序列相似度最高, 达到99%。将这13株浮丝藻的mcyT序列与GenBank上下载的浮丝藻的序列经比对及对位排列整理, 以 Nostoc sp.PCC 7120编码硫酯酶基因序列为外类群, 构建关于mcyT序列的分子系统树, 得到结果如图3所示。该分子系统树分为两大分支, 本研究中的13株阿氏浮丝藻位于第I分支, 并且第I分支中除这13株藻株之外的其他序列均来自不产微囊藻毒素的阿氏浮丝藻; 而第II分支中所有序列均来自产微囊藻毒素的阿氏浮丝藻和粉红浮丝藻, 两分支之间的最低相似度分别为99.1%和98.5%, 这说明基于mcyT序列的分子系统树可以将产毒与不产毒的阿氏浮丝藻区分开来。我们通过序列比对分析发现我国不产毒浮丝藻与国外产毒阿氏浮丝藻的序列存在一定的碱基差异, 其差异处如图 4所示, 从图中可以看出产毒与不产毒浮丝藻的碱基差异没有一个固定的区间, 只是零散的分布于该序列中, 而正是这些差异使得浮丝藻的 mcyT序列呈现一定的多样性, 从而在分子系统树中将产毒与不产毒浮丝藻区分开来。
3 讨论

图3 基于mcyT基因序列的NJ分子系统树(节点处的数字代表1000重复Bootstrap值的百分数, 低于50%的未显示)Fig. 3 Phylogenetic tree (NJ) based on mcyT sequences (The numbers at each node represent the percentage of bootstrap setting as 1000,only showing those above 50%)

图4 浮丝藻的部分mcyT基因序列(黑色方框代表差异位点)Fig. 4 Partial sequence alignment of the mcyT gene from strains of Planktothrix species (The box represents the variable sites)
在欧洲, 浮丝藻是一种常见产微囊藻毒素的丝状水华蓝藻, 其中阿氏浮丝藻和粉红浮丝藻是欧洲水体中最常见的两种浮丝藻种类, 两者可分别占据不同生境水域的优势地位[17]。粉红浮丝藻一般出现在分层、贫营养或中营养的深水湖泊, 而阿氏浮丝藻则分布更为广泛, 常与其他类型的浮游蓝藻如微囊藻、鱼腥藻等共存于富营养化的浅水湖泊中, 并且这两种浮丝藻种类均能够产微囊藻毒素[7,25]。Christiansen, et al.在2003年就报道过阿氏浮丝藻的微囊藻毒素合成基因簇完整序列[13], Rounge, et al.也研究报道了粉红浮丝藻的微囊藻毒素合成基因簇[14]。在我国, 浮丝藻广泛存在于我国不同地域的水体中, 甚至是某些水体的优势种类, 但是至今我国没有粉红浮丝藻的相关报道[17]。桂佳等报道过我国某水库的一株阿氏浮丝藻产微量微囊藻毒素, 但是该文献没有对其微囊藻毒素合成基因进行序列测序研究[26]。除此之外, 几乎没有其他关于浮丝藻的微囊藻毒素及毒素合成基因的相关报道。因此研究我国浮丝藻的产毒情况及毒素合成基因序列多样性十分必要, 可以为评估水体中有害蓝藻的组成及潜在危害提供一定的参考。
微囊藻毒素作为蓝藻的次级代谢产物, 其合成受到毒素合成酶基因的调控, 研究表明有些微囊藻毒素合成关键基因的缺失会导致微囊藻毒素无法正常合成, 如mcyA, -B, -D, -E和-F基因等, 而有些毒素基因如修饰酶基因 mcyJ的缺失仅能影响到藻株无法合成特异的甲基化的微囊藻毒素异构体[13]。因此可以通过特异性引物PCR检测微囊藻毒素合成的关键基因来检测蓝藻的产毒情况。该方法可用来检测纯藻株的产毒特性, 也可应用于监测环境水样毒素基因的拷贝数从而评估水华暴发期有害蓝藻的危害性。本文对分离自我国不同省份水体中的13株浮丝藻进行了16S rRNA基因序列分析及微囊藻毒素合成酶基因的检测, 分别选取毒素合成的关键酶基因序列 mcyA、mcyE及浮丝藻所特有的硫酯酶基因mcyT序列进行PCR检测。根据16S rRNA基因序列扩增及测序结果, 可以鉴定这13株藻株均为阿氏浮丝藻种类。微囊藻毒素合成酶基因检测研究结果表明这13株阿氏浮丝藻缺失mcyA及mcyE基因序列,均能检测到mcyT基因序列。mcyT是浮丝藻微囊藻毒素合成基因簇中所特有的编码硫酯酶的基因, 该基因在微囊藻、鱼腥藻等的微囊藻毒素合成基因簇中并不存在[5], 并且已有文献报道该基因在不产毒浮丝藻的基因组中也可能存在[18]。由于这13株浮丝藻仅能检测到mcyT基因, 说明这13株浮丝藻没有完整的微囊藻毒素合成酶基因簇, 属于毒素合成基因缺陷型藻株, 不具备产微囊藻毒素的能力。根据分子系统分析, 发现这13株藻的mcyT基因序列与GenBank中的无毒浮丝藻序列在NJ树中聚为一簇,与产毒浮丝藻相区分, 这与我们所检测的结果相一致。而这13株藻的16S rRNA序列在分子系统树中与产毒浮丝藻是聚在一簇的, 可以说明浮丝藻的mcyT基因序列在一定范围内能够将产毒浮丝藻与不产毒浮丝藻通过分子系统关系区分开来。关于产毒浮丝藻与不产毒浮丝藻的微囊藻毒素合成基因的序列差异性相关报道很多, Christiansen, et al.研究发现 25株无毒型浮丝藻的微囊藻毒素合成基因簇缺陷型存在 4种类型, 其中 3种缺陷型含有完整的mcyT基因序列, 而只有1株浮丝藻没有mcyT基因而独立成为一种缺陷类型[18], 这说明 mcyT序列在不产毒浮丝藻中是广泛存在的, 该结果与本实验的结果一致。
随着浮丝藻在我国蓝藻水华现象中越来越普遍,研究不同水体中的浮丝藻组成、产毒特性及毒素基因序列多样性是十分必要的。本研究中的13株阿氏浮丝藻分离自我国中东西北部部分省份, 均没有检测到毒素合成酶基因。但是由于本研究所用藻株数量偏少, 并不能代表我国大部分水体中的浮丝藻均是毒素合成基因缺陷型。因此仍然需要大量检测分离于我国不同水环境的浮丝藻的产毒特性, 以期更全面更有效地评估我国不同类型的有害蓝藻水华的危害性。而在本研究中13株浮丝藻mcyT基因之间的序列相似度很高, 并且可以与国外产毒浮丝藻序列相区别开来, 该结果可以为后续研究我国浮丝藻的毒素相关基因的多样性以及它们的分子监测提供参考。
