利用RFLP分析DO对附积床系统中AOB群落结构的影响
2014-08-07刘焕光孙凤侠甘志明谢杭冀北京工业大学北京市水质科学与水环境恢复工程重点实验室北京100124
张 岩,朱 敏,刘焕光,孙凤侠,甘志明,陈 敬,史 杨,谢杭冀 (北京工业大学,北京市水质科学与水环境恢复工程重点实验室,北京 100124)
利用RFLP分析DO对附积床系统中AOB群落结构的影响
张 岩*,朱 敏,刘焕光,孙凤侠,甘志明,陈 敬,史 杨,谢杭冀 (北京工业大学,北京市水质科学与水环境恢复工程重点实验室,北京 100124)
为了解析DO浓度对附积床反应器脱氮系统中COD、NH4+-N、TN去除效率的影响,以及对氨氧化菌群(AOB)结构及多样性的影响,分析了DO分别为1.0~2.0,2.0~3.0,3.0~4.0mg/L时COD、NH4+-N、TN去除效率,并采用针对AOB功能基因氨单加氧酶(amoA)的限制性内切酶片段长度多态性技术(RFLP)分析了三组DO浓度下反应器中AOB的群落结构及多样性.结果表明,不同DO条件下,系统均取得较高的COD和NH4+-N的去除效果, NH4+-N的去除效率随着DO的增加而提高.不同DO浓度下反应器生物膜上AOB菌群多样性丰富,且与DO对AOB菌群的多样性影响较小相比,DO对AOB的菌群结构及种类的影响较大.
氨氧化细菌(AOB);amoA基因;RFLP;生物膜
同时硝化反硝化(SND)的硝化过程首先在氨氧化细菌(AOB)的作用下,NH4+被氧化成 NO2-,继而通过亚硝酸氧化菌(NOB)将NO-氧化为NO-.23由于AOB细菌是影响硝化过程稳定性的主要微生物[1],且氨氧化作用成为硝化过程的起始和限速步骤[2],因此深入了解氨氧化细菌种类、种群结构及数量变化,有针对性地对系统进行优化调控,是确保脱氮处理系统稳定、高效运行的关键.
AOB生长速度极为缓慢,传统分离纯化方法限制了对硝化细菌的研究与应用.由于荧光原位杂交技术(FISH)、荧光实时定量 PCR、PCR-DGGE、T-RFLP等分子生物学技术避开了传统微生物培养分离环节,采用直接从样品中提取所含微生物总DNA,对DNA进行分析,这些技术已经成为现代环境微生物学研究的重要手段.如Dong等[3]利用技术研究了湿地处理猪废水系统中氨氧化细菌的种类. Winkler等[4]利用FISH、定量PCR技术分析了好氧颗粒污泥中AOB与NOB细菌比例不同的原因.Park等[5]利用 FISH技术研究了溶解氧对活性污泥中AOB菌群结构的影响.但是目前对于SND反应器中DO对AOB菌群结构以及AOB细菌的种类组成影响的研究报道较少.
附积床系统是在反应器中悬挂Bio-fix填料的生物膜与活性污泥并存的系统,Bio-fix填料可以形成厚度适中的生物膜.本课题组在前期研究中,采用 PCR-DGGE、FISH技术研究了不同C/N[6]、碳源[7]、HRT[8]、DO[9]等条件下附积床系统中微生物菌群群落结构及空间分布,但对附积床系统中AOB菌群结构、多样性及种类组成还未有研究.
DO是 SND的重要影响因素之一,研究表明
[10-12],SND系统中DO浓度为1~3mg/L时,系统脱氮效果较好.本研究将反应器 DO分别设定为1.0~2.0mg/L、2.0~3.0mg/L、3.0~4.0mg/L,对不同DO浓度下附积床反应器的COD、NH4+-N、TN去除效果进行研究,并利用限制性内切酶段长度多态性技术(RFLP)技术分析不同 DO条件下反应器中AOB的群落结构、多样性及种类的组成和变化.
1 材料与方法
1.1 实验装置与填料
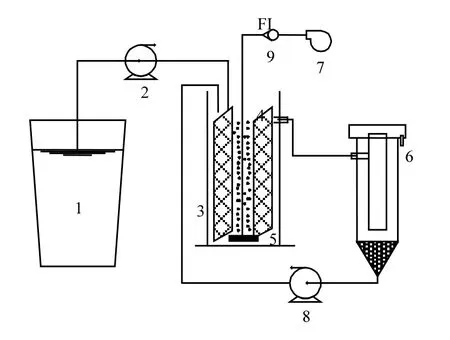
图1 附积床生物膜反应器Fig.1 Schematic diagram of catching bed biofilm reactor
如图 1所示.反应器装置主体包括原水箱、SND反应器、二沉池.反应器主体及二沉池有效容积分别为12L和16L.SND反应器内部为单槽结构,进水由蠕动泵供入底部,中间悬挂两片bio-fix填料,上部设出水口,依靠重力作用流入二沉池.二沉池为竖流式二沉池中间进水,外围出水,池底污泥依靠蠕动泵回流到反应器中.实验中采用的高亲水性生物填料 Bio-fix(BX) 主要材质为丙烯酸树脂纤维,具有很高的孔隙率和比表面积,填料的特征参数如表1所示.其表面粗糙的纹理及填料内部所撑开的空间结构,使填料能很好地固定生物膜,其上黏附的生物膜不易大面积脱落.同时,在填料中气水可以在其中自由流动,可有效防止污泥在填料上堵塞结块,保证生物膜内部微生物活性.
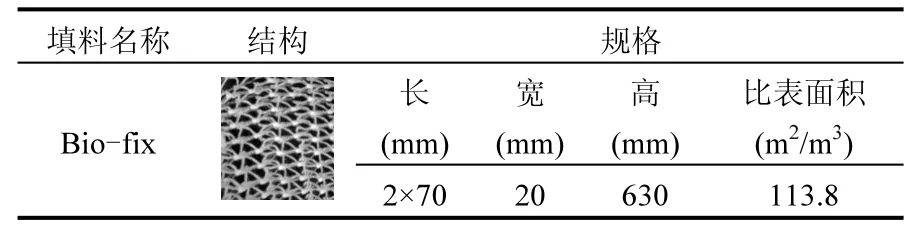
表1 填料技术参数Table 1 Characteristics of the carrier
1.2 原水水质、运行参数及分析方法
实验用原水为生活用水,取自北京工业大学西区教工生活小区化粪池上清液,ρ(COD)=159.5~675.7mg/L,ρ(NH4+-N)=55.4~116.4mg/L,ρ(TN)=61.0~119.6mg/L.
运行参数为 HRT=8h,污泥回流量为 100%,三组平行反应器 DO 分别为 1.0~2.0, 2.0~3.0, 3.0~4.0mg/L.
COD采用5B-3型快速测定仪;NH4+-N采用纳氏试剂光度法测定;NO-2-N采用N-(1-萘基)-乙二胺光度法测定;NO3--N采用紫外分光光度法测定;DO、温度采用WTW DO测定仪测定;pH值测定采用METTLER TOLEDO LE438pH计.
1.3 DNA提取
将取自生物膜上的污泥样品在5000g下离心3min,取离心后样品0.3g置于2mL离心管中,DNA样品采用土壤DNA提取试剂盒(生工生物,上海)提取,最终DNA提取液为80μL.电泳前用核酸染料Gelred将琼脂糖染色,130V,30min电泳完成后用去离子水漂洗,在254nm的紫外下观察.
1.4 PCR扩增
对于AOB的扩增,选用针对功能基amoA的引物amoA-1F(5’-GGGGTTTCTACTGGTGGT-3’)和 amoA-2R (5’-CCCCKCKGSAAAGCCTTCTTC-3’;K表示G或T;S表示C或G)[13].PCR反应体系为50μL,包括2μL模板DNA,5μL 10× PCR buffer,4μL dNTP混合液(各2.5mmol/L),上、下游引物(10pmol/μL)各 2μL,0.25μL Tap酶(5U/ μL,TaKaRa,日本),加双蒸灭菌水至50μL.PCR扩增程序如下:94℃预变性5min;94℃变性60s,50℃退火 90s,72℃延伸 90s,共 40个循环;72℃延伸10min.扩增产物采用 Agarose Gel DNA purification kit A剂盒(生工生物,上海)进行纯化,纯化过程按照试剂盒说明书进行.
1.5 amoA基因克隆及RFLP分析
将纯化后的PCR产物链接到pMD18-T载体(生工生物,上海)上,再转化到 Escherichia coli JM109(TaKaRa,日本)细胞中,质粒的转化、克隆方法参考pMD18-T Simple Vector说明书.蓝白斑筛选分别挑取80个白色菌落转接到另一培养基平板上.以挑选的克隆菌落为模板,用pMD18-T载体引物RV-M和M13-47作为PCR引物,检验筛选的克隆是否插入正确长度的片段.片段大小正确的amoA基因PCR产物用MboI (TaKaRa,日本)限制性内切酶进行酶切.酶切体系为10μL,包括PCR产物5μL, MBoI 1μL,ddH2O 4μL,酶切反应于 37°C条件下进行 1h.酶切产物用浓度为2.0%的琼脂糖凝胶电泳检验,在凝胶成像系统中凝胶成像.凝胶成像结果中 RFLP分型相同的划分为一个操作单元(OUT).
1.6 测序与序列系统发育学分析
每个OTU选取一个代表菌株送往上海生工生物公司进行测序.获得的 amoA基因序列与GenBank 进行比对(http://www.ncbi.nlm.nih. gov/blast),选取具有代表性且同源性较高的序列,利用MEGA4.0构建系统进化树.
1.7 物种多样性分析
基于限制性酶切分析结果,对3组反应器中群落的α多样性指数进行分析.3种α多样性指数Shannon Wiener指数、Simpson指数和均匀度指数的计算公式分别为:H=-∑(Pi)(log2Pi);D=1-∑Pi2,Pi=Ni/N;E=H/lnS.式中H:Shannon Wiener指数,D:Simpson指数,E:均匀度指数,Ni:为第i种酶切类型所包含的克隆数,N:总克隆数,Pi=Ni/N,S:群落中总物种数.
2 结果与讨论
2.1 反应器运行情况
如表 2所示,DO=1.0~2.0mg/L时,反应器中COD去除率为86.4%,NH4+-N去除率为89.21%, TN去除率为 33.37%;当 DO=2.0~3.0mg/L时, COD、NH4+-N和 TN的去除率均有所增加;当DO增加至3.0~4.0mg/L时,COD、NH4+-N和TN去除率分别为 87.3%、99.31%和 29.67%.在反应器运行过程中,系统受到持续性的冲击负荷,期间进水 COD最高达 675.1mg/L,NH4+-N最高达116.4mg/L,这种情况持续了17d,造成系统COD、NH4+-N、TN处理能力有所降低,但在冲击负荷消失后,系统处理能力恢复较快,说明该系统承受冲击负荷能力强,且调节能力强.从整体上看,不同DO条件下系统都取得了良好的有机物及氨氮去除效果,氨氮去除效率随着DO的增加而提高.
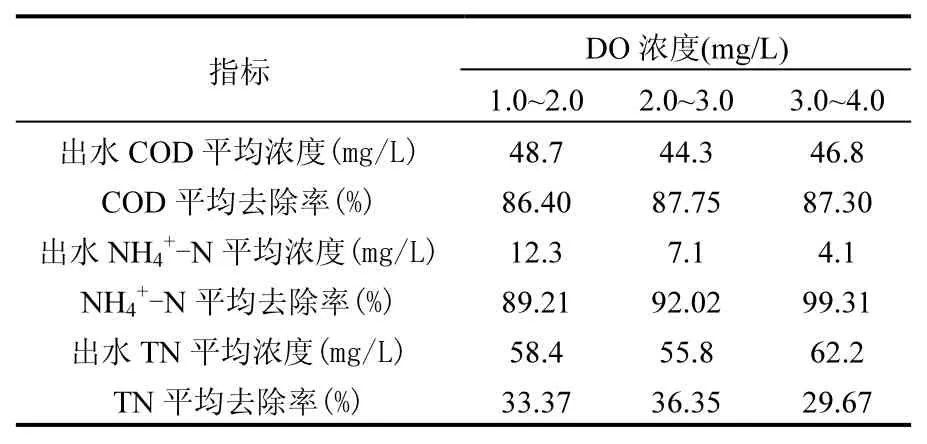
表2 不同DO条件下COD、NH4+-N、TN去除效果Table 2 COD, NH4+-N and TN removal efficiency under different DO concentrations
2.2 amoA基因的RFLP分析
在3组反应器的187个阳性克隆子中,通过酶切分型,一共得到 29个 OTUs,其中 DO=1~2mg/L时:61个阳性克隆子,8个OTUs; DO=2~3mg/L时:63个阳性克隆子,12个OTUs; DO=3~4mg/L时:60个阳性克隆子,9个OTUs.
从图2可以看出,同时存在于3组反应器的OTU只有1个,同时存在于两个反应器中的OTU有 7个.此外,每个反应器中都存在独有的 OTU类型, DO=1~2mg/L(图中M1)、DO=2~3mg/L(图中M2)、DO=3~4mg/L(图中M3)反应器中独有的OTU分别为2个、7个和5个,这些数据表明,不同 DO条件下微生物群落构成有较大的变化.3组反应器中独有 OTU分别占各组反应器 OTU总数的25%(M1)、58.3%(M2)和55.6%(M3),这些独有 OTU的克隆数占其文库的 41.0%(M1)、25.4%(M2)和35.0%(M3),说明3组反应器在氨氧化细菌构成方面相差较大,每组反应器都含有其特有的操作单元,然而从克隆数来看,这些特有的操作单元并没有成为系统中的优势氨氧化菌群.对每组反应器中含有阳性克隆子数目前三位的OTU进行分析,其阳性克隆子数总和分别占各组反应器中总克隆子数的70.5%(M1)、69.8%(M2)、71.7%(M3),其中,M1反应器中30.2%的克隆子为独有OTU,M3反应器中仅有 23.3%的克隆子数属于独有OTU,其余克隆子同时存在于两个反应器中或同时存在于 3个反应器中,说明本实验中各组反应器均存在优势氨氧化菌群,DO对氨氧化菌群有一定的筛选功能.
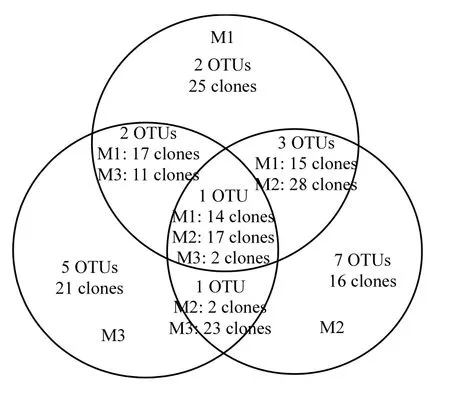
图2 不同DO浓度下反应器中氨氧化细菌amoA基因酶切类型关系的文氏图Fig.2 Venn diagram showing relationships between three different DO concentrations in RFLP pattern-based amoA gene OTUs
2.3 amoA基因文库的多样性分析
评价生物多样性的高低时,同时要考虑到物种的丰富度与均一度.环境种类丰富度越高均一度越高,则物种的多样性越高.Shannon-Wiener指数和Simpson指数既考虑到了物种数,同时也考虑到每个物种的相对多度.

表3 不同DO浓度下样品amoA基因文库α多样性指数Table 3 α diversity index of the amoA gene libraries from three samples collected at different DO concentrations
由表 3可见,DO=2.0~3.0mg/L时,Shannon-Wiener指数和Simpson指数均为最高,但是三组反应器变化不明显,说明不同DO浓度下,反应器中的物种多样性均较高,DO对物种多样性影响较小.该结果与在现有的研究报道相似, Magdalena等[14]采用 PCR-DGGE研究发现当DO从0.5mg/L提高到1.5mg/L,反应器中AOB菌群相似度基本不变,Princic等[15]通过对菌群rDNA进行限制性酶切分析发现1%、7%、21%的氧饱和度下反应器中AOB生物多样性基本相同.从均匀度指数看,DO为2.0~3.0mg/L时,均匀度指数最高,DO=1.0~2.0mg/L 和 DO=3.0~4.0mg/L时均匀度指数差异不大,说明AOB群落结构能较好地适应 DO为 2.0~3.0mg/L的环境,物种较为均匀.
通过比较不同DO条件下NH4+-N的去除效果发现,NH4+-N的去除效率随着DO的增加而提高,说明氨氧化作用基本不受氨氧化细菌多样性的影响,有研究表明氨氧化作用主要与氨氧化细菌在活性污泥中占的比例、活性以及种类有关[14,16].
2.4 AOB系统发育分析
将测序后的克隆子基因片段在 NCBI网站上进行 BLAST比对,发现大部分克隆子比对后与最近物种相似度在 90%以上.比对得到的菌群均属于Nitrosomonas,这是因为Nitrosomonas有较快的增长速度,如 N.europaea,其µmax为0.088h-1,而 Nitrosospira 的µmax 为 0.033~0.035h-1[17],这种生长优势可能使Nitrosomonas更容易成为活性污泥和生物膜系统中优势AOB[18],这与已有的研究报道结论相似[19-21].由图 3~图 5可以看出不同DO浓度下,系统中AOB细菌种类发生了较大的变化,当DO=1.0~2.0mg/L时,OTU经对比找出7种AOB细菌,当DO=2.0~3.0mg/L时,OTU经对比找出9种AOB细菌种类,而当DO提高到3.0~4.0mg/L时,OTU经对比只找出4种AOB细菌.这可能是因为DO=1.0~2.0mg/L的低溶解氧浓度环境限制了部分 AOB菌群的生长,不适应环境的菌群被淘汰,DO为2.0~3.0mg/L是AOB菌群最佳生长环境,更多的AOB菌群逐渐适应这种生长环境,AOB菌群种类丰富度提高,而当DO为3.0~4.0mg/L时,AOB菌群之间的内部竞争激烈,不适应外界环境的菌种被逐渐淘汰,其空出的生存空间被其他种类的AOB菌群所替代,AOB种类减少.
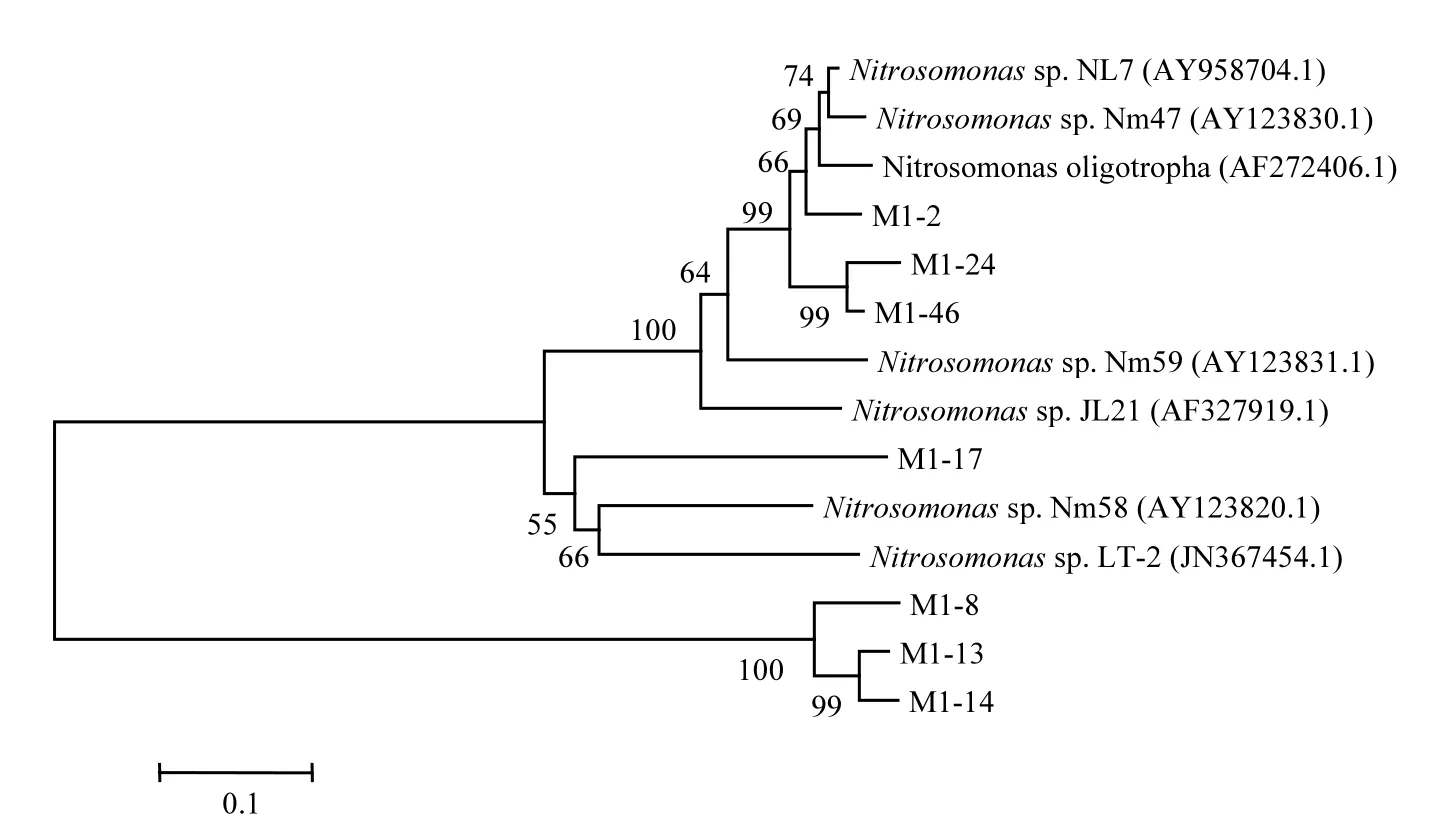
图3 DO=1.0~2.0mg/L amoA基因的系统发育树Fig.3 Phylogenetic tree based on amoA gene at DO =1.0~2.0mg/L
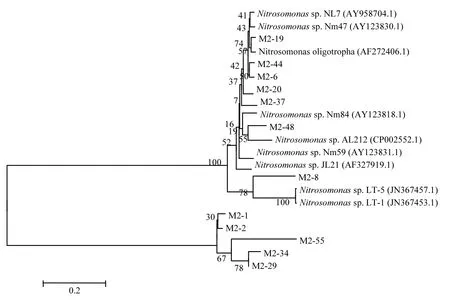
图4 DO=2.0~3.0mg/L amoA基因的系统发育树Fig.4 Phylogenetic tree based on amoA gene at DO =2.0~3.0mg/L
3组反应器中,AOB组成存在一定的差别,DO为1.0~2.0mg/L时,主要由N. oligotropha cluster(约占 88.5%)组成,同时还存在 N. communis cluster(约 占 4.9%)和 unknown Nitrosomonas cluster(约占6.6%),在DO为2.0~3.0mg/L的反应器中,N. oligotropha cluster(约占73.8%)占绝对优势,同时存在部分 unknown Nitrosomonas cluster(约占 16.2%),而在 DO 为3.0~4.0mg/L的反应器中,仅存在 N. oligotropha cluster,这种差异是由 DO浓度引起的,说明 N. communis cluster具有在低DO环境中生存的优势,这与已有的研究的结论相似[22],同时也表明,在 3组反应器中,N. oligotropha可能是优势菌群.有文献指出,N. oligotropha cluster对NH4+-N的Ks 较低,在0.027~0.059mg/L之间[23],此类AOB细菌适宜在低NH4+-N浓度的污水处理系统中,但本实验进水平均 NH4+-N浓度达 85.5mg/L,说明 N. oligotropha cluster的AOB细菌对NH4+-N浓度的耐受度不同,部分N. oligotropha cluster的AOB细菌能适应高浓度NH4+-N环境,具体种类和原因还需进一步的研究.
3组反应器均有较高的氨氮去除效率,且各反应器中AOB菌群主要由N. oligotropha cluster的AOB组成(3组反应器分别占总阳性克隆子数的 88.5%、73.8%、100%),说明 N. oligotropha cluster的AOB可能是去除氨氮的主要菌群.
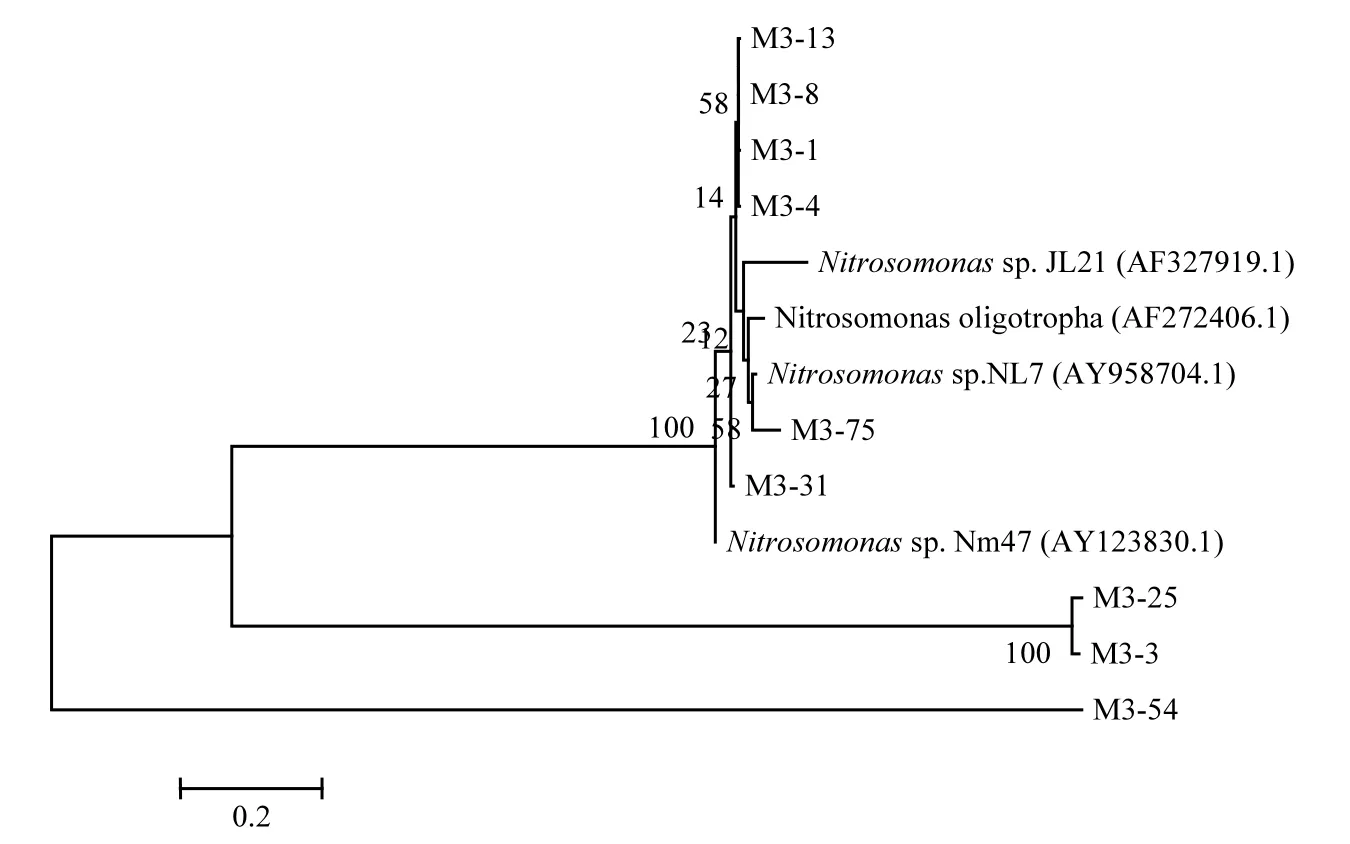
图5 DO=3.0~4.0mg/L amoA基因的系统发育树Fig.5 Phylogenetic tree based on amoA gene at DO =3.0~4.0mg/L
3 结论
3.1 DO浓度为1.0~2.0,2.0~3.0和3.0~4.0mg/L的反应器中,COD去除效率分别为86.4%,87.5%和 87.3%,NH4+-N 去除效率分别达 89.21%, 92.02%和99.31%,说明不同DO浓度下,系统均取得较高的COD和NH4+-N的去除效果, NH4+-N的去除效率随着DO的增加而提高.
3.2 对不同DO条件下反应器中AOB细菌进行多样性分析,由α多样性指数可以看出,3组反应器生物膜上AOB菌群多样性丰富,DO对AOB菌群的多样性影响较小.
3.3 DO对AOB的菌群结构和种类组成产生了较大的影响.在 3组反应器中,均存在 N. oligotropha cluster,且为优势菌群,在低浓度 DO条件下,还存在N. communis cluster和unknown Nitrosomonas cluster,均属于Nitrosomonas.
[1]Satoh H, Yamakawa T, Kindaichi T, et al. Community structures and activities of nitrifying and denitrifying bacteria in industrial wastewater-treating biofilm [J]. Biotechnology and Bioengineering, 2006,94(4):762-772.
[2]Hallin S, Lydmark P, Kokalj S, et al. Community survey of ammonia-oxidizing bacteria in full-scale activated sludge processes with different solids retention time [J]. Journal of Applied Microbiology, 2005,99(3):629-640.
[3]Dong Xiuli, Redd G B. Ammonia-oxidizing bacterial community and nitrif i cation rates in constructed wetlands treating swine wastewater [J]. Ecological Engineering, 2012,40:89-197.
[4]Winkler M K, Bassin J P, Kleerebezem R. Unravelling the reasons for disproportion in the ratio of AOB and NOB in aerobic granular sludge [J]. Applied Microbiology and Biotechnology, 2012,94(6):1657-1666.
[5]Park H D, Noguera D R. Evaluating the effect of dissolved oxygen on ammonia-oxidizing bacteria communities in activated sludge [J]. Water Res., 2004,38:3275-3286.
[6]Zhang Yan, Yang Zhengyang, Wang Lili, et al. Influence of C/N ratio on SND and microbiological analysis in catching bed biofilm reactor using acrylic resin fiber as carrier materials in civil engineering [C]//Advanced Materials Research, 2012,568: 89-93.
[7]Zhang Yan, Wang Lili, Yang Zhengyang, et al. A PCR-DGGE approach to evaluate the influebce of environment material for civil engineering-different carbon sources on a catching bed biofilm reactor in start-up phase [C]//Advanced Materials Research, 2012,568:260-264.
[8]张 岩,郭 岩,白玉华,等.附机床生物膜反应器同步硝化反硝化脱氮特性 [J]. 环境科学, 2010,31(1):134-139.
[9]Zhang Yan, Zhu Min, Liu Huanguang, et al. Influence of DO conentration on SND and microorganisms under catching bed biofilm reactor [C]//International conference on Material Science and Environmental Engineering [MSEE 2013].
[10]徐伟锋,孙力平,古建国,等.DO对同步硝化反硝化影响用动力学 [J]. 城市环境与城市生态, 2003,16(1):8-10.
[11]阮文权,卞庆荣,陈 坚.COD与 DO对好氧颗粒污泥同步硝化反硝化脱氮的影响 [J]. 应用与环境生物学报, 2004,10(3):366-369.
[12]王学江,夏四清,陈 玲,等.DO对MBBR同步硝化反硝化生物脱氮影响研究 [J]. 同济大学学报(自然科学版), 2006,34(4): 514-538.
[13]Hornek R, Pommerening-Roser A, Koops H P, et al. Primers containing universal bases reduce multiple amoA gene specif i c DGGE band patterns when analyzing the diversity of betaammonia oxidizers in the environment. [J]. Microbiol. Meth., 2006,66(1):147-155.
[14]Magdalena Z, Katarzyna B, Agnieszka C K, et al. Nitrogen removal from wastewater and bacterial diversity in activated sludge at different COD/N ratios and dissolved oxygen concentrations [J]. Journal of Environment Sciences, 2012,24(6): 990-998.
[15]Princic A, Marne I, Megusar F, et al. Ef f ects of pH and oxygen and ammonium concentrations on the community structure of nitrifying bacteria from wastewater [J]. Applied and Environmental Microbiology, 1998,64(10):3584-3590.
[16]Ebie Y, Matsumura M, Noda N, et al. Community analysis of nitrifying bacteria in an advanced and compact Gappei-Johkasou by FISH and PCR-DGGE [J]. Water Science and Technology, 2002,46(11/12):105-111.
[17]王晓慧,文湘华,杨宁宁,等.污水处理工艺对氨氧化菌及细菌群落的影响 [J]. 中国环境科学, 2009,29(6):622-628.
[18]Siripong S, Rittmann B E. Diversity stuy of nitrifying bacteria in full-scale municipal wastewater treatment plants [J]. Water Research, 2007,41(5):1110-1120.
[19]Xiaohui W, Xianghua W, Craig C, et al. Community analysis of ammonia-oxidizing bacteria in activated sludge of eight wastewater treatment systems [J]. Journal of Environment Sciences, 2010,22(4):627-634.
[20]Lapara T M, Ghosh S. Population dynamics of the ammonia-oxidizing bacteria in a full-scale municipal wastewater treatment facility [J]. Environment Engineering Science, 2006, 23(2):309-319.
[21]Rowan A K, Snape J R, Fearnside D, et al. Composition and diversity of ammonia-oxidizing bacteria community in wastewater treatment reactors of different design treating identical wastewater [J]. FEMS Microbiology Ecology, 2003, 43(2):195-206.
[22]Limpiyakorn T, Shinohara Y, Kurisu F, et al. Communities of ammonia-oxidizing bacteria in activated sludge of various sewage treatment plants in Tokyo [J]. FEMS Microbiology Ecology, 2005,54(2):205-217.
[23]Lydmark P, Almstrand R, Samuelsson K, et al. Effects of environmental conditions on the nitrifying population dynamics in a pilot wastewater treatment plant [J]. Environmental Microbiology, 2007,9(9):2220-2233.
Analysis of ammonia-oxidizing bacteria in catching bed reactor at different DO concentrations by RFLP
Z HANG Yan*, ZHU Min, LIU Huan-guang, SUN Feng-xia, GAN Zhi-ming, CHEN-Jing, SHI-Yang, XIE Hang-ji (Key Laboratory of Water Quality Science and Water Environment Recovery Engineering, Beijing University of Technology, Beijing 100124, China). China Environmental Science, 2014,34(9):2387~2393
To investigate the influence of DO concentration on the characteristics of catching bed reactor, the removal efficiency of COD, NH4+-N and TN were analyzed under the DO concentrations at 1.0~2.0mg/L, 2.0~3.0mg/L and 3.0~4.0mg/L, respectively. Meanwhile, ammonia-oxidizing bacteria (AOB) community and diversity in each reactor were examined using restricted fragment length polymorphism (RFLP), and sequencing of amoA genes. The results showed that higher COD and NH4+-N removal efficiency were
under different DO concentrations, NH4+-N removal efficiency increased with the DO concentration increase. The diversity of AOB was abundant in different DO concentrations, but the communities and species of AOB were effect greatly by DO concentration.
ammonia-oxidzing bacteria (AOB);amoA gene;RFLP;biofilm
X172
A
1000-6923(2014)09-2387-07
张 岩(1962-),女,山东烟台人,副教授,博士,主要从事污水处理与资源化方面的研究.
2014-01-22
国家自然科学基金资助项目(50978004)
* 责任作者, 副教授, yzhang@bjut.edu.cn
