豚鼠基因组26个多态性微卫星标记的筛选
2014-07-25刘迪文杨伟伟吴宝金
刘迪文,杨伟伟,吴宝金
(1.浙江大学实验动物中心,杭州 310058;2.杭州师范大学实验动物科学实验室,杭州 310036)
豚鼠(Cavia porcellus)又名荷兰猪或荷兰鼠。1780年Laviser首次用豚鼠作热源实验,此后开始实验动物化并遍布世界。因其特殊的解剖学及生物学特征,豚鼠广泛应用于生物医学研究中的各个领域。目前,国内豚鼠主要为封闭群Hartley品系,由于引进年代较长,饲养规模及繁殖过程中未能实现完全的随机交配,封闭群豚鼠的近交现象比较严重[1]。为了保持豚鼠的基因杂合度及遗传稳定性,对豚鼠遗传质量实行监测成为重要的课题,而开发一套基因组分子标记是检测的前提[2-3]。此外,在豚鼠饲育过程中,研究人员发现并分离出多个豚鼠突变株,如刘迪文等[4-5]筛选培育Zmu-1:DHP豚鼠,具有特殊的生物学性状,对口蹄疫病毒的易感性达100%,更适于制作该病的感染模型[6]。但是Zmu-1:DHP豚鼠特殊性状的分子机制尚不清楚,要解答这一问题,定位鉴定相关突变基因十分必要,但豚鼠基因组遗传标记的缺乏使得相关工作无法深入。
微卫星作为第二代分子标记,以其较高的多态性及简便的操作,能够满足一般实验室对遗传研究的要求,而得到较为广泛的应用[7-10]。对于基因组序列完全未知的物种,磁珠富集法筛选微卫星标记的报道很多,是目前微卫星标记开发广泛采用的方法。与此同时,已有不少物种的基因组序列登录在GenBank等数据库中,采用生物信息学手段为获取并筛选分子标记提供了可能。鉴于以上原因,本实验先后采用磁珠富集法和数据库筛选法对豚鼠基因组微卫星位点进行筛选及多态性探索,从而为微卫星标记在豚鼠遗传质量控制及突变基因定位等方面的应用奠定基础。
1 材料和方法
1.1 材料
1.1.1 实验动物
普通级豚鼠,雌性,6~8周龄,①DHP系,花色伴腿部黑色;②Zmu-1:DHP远交系,纯白色;③Zmu-1:DHP近交系,纯白色;④DHP系,全黑色;由浙江大学实验动物中心提供【SCXK(浙)2012-0052】;⑤卷毛豚鼠购于浙江武义县;共计5种,2-3只/种。实验在浙江大学实验动物中心进行【SYXK(浙)2012-0178】,并按实验动物使用的3R原则给予人道的关怀。
1.1.2 主要试剂与仪器
链霉亲和素磁珠#S1420S(NEB,英国);pGEM-T vector载体(Promega,美国);限制性内切酶Bsp143I及MboI(Fermentas,美国)。磁性分离架(S1506S,美国);生化培养箱(SHP-250,中国);凝胶成像系统(Tanon2500,中国);PCR 仪(Bio-Rad T100,美国)等。
1.2 方法
1.2.1 磁珠富集法获取微卫星序列
采用酚氯仿法提取豚鼠基因组 DNA,经Bsp143I和MboI酶切,回收小DNA片段,与Linker-1(P-5'-GATCGCAGAATTCGCACGAGTACTAC-3')和Linker-2(5'-GTAGTACTCGTGCGAATTCTGC-3')连接,PCR扩增,回收产物与生物素标记的 (AC)16A和(AG)16A探针进行杂交,再将杂交液和预先平衡的磁珠轻轻混合均匀,25℃孵育30 min,依次清洗,最后取 50 μL 0.1 × TE,95℃变性10 min,放到磁力架上,上清即为捕获微卫星序列,PCR扩增回收,与pGEM-T载体连接转化,筛选阳性克隆,进行PCR检测及DNA测序鉴定。
1.2.2 数据库筛选法
数据库筛选的微卫星序列来自Genome Browser Home(http://genome.ucsc.edu/)网站。操作步骤如下:1)豚鼠基因组序列的查找:点击“Genomes”,选择物种“Mammal”,“Guinea pig”;2)微卫星核苷酸重复单元的查找:点击“Tools”选择“Table Browse”,设置参数,查找并保存序列及位置信息;3)微卫星核苷酸重复单元两侧序列的查找:返回到Sequence Retrieval Region Options,在两侧各设置200 bp序列,获得含有两侧序列的微卫星序列并保存。
1.2.3 引物设计
根据微卫星两侧高度保守的序列,设计特异性引物。本实验选用两碱基重复7次及以上,三碱基重复4次以上的序列,设计引物,选择扩增长度在100~300 bp的序列。送上海生物工程有限公司合成。
1.2.4 初步及多态性检测
首先进行常规PCR初步筛选,其次选择性地进行梯度PCR扩增,通过优化Mg2+浓度(1~4 mol/L)、退火温度(45~65℃)等条件,建立最佳PCR反应体系。以五种豚鼠基因组DNA为模板,进行PCR扩增,8%聚丙烯酰胺凝胶电泳银染显色,筛选多态性微卫星。
2 结果
2.1 磁珠富集法筛选微卫星标记
2.1.1 微卫星序列的获得及类型统计
磁珠富集捕获300~1000 bp目的片段,连接转化,筛选阳性克隆,经菌液PCR初步检测(部分结果如图1),挑选阳性克隆312个,DNA测序,304个含有微卫星序列,阳性率为97.5%。经SSHunter软件统计分析:304个序列均为二核苷酸重复,类型较为单一。AC/CA占62.4%;GT/CA占20.8%;TC/AG占16.8%。
2.1.2 引物合成及多态性筛选
使用DNAStar软件针对304个微卫星序列,设计引物125条(41.12%);初步检测,特异性引物22条;多态性检测,多态性位点1个,暂未发现多态性的位点17个(结果见图2),其余4个均出现杂带,无法进行多态性判断。微卫星位点信息引物编号HZ-001;引物序列:CGTGATATGGCCTGGTCGTG ATGAAAAAGATGGGCTGGTT;微卫星类型:(AC)17(AC)21;退火温度:59℃;产物长度:208 bp;有多态性。
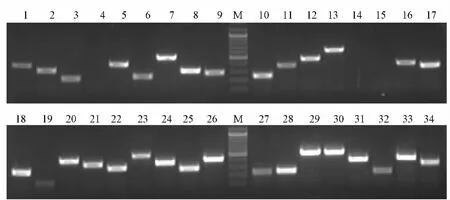
图1 菌落PCR鉴定结果Fig.1 Results of colony PCR amplification
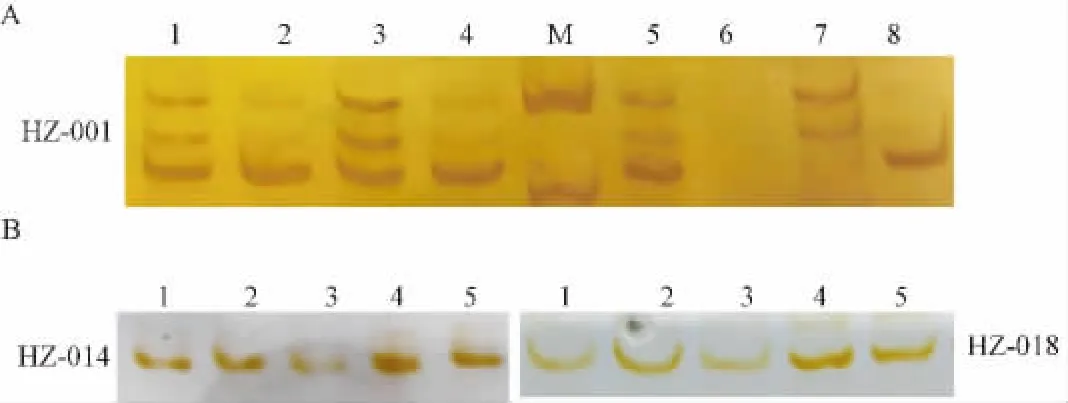
图2 磁珠富集法筛选微卫星位点PAGE电泳结果Fig.2 PAGE electrophoresis results of microsatellite loci screened using a magnetic bead enrichment protoclol
2.2 数据库筛选微卫星标记
2.2.1 基因组数据检索及类型统计
利用豚鼠基因组数据库进行部分筛选,获得微卫星序列292个,以二核苷酸重复单元为主(共计216条)占73.97%,其中TC/AG为主(149条),占二核苷酸重复单元的68.98%;三核苷酸重复单元(共计30条)占10.27%,其中以TCT较多(7条),占三核苷酸重复单元的23.33%;四核苷酸重复单元(共计39条)占13.36%,其中以TCTT较多(10条),占四核苷酸重复单元的25.64%;此外,五核苷酸重复单元和六核苷酸重复单元分别为2.06%(5条)和0.34%(1条)。
2.2.2 引物设计及多态性筛选
使用DNAStar软件针对292个序列,设计引物178条(60.96%);初步检测,特异性引物90条(50.56%);多态性检测,共获得多态性位点25个(27.78%)(如图3),暂未发现多态性的特异性扩增位点28个(31.11%),其余37个均出现杂带,有待进一步优化。微卫星位点信息(见表1)。

表1 数据库筛选25个豚鼠微卫星位点相关信息Tab.1 Characteristics of 25 microsatellite loci screened from the genome database of guinea pig

图3 数据库法筛选多态性微卫星标记PAGE电泳结果Fig.3 PAGE electrophoresis results of polymorphic microsatellite loci screened from the genome database of guinea pig
3 讨论
3.1 磁珠法和数据库筛选法筛选微卫星标记的对比
微卫星标记的开发过程需要耗费相当的人力财力,选择合适的开发方法会起到事半功倍的效果。对于基因组序列完全未知的物种,磁珠富集法筛选微卫星标记的报道很多,是目前微卫星标记开发广泛采用的方法。朱亮[11]采用磁珠富集法获得98个豚鼠微卫星序列,合成引物35对,多态性位点17个。本实验磁珠富集法获得微卫星序列304个,合成引物125条(125/304,41.12%),最终获得多态性位点1个,暂未发现多态性的位点17个。本文采用豚鼠基因组数据库筛选法进行部分搜索,其核苷酸重复单元类型较多,引物设计(178/292,60.96%)及特异性引物筛选(90/178,50.56%)的比例相对较高,多态性位点25个,未发现多态性的位点28个。总体来讲,磁珠富集法存在操作流程复杂,费用高,获得理想的微卫星序列相对较少等缺点。相比之下,数据库法具有极大的筛选利用空间。利用生物学软件从 GenBank、EMBL、DDBJ、Genome Browser Home等数据库中检索微卫星序列无疑是一个经济、简便,快速有效的方法。
3.2 豚鼠基因组分子标记技术的研究现状
豚鼠作为常用实验动物,具有十分显著的特征,如血清补体含量最高、体内不能合成维生素C,耳蜗大且听觉灵敏等,但其遗传学研究一直远远落后于其它物种,而其相关机制的研究受制于其遗传学的发展。Genbank数据库中豚鼠微卫星标记仅有6个[12],国内除朱亮等[11]成功获得豚鼠微卫星引物17对外,未见其他报道。目前,豚鼠基因组分子标记的数量尚不能满足对豚鼠遗传检测及突变基因定位的需要。本文筛选的26个多态性微卫星位点及45个候选标记是对相关工作的极好补充,候选标记在扩大样本来源及数量的基础上进行多态性检测,很可能成为多态性标记。研究者可以利用现有的豚鼠基因组序列信息进行分子标记的筛选(包括微卫星及SNP),待基因组序列组装后,可依据微卫星序列将其锚定到对应的染色体区域,故豚鼠分子标记的开发在基因组测序公布前后均应该得到重视,以促进豚鼠遗传学的发展。
3.3 关于豚鼠微卫星标记的多态性及群体特征
Burgos-Paz等[13]利用数据库中6个豚鼠微卫星标记,对哥伦比亚7个地区3个品系(本土种,改良种和宠物种共计384只)豚鼠的遗传多样性及群体结构进行研究,结果显示:等位基因丰度在3.0-6.56,观察杂合度在0.33-0.60,提示种群间的遗传分化很低。朱亮等[1]利用自主筛选的8个微卫星标记对封闭群豚鼠(1000个样本中随机选择72个)的遗传情况进行检测,等位基因丰度在2.0-5.0,杂合度在0.2222-0.5833,群体的遗传多态性处于中等水平,在繁殖过程中存在一定程度上的近交现象。以上结果提示封闭群豚鼠的遗传分化较低,群体结构单一,近交现象比较严重。
本文用26个微卫星标记检测现有5种来源的豚鼠群体,发现这些位点复等位基因数目远远低于朱亮等结果,分析原因可能是我们5种豚鼠有三个远交系来自浙江大学,亲缘关系较近,一个近交系又是由浙江大学的远交系培育而成,产生突变的可能性不大,而外购豚鼠的数量较少,这些问题都可能导致豚鼠群体的近交系数上升,微卫星标记的多态性下降,以至于本研究结果中多态性位点的比例较低。作者05年曾用DNA指纹技术分析Zmu-1:DHP和DHP品系豚鼠,发现近交系数位于0.7左右,处于中高度近交水平[14],与本文研究结果近似。关于国内豚鼠的遗传背景及检测标准有待进一步研究。
[1]朱亮,蔡月琴,屠珏,等.应用微卫星标记研究Dunkin Hartley豚鼠封闭群的遗传背景[J].中国实验动物学报,2011,19(1):51-55.
[2]李薇,江其辉,杜小燕,等.封闭群长爪沙鼠遗传标准的建立[J].中国实验动物学报,2011,28(2):31-36.
[3]李瑞生,陈振文,李晓娟,等.微卫星DNA标记在常用实验动物遗传检测中的分析[J].中国比较医学杂志,2010,20(10):75-76.
[4]刘迪文.Zmu-1:DHP豚鼠部分生物学特性研究[J].畜牧兽医学报,2006,37(5):492-495.
[5]刘迪文.Zmu-1:DHP豚鼠免疫学功能研究[J].浙江农业学报,2006,18(4):246-249.
[6]刘迪文.2个品系豚鼠对口蹄疫疫苗的免疫反应[J].中国兽医学报,2008,28(3):272-275.
[7]陈振文,赵太云,孙贺娟,等.微卫星DNA与生化标记分析对长爪沙鼠群体遗传分析的比较[J].中国实验动物学报,2006,14(4):276-279.
[8]吴宝金,茅慧华,曾咏梅,等.snthr-1Bao稀毛小鼠突变基因的精确定位及克隆鉴定[J].动物学研究,2009,30(3):267-275.
[9]施美莲,徐平,殷筱舒,等.腹侧黄斑小鼠的部分特征及其突变基因的染色体定位[J].动物学研究,2012,33(3):290-297.
[10]闻旭阳,戴广海.hMLH1基因启动子CpG岛甲基化,微卫星不稳定与胃癌[J].中国医药导报,2011,8(7):9-11.
[11]朱亮,蔡月琴,屠珏,等.磁珠富集法筛选实验豚鼠微卫星分子标记[J].中国比较医学杂志,2010,20(6):29-34.
[12]Asher M,Lippmann T,Epplen J,et al.Large males dominate:ecology,social organization,and mating system of wild cavies,the ancestors of the guinea pig [J].Behav Ecol Sociobiol,2008,62:1509-1521.
[13]Burgos-Paz W,Cerón-Muñoz M,Solarte-Portilla C.Genetic diversity and population structure of the Guinea pig(Cavia porcellus,Rodentia,Caviidae)in Colombia [J].Genet Mol Biol.2011,34(4):711-718.
[14]刘迪文,母连志.用DNA指纹技术研究Zmu-1:DHP豚鼠的群体遗传结构[J].中国兽医学报,2005,25(6):661-663.
