大豆耐盐基因的预测与筛选
2014-07-10原野
原 野
(复旦大学 生命科学学院,上海 200433)
大豆耐盐基因的预测与筛选
原 野
(复旦大学 生命科学学院,上海 200433)
本研究运用生物信息学方法和实验验证,对大豆的3号染色体上Satt339~Sat304区间内耐盐相关基因进行了结构和功能预测,筛选大豆的耐盐性状相关基因。通过实时荧光PCR技术,定量分析候选基因响应于盐胁迫。
大豆;耐盐基因;生物信息学;基因筛选
作为我国重要的经济作物之一,大豆的耐盐性较敏感,尤其在我国北方地区,可用耕地多为盐性土壤,对大豆的生长影响较大。因此,找到耐盐基因,解决大豆的耐盐问题是克服土壤盐渍化的重要前提[1],是我国农业经济可持续发展的关键。已有的研究表明,植物的抗盐性是一个由多基因控制的数量性状,其抗性机制是一个十分复杂的问题[2],涉及植物器官、组织、细胞、细胞器直至分子,既有蛋白质、核酸、碳水化合物等结构和能量物质的代谢,还有激素合成、抗氧化酶活化、离子调控与区域化、渗透调节物质合成及基因表达[3],许多基因会随环境中盐浓度的改变而改变表达量[4]。而过高的土壤含盐量会对植株造成包括水分胁迫、离子胁迫、光合作用下降等多方面不利影响[5-12],等。国外文献中刊载的相关研究[13-16]是针对农作物耐盐性的研究[12,15,17,18],Mansour(2004)等主要是和对植物耐盐性状分子机理的研究。以往的研究中,对于大豆的耐盐性状研究尚显薄弱。正是基于此现状的启发,确定以大豆为研究对象,应用Softberry的ORF预测功能和美国国立生物技术信息中心(NCBI)的BLAST检索系统获取大豆基因的基因结构并进行生物信息学分析,了解基因可能的功能,高效地缩小大豆耐盐候选基因的范围。在得到大豆耐盐候选基因后,通过对大豆植株进行DNA酶切和PCR扩增,验证候选基因的存在。继而对大豆植株进行盐胁迫,通过运用实时荧光PCR技术,定量验证目的基因响应于大豆植株受到的盐胁迫。
1 材料与方法
1.1 实验材料
实验所用合丰39大豆种由上海交通大学生命科学学院提供。
1.2 大豆耐盐基因的筛选
大豆耐盐候选基因的筛选即通过生物信息学方法,分析大豆基因序列,在不少于1.4Mbp的序列内分析基因功能,预测与大豆耐盐性相关的基因。通过phytozome网站,得到大豆的全基因组序列,并在标记基因JD33前后,在Gm03染色体上标记基因Satt339~Sat304之间,选取约1.4Mbp的DNA序列,通过Softberry的FGENESH分析,得到预测基因序列。
通过NCBI的BLAST分析 (蛋白质序列),将预测的ORF(开放阅读框)与基因库(Gene Bank)的双子叶植物数据库进行比对,筛选同源序列。根据序列相似程度(E值、Score值)以及基因组成,对基因进行综合分析,从而排除不完整的基因片段。
根据已注释的直系同源基因的功能对大豆ORF的功能进行预测,确定与耐盐相关的候选基因。
1.3 候选基因的PCR验证
大豆耐盐阈值,即当外界土壤或水中盐浓度 (以NaCl计)超过时植株生长开始受到严重影响,如枝叶萎靡、生长骤缓、叶片出现斑点等,是大豆耐盐性的重要指标。测定合丰39大豆耐盐阈值,即作为植株盐胁迫实验的标准浓度。
选饱满、色泽亮黄的大豆种,分组置于培养皿中,置于干燥器中,加100ml NaClO溶液(84消毒液)与1.5ml 37%HCl,灭菌24小时。将大豆种置于1.2%水琼脂培养基上,4℃春化24小时;转入光照培养箱中,培养3~5天。大豆出芽后,转入50% 霍格兰氏培养液,光照培养箱中,光照16小时/天,23℃持续水培。
配置含盐霍格兰氏工作液 (50%浓度),NaCl浓度梯度为 0mmol/L、0mmol/L ( 对 照 组 )、20mmol/L、40mmol/L、60mmol/L、80mmol/L、100mmol/L、120mmol/L、150mmol/L, 共9组,每组9或10株,光照16小时/天,24~25℃,每3日更换含盐培养液,持续水培。
第二周期在前一周期试验的基础上,NaCl浓度梯度为0mmol/L(对照组)、40mmol/L、45mmol/L、50mmol/L、55mmol/L、60mmol/L,共六组,其余条件同上。
盐胁迫两周后取样。随机抽取每组植株一株,记录植株生长情况,包括第一、第二对真叶叶长、根长、茎长和植株出现的特殊状况,如叶上长癍,茎叶萎靡等。观察分析得到实验中合丰39大豆的耐盐阈值。
通过生物信息学方法,我们已经获得了大豆耐盐相关的候选基因。但其中很有可能仍包括一些不完整的基因片段。于是,我们对候选基因进行实验验证。
在对候选基因的进一步分析、选优过程中,我们发现个别基因过短、保守域在基因中所占比例过大,难以设计合适的PCR引物的情况。借助计算机软件Primer Premier 5.0,设计CG含量介于40%~60%、一对3’~5’和5’~3’引物退火温度相差不超过5℃、引物长度介于18~25bp之间、PCR产物在300bp以上的对应引物,并尽量确保没有发卡或回文结构。
采用合丰39大豆耐盐阈值浓度50mmol/L NaCl的50%霍格兰氏培养液,对水培植株进行盐胁迫12小时,共5株。
取每棵植株第一片、第二片真叶各一片,一部分根。剪下植株组织后迅速置于10ml离心管中,标记后置于液氮中保存。
使用康为RNA pure Plant Kit试剂盒提植物RNA,将获得的 RNA溶液10μL, 向其中加入 1μL oligodT(50mmol/L),混匀离心,置于70℃水浴5分钟。取出后立刻冰浴5分钟。后向其中加入:
4μL 5×反转录酶的 Buffer;1μL反转录酶;0.5μL RNA酶 抑 制 剂 ;2.4μL Mg离 子 溶 液 (2.5mM);1.1μL dNTPs(2.5mM)混匀后,25℃水浴5分钟,再置于42℃水浴1小时,得到cDNA。
使 用 温度梯度 PCR方 法, 从 51、53、55、56、57℃至59℃,共六个温度梯度,40个循环,平行进行PCR扩增:
25μL System:2.5μL dNTPs;2.5μL 10×Taq buffer;0.5μL模板 cDNA;0.25μL正向引物S;0.25μL反向引物A;18.5μL水;0.5μL Taq酶。
配置1%琼脂,冷凝后加入样本、Marker,110V电泳,30分钟后用EB染色,拍照。根据Marker条带,估计PCR产物大小。并根据样本条带亮度,与对照组对比,估算浓度。
通过实验,排除未能正常扩增的基因片段或不响应与大豆盐胁迫的基因。
1.4 实时荧光定量PCR测定预测基因表达量
通过普通PCR扩增与琼脂糖凝胶电泳实验,可以证明实验选取的一部分基因是在大豆植株中存在并与耐盐有关的。而实时荧光定量PCR实验可以定量检测预测基因在大豆植株中的表达量,并测定其改变量。因此,我们可以分析检测同一基因在受不同时间盐胁迫植株中的表达量,确定预测基因与大豆耐盐的相关性。(步骤同验证候选基因)
采用合丰39大豆耐盐阈值浓度,含50mmol/L NaCl的50%霍格兰氏培养液,对水培大豆植株进行盐胁迫,分别进行盐胁迫0小时、3小时、12小时、24小时、3天、5天,共6组,每组5株。按照相应胁迫时间进行取样 (取样方法同1.3.2)。
使用康为RNApure Plant Kit提植物RNA,反转录为cDNA。(提取、反转录方法同步骤同验证候选基因)PCR退火温度为57℃,40个循环,每组5个样品,分别将模板中cDNA浓度稀释1倍、10倍、100倍、1000倍、10000倍,加入SYBR Green荧光染料,平行进行实时荧光定量PCR扩增。
最终确定预测基因的表达式样与大豆所受盐胁迫的相关性。
2 结果与讨论
2.1 大豆耐盐基因的筛选结果
2.1.1 ORF预测结果 通过SoftBerry网站的相关软件,我们以大豆基因组中1167011bp的DNA序列为基础。预测了ORF共211个,其中113个位于正链,98个位于负链,平均长度1007bp;967个外显子,平均长度220bp。
2.1.2 同源基因检索结果 通过在NCBI双子叶植物基因数据库中进行BLAST检索,211个预测基因中86个ORF没有发现相似的序列,被淘汰;125个ORF与其它双子叶植物基因有较强的相似性。根据已注释的大豆及其它双子叶植物基因的功能,从125个ORF中筛选出25个可能与大豆耐盐相关的抗盐候选基因。其功能涉及包括离子交换和调控基因表达等。
2.2 大豆耐盐候选基因PCR验证结果
2.2.1 合丰39大豆的耐盐阈值测定结果
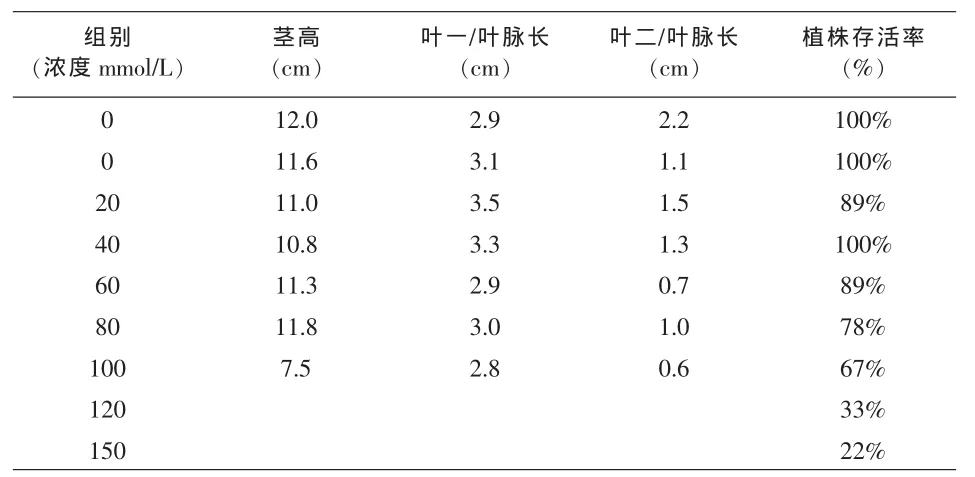
表1 第一周期耐盐范围测定的实验结果
通过分析表1数据,盐浓度在120mmol/L及以上的情况下,植株死亡过半,这种环境对于植株的胁迫作用相当明显,极大抑制植株的生长。盐浓度在60mmol/L及以上的情况下,植株的叶子生长情况较差,叶片普遍偏小且下垂、黄萎,这种环境对于植株生长有比较大的胁迫作用。20mmol/L组共有9株样品,其中有一株从第一对真叶展开后生长明显迟缓,而后逐渐黄萎,在培养两周后枯萎,个体异常造成该组存活率异常。综合以上结果,可以分析得到合丰39大豆的耐盐极限大致在40mmol/L—60mmol/L NaCl。

表2 第二周期耐盐范围测定的实验结果
通过表2我们可以看出,当盐浓度超过50mmol/L后,第一片真叶黄萎、枯死明显,第二片真叶虽都存活,但植株叶与根的生长都受到一定影响。经过第二周期实验,合丰39型大豆的耐盐阈值可以确定在50-55mmol/L NaCl。
2.2.2 候选基因PCR验证结果
通 过 琼 脂 糖 凝 胶 电 泳 实 验 ,ORF20、ORF31、ORF33、ORF42、ORF62、ORF63和ORF79,都没有明显可见条带,很有可能是不直接响应于大豆盐胁迫,遂将其排除;其它基因都基本正常扩增,证实它们在植株中能够正常表达,确定候选基因13个。
2.3 实时荧光定量PCR结果
实验一共进行了两组,第一组将13个精筛后的候选基因和Tube、AS,共15个样品进行实验操作,绘制扩增曲线,分析DNA溶解温度,最终确定10个候选基因,)进行第二组定量PCR扩增,精确测定表达量。
实验数据采用2-ΔΔCt法处理,即以基因在每组胁迫0小时植株的根中的表达量为标准量1,计算相对表达量。实验结果按根中的表达量可分为3种:
(1)单调增加。ORF179和ORF202在根中的表达量都是随盐胁迫时间成线性上升,说明该基因的表达是与植株耐盐性状正向相关的。如ORF179是PHD finger蛋白家族相关基因,参与到染色体状态的调控和基因的转录调控等[9],该基因在盐胁迫下的表达量增加,很可能是植株克服逆境的重要机制。
(2)先增高后降低。这些基因可能是植株的某种应急机制。在受到盐胁迫时,植株立即响应,提高某些基因的表达,对抗不利环境,随后在其它基因的陆续作用,或外界不利条件加剧的影响下,基因表达量再降低。如ORF160是亮氨酸拉链相关基因,是植物基因调控的重要因子,其在根中表达量上升又下降,很可能是受其他基因的影响,在短时的高表达之后,作用渐渐被其他基因所取代。
(3)单调下降。对于盐胁迫时间长而表达量单调下降的基因而言,是与盐胁迫反向相关。这样的基因可能是受到盐胁迫,表达被抑制。

图1 实时荧光定量PCR
3 结论
实时荧光定量PCR的结果显示,ORF179和ORF202是与大豆所受盐胁迫成正相关的。说明其不仅响应于大豆植株所受的盐胁迫,还很有可能调控大豆其它相关基因的表达以对抗高盐浓度对植株造成的损害。
ORF123等基因在根中的表达量都呈现先增高,后降低的过程。如ORF123,基因功能可能是Na+/H+逆向运输蛋白相关的基因,能降低胞质Na+浓度,减轻Na+对酶类和膜系统的伤害,但之后其表达量又有所下降,推测是因为随着盐胁迫时间的增长,盐溶液对植株的毒害作用逐渐增大,对其表达产生了影响;而其它基因功能可能为转录因子,其先增高后降低的趋势,可能是一种预警或应激机制,与耐盐性状并非直接相关。
ORF61在根中的表达量随着盐胁迫时间的增长而下降,属于与盐胁迫反向相关。
综上所述,与盐胁迫正向相关的基因ORF179 PHD finger transcription factor-like protein,位于大豆基因负链上;ORF202 ccaat-binding transcription factor subunit A,位于大豆基因正链上,是两个调控基因。预测基因功能与离子转运有关的基因ORF123 Na+/H+antiporter-like protein,位于大豆基因正链上,是结构基因。根据多步实验的逐级验证,这三个基因在大豆植株中与其耐盐性状相关。
[1]余玉雯.大豆耐盐相关基因的分离及其功能鉴定[J].深圳大学学报理工版,2004,21(4):324-326.
[2]孙文喜,孙文鑫,周大旺,等.植物抗盐性生理生化研究进展及提高植物抗盐性对策[J].农业科技通讯,2013:174-177.
[3]吴敏.植物盐胁迫适应机制研究进展[J].林业科学,2007,43(8):111-117.
[4]佟友丽.植物抗盐胁迫研究进展[J].生物技术通讯,2008,19(1):138-140.
[5]曾洪学.盐害生理与植物抗盐性[J].生物学通报,2005,40(9):1-2.
[6]管清杰.水稻质膜H+-ATPase基因对拟南芥遗传转化及其抗盐性分析[J].分子植物育种,2007,5(4):451-454.
[7]陈敏 植物质膜H+-ATPase响应盐胁迫的分子机制[J].植物生理学通讯,2006,42(5):805-811.
[8]常红军.植物的盐胁迫生理 [J].安阳师范学院学报,2006:149-152.
[9]王晓栋.10份豆科牧草种质材料耐盐性研究[J].内蒙古农业大学,2008.
[10]张楠楠.植物抗盐机理的研究[J].哈尔滨师范大学:自然科学学报,2005,21(1):65-68.
[11]Tsui-Hung Phang.Salt Tolerance in Soybean[J].Journal of Integrative Plant Biology,2008,50(10):1196-1212.
[12]Huang J.Genetic engineering of giycine betaine production toward enhancing stress tolerance in plants:metabolic limitations[J].Plant Physiology,2000,122:747-756.
[13]Baoshan Wang,Kefu Zhao,Qi Zou.Advances in mechanism of crop salt tolerance and strategies for raising crop salt tolerance[J].Chinese Bulletin of Botany,1997,14:25-30.
[14]Mansour M M F,Salama K H A.Cellular basis of salinity tolerance in plants[J].Environmental and experimental botanty,2004,52:113-122.
[15]Xiong L,Schumaker K S,Zhu J K.Cell signaling during cold,drought and salt stress[J].Plant Cell,2002,14(Suppl):165.
[16]Kawasaki S J,Borchert C,Deyholos M,et al.Gene expression profiles during the initial phase of salt stress in rice[J].Plant Cell,2001,13(4):889.
[17]Ma S,Gong Q,Bohnert H J.Dissecting salt stress pathways[J].J Exp Botany,2006,57(5):1097.
[18]Zhu Jiankang.Regulation of ion homeostasis under salt stress[J].Cur Poin Plant Bio,2003,6:441-445.
[19]张帆,杨仲南,张森.拟南芥PHD-finger蛋白家族的全基因组分析[J].云南植物研究,2009,31(3):227-238.
[责任编辑:钱立武]
Q344
A
1674-1104(2014)03-0158-03
10.13420/j.cnki.jczu.2014.03.050
2014-03-14
原野(1992-),男,山西黎城人,复旦大学生命科学学院学生,研究方向为植物生理及生物进化。
