基于SVM的氨基酸频率计算预测水稻蛋白质磷酸化位点
2014-07-10何华勤
王 伟,何华勤
(福建农林大学,福建 福州 350002)
基于SVM的氨基酸频率计算预测水稻蛋白质磷酸化位点
王 伟,何华勤
(福建农林大学,福建 福州 350002)
本文从swiss-prot中选取经过试验验证的水稻蛋白质磷酸化位点数据作为训练集合,应用蛋白质序列的氨基酸频率计算方法来进行特征提取,再利用SVM算法构建专门针对水稻蛋白质磷酸化位点的预测新工具.氨基酸频率算法指的是计算出相应待预测磷酸化位点附近氨基酸的出现频率,进一步反映了残基之间的相关性.本文利用LibSVM软件包对已通过氨基酸频率算法特征提取出来的数值特征对磷酸化位点进行预测,从而为之后构建水稻蛋白质磷酸化位点的预测工具做准备.结果表明,本文基于SVM和氨基酸频率方法的水稻蛋白质磷酸化位点预测在丝氨酸,苏氨酸和酪氨酸的平均预测准确性为77.665%,马修斯系数为0.571.与Plant Phos和Musite的预测性能的对比结果显示,在磷酸化苏氨酸位点的预测性能显著高于Plant Phos及Musite.
LIBSVM;SVM;氨基酸频率计算;磷酸化位点
1 水稻蛋白质磷酸化位点的预测
由于蛋白质领域研究的日益进步以及基因测序、编码技术的普及,各大数据库中已经大量收集了各种蛋白质的氨基酸序列.因为蛋白质组学研究的重要领域是蛋白质功能,因此研究蛋白质序列已经成为生物信息学中不可或缺的部分[1][3].Vapnik和Cortes于1995年首先提出支持向量机(全名Support Vector Machine)这一概念,它的基本原理是在线性可分的基础上,通过自身的算法将线性可分变为线性不可分[2].通过此转变我们可以在非线性函数中进行使用和计算,这种分类算法被称为支持向量机,即SVM.将支持向量机算法应用到水稻蛋白质磷酸化位点的预测当中去,是现在研究水稻蛋白质磷酸化的一个重要方向.
研究水稻蛋白质磷酸化的三个主要目的:
(1)对位于某一特定状态下水稻细胞内磷酸化蛋白质的序列及磷酸化氨基酸残基定位;
(2)鉴定与磷酸化过程有关的激酶;
(3)分析所观察到的磷酸化现象对功能的影响.其中,第一个目的是磷酸化研究的主要任务和基础.
所以研究蛋白质序列已经成为生物信息学中一个重要的、不可或缺的部分.
2 SVM简介
支持向量机在应对高维模式识别、非线性及小样本中展现出了它的不可比拟的优势,并在其他机器学习问题、函数拟合等问题中都能够得到很好的应用.
SVM方法是在统计学理论中的VC维理论以及结构风险最小原理的基础上建立的,根据有限的样本信息在模型的复杂性,即对以经过选定的训练样本的学习精度,准确度以及学习能力,即无错误地识别任意样本的能力,之间寻找到最合理和最稳定的方案,从而能够有机会获得最好的推广能力,也可称作泛化能力[5].
3 LIBSVM简介
LIBSVM是一款涉及回归算法与模式识别的软件包,并具有高效快捷、简单易用等特点,该软件由台湾大学林智仁副教授等研制开发的.由于LIBSVM中对SVM的参数筛选方面的支持较少,因此使用了经过大量验证的默认参数进行替代,而大多数相关问题都可以通过这些默认参数进行解决;交叉检验(Cross-Validation)功能还被该软件包集成在其中.同时还可以解决包括基于1对1算法的多类模式识别问题,以及c-SVM、V-SVM、ε-SVR和V-SVR等问题.
4 基于氨基酸频率的特征提取算法
首先我们将所获得的数据集进行excel表格化整理,把蛋白质序列一一存储到表格中.在正样本中每一行必须标有已被磷酸化的位点信息,即已被磷酸化的位点在序列中的位置.通过编程写出函数,该函数的功能是截取该序列的25个残基.即以磷酸化位点为中心截取该片段的上游和下游各12个氨基酸,此片段包括磷酸化位点共计25个氨基酸.到此为止我们拥有了计算过程中所要的重要数据.
然后将这包含有25个氨基酸的残基片段放进一个数组中,该数组放在单独计算频率的子函数中,为后面算出每段包含有25个氨基酸残基的氨基酸频率作准备.最终经由以上过程,可算出该残基序列中的上游和下游各12个氨基酸出现的频率,并将这25个所提取出来的特征数值作为后面将要预测磷酸化位点的特征值.
5 SVM模型的建立
本文用到的SVM核类型为RBF,并且使用的SVM类型为C-SVC[5].
RBF的核函数为:
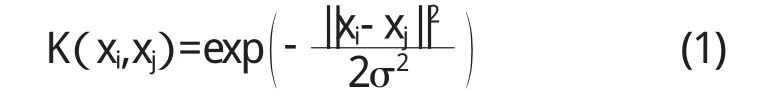
σ代表串口的宽度
(2)C-SVC即C-支持向量分类.给定(xi,xj), i=1,2,…,L,y∈{1,-1}.SVM需要以上优化问题的解决方法,其中ξi≥0
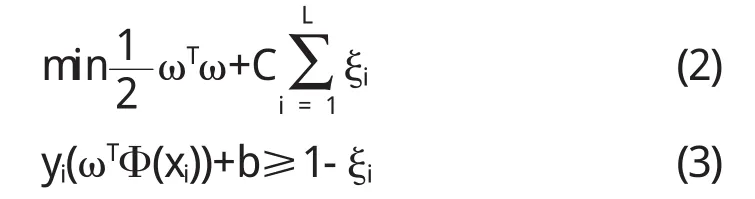
更高维空间中具有最大化边缘的线性分离超平面我们使用SVM算法可以找到.错误项的惩罚函数我们用C<0来表示[5].决策功能为:
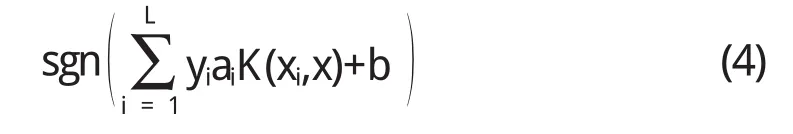
6 模型的建立与评估
首先我们从已获得数据集合中提取一部分作为测试集,也就是选取部分数据来进行训练.对于要进行预测的数据,为了避免人为干扰,我们分别从总数据集合的数据中随机抽取十次正负样本,选取的正负样本比例为1:1.
在利用libSVM进行预测之前,使用交叉验证对所提取的特征值进行评估和测试,得到不同的Cost值和Gamma值后,从中选取模型所需的最优参数.通过比对我们选取rbf核类型和c-svc类型来创建模型.SVM中模型是通过正负样本集来构建的,并且正负样本比例为1:1.对于易为磷酸化的S(丝氨酸)、T(苏氨酸)和Y(酪氨酸)的子集,分别从相应总训练集的正负位点数据中随机抽取十次正负样本[7].
分别对每个序列子集的10个SVM模型进行交叉验证,通过对结果的比对和分析分别从中选取交叉验证性能最高的模型作为SVM的子模型.通过libsvm中的grid.py进行参数优选得出最优参数训练出最终模型.再通过此模型,应用svm_predict进行预测.
预测结果:
虽然参数优选中的最佳准确率accuracy=76.965%,但实际中预测的准确率为accuracy=77.665%.
7 评价指标
通过Sn(灵敏度)、Sp(特异性)、ACC(准确度)和MCC(马修斯系数)对该算法的性能进行评价.
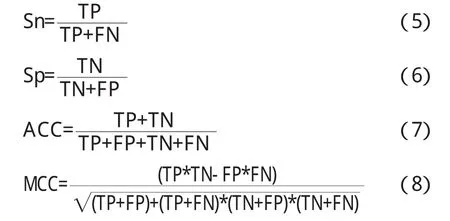
其中,TN表示的是实际为负样本的序列数目在预测结果中也为负样本.TP表示实际为正样本的序列数目在预测结果中也为正样本.FP表示实际为负样本的序列数目却在预测结果中为正样本.FN表示实际为正样本的序列数目却在预测结果中为负样本[7].MCC的值越大表示预测结果越好,其取值范围为-1至1.
通过在Python编程环境下,自己编写的评价指标函数得出个评价参数
该方法的各评价指标:SN=0.789,SP=0.761,ACC=77.6%,MCC=0.495
8 主要工具的对比
磷酸化位点预测工具有很多,但正式的专门针对水稻蛋白质磷酸化位点的预测工具和方法却是空白,而前人开发了针对植物蛋白质的磷酸化位点的预测工具,然而如phosPhAT以及2008年才研制的Gaoetal工具.它是一款基于SVM的蛋白质磷酸化位点预测工具,该工具是整合K近邻信息(KNN)、蛋白质序列信息和蛋白质无序区域而构建的.然而唯独phosPhAt提供可靠并且较为稳定的在线预测服务.数据测试方面,本文使用的是自己构建的独立测试集来,使用此数据来测试本文方法与Plantphos和Musite的预测性能.
Plantphos:
Plantphos应用MDD,即最大依赖性分解方法,把所有的磷酸化片段进行聚类,形成具有显著位点特异性的磷酸化片段子集.为了搜索HMM的采样数,HMMER会返回一个HMMER值和期望值,即E值[8-10].
Musite:
Musite是一款几乎适合于所有或特定激激酶的磷酸化位点的预测工具.它能够将磷酸化位点的预测作为为一个失衡的分类问题来看待,使用的是机器学习的方法.该工具收集了多种生物体磷酸化蛋白质组的可靠实验数据,用这些数据来训练磷酸化位点的预测模型.Musite工具中使用到了k最近邻方法(KNN)和蛋白质无序区域特征提取的方法.所谓无序区域,即缺乏一个稳定的第三结构蛋白质的部分[11].
9 不同预测方法的性能比较
依照上述,本文应用自己构建的测试数据集来与Plant Phos和Musite的预测性能进行对比.我们将本文的预测方法和Plant Phos、Musite对同一测试集数据进行预测,首先将数据分成1:1的正负样本集,即磷酸化和非磷酸化位点.然后算出这三种方法的Sn(灵敏度)、Sp(特异性)、ACC(准确度)和MCC (马修斯系数)来比较各自的预测性能,结果见表1.
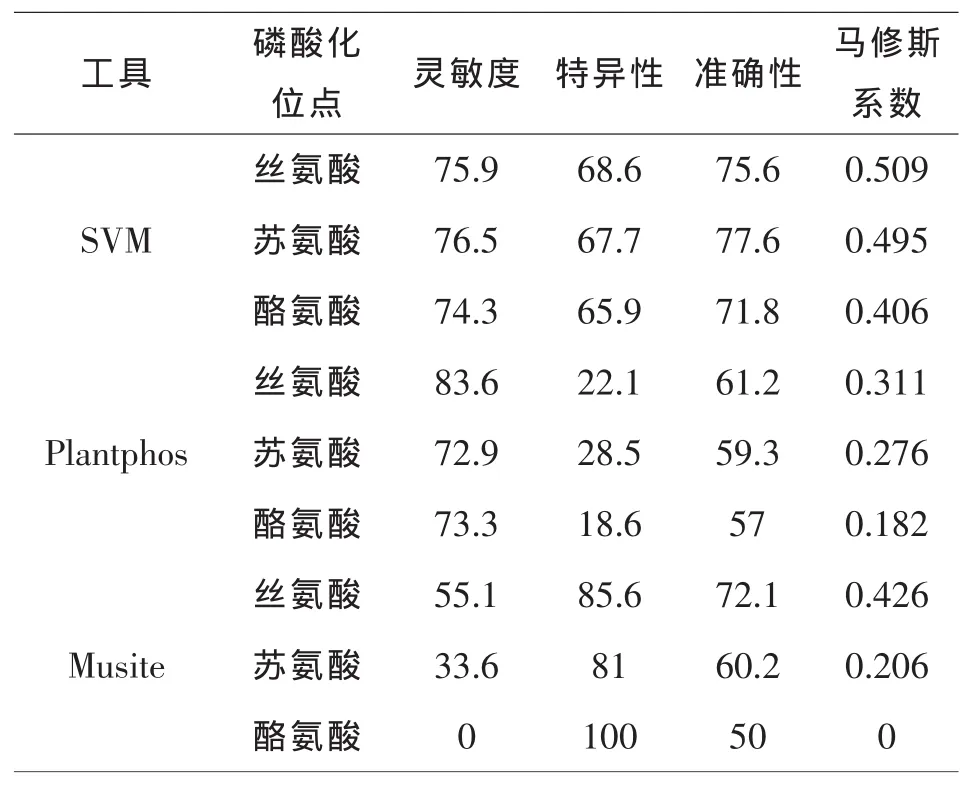
表1 为本文方法和各方法对独立测试数据集的预测结果
由表可知,本文的预测工具对丝氨酸预测的准确性ACC和马修斯系数MCC分别为75.6%和0.509,plantPhos的准确性ACC为61.2%和马修斯系数MCC为0.311,而Musite预测的准确性ACC和马修斯系数MCC分别为72.1%和0.426.表明本文的预测工具对磷酸化丝氨酸位点的预测性能高于PlantPhos及Musite.
而本文的预测工具对酪氨酸位点预测的准确性ACC和马修斯系数MCC分别为71.8%和0.406,plantPhos的准确性ACC为57.0%和马修斯系数MCC为0.182,而Musite预测的准确性ACC为50%,而马修斯系数MCC却为0.表明本文的预测工具对磷酸化苏氨酸位点的预测性能高于PlantPhos及Musite.
本文的预测方法在预测苏氨酸位点的准确性ACC和马修斯系数MCC分别为77.6%和0.495,显著高于PlantPhos的准确性ACC为59.3%和马修斯系数MCC为0.276,以及Musite的准确性ACC为60.2%和马修斯系数MCC为0.206.说明本文的预测工具对磷酸化苏氨酸位点的预测性能显著高于PlantPhos及Musite.
〔1〕张颖,罗辽复,吕军.使用多样性增量预测磷酸化位点.内蒙古大学学报(自然科学报)2008(1).
〔2〕朱玉贤,李毅,郑晓峰.现代分子生物学(第三版).
〔3〕蔡津津.蛋白质磷酸化位点预测与规则抽取方法研究.中国科学院计算技术研究所.
〔4〕姜铮,王芳,何湘,等.蛋白质磷酸化修饰的研究进展.中国人民解放军疾病预防控制研究所,2009.
〔5〕赵凌志,刘颖,等.WeightedSVM在蛋白质磷酸化位点预测中的应用.清华大学软件学院,2006.
〔6〕白海燕,吕军,张颖,等.蛋白质磷酸化位点的识别.内蒙古工业大学学报,2011(2).
〔7〕Koenig M ,Grade N.Highly specific prediction of phosphorylation sites in proteins [J].Bioinformatics, 2004.
〔8〕Lee TY, Lin ZQ, Hsieh SJ, Bretana NA, Lu CT: Exploiting maximal dependence decomposition to identify conserved motifs from a group of aligned signal sequences.Bioinformatics 2011, 27(13):1780-7, 1.
〔9〕Burge C, Karlin S: Prediction of complete gene structures in human genomic DNA.J Mol Biol 1997, 268(1):78-94.
〔10〕Diella F, Gould CM, Chica C, Via A, Gibson TJ: Phospho.ELM: a database of phosphorylation sites-update 2008.Nucleic Acids Res 2008, 36 Database: D240-244.
〔11〕Jianjiong Gao, Jay J.Thelen, A.Keith Dunker and Dong Xu.Musite, a Tool for Global Prediction of General and Kinase-specific Phosphorylation Sites.Molecular & Cellular Proteomics 2010, 9: 2586 –2600.
S511
A
1673-260X(2014)03-0011-03
