菊花脑热激蛋白70基因的克隆及表达分析
2014-07-02张杨孙明杨海燕张启翔
张杨,孙明,2,3*,杨海燕,张启翔,2,3
(1.北京林业大学园林学院,北京 100083;2.国家花卉工程技术研究中心,北京 100083;3.花卉种质创新与分子育种北京市重点实验室,北京 100083)
菊花脑热激蛋白70基因的克隆及表达分析
张杨1,孙明1,2,3*,杨海燕1,张启翔1,2,3
(1.北京林业大学园林学院,北京 100083;2.国家花卉工程技术研究中心,北京 100083;3.花卉种质创新与分子育种北京市重点实验室,北京 100083)
采用同源克隆结合RACE技术从菊花脑叶片中克隆菊花脑(Chrysanthemum nankingense)热激蛋白70基因 (hsp70) 的cDNA序列,并通过荧光定量PCR(qRT–PCR)分析菊花脑在40 ℃高温下热激不同时间(0、0.5、1、2、3、6 h)后叶片中hsp70基因的表达差异。结果表明,克隆的cDNA序列全长2 224 bp,与水母雪莲花、紫茎泽兰的 hsp70基因在核苷酸和氨基酸水平的同源性分别为 88%、98%,表明该序列为菊花脑的 hsp70基因序列(GenBank登录号,KJ561911),命名为Cnhsp70。Cnhsp70基因的开放阅读框为1 944 bp,其编码的蛋白(647个氨基酸) 的相对分子质量约为70 900,含有HSP70家族序列标签。qRT–PCR分析的结果表明,Cnhsp70基因的表达在热激后短时间内迅速上调,热激3 h达到最高,热激6 h时表达量迅速下调。
菊花脑;热激蛋白70;基因克隆;高温胁迫;表达分析
℃
热激蛋白(heat shock protein, HSP)是生物体在应对高温或其他胁迫环境下所产生的应激蛋白,能在短时间内大量合成和累积,以提高生物体对高温等不利环境的耐受性[1]。不同物种 HSPs的氨基酸序列与功能都很保守。Carper等[2]根据相对分子质量大小,将热激蛋白分为HSP110、HSP90、HSP70、HSP60、小分子sHSP和泛素等6个家族,其中,HSP70最为保守和普遍[3],也是研究得最为广泛的热激蛋白之一。目前,已从拟南芥[4]、玉米[5]、菠菜[6]、紫茎泽兰[7]等植物中克隆到 hsp70基因,但有关菊花的hsp70基因还未见报道。
菊花(Chrysanthemum morifolium)是中国十大传统名花和世界四大切花之一,品种丰富,花型花色多样,观赏价值极高。菊花生长和发育的最适温度是15~25 ℃,超过25 ℃即不利于菊花的生长。菊花脑(Chrysanthemum nankingense)为菊科菊属菊花的野生种,有较强的耐热能力[8]。本研究运用同源克隆技术,拟从菊花脑中克隆出hsp70基因,并对其在高温下的表达情况进行分析,旨在为菊花耐热育种及种质发掘提供参考依据。
1 材料与方法
1.1 供试菊花
从北京林业大学小汤山基地菊花资源圃选择无病虫害、长势一致的菊花脑脚芽进行扦插繁殖,所用基质为草炭和珍珠岩(质量比为1∶1),温室培养2个月后,转移至人工气候箱中。于温度20 /℃18 ℃(白天/黑夜),相对湿度75%,光周期12 h/12 h(光照/黑暗)的条件下缓苗 1 d后,将温度设成40 ℃,热激6 h,收集热激前(热激0 h)、热激后0.5 1、2、3、6 h的上部新鲜叶片置于液氮中冷冻后,–70℃保存、备用。
1.2 方 法
1.2.1 总RNA的提取
用EASYspin Plus植物RNA快速提取试剂盒(艾德莱)分别提取收集的叶片的总RNA。用NanoDrop 2000 超微量分光光度计(Thermo Scientific)测定RNA的A260 nm/A280 nm值,判断质量,并用1%的琼脂糖凝胶电泳检测RNA的完整性。
1.2.2 hsp70基因的克隆
1) 中间保守序列的扩增。 选取热激3 h的叶片的总RNA,用TIANscript cDNA 第一链合成试剂盒(天根生化科技有限公司)合成总cDNA。根据GenBank蛋白数据库中紫茎泽兰(登录号为 ACM42161)、烟草(登录号为 AAR17080)、水母雪莲花(登录号为AAV97978)、杜鹃(登录号为ADD71975)、仙客来(登录号为ABP35942)等hsp70基因的序列,利用在线网站Icodehop设计简并引物HSP1(5′–CTTATTCTT GTGTTGGAGTTTGGCARCAYGAYMG–3′)和HSP2 (5′–ATCTCTCAGACACTTTTCAACAGGYTCCAT RCAYT–3′),以扩增hsp70基因的中间保守序列。PCR反应体系 (25 μL)为:cDNA 2 μL,2×Taq PCR MasterMix(BIOMIGA)12.5 μL,10 μmol/L HSP1 0.3 μL,10 μmol/L HSP2 0.3 μL。PCR反应程序为:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,30个循环;最后72 ℃延伸4 min。将扩增得到的PCR产物经1%琼脂糖凝胶电泳检测,切胶回收后经T载体克隆,测序。
2) 3′RACE的扩增。选取热激3 h的叶片的总RNA,用SMARTer™ RACE cDNA Amplification Kit试剂盒(Clontech公司)合成cDNA。根据测序获得的中间保守序列设计特异引物3GSP1(5′–GACATCAC TGGTAACCCCAGAGCCC–3′)和3GSP2(5′–CCCTC TCATCAACTGCTCAAACCACC–3′),按照SMARTer™RACE cDNA Amplification Kit说明书进行两轮扩增,以获得hsp70基因3′–端序列。将扩增得到的PCR产物经1%琼脂糖凝胶电泳检测,切胶回收后经T载体克隆,测序。
3) 5′RACE的扩增。根据测序获得的中间保守序列设计引物GSP(5′–TCGC CTACCGATGAGA–3′),以热激3 h的叶片的总RNA为模板,用5′RACE System for Rapid Amplification of cDNA Ends Version 2.0试剂盒(Invitrogen公司)合成扩增hsp70基因5′–端序列所需的 cDNA,然后分别以根据中间保守序列设计的引物5GSP1(5′–GTGTTGGTAGGGTTCAT GGC–3′)和5GSP2(5′–CTCAGAATCAGTAAAGGCA AC–3′)进行第一轮和第二轮扩增。将扩增得到的PCR产物经1%的琼脂糖凝胶电泳检测,切胶回收后经T载体克隆,测序。
4) 全长 cDNA的拼接与验证。使用 Contig-Express序列拼接软件将扩增得到的 3段目的基因片段进行拼接,获得hsp70基因全长cDNA。根据拼接所得序列设计特异引物 70U1(5′–AAACATTA CACTCTCTCATCAC–3′)和70D1(5′–TTGTATTGT ATCACAAGCAAC–3′),以热激3 h的叶片的总cDNA为模板进行PCR扩增。将扩增得到的PCR产物经1%琼脂糖凝胶电泳检测,切胶回收后经 T载体克隆,测序。将测序结果与拼接序列用DNAman软件进行比对。
1.2.3 序列分析
用 NCBI网站(http://www.ncbi. nim.nih.gov/) BLASTn、BLASTp软件及DNAman软件进行核苷酸序列、氨基酸序列的比对和保守域的预测。
1.2.4 基因表达分析
用TIANscript cDNA 第一链合成试剂盒,以热激后0、0.5、1、2、3、6 h的叶片的总RNA为模板合成cDNA。根据获得的全长基因序列设计上游引物(5′–GAAGTAGGCTGGGA CTGTAAC–3′)和下游引物(5′–TAACTACAAGGGTGAGGAGAA–3′)。在 Piko®Thermal Cycler 96–well system (Thermo Scientific),用SYBR®Premix EX TaqTM(Til RNaseH Plus)试剂盒(TaKaRa公司)进行定量PCR检测,PCR扩增体系为10 μL (cDNA模板1 μL,10 μmol/L上、下游引物各0.2 μL,SYBR Premix Ex Taq 5 μL,ddH2O 3.6 μL)。反应条件:95℃预变性1 min,95℃变性15 s,55℃退火15 s,72℃延伸45 s,40个循环后进行熔解曲线分析。每个样品重复3次,结果取平均值。选预试验中表达量较稳定的PP2Acs基因为相对定量的内参基因[9],采用 2–△△Ct方法[10]计算相对表达量,比较不同热激时间下目的基因的表达情况。
2 结果与分析
2.1 RNA的质量检测
用 NanoDrop 2000 超微量分光光度计对提取的RNA的A值进行测定,其A260 nm/A268 nm略大于2.0,表明RNA的纯度较高。凝胶电泳结果也显示,总RNA的条带清晰(图1)。可见,所提取的RNA纯度高,完整性好,可以满足后续试验的要求。
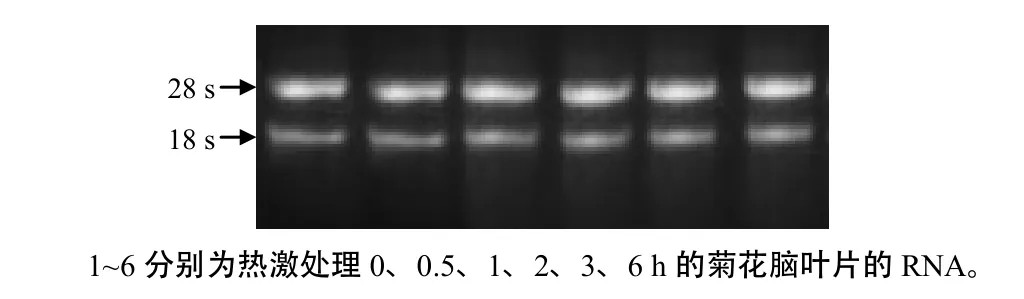
图1 菊花脑叶片的总RNA电泳结果Fig.1 Electrophoresis of RNA isolated from leaves of Chrysanthemum nankingense
2.2 菊花脑hsp70的cDNA克隆
以菊花脑叶片 cDNA为模板,使用引物组合HSP1、HSP2扩增,获得大小为831 bp的hsp70基因的中间片段(图 2–A)。根据中间片段序列,进行3′Race、5′Race扩增,获得1 323 bp的3′序列(图2–B) 和252 bp的5′序列(图2–C)的cDNA片段。将3个片段的序列拼接后得到大小为2 224 bp的基因全长序列。根据拼接的全长序列设计引物,扩增获得了2 224 bp(图2–D)的片段,验证了拼接序列的正确性。Blastn分析发现,克隆的序列与水母雪莲花(GenBank登录号为AF509336)、紫茎泽兰(GenBank登录号为EU269069) hsp70基因的同源性均为88%,表明获得的基因为菊花脑hsp70基因,命名为Cnhsp70,将序列登录GenBank,获得的登录号为KJ561911。

图2 菊花脑hsp70的PCR扩增电泳结果Fig.2 Electrophoresis of PCR products for hsp70 of Chrysanthemum nankingense
2.3 菊花脑Cnhsp70序列分析
Cnhsp70的cDNA含1 944 bp的完整开放性阅读框,编码647个氨基酸,与水母雪莲花(GenBank登录号:AAV97978)、紫茎泽兰(GenBank登录号为ABX76301) hsp70基因的编码氨基酸的同源性均高达98%,并包含HSP70高度保守的3个家族标签序列 DLGTTYS(13–19)、IFDLGGGTFD VSLL(203–216)和VVLVGGSARIPKVQQ(340–354);1个非细胞器保守基序RARFEEL(305–311);1个糖基化位点NVSA(493–496)和胞质HSP70的特征基序EEVD (644–647)[11]。
2.4 Cnhsp70表达特性分析
定量PCR检测结果表明,当菊花脑受到高温胁迫时,Cnhsp70的表达量呈现先升高再下降的趋势。高温胁迫0.5 h时,Cnhsp70的表达量为对照的3倍多;之后其表达保持平缓的上升趋势,于高温胁迫3 h时达到最大,接近对照的5倍;但当热激6 h时,Cnhsp70基因表达量急剧下降,甚至略低于对照水平。
3 讨 论
热激蛋白能提高植物的耐热性,减少高温对生物体的伤害[12–13]。本试验利用同源克隆技术获得了菊花脑热激蛋白70基因(Cnhsp70),并对其表达进行了分析,得出Cnhsp70基因在常温下有少量表达,且表达量稳定,而在受到短暂高温刺激后表达量明显提高。可见,HSP70能在短时间内对高温胁迫进行响应。然而,Cnhsp70的表达量在热激处理6 h后出现显著性下降,这与同为热激蛋白家族的Lshsp90[14]及老鼠的hsp70基因[15]表现一致,这可能是由于热激基因的长时间表达不利于生物体,生物体在适应了高温环境后其表达即下调[16]。
本试验的结果表明,菊花脑的热激蛋白 70主要在热胁迫初期起作用,以短时间内大量表达来帮助植物体应对不利环境,表明可根据hsp70短时表达量的多少来初步判断菊花脑的耐热情况。
[1] Ballinger D G.The control of protein synthesis during best shock in Drosophila cells involves altered polypeptide elongation rates[J].Cell,1983,33(1):103–114.
[2] Carper S W,Duffy J J,Gerner E W.Heat shock proteins in thermotolerance and other cellular processes[J].Cancer Res,1987,47(20):5249–5255.
[3] Wahid A,Gelani S,Ashraf M,et al.Heat tolerant in plants:An overview[J].Environ Exp Bot,2007,61(3):199–223.
[4] Wu C H,Caspar T,Browse J,et al.Characterization of an hsp70 cognate gene family in Arabidopsis[J].Plant Physiol,1988,88(3):731–740.
[5] Rochester D E,Winer J A,Shah D N.The structure and expression of maize genes encoding the major heat shock protein,hsp70[J].EMBO J,1986,5(3):451–458.
[6] Guy C L,Li Q B.The organization and evolution of the spinach stress 70 molecular chaperone gene family[J]. Plant Cell,1988,10(4):539–556.
[7] Gong W N,Xie B Y,Wan F H,et al.Molecular cloning,characterization and heterologous expression analysis of heat shock protein genes (hsp70 and hsp90) of invasive alien weed Ageratina adenophorum (Asteraceae)[J]. Weed Biol Management,2010,10(2):91–101.
[8] 王亚,孔志新,孙明,等.湿热胁迫对地被菊及野生菊生理生化特性的影响[J].西北农业学报,2012,21(9):133–138.
[9] Gu C,Chen S,Liu Z,et al.Reference gene selection for quantitative real–time PCR in Chrysanthemum subjected to biotic and abiotic stress[J].Mol Biotechnol,2011,49(2):192–197.
[10] Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2(–Delta Delta C(T)) Method[J].Methods,2001,25(4):402–408.
[11] Sun S S,Chung M C,Lin T Y.The structure and expression of an hsc70 gene from Lycopersicon esculentum[J].Gene,1996,170(2):237– 241.
[12] Sung D Y,Guy C L.Physiological and molecular assessment of altered expression of Hsc70–1 in Arabidopsis:Evidence for pleiortopic consequences[J]. Plant physiol,2003,132(2):979–987.
[13] Srikanthbabu V,Ganeshkumar,Krishnaprasad B,et al. Identification of pea genotypes with enhanced thermotolerance using temperature induction response technique (TIR)[J].J Plant physiol,2002,159(5):535–545.
[14] 任月,韩莹琰,李婷,等.叶用莴苣热激蛋白90(LsHsp90)基因的克隆及其在热激下的表达[J].中国农业科学,2013,46(16):3514–3522.
[15] 陆蓓玲,王兰芳.急性热应激后小鼠的抗氧化能力和Hsp70 基因的表达[J].湖南农业大学学报:自然科学版,2011,37(6):650–653.
[16] Krebs R A,Holbrook S H.Reduced enzyme activity following Hsp70 over expression in Drosophila melanogaster [J].Biochemical Genetics,2001,39(1/2):73–82.
责任编辑:罗 维
英文编辑:罗 维
Cloning and expression analysis of heat shock protein 70 gene in Chrysanthemum nankingense
ZHANG Yang1, SUN Ming1,2,3*, YANG Hai-yan1,ZHANG Qi-xiang1,2,3(1.College of Landscape Architecture, Beijing Forestry University, Beijing 100083, China; 2.National Engineering Research Center for Floriculture, Beijing 100083, China; 3.Beijing Key Laboratory of Ornamental Plants Germplasm Innovation and Molecular Breeding, Beijing 100083, China)
Homology cloning combined with RACE techniques was applied to clone the cDNA of heat shock protein 70 gene (hsp70) from Chrysanthemum nankingens and real–time quantitative PCR (qRT–PCR) was conducted to analyze the expression patterns of this gene under 40 for 0 h, 0.5 h, 1 h, 2 h, 3 h and 6 h. The results showed that the full cDNA sequence cloned is 2 224 bp, which is determined to be hsp70 gene of Chrysanthemum nankingens, and subsequently named Cnhsp70 as it shares 88% homology with Saussurea medusa at nucleotide level and 98% homology with Ageratina adenophora at amino acid level. Cnhsp70 contains a 1 944 bp open reading flame (ORF), which encodes 647 amino acids containing the HSP70 family sequence tags with a predicted molecular mass of 70 900. Real–time quantitative PCR presented that the expression of Cnhsp70 was up-regulated quickly in a short time after heat stress and get to the highest level 3 h after stress, which was rapidly down-regulated 6 h after stress.
Chrysanthemum nankingense; heat shock protein 70; gene cloning; heat stress; expression analysis
S682.1+1
A
1007−1032(2014)02−0153−04
10.13331/j.cnki.jhau.2014.02.009
投稿网址:http://www.hunau.net/qks
2013–12–09
国家“十二·五”科技支撑计划(2013BAD01B07);中央高校基本科研业务费专项(YX2011-32)
张杨(1989—),女,湖南常德人,硕士研究生,主要从事园林植物遗传育种研究,zhangyang8144@163.com;*通信作者, sun.sm @163.com
