SNPscan法用于新疆主要少数民族非综合征型聋患者GJB2基因突变筛查的研究
2014-06-12陈兴健徐百成陈迟朱一鸣刘晓雯杨小龙王艳莉边盼盼郭玉芬
陈兴健 徐百成 陈迟 朱一鸣 刘晓雯 杨小龙 王艳莉 边盼盼 郭玉芬,2
耳聋是临床上最常见的遗传性疾病之一 ,据统计每1 000名新生儿中有1~3 例为听力障碍儿童,其中至少一半与遗传因素有关[1, 2]。研究发现非综合征型聋(nonsydromic hearing loss,NSHL)患者大多为常染色体隐性遗传,而在被定位和克隆的耳聋相关基因位点中,缝隙连接蛋白B2(gap junction protein bate-2,GJB2)基因突变最为常见,约有50%的常染色体隐性遗传非综合征型聋患者存在该基因的突变[3]。既往针对耳聋相关基因的研究表明,GJB2 基因突变导致的非综合征型聋具有明显的种族差异性,且突变复杂多样[4, 5]。新疆是一个多民族聚居的地方,地域背景民族特点独特,为了解新疆各民族耳聋基因突变谱和热点突变的特点,本研究应用多重单核苷酸多肽性(single nucleotide polymorphism,SNP)分型技术(SNPscan)对新疆四个主要少数民族非综合征型聋患者进行了GJB2基因的突变检测,报告如下。
1 资料与方法
1.1研究对象 从新疆地区12个地州市的残联、聋儿语训中心、聋哑学校及特殊教育学校耳聋患者中筛选出非综合征型聋患者(先证者)565例作为研究对象,其中,维吾尔族428例,男218例,女210例,年龄3~57岁,平均14.87±9.66岁;回族41例,男19例,女22例,年龄3~22岁,平均12.07±5.17岁;哈萨克族64例,男36例,女28例,年龄4~55岁,平均19.15±12.23岁;柯尔克孜族32例,男20例,女12例,年龄7~45岁,平均22.63±12.17岁。均排除外、中耳病变引起的听力损失,听力检测均显示为双侧中重度以上感音神经性听力损失;按照听力损失的国际标准分级(WHO-1997):维吾尔族患者中,重度听力损失者16例,重度44例,极重度368例;回族患者中,重度听力损失者5例,极重度36例;哈萨克族患者中,重度听力损失者5例,极重度59例;柯尔克孜族患者中,重度听力损失者6例,极重度26例。
1.2研究方法
1.2.1病史采集 采用问卷调查及当场询问(家长或老师)为主的方式采集病史资料,并对患者进行全身体格检查及耳鼻咽喉专科检查后填写调查问卷,问卷内容包括一般情况、耳聋发病史、耳部外伤史、药物史、新生儿期听损伤高危因素、全身其他系统(心脏病、骨骼发育、视力)及智力发育状况、母亲分娩史、家族史、其他遗传疾病史、外耳和中耳疾病史和听力学检查资料,并对有耳聋家族史的患者绘制家系图谱,建立详尽的病例资料档案。
1.2.2听力检查及采血 ①纯音听阈、ABR及声导抗检查:对大龄及成年耳聋患者应用Madsen纯音听力计行纯音听阈检查,对所有患者用美国智听公司SmartEP听觉诱发电位仪行听性脑干反应测试,同时行声导抗检查以排除中耳病变。②在知情同意的情况下抽取所有患者外周静脉血5~10 ml,ACD—EDTA抗凝,4 ℃运送,避免震荡、溶血,-20 ℃冷柜保存。
1.2.3基因组DNA的提取与保存 所有标本采用磁珠法提取基因组DNA,0.8%琼脂糖凝胶电泳定性检测,基因组DNA-80 ℃保存。
1.2.4SNPscan法GJB2基因突变筛查 SNPscan法是由上海天昊生物科技有限公司自主专利开发的多重SNP分型专利技术。根据美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)和人类基因突变数据库(Human Gene Mutation Datolase,HGMD)报道的GJB2基因突变位点,结合亚洲人群该基因突变特点[4~6],选取GJB2基因40个已知突变位点用SNPscan方法进行筛查。
1.3统计学方法 各民族间的致病突变位点的等位基因频率运用SPSS17.0软件进行统计学检验,当四格表资料中样本含量大于40且每个格子中的理论频数≥5时,使用卡方检验;当样本含量大于40但有1≤理论频数<5时,卡方值进行校正,当样本含量<40或理论频数<1时用确切概率法计算概率。行×列资料中当 小于5的格子数的比例大≥20%的情况下使用确切概率法,余使用卡方检验,检验水准α=0.05。
2 结果
2.1新疆四个主要少数民族NSHL患者中GJB2基因致病突变位点的等位基因频率检出结果 四个少数民族565例NSHL患者中,GJB2基因40个致病突变位点的等位基因频率为10.09%(114/1 130),共检测到11种突变形式,其中7种为致病突变(表1),其余c.1A>G、c.11G>A、c.23C>T、c.34_35insG、c.95G>T、c.5G>A、c.134G>A、c.139G>A、c.139G>T、c.157T>A、c.164C>A、c.167delT、c.187G>T、c.230G>A、c.232G>A、c.257C>G、c.283G>A、c.287C>G、c.358_360delGAG、c.382A>G、c.408C>A、c.416G>A、c.439G>A、c.493C>T、c.511_512insAACG c.571T>C、c.583A>G、c.598G>A和c.605ins46未检出阳性结果。共明确48例NSHL患者耳聋病因为GJB2基因突变引起,其中33例为纯合突变,15例为复合杂合突变。
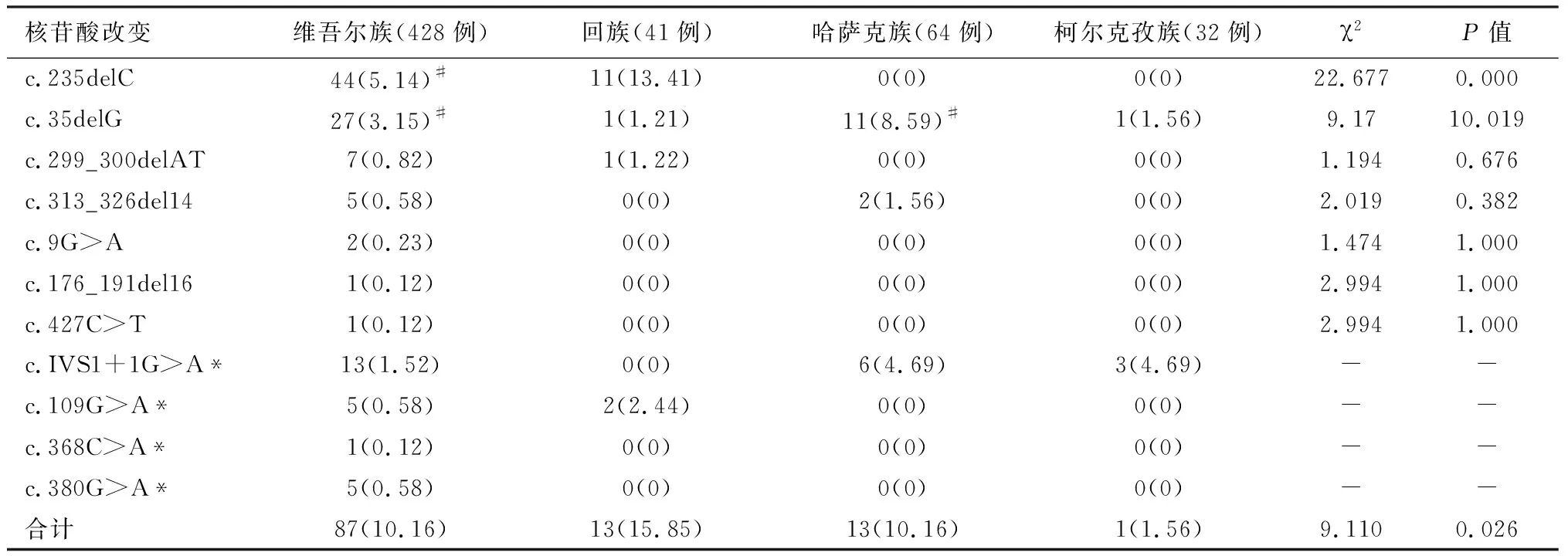
表1 GJB2基因各核苷酸改变在四个少数民族NSHL患者中的等位基因数及频率(例,%)
注:等位基因频率=(2×纯合子个数+杂合子个数) / 总人数×2;*为非致病突变,不做最后统计;#与汉族[10]比较,P<0.05
2.2维吾尔族NSHL患者GJB2 基因突变的检测结果 428例维吾尔族NSHL患者中,共检测到11种突变形式,其中7种为致病突变,分别为c.235delC、c.35delG、c.299_300delAT、c.313_326del14、c.9G>A、c.176_191del16、c.427C>T,等位基因频率为10.16%(87/856),其中c.235delC和c.35delG的等位基因频率最高,分别为5.14%和3.15%,占所有GJB2致病等位基因数的81.61%(71/87),是该民族NSHL患者GJB2基因的热点突变(表1)。所有维吾尔族NSHL患者中,37例明确为GJB2基因突变所致(24例纯合突变,13例复合杂合突变),突变频率为8.64%(37/428);21例为单个GJB2杂合突变携带者,携带频率为4.91%(21/428)(表2)。
2.3回族NSHL患者GJB2基因突变的检测结果 41例回族NSHL患者中,共检测到4种突变形式,其中3种为致病突变,分别为c.235delC、c.35delG、c.299_300delAT,等位基因频率为15.85%(13/82),其中c.235delC等位基因频率最高,为13.41%,占所有GJB2致病等位基因数的84.62%(11/13),是该民族NSHL患者GJB2基因的热点突变(表1)。41例回族NSHL患者中,6例明确为GJB2基因突变所致(5例纯合突变,1例复合杂合突变),突变频率为14.63%(6/41)(表2)。
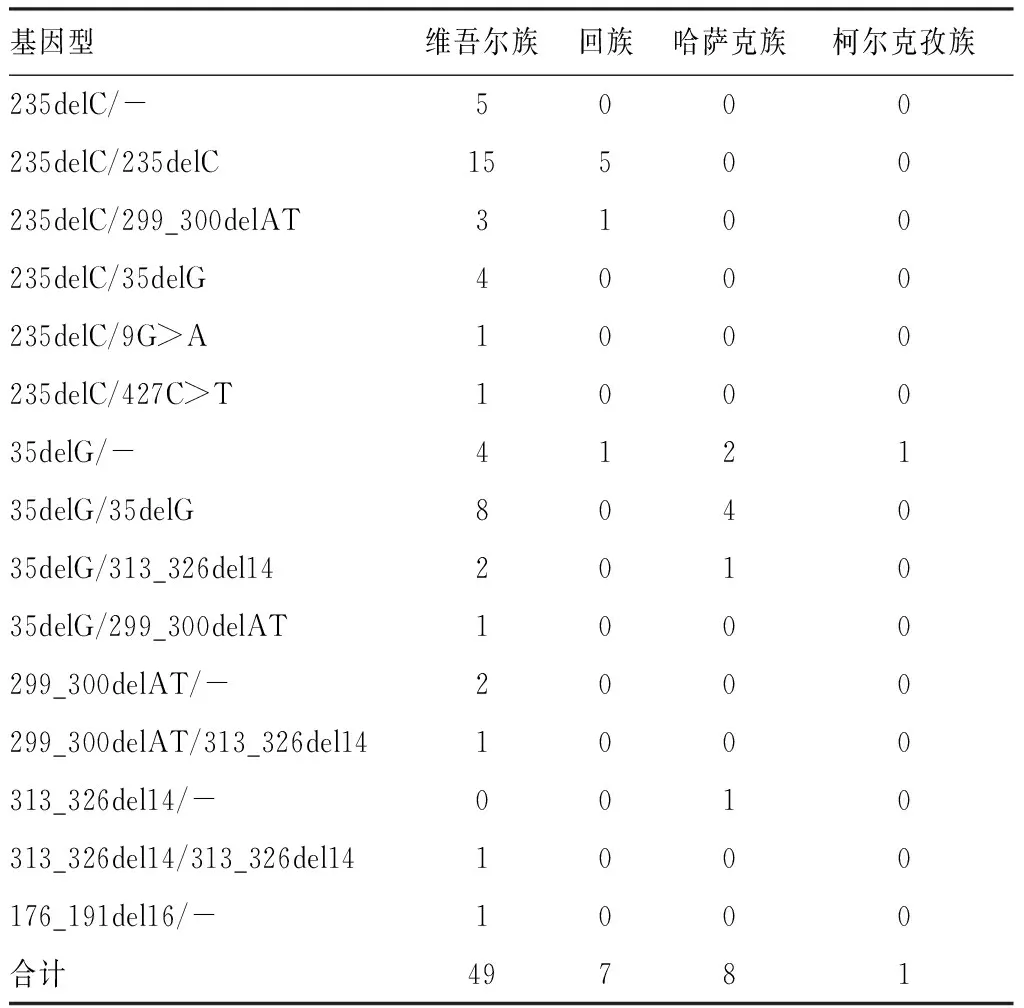
表2 565例NSHL患者GJB2基因致病突变基因型在各民族中的分布(例)
2.4哈萨克族NSHL患者GJB2 基因突变的检测结果 64例哈萨克族NSHL患者中,共检测到3种突变形式,其中2种为致病突变,分别为c.35delG、c.313_326del14,等位基因频率为10.16%(13/128),其中c.35delG等位基因频率最高,为8.59%,占所有GJB2致病等位基因数的84.62%(11/13),是该民族NSHL患者GJB2基因的热点突变(表1)。64例哈萨克族NSHL患者中,5例明确为GJB2基因突变所致(4例35delG/35delG纯合突变,1例35delG/313_326del14复合杂合突变),突变频率为7.81%(5/64)(表2)。
2.5柯尔克孜族NSHL患者GJB2 基因突变的检测结果 32例柯尔克孜族NSHL患者中,共检测到2种突变形式,其中1种为致病突变,为c.35delG,等位基因频率为1.56%(1/64),c.35delG是该民族NSHL患者GJB2基因的热点突变(表1)。
2.6统计学分析结果 维吾尔族、回族、哈萨克族和柯尔克孜族NSHL患者中GJB2基因的致病突变位点等位基因频率分别为10.16%(87/856)、15.85%(13/82)、10.16%(13/128)、1.56%(1/64),其中回族患者突变检出率最高,维吾尔族和哈萨克族患者突变检出率次之,均低于回族,柯尔克孜族患者突变检出率最低,差异有统计学意义(χ2=8.140,P=0.043)。
3 讨论
不同国家、不同地区、不同民族耳聋基因突变频率有很大的差异,GJB2基因突变最为常见且差异明显[5, 6]。随着人类后基因组时代的开始,单核苷酸多态性(single nucleotide polymorphism,SNP)作为第三代遗传标记被越来越广泛的应用于疾病遗传学、药物基因组学及生物多样性分析中,同时,SNP基因分型技术也取得了令人瞩目的进展[7]。
本研究中应用的SNPscan技术是一种多重SNP分型技术,该技术采用连接酶连接反应的高特异性实现对SNP位点等位基因的识别,然后通过在连接探针末段引入不同长度的非特异序列以及通过连接酶加接反应获得位点对应的不同长度连接产物,利用标记荧光的通用引物对连接产物进行PCR扩增,通过荧光毛细管电泳对扩增产物进行电泳分离,最后通过对GeneMapper软件分析获取各个SNP位点的基因型。本研究结果显示本组新疆维吾尔族、回族、哈萨克族和柯尔克孜族NSHL患者中GJB2基因的致病等位基因频率分别为10.16%、15.85%、10.16%、1.56%,回族高于维吾尔族和哈萨克族,柯尔克孜族检出率最低。从人种角度分析,c.235delC是新疆地区维吾尔族和回族耳聋患者的热点突变,与蒙古利亚人种的热点突变一致[8];c.35delG是维吾尔族、哈萨克族和柯尔克孜族的热点突变,与高加索人种的热点突变一致[9]。
满荣军等[10]用直接测序法对维吾尔族NSHL患者GJB2第2外显子进行检测,检出率为13.68%(29/212);陈俞等[11]应用基因芯片法对维吾尔族GJB2基因c.235delC、c.35delG、c.299_300delAT、c.176_191del16进行检测,检出率为9.24%(17/184);本研究用SNPscan方法对新疆维吾尔族NSHL患者进行GJB2基因2个外显子40个已知突变位点的筛查,检出率为11.45%(49/428);上述结果差异均无统计学意义。GJB2只有2个外显子,编码区位于第2外显子,虽然基因测序方法和检测位点不同,但对GJB2常见致病突变位点的检出率无明显差异,提示通过对GJB2基因常见位点的筛查就可以得到较高的检出率,可为新疆维吾尔族耳聋基因筛查在突变位点选择上提供指导。
维吾尔族是一个多源民族,且地理位置靠近中亚,长期的迁移、婚配等原因使维吾尔族融合了约30%的高加索人种的血缘[12]。既往报道显示GJB2在该民族中的突变频率为9.24%,c.235delC和c.35delG是维吾尔族GJB2基因的突变热点[10, 11]。本研究428例维吾尔族NSHL患者中c.235delC的等位基因频率为5.14%,c.35delG等位基因频率为3.15%,是该民族的突变热点,与汉族[10]具有统计学差异,提示汉族与维吾尔族NSHL患者GJB2基因突变谱存在差异。研究显示,在欧美国家高加索人种中,c.35delG为NSHL患者的主要突变形式,占总突变的60%~85%[13],而在亚洲蒙古利亚人中,c.35delG很少发现,c.235delC突变是中国、日本、韩国最常见的突变形式[14, 15]。维吾尔族NSHL患者中GJB2基因的突变表现可能与该民族融合了约30%的高加索人种的血缘有关。
本研究中41例回族NSHL患者c.235delC和c.35delG的等位基因频率分别为13.41%和1.21%,其中c.235delC等位基因频率最高,是该民族NSHL患者GJB2基因的热点突变,与汉族患者[10]比较,差异无统计学意义(P>0.05),也与马建鹂等[16]报道的中国西北地区回族NSHL患者GJB2基因突变的结果一致。回族散居全国,从民族起源来说,是一个由众多民族融合而成的融生性民族,本研究结果也符合回族融生性民族的特征。
新疆地区哈萨克民族属于蒙古人种北亚类型和欧罗巴人种及印度地中海类型之间的混合类型,属于混血民族;柯尔克孜族多数人属于蒙古人种北亚类型,少数人兼有欧罗巴特征,大体属于混合民族[12]。本研究中哈萨克族和柯尔克孜族NSHL患者c.35delG的等位基因频率分别为8.59%和1.56%,c.235delC和c.299_300delAT是蒙古利亚人种GJB2基因最常见的突变位点[4,5],在这个两个民族中均没有发现,可能与其民族起源和样本量较少有关,还有待于进一步的研究。
综上所述,本研究进一步证实新疆地区维吾尔族、回族、哈萨克族、柯尔克孜族NSHL患者GJB2基因的突变热点不同,且具有民族特异性,可能与民族起源、长期人种间基因交流有关。SNPscan技术以SNP位点检测为基础,可以在一个检测流程中同时实现对多个SNP位点进行分型,与直接测序法相比较,省去了实验过程中较多繁琐的操作,且能对多个样本多个位点同时检测,也降低了成本;与基因芯片法相比,目前国内仅针对GJB2四个常见位点的检测[17],而SNP检测位点多,可针对已知目标基因突变位点灵活设计,真正实现高通量。此外,SNPscan已成功应用于冠状动脉疾病相关基因和乳腺癌易感基因的研究中,同时证实了该方法的准确性和高成功率[18, 19]。因此,SNPscan是一种适合大规模的耳聋基因筛查的方法。
4 参考文献
1 Hilgert N, Smith RJ, Van Camp G. Forty-six genes causing nonsyndromic hearing impairment: which ones should be analyzed in DNA diagnostics[J]? Mutation Research/Reviews in Mutation Research,2009,681:189.
2 Dalamón V, Lotersztein V, Béhèran A, et al. GJB2 and GJB6 genes: molecular study and identification of novel GJB2 mutations in the hearing-impaired Argentinean population[J]. Audiology and Neurotology,2009,15:194.
3 Kelley P, Harris D, Comer B, et al. Novel mutations in the connexin 26 gene (GJB2) that cause autosomal recessive (DFNB1) hearing loss[J]. The American Journal of Human Genetics,1998,62:792.
4 Tekin M, Xia XJ, Erdenetungalag R, et al. GJB2 mutations in Mongolia: complex alleles, low frequency, and reduced fitness of the deaf[J]. Ann Hum Genet,2010,74:155.
5 Guo YF, Liu XW, Guan J, et al. GJB2, SLC26A4 and mitochondrial DNA A1555G mutations in prelingual deafness in Northern Chinese subjects[J]. Acta Oto-laryngologica,2008,128:297.
6 Ohtsuka A, Yuge I, Kimura S, et al. GJB2 deafness gene shows a specific spectrum of mutations in Japan, including a frequent founder mutation[J]. Hum Genet,2003,112:329.
7 Crawford A, Fassett RG, Geraghty DP, et al. Relationships between single nucleotide polymorphisms of antioxidant enzymes and disease[J]. Gene,2012,501:89.
8 Dai P, Yu F, Han B, et al. The prevalence of the 235delC GJB2 mutation in a Chinese deaf population[J]. Genetics in Medicine,2007,9:283.
9 Abidi O, Boulouiz R, Nahili H, et al. GJB2(connexin 26) gene mutations in Moroccan patients with autosomal recessive non-syndromic hearing loss and carrier frequency of the common GJB2-35delG mutation[J]. Int J Pediatr Otorhinolaryngol,2007,71:1 239.
10 满荣军, 郭玉芬, 刘晓雯, 等. 新疆少数民族和汉族聋哑学生 GJB2 基因和线粒体 DNA 12SrRNA A1555G 突变研究[J]. 中国耳鼻咽喉头颈外科,2009,16:190.
11 陈俞, 赵娟, 皮力东·库亚西, 等. 新疆不同民族耳聋人群耳聋基因常见突变的筛查[J]. 新疆医科大学学报,2011,8:850.
12 杜若甫. 我国的人类群体遗传学研究[J]. 生物学通报,1997,32:9.
13 Kenneson A, Braun KVN, Boyle C. GJB2 (connexin 26) variants and nonsyndromic sensorineural hearing loss: a HuGE review[J]. Genetics in Medicine,2002,4:258.
14 Abe S, Usami SZ, Shinkawa H, et al. Prevalent connexin 26 gene (GJB2) mutations in Japanese[J]. J Med Genet,2000,37:41.
15 Dai P, Yu F, Han B, et al. GJB2 mutation spectrum in 2063 Chinese patients with nonsyndromic hearing impairment[J]. J Transl Med,2009,7:1.
16 马建鹂, 徐百成, 边盼盼, 等. 中国西北地区回族非综合征型聋患者耳聋相关基因研究[J]. 听力学及言语疾病杂志,2013,21:451.
17 王国建, 戴朴, 韩东一, 等. 基因芯片技术在非综合征性耳聋快速基因诊断中的应用研究[J]. 中华耳科学杂志,2008,6:61.
18 Tang LL, Chen FY, Wang H, et al. Haplotype analysis of eight genes of the monoubiquitinated FANCD2-DNA damage-repair pathway in breast cancer patients[J]. Cancer Epidemiology,2013,37:311.
19 Chen X, Li S, Yang Y, et al. Genome‐wide association study validation identifies novel loci for atherosclerotic cardiovascular disease[J]. Journal of Thrombosis and Haemostasis,2012,10:1 508.
