家蚕光泽小眼(varnished eye)突变基因的精细定位
2014-05-25刘春李琼艳荀利杰胡文波吕金风刘茹凤夏庆友
刘春,李琼艳,荀利杰,胡文波,吕金风,刘茹凤,夏庆友
家蚕基因组生物学国家重点实验室,农业部蚕桑重点实验室,西南大学,重庆 400715
家蚕光泽小眼(varnished eye)突变基因的精细定位
刘春,李琼艳,荀利杰,胡文波,吕金风,刘茹凤,夏庆友
家蚕基因组生物学国家重点实验室,农业部蚕桑重点实验室,西南大学,重庆 400715
家蚕(Bombyx mori)光泽小眼(varnished eye,ve)突变体是家蚕突变品种中少数关于家蚕复眼形态异常的自然隐性突变品种之一,表现为成虫复眼小,表面富有光泽,有瘤状突起;小眼数量少且不规则。经典遗传学连锁图谱将ve基因定位在第6连锁群32.2座位,但到目前为止,该突变性状的形成机理仍不清楚。文章以突变品种ve和对照品种Dz为亲本,杂交产生的F1代雄个体与轮回亲本ve雌个体回交产生的BC1代为材料进行ve基因的精细定位。通过对ve低密度连锁图谱分析发现,ve基因被定位在SNP3~SNP6之间,物理图谱距离大约为1.2 Mb。利用1563个BC1代个体构建ve高密度连锁图谱,最终将ve基因定位在SNP5~SNP61之间,物理图谱距离大约为221.8 kb。通过家蚕基因数据库对ve最终定位区域内进行预测基因检索,发现有6个预测基因。对6个预测基因进行生物信息学分析和测序,这些候选基因都有可能参与家蚕复眼形成,但在编码区没有发现ve特异的突变位点;对这些候选基因进行表达分析,发现编号为BGIBMGA013642的候选基因在ve复眼形成过程中的表达量明显比正常对照Dz低,推测该基因为最佳候选基因。
家蚕;光泽小眼;SNP标记;精细定位;候选基因
昆虫视觉器官能够感受波长范围为253~700 nm的光波,光波受体细胞刺激产生生物电位,传播到中枢神经系统引起视觉反应。一般昆虫的视觉器官包括单眼和复眼两种,单眼的视觉中心位于单眼柄顶端内,复眼的视觉中心位于视叶。昆虫的视觉器官对昆虫的觅食、求偶、休眠、滞育、避害、决定行为方向等都起着重作用[1~3]。
家蚕是一个完全变态昆虫,一生经历卵、幼虫、蛹、成虫 4个发育阶段。家蚕幼虫期视觉器官由 6对单眼组成,成虫期视觉器官由一对复眼组成。家蚕成虫复眼黑色,由3000多个规则的六角形小眼排列成蜂窝状结构。每个小眼由角膜、晶锥、色素细胞、感杆束和视神经等结构组成[4],并且这些组成和果蝇、哺乳动物以及人类视觉系统基本组成具有较高的相似性。家蚕有许多关于复眼的突变体,大多数是与色素相关的眼色突变体,如第1白卵(w-1),复眼白色[5,6];第2白卵(w-2),复眼白色[7];第3白卵油蚕(w-3oe)[8,9],复眼白色;第5白卵(w-5),复眼红色[10];红眼红卵(red egg, re),复眼深赤褐色[11]等突变体;而且,大部分突变基因已经被相继克隆鉴定,其突变机理也被阐明。目前,关于家蚕复眼眼形突变的突变体只有两种,光泽眼(lustrous, lu)和光泽小眼(varnished eye, ve),经典遗传学分析将lu突变基因定位在家蚕第16连锁群0.0座位,将ve突变基因定位在家蚕第6连锁群32.2座位[12,13],这两个突变体都是由单基因控制的的隐性突变。
家蚕光泽小眼突变体的表型为成虫复眼小,表面富有光泽,小眼形状不规则、成瘤状突起、数量显著减少。本实验室通过前期的形态观察,发现ve复眼的突变不仅表现在外部形态,复眼内部结构也发生巨大的变化。与正常复眼相比,相应的角膜、晶锥、感杆束及色素细胞都发生了变化[14]。但目前为止,其突变基因及突变机理还未知。因此,本研究利用SNP标记对ve突变基因进行了精细定位,希望能克隆鉴定ve突变基因,进而阐明ve的突变机理,为家蚕和昆虫的视觉器官的变态发育研究提供参考,同时为人类眼疾病研究提供信息。
1 材料和方法
1.1 材料
本实验所用的正常蚕品种Dz及突变品系ve复眼表型如图 1所示。用于验证突变位点特异性的其他蚕品种及突变品种,包括碧波、C108、绵茧、狭胸、黑缟、鹑斑、伴性赤蚁、夏芳、缩蚕及裸蛹等蚕卵均由西南大学家蚕基因库提供,分别按常规桑叶育进行饲养。

图1 正常蚕蛾复眼和ve突变蚕蛾复眼比较图
1.2 方法
1.2.1 ve定位材料的配置
本研究以ve突变个体和Dz对照个体为亲本,杂交F1代雄个体与轮回亲本ve雌个体回交,产生的BC1代为材料进行精细定位。整个材料饲养及配置由西南大学家蚕基因资源库提供。所有材料收集、统计、贴上标签后保存于-80℃冰箱中保存,材料统计结果见表1。
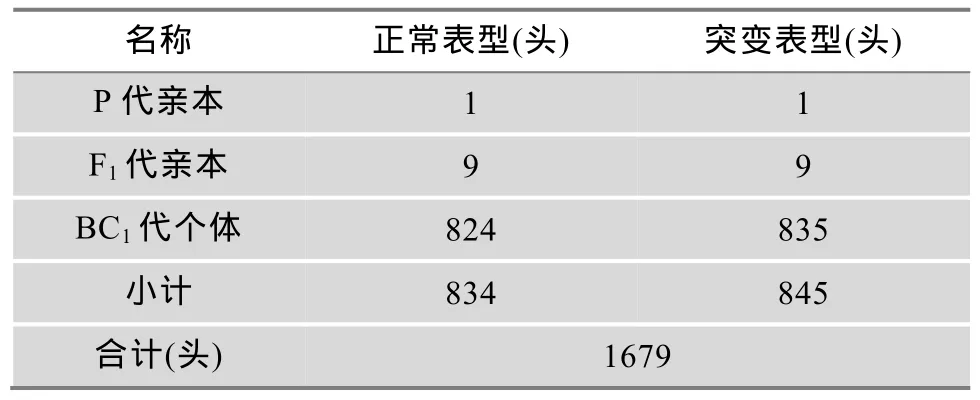
表1 所用材料数目统计
1.2.2 基因组DNA提取
家蚕基因组的提取方法按 DNA自动提取仪(Kurobo-PI1200,日本)提取小鼠尾巴基因组DNA的提取方案说明书进行。样品DNA溶解在TE,利用分光光度计抽样检测基因组DNA浓度和纯度,并利用家蚕看家基因核糖体蛋白L3(ribosomal protein L3 of Bombyx mori,Bmrp3)基因引物(正向引物:CGGTGTTGTTGGATACATTGAG;反向引物:GCTCATCCTGCCATTTCTTACT)调整基因组DNA的最适模板浓度,便于后期大规模筛选SNP标记。
1.2.3 SNP标记的筛选
从已构建的家蚕SNP 分子标记连锁图中的第6连锁群中等间距挑取SNP标记[15,16],以两个亲本和F1个体为模版进行PCR扩增反应。PCR扩增体系(10 μL):1 μL 10×r Tag buffer; 2.5 mmol/L dNTP 0.8 μL;rTag (TaKaRa) 0.1μL,2 μL基因组模板;2 μmol/L引物(F/R) 2 μL;4.1 μL ddH2O。扩增条件:94℃预变性5 min;94℃ 10 s,55℃ 15 s,72℃ 30 s,共35个循环;最后再72℃延伸5 min。经电泳检测后存在片段长度多态性的直接用于下一轮连锁分析;对片段长度没有差异的则直接进行PCR产物测序。PCR产物首先利用SAP酶和核酸外切酶Ⅰ按试剂说明书进行消化处理,然后利用 BigDye® Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems, Foster City, CA, USA) 进行测序PCR反应,并利用无水乙醇进行纯化,最后利用3730XL DNA analyzer (Applied Biosystems)进行测序。测序反应按 ABI3700仪器 PCR产物测序操作说明进行。利用 Sequener 4.0软件对序列进行 SNP位点的鉴定和分析,筛选出可用的SNP标记。本实验用于定位的SNP标记引物信息见表2。
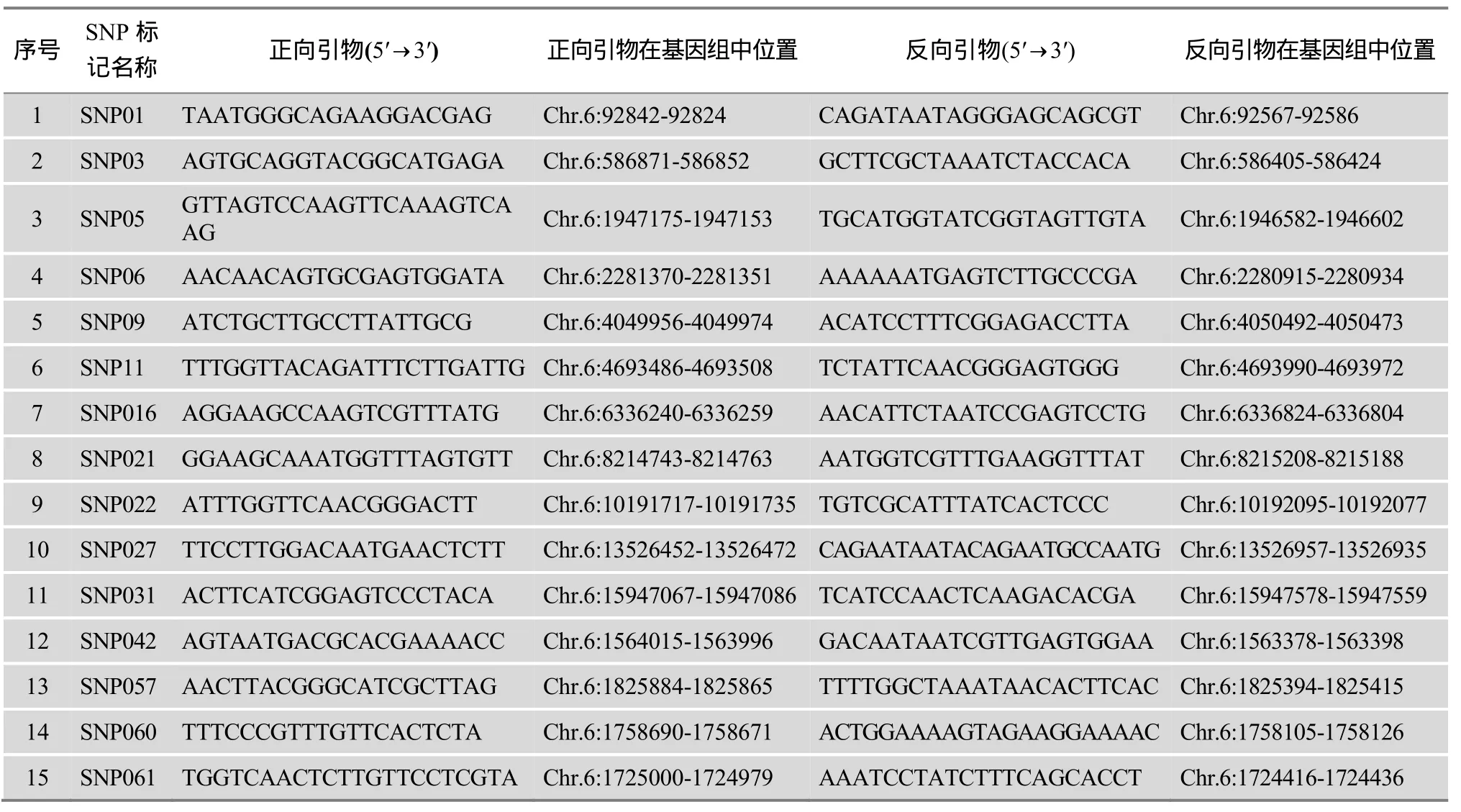
表2 ve精细定位所用SNP标记引物及在日本家蚕基因组数据中的位置
1.2.4 家蚕ve基因连锁定位
将筛选出的SNP标记,利用BC1中92个个体进行SNP分析,构建ve低密度遗传连锁图谱。利用与表型最紧密连锁的2个SNP标记进行大规模BC1群体(1563个个体)的基因型分析,筛选交换个体;同时,在最紧密连锁的两个SNP标记间根据家蚕遗传多态性图谱设计新的引物,发展新的 SNP标记,构建目标基因ve的高密度遗传连锁图谱。
1.2.5 ve连锁区域内候选基因分析
利用西南大学家蚕基因组数据库 SilkDB(http: //silkworm.swu.edu.cn/silkdb)和日本基因组数据库(http://sgp.dna.affrc.go.jp/KAIKObase/)对连锁区域进行基因检索并下载候选基因序列。利用家蚕数据库和Pfam在线预测候选基因编码蛋白质结构域。
1.2.6 ve候选基因的测序鉴定及表达差异比较
为了比较候选基因的表达差异,收集Dz和ve5龄第 3 d及上蔟至化蛾期材料,利用 Triozl(Roche)试剂提取头部RNA,提取方法参考试剂说明书进行。采用反转录试剂盒(Promega)对纯化后的 RNA进行cDNA合成。根据候选基因序列设计引物进行cDNA扩增,并进行测序鉴定;同时进行表达差异分析。反应条件:94℃预变性5 min;94℃ 10 s,55℃ 15 s,72℃ 30 s,共35(30)个循环;最后72℃ 5 min。对测序分析出Dz和ve有氨基酸差异的突变位点在碧波、C108、绵茧、狭胸、黑缟、鹑斑、伴性赤蚁、夏芳、缩蚕及裸蛹10个蚕品种基因组中进行序列突变特异性鉴定。候选基因测序及表达谱分析所用引物序列见表3。

表3 ve候选基因鉴定所涉及的引物名称及序列

引物编号 基因编号 引物序列(5′→3′)ve-can3-F2BGIBMGA013643ATTCGGGAAGGTCTAGTTTGve-can3-R1BGIBMGA013643TTTTGGCTAAATAACACTTCACve-can3-R2BGIBMGA013643TTTTGGCTAAATAACACTTCACve-can4_F1BGIBMGA013642TTTTATCGTCCATCATTCGTGve-can4_F2BGIBMGA013642GCCGCTGCTTTTATTGGTATve-can4_F3BGIBMGA013642AAAGAGTTGGAGGATTCGTCGve-can4_F4BGIBMGA013642ATGAAAACCGAAGACGAAAGAACve-can4_F5BGIBMGA013642GTGTATGCTTACCTCGGCGAve-can4_F6BGIBMGA013642TCTGTCTCCGTGTTCCCGATve-can4_R1BGIBMGA013642TTATGTTCTGATCGGTCGAGTCve-can4_R2BGIBMGA013642TCCTGAGCACAGCGAGACTATve-can4_R3BGIBMGA013642ACCTGTAACCGTAGCAGTCTTGAve-can4_R4BGIBMGA013642 GATAGCCCCACAAGAACGACve-can4_R5BGIBMGA013642GTGGCTTGAACATTAGGGCAve-can4_R6BGIBMGA013642AACCGTGAAGGCTGTTTGACve-can5_F1BGIBMGA013574GTCAGACGTATCGCGTGTTCCBGIBMGA013574 ve-can5_F2TTGGAGGTTGCTCATCATCGve-can5_F3BGIBMGA013574CACGAGGCGAATGTTGTAGTve-can5_R1BGIBMGA013574ATTTCCTGTTGCTTCCGTATGAve-can5_R2BGIBMGA013574GCTTTCGCTTTCTAGTACTTCAGve-can5-F4BGIBMGA013574GGGCAGGAGTTCGGCATTCAve-can5-R3BGIBMGA013574TAAGAAGCAAGGATGATACCAAve-can5-R4BGIBMGA013574TTCCCTTCAGCACCTCCATAGCve-can5-R5BGIBMGA013574GATACTTTACCATTATTTACTAve-can6_F1BGIBMGA013641TTTTGTTAGTGTAAATGTGCCTTve-can6_F2BGIBMGA013641AAAGCACCGTTCCAGACTACAve-can6_F3BGIBMGA013641AACGATTACTTTGCTGATGGGve-can6_F4BGIBMGA013641ATTTACTCGTGGAGGTTGTTCve-can6_F5BGIBMGA013641ATGCCACTATGGATGACGAGve-can6_F6BGIBMGA013641CTGCTCCAACTCCAACAAGATAve-can6_R1BGIBMGA013641ACACGAAAAGTATGAGACTGGATTve-can6_R2BGIBMGA013641GCTGCTACGGCAATAAAAGGve-can6_R3BGIBMGA013641ATTAATCGATTTATTCTTGCAGTve-can6_R4BGIBMGA013641CTACATGTAAGTTGATATGTATve-can6_R5BGIBMGA013641CACTCCTGATGCGAAGAAAGve-can6_R6BGIBMGA013641AACAAAACTCATTCCCACCAve-can6_R7BGIBMGA013641TTGTCTTCTTCTTTTCGCGT
2 结果与分析
2.1 SNP标记的筛选
本研究所用的标记为 SNP标记,目前已知的SNP标记是基于 Dz与 C108之间的分子标记[15,16],这些分子标记是否存在于ve与Dz两品种之间需进行进一步的实验验证。因此,本研究对SNP标记进行了电泳筛选和测序筛选。利用电泳筛选主要是去除不能在亲本间获得PCR产物的或者PCR产物不理想的SNP标记;对于PCR产物在两品种间具有片段长度多态性的,则直接利用电泳检测的方法对 BC1代进行基因分型。对于没有片段长度的PCR产物则进入测序分析;通过电泳检测和测序分析,发现检测的38个SNP标记中有20个在Dz和ve中存在PCR产物长度多态性或者SNP位点,可以用于下一步实验。
2.2 ve突变基因的精细定位
根据可用SNP标记在染色体上的位置,选择了10个SNP标记(图2A)和92个回交个体构建低密度连锁图谱。将电泳和测序结果统计在excel表中,排序整理,删除未发生交换、双交换,构建获得低密度连锁图谱。通过对图谱的分析,发现ve被锁定在SNP3和SNP6标记之间,物理距离约为1.2 Mb(图2B)。为了更进一步缩小ve的连锁区域,对低密度连锁图谱区域重新开发新的 SNP标记,获得可用的 SNP标记8个。利用1563个回交个体构建ve高密度连锁图谱,最终将ve突变基因锁定在SNP5和SNP61两个分子标记之间,物理图距为221.8 kb(图2C)。
2.4 连锁区域候选基因分析和鉴定
利用西南大学家蚕基因组数据库 SilkDB(http: //silkworm.swu.edu.cn/silkdb)和日本基因组数据库(http://sgp.dna.affrc.go.jp/KAIKObase/)对 ve的最小连锁区域进行基因检索,发现该区域有 6个预测基因(图2C)。利用家蚕数据库和在线软件Pfam对候选基因进行蛋白质结构域分析,结果如表4所示。
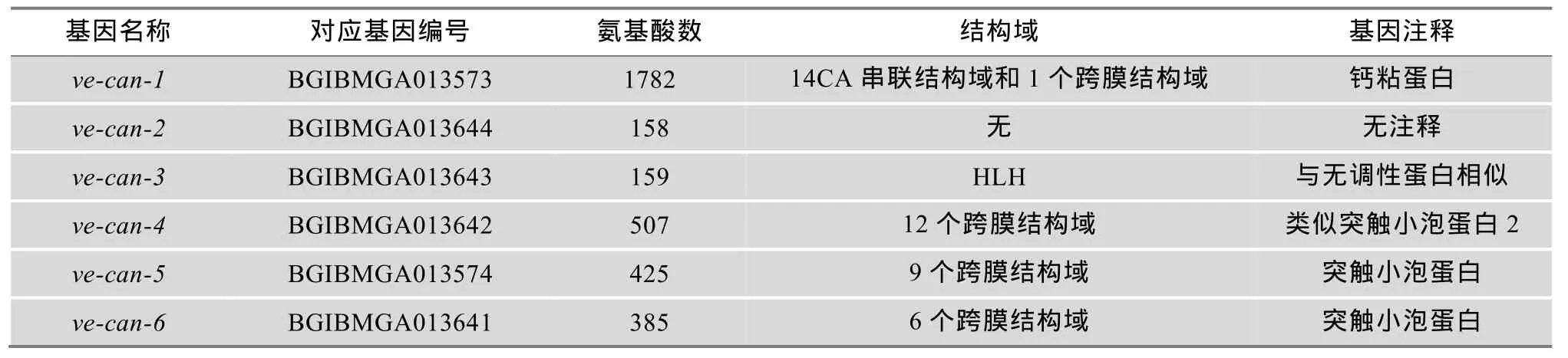
表4 ve连锁区域候选基因及编码蛋白结构域分析
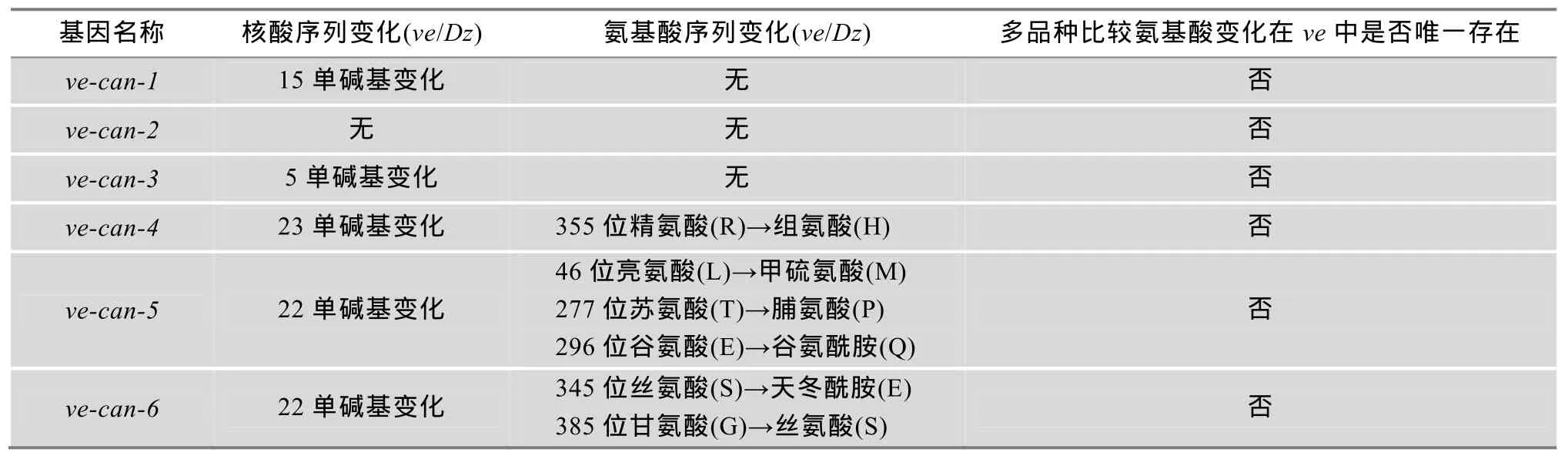
表5 预测基因在ve与Dz品种间的突变位点分析
根据基因注释分析得出ve-can-1基因编码钙粘蛋白,参与细胞与细胞之间及细胞与细胞外基质之间相互粘连过程;ve-can-2无任何功能注释;ve-can-3基因编码HLH类型的转录调控因子,对生长发育起调控作用;ve-can-4、ve-can-5及ve-can-6都编码突出囊泡蛋白,在细胞转运膜泡运输当中起重要作用。对上述 6个候选基因编码区进行测序,发现大部分基因都存在单碱基的变化。将其翻译成蛋白序列,发现有3个基因存在单个或者多个氨基酸的变化(表5)。为了验证这些氨基酸的变化是否在 ve中特异存在,利用 10个其他复眼正常的品种进行进一步的测序研究,测序结果表明这些突变位点都不是ve唯一的(表5)。对这些候选基因进行了表达差异比较,发现ve-can-4(BGIBMGA013642)基因在ve复眼形成时期表达量较正常低(图 3),将其认为是 ve的最佳候选基因。

图3 ve-can-4基因在Dz和ve复眼形成期的表达情况1:5龄第3 d;2:刚上簇;3:上簇第1 d; 4:上簇第2 d;5:刚化蛹;6:化蛹第1 d;7:化蛹第2 d;8:化蛹第3 d;9:化蛹第4 d;10:化蛹第5 d;11:化蛹第6 d;12:化蛹第7 d。
3 讨 论
本研究通过基于SNP标记的定位克隆技术将家蚕ve基因定位在了第6染色体的SNP5和SNP61两个分子标记之间,物理距离约为 221.8 kb,对定位区域内染色体序列进行了信息学分析发现在这个定位区域内有6个预测基因。本文对这6个预测基因进行了结构预测和功能分析,发现这 6个基因都有可能参与眼的形成。对 6个预测基因编码区进行测序,却未发现ve特异的氨基酸序列变化。本研究对候选基因进行了蛹期表达谱分析,从每个基因表达谱结果发现BGIBMGA013642基因表达量在ve中显著降低,可能与 ve的突变表型有关。2009年,Catherine等[17]在小鼠中对BGIBMGA013642基因的同源基因SV2B进行基因敲除实验,敲除后小鼠视网膜发育异常,推测 BGIBMGA013642基因在家蚕眼部发育也起着重要作用,是最佳的候选突变基因。
从目前家蚕定位克隆研究进展可以看出,导致家蚕突变的原因形形色色,既有大片段序列的插入或缺失,又有单碱基的改变;既有编码区域序列的改变,又有非编码区域序列的改变;既有基因改变导致的蛋白功能的变化或丧失,又有基因表达量的降低导致编码蛋白功能不足等等。根据ve的候选基因分析情况,推测有可能其突变位点发生在基因的非编码区,即基因的启动子、内含子或者UTR区域。这些区域的变化虽然不会引起蛋白结构的变化,但会导致基因表达量的变化,同样也会因为蛋白表达量不够而引起形态上的变化;同时,也不排除另一种可能,在锁定区域内,还有未被预测的基因或者基因组序列在该区域被组装错误,导致基因预测不完全。Quiring等[18]证实,果蝇的 eyeless基因和小鼠的small eye基因以及人类的aniridia基因属于同源基因,其突变性状具有很高的相似性,并且证实这3个基因属于pax-6基因家族成员,而它们的功能主要是控制眼睛的发育[19]。Kawguchi等[20]推测ve突变可能是由于pax-6基因突变引起。本研究对家蚕pax基因家族进行分析后发现,编号为BGIBMGA-009545与eyeless基因同源,然而BGIBMGA0009545基因却未被成功定位到染色体上。因此,下一步我们将对该基因进行分析,确定其是否在二者间发生了变化。
[1] 刘红霞, 彩万志. 昆虫单眼的结构和功能. 昆虫知识, 2007, 44(4): 603-607.
[2] 冷雪, 那杰. 昆虫复眼的结构和功能. 沈阳师范大学学报 (自然科学版), 2009, 27(2): 241-244.
[3] 彩万志. 普通昆虫学. 北京: 中国农业大学出版社, 2001: 286-291.
[4] 吴载德. 蚕体解剖生理学. 北京: 农业出版社, 1989: 137-139.
[5] Quan GX, Kobayashi I, Kojima K, Uchino K, Kanda T, Sezutsu H, Shimada T, Tamura T. Rescue of white egg 1 mutant by introduction of the wild-type Bombyx kynurenine 3-monooxygenase gene. Insect Sci, 2007, 14(2): 85-92.
[6] Quan GX, Kim I, Kômoto N, Sezutsu H, Ote M, Shimada T, Kanda T, Mita K, Kobayashi M, Tamura T. Characterization of the kynurenine 3-monooxygenase gene corresponding to the white egg 1 mutant in the silkworm Bombyx mori. Mol Genet Genomics, 2002, 267(1): 1-9.
[7] Tatematsu K, Yamamoto K, Uchino K, Narukawa J, Iizuka T, Banno Y, Katsuma S, Shimada T, Tamura T, Sezutsu H, Daimon T. Positional cloning of silkworm white egg 2 (w-2) locus shows functional conservation and diversification of ABC transporters for pigmentation in insects. Genes Cells, 2011, 16(4): 331-342.
[8] Quan G, Kanda T, Tamura T. Induction of the white egg 3 mutant phenotype by injection of the double-stranded RNA of the silkworm white gene. Insect Biochem Molec, 2002, 11(3): 217-222.
[9] Kobayashi I, Uchino K, Iizuka T, Tatematsu KI, Yonemura N, Sezutsu H, Tamura T. Rescue of the Aojuku white-egg translucent (w-3ol) Bombyx mori mutant by transgenic expression of the wild-type Bmwh3 gene. J Insect Biotechnol Sericol, 2011, 79(3): 3-111-3-116.
[10] 蒋同庆, 向仲怀. 家蚕第5白卵 (w-5) 红眼白卵系遗传学的研究. 中国农业科学, 1981, 14(3): 81-89.
[11] Osanai-Futahashi M, Tatematsu K, Yamamoto K, Narukawa J, Uchino K, Kayukawa T, Shinoda T, Banno Y, Tamura T, Sezutsu H. Identification of the Bombyx red egg gene reveals involvement of a novel transporter family gene in late steps of the insect ommochrome biosynthesis pathway. J Biol Chem, 2012, 287(21): 17706-17714.
[12] 向仲怀. 蚕丝生物学. 北京: 中国林业出版社, 2005: 131-132.
[13] Doira H. Genetical studies of the varnished eye mutant in Bombyx mori. Jpn J Genet, 1975, 50(2): 115-120.
[14] 李琼艳, 刘春, 荀利杰, 李栋, 党增强, 吕金凤, 夏庆友.家蚕复眼突变系光泽眼 (lu) 和光泽小眼 (ve) 的复眼形态观察. 昆虫学报, 2013, 56(4): 350-357.
[15] Yamamoto K, Narukawa J, Kadono-Okuda K, Nohata J, Sasanuma M, Suetsugu Y, Banno Y, Fujii H, Goldsmith MR, Mita K. Construction of a single nucleotide polymorphism linkage map for the silkworm, Bombyx mori, based on bacterial artificial chromosome end sequences. Genetics, 2006, 173(1): 151-161.
[16] Yamamoto K, Nohata J, Kadono-Okuda K, Narukawa J, Sasanuma M, Sasanuma S, Minami H, Shimomura M, Suetsugu Y, Banno Y, Osoegawa K, de Jong PJ, Goldsmith MR, Mita K. A BAC-based integrated linkage map of the silkworm Bombyx mori. Genome Biol, 2008, 9(1): R21.
[17] Morgans CW, Kensel-Hammes P, Hurley JB, Burton K, Idzerda R, Mcknight GS, Bajjalieh SM. Loss of the Synaptic Vesicle Protein SV2B results in reduced neurotransmission and altered synaptic vesicle protein expression in the retina. Plos ONE, 2009, 4(4): e5230.
[18] Quiring R, Walldorf U, Kloter U, Gehring WJ. Homology of the eyeless gene of Drosophila to the Small eye gene in mice and Aniridia in humans. Science, 1994, 265(5173): 785-789.
[19] Jordan T, Hanson I, Zaletayev D, Hodgson S, Prosser J, Seawright A, Hastie N, van Heyningen V. The human PAX6 gene is mutated in two patients with aniridia. Nat Genet, 1992, 1(5): 328-332.
[20] Kawaguchi Y, Tatsuke T, Kusakabe T, Lee JM, Koga K. Singular compound eye architecture of the varnished eye mutation, ve, in Bombyx mori. Journal of Insect Biotechnology and Sericology (Japan), 2008, 77(1): 153-157.
(责任编委: 黄 原)
Fine mapping of the mutant gene varnished eye in the silkworm
Chun Liu, Qiongyan Li, Lijie Xun, Wenbo Hu, Jinfeng Lv, Rufeng Liu, Qingyou Xia
State Key Laboratory of Silkworm Genome Biology, the Key Sericultural Laboratory of the Ministry of Agriculture, Southwest University, Chongqing 400715, China
The varnished eye (ve) mutant is one of the rare natural recessive mutants related to the abnormal phenotypes in the compound eye of silkworm (Bombyx mori). The compound eyes of the ve adults are smaller and glossier than those of the wild-type Dazao (Dz) and many tumor-like bumps are present on their surface; their small eyes are irregularly arranged and are smaller and fewer than those of the wild-type. The mutant gene ve has been located at the 32.2 locus of chromosome 6 in the classic genetic linkage map. However, it still remains unknown about the mechanism responsible for the mutant. In this study, we got a BC1generation using male F1(ve×Dz) with female ve for fine mapping of this mutant gene. The results showed that ve was located in a region between SNP3 and SNP6. The physical distance is approximately 1.2 Mb in the low densitylinkage map. By constructing a high-density map using 1563 BC1individuals, the ve mutant gene was further mapped into a region between the SNP5 and SNP61. The physical region is about 221.8 kb and contains six potential genes but no specific mutations were found in the CDSs of these candidate genes. RT-PCR showed that the expression of BGIBMGA013642 was decreased obviously compared with that in wild type, suggesting it might be a key candidate gene for further studying the ve mutant.
silkworm (Bombyx mori); varnished eye; SNP marker; fine mapping; candidate genes
2014-03-26;
2014-06-03
国家高技术研究发展计划(863计划)项目(编号:2013AA102507),教育部“新世纪优秀人才支持计划”项目(编号:NCET-11-0699)和国家自然科学基金面上项目(编号:31372380)资助
刘春,博士,研究方向:家蚕功能基因组生物学。E-mail: mlliuchun@163.com
夏庆友,教授,博士生导师,研究方向:家蚕基因组生物学。E-mail: xiaqy@swu.edu.cn
致 谢: 特别感谢家蚕基因组生物学国家重点实验室的代方银教授为本实验提供实验材料。
10.3724/SP.J.1005.2014.1006
时间: 2014-9-2 10:38:24
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140911.1046.002.html