利用传递不平衡分析在多群体中鉴别猪脐疝易感基因
2014-05-25宿英龙毅阮国荣吴丽花张志燕肖石军邓玮芸吕显山胡豆吴国灶沈虎群廖信军丁能水黄路生
宿英,龙毅,阮国荣,2,吴丽花,张志燕,肖石军,邓玮芸,吕显山,胡豆,吴国灶,沈虎群,廖信军,3,丁能水,黄路生
1. 江西农业大学,动物生物技术国家重点实验室培育基地,南昌 330045;
2. 福建农业职业技术学院,福州 350119;
3. 井冈山大学生命科学学院,吉安 343009
利用传递不平衡分析在多群体中鉴别猪脐疝易感基因
宿英1,龙毅1,阮国荣1,2,吴丽花1,张志燕1,肖石军1,邓玮芸1,吕显山1,胡豆1,吴国灶1,沈虎群1,廖信军1,3,丁能水1,黄路生1
1. 江西农业大学,动物生物技术国家重点实验室培育基地,南昌 330045;
2. 福建农业职业技术学院,福州 350119;
3. 井冈山大学生命科学学院,吉安 343009
在本实验室前期利用白色杜洛克×二花脸F2资源家系开展脐疝易感位点全基因组扫描定位的基础上,文章在7号染色体上的SWR1928和10号染色体上的SW830易感标记区域,结合脐疝发病机制在多群体中进行脐疝位置功能候选基因的筛选和易感位点的精细定位。在两个显著关联的微卫星位点区域搜寻到 12个位置功能候选基因,采用比较测序法,选取12个候选基因内共计40个SNP位点在白色杜洛克×二花脸资源家系F2/F3脐疝群体中进行基因分型,利用Plink v1.07软件对基因型数据进行质量控制和传递不平衡(Transmission disequilibrium test,TDT)分析。结果表明,IL16(Interleukin 16)基因中的g.708C>T位点和CDC73(Cell division cycle 73)基因中的 g.10664G>A位点与脐疝的关联性达到显著水平(P<0.05)。对这两个位点在西方商业猪种脐疝患病家系中进行基因分型和TDT验证分析,发现CDC73基因中的g.10664G>A位点仍与猪脐疝呈显著关联(P<0.05)。同时对CDC73基因中与资源家系脐疝呈弱相关的两个SNP位点g.10546A>G和g.10811A>G在西方商业猪种中进行TDT验证分析,发现这两个SNP位点与商业猪种脐疝发生的关联性达到极显著水平(P<0.01)。根据文章的分析结果,结合脐疝发生的生理机制及CDC73基因的生物学功能,推测CDC73基因可能为猪脐疝发生的易感基因。
猪;脐疝;位置功能候选基因;TDT分析;CDC73
脐疝(Umbilical hernia,UH)是猪生产中最常见的生长发育缺陷之一,主要表现为因疝环异常扩大导致腹腔内容物(如小肠)脱至皮下形成囊状物,其发病率一般在0.13%~5%之间[1~5],给养猪产业造成巨大经济损失并降低猪只福利[3]。该疾患不但影响猪只体形外貌,而且妨碍生长发育、降低饲料转化率、损害种猪使用性能(一般不留作种用)、影响商品猪品质。猪脐疝发生的病因复杂,主要有先天遗传因素和后天环境因素,脐疝家系聚集性发病、品种间发病率和遗传易感性差异提示遗传因素在其发病机制中具有重要作用[5]。通过表型选择通常难以降低猪脐疝的发生率,因此分离和鉴别脐疝主效致病基因或与之紧密连锁的分子标记成为猪脐疝抗病育种的关键,对现代规模化养猪生产具有十分重要的意义。同时,猪还是人类疾病研究的极好模型,猪脐疝发生的分子遗传机理研究也将推动人类脐疝(脐带疝、脐膨出或腹壁疝)发生的分子遗传解析。
脐疝发病的分子病理机制目前尚不清楚,但综合人类和小鼠腹壁缺陷发生机制、动物胚胎发生及脐疝解剖学和病理学研究成果,脐疝发生可能与腹壁细胞增殖和凋亡紊乱[6~8]、上皮细胞-间叶细胞互作和表型转化异常[9~11]以及胶原代谢紊乱[12~14]相关。
近年来,研究者普遍采用基因组扫描和全基因组关联分析等策略对复杂疾病进行相关基因及易感区域的鉴别。迄今为止,有关猪脐疝易感位点定位的研究仅见于本实验室的前期报道,Ding等[15]利用涵盖猪基因组范围内的194个微卫星标记对F2资源家系脐疝群体进行全基因组扫描,在12条染色体上鉴别到影响猪脐疝发生的易感位点。用传递不平衡分析(Transmission disequilibrium test,TDT)和非参数连锁分析(Non-parametric linkage analysis,NPL)两种方法同时检测到 7号染色体(SSC7)上的SWR1928标记和10号染色体(SSC10)的SW830标记与脐疝发生呈显著相关(P<0.05)。Ron等[16]在一个脐疝发病率高达9.7%的荷斯坦奶牛家系中,利用覆盖牛29条染色体的59个微卫星标记对包含49头脐疝患病个体在内的 119头奶牛进行全基因组扫描及单倍型分析,最终将脐疝易感位点定位在牛8号染色体(BTA8)着丝粒附近的标记 AR071/AR072与UWCA47之间,并在该区域内筛选到功能候选基因GATA47,该基因的表达产物是 Ca2+/钙调磷酸酶NF-AT通路中的信号元件,而NF-TA信号通路阻滞会影响小鼠脐环的形成[17]。小鼠的腹壁缺陷研究中,AP2-α家族基因及TGF-β家族基因敲除的小鼠胚胎均表现出不同程度的腹壁发育缺陷[10,18,19]。
本研究在SSC7和SSC10上两个已定位的猪脐疝易感基因组区域内筛选到 12个位置候选基因并据此开发 SNP标记。在白色杜洛克×二花脸资源家系 F2/F3脐疝群体中对这些 SNP位点开展相关性分析,显著关联的SNP在远缘商业脐疝群体中进一步进行重复验证,从而锚定易感基因。本研究结果将有助于解析脐疝发生的分子病理机制,并为建立“抗脐疝”猪的分子育种提供重要参考数据。
1 材料和方法
1.1 动物及病理诊断
1.1.1 资源群体
资源家系脐疝群体中有 58个脐疝患病家系共计160个个体,其中脐疝患病个体75头,每个家系患病个体数有1~6个不等。该群体是以2 头白色杜洛克公猪和 17 头二花脸母猪为祖代,随机选取 9头F1公猪和59头F1母猪自群繁育(避免全同胞交配)产生1912头F2个体,其中脐疝患病个体50头,随机选取F2代脐疝个体的正常全同胞公猪与母猪杂交繁育产生 F3,其中脐疝患病个体 25头[15]。每头患病个体详细记录系谱、性别并拍照留档。因仔猪出现脐疝的发生是遗传和环境共同作用的结果,因此脐疝表型判定均在猪只 21~72 d时由专业兽医师进行仔细判定,从而减小环境致病因素的干扰作用。
1.1.2 远缘群体
远缘商业群体来自于全国15个核心育种场,品种包括纯种杜洛克、长白、大白、皮特兰、斯格及部分长大二元猪(表1),共计113个家系,每个家系中患病个体的数目为1~2个不等。样本采集标准与资源家系相同。
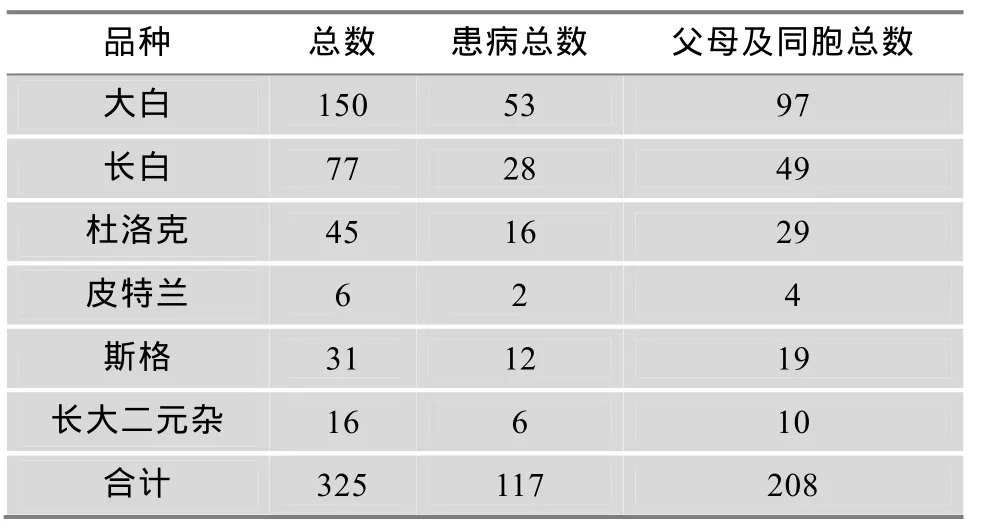
表1 远缘群体中猪脐疝患病个体的品种分布情况
1.2 方法
1.2.1 基因组DNA提取及质量检测
采集0.5~1.0 g猪耳组织,按照标准酚/氯仿法提取DNA,然后将其溶于TE缓冲液中。用Nanodrop 1000核酸蛋白分析仪(Thermo Scientific, USA)测定DNA的浓度和质量,A260/A280的比值在1.8~2.0之间判定为合格。将浓度统一稀释至50 ng/μL,用琼脂糖凝胶电泳检测DNA片段的完整性,片段长度大于10 kb视为合格。质量合格的DNA样品用于多态位点的搜寻及基因分型。
1.2.2 多态位点的搜寻及基因分型
1.2.2.1 运用比较测序法搜寻 SNP位点 在 Ensembl(http:/www.ensembl.org/index.html)网站搜寻获得猪12个基因(表2)的文库序列,对这些基因的部分外显子和内含子使用在线引物设计软件(http: //frodo.wi.mit.edu/cgi-bin/primer3/primer3.cgi)设计引物(部分引物见表2)并进行PCR扩增。PCR模板来自资源家系中患病个体与正常个体分别混合形成的两个DNA池。PCR扩增体系为25 μL,包括:基因组DNA 20 ng,ddH2O 17.5 μL,25 mmol/L Mg2+1.5 μL,10 mmol/L dNTPs 0.5 μL,上、下游引物(10 μmol/L)各0.5 μL,10 × PCR buffer 2.5 μL,Taq DNA 聚合酶2.5 U(博彩生物科技有限公司,上海)。PCR扩增反应于PTC-200热循环仪(BIO-RAD,USA)上进行。反应条件为:94℃预变性3 min;94℃变性30 s,复性(部分引物复性温度见表2) 30 s,72℃延伸40 s,共35个循环;72℃延伸10 min。PCR产物经2%琼脂糖凝胶电泳检测后,用QIAquick PCR纯化试剂盒(QIAGEN,Germany)纯化,委托深圳华大中生科技发展有限公司双向测序。通过BLAST分析,验证其特异性,再用DNAStar软件包的Seqman程序分析鉴别SNPs。
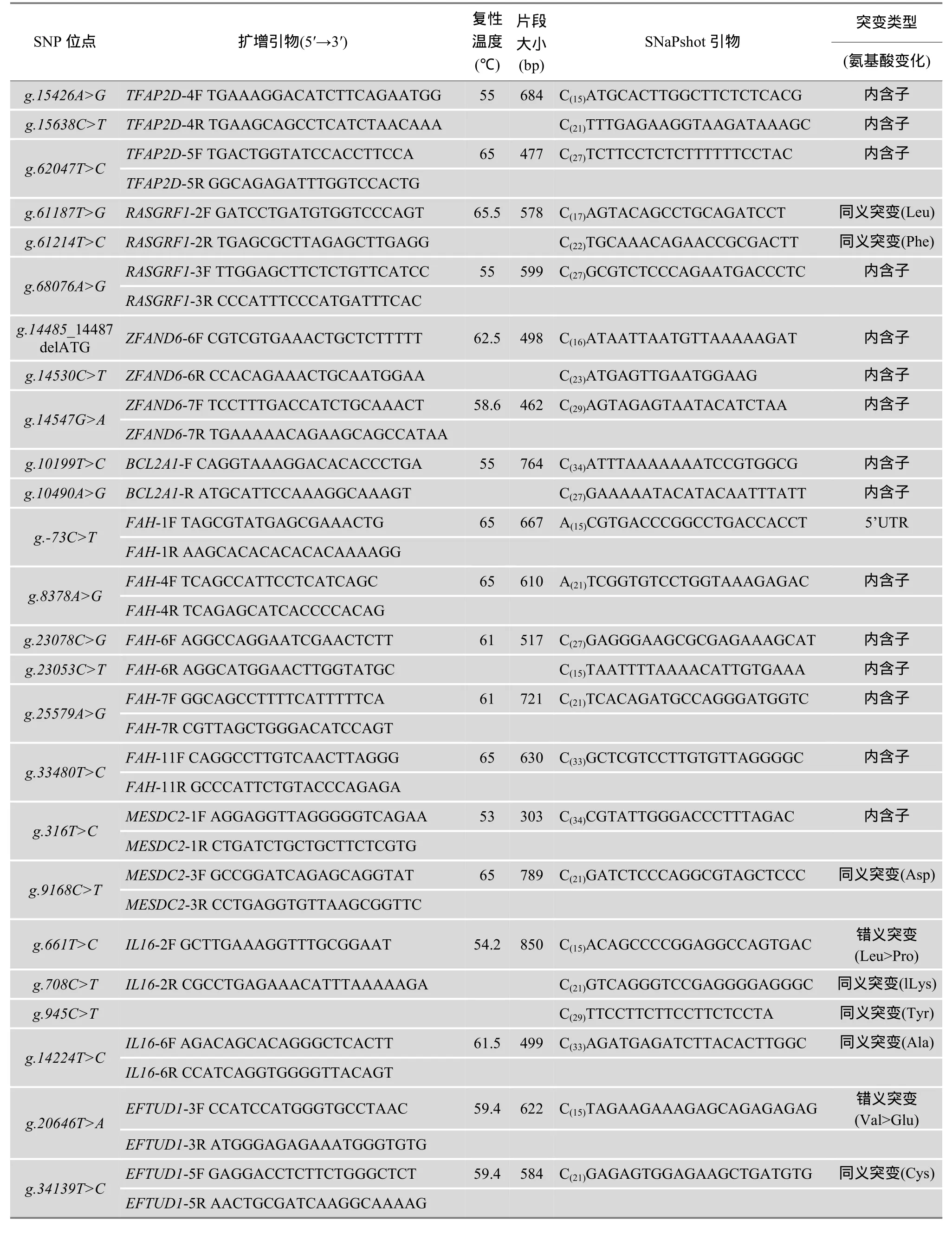
表2 利用SNaPshot技术检测的38个SNP位点的突变类型及所用引物
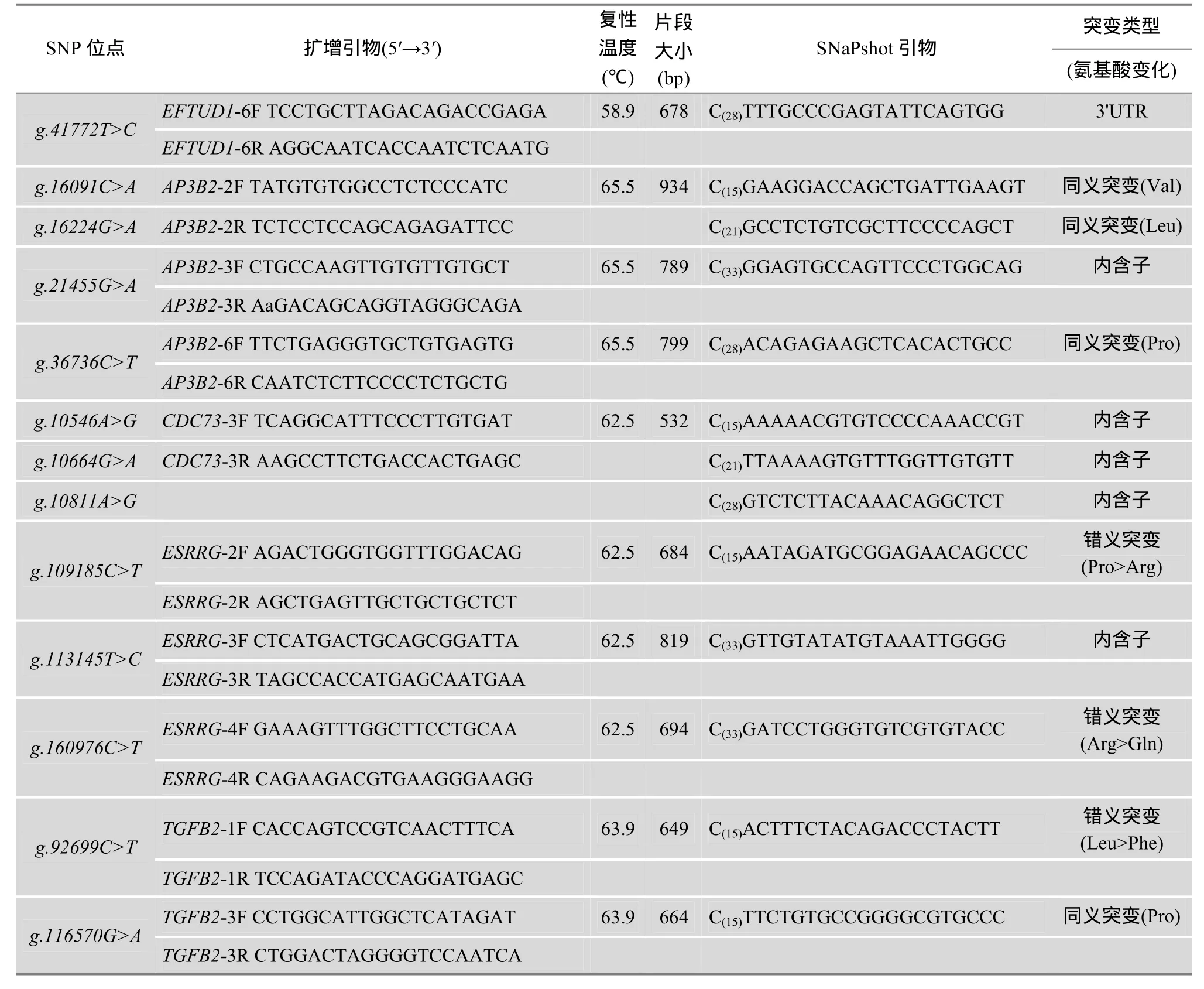
续表2
1.2.2.2 SNaPshot基因分型 通过比较分析,在12个候选基因中共检测到 99个多态位点。选取在正常池与患病池存在差异或位于外显子上的 38个SNP位点,在资源家系中利用 SNaPshot技术[20,21]进行基因分型。SNaPshot反应前PCR扩增所用引物与SNP搜寻引物(表2)相同,SNaPshot 技术流程依照 SNaPshot ® Multiplex 试剂盒(ABI,USA)说明书进行。具体过程包括PCR产物纯化、 SNaPshot反应、SNaPshot反应产物纯化和纯化产物变性及上样4个步骤。利用3130 xl遗传分析仪(ABI,USA)对产物进行毛细管电泳检测,经 Gene Mapper Software version 3.7 (ABI,USA)分析后得到基因型数据。
1.2.2.3 PCR-RFLP方法进行基因分型ZFAND6基因中 g.2395 C>T和 g.15060 A>G 两个位点用PCR-RFLP方法进行基因分型。过程如下:在3 μL PCR产物(引物见表3)中加入0.2 μL限制性内切酶(10 U/μL)、1.2 μL10×缓冲液和7.6 μL去离子水,于37℃温育6 h或过夜。上述反应产物经3.0%的琼脂糖凝胶电泳检测并记录不同片段所代表的基因型结果。
1.2.3 统计分析
基于家系的传递不平衡检验(TDT)是研究疾病性状的重要方法,可有效避免复杂的群体分层效应导致的结果偏差。本研究采样来源广泛、遗传背景复杂,具有一定程度的群体分层效应,因而选用plink v 1.07程序中的TDT[22]命令进行关联分析。其原理是:分析某个等位基因从杂合子父母传递给患病子代的机率是否高于预期值(50%),如果这个家系至少有一个亲本是杂合子,则进行经典的TDT分析;如果双亲的基因型缺失,则进行同胞TDT(Sib transmission disequilibrium test,S-TDT)检验[23,24]。当连锁不平衡程度很高的情况下,标记与致病基因紧密
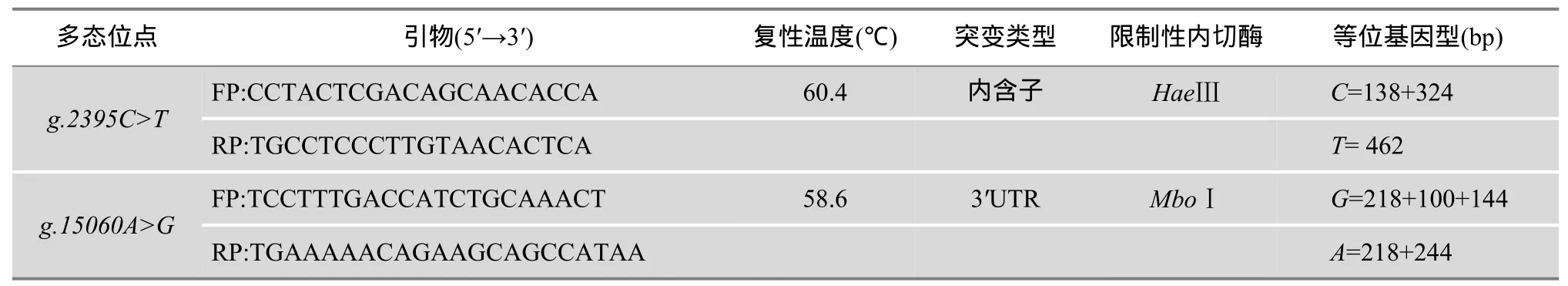
表3 ZFAND6基因内两个SNPs的突变类型及分型方法
连锁时,它的检测效率很高。计算公式如下:
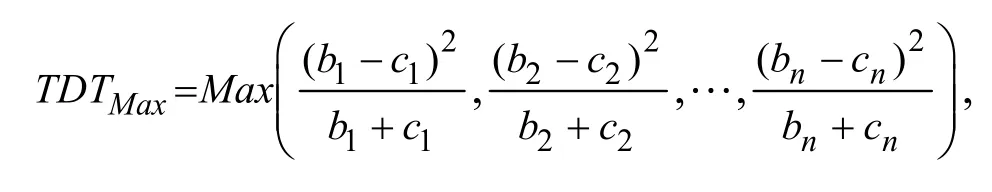
其中n是SNP的数目,bi为传递的SNP数目,ci为未传递的SNP的数目。
2 结果与分析
2.1 SNP搜寻结果
通过比较分析,在 12个候选基因中共检测到99个多态位点。选择在正常池与患病池存在差异或位于外显子上的40个SNP位点(表4)作为候选SNP在资源家系中进行基因分型,40个SNPs在易感区域内的分布情况见图1。
2.2 PCR-RFLP方法进行基因分型结果
利用PCR-RFLP方法对ZFAND6基因中g.2395 C>T和g.15060 A>G两个位点进行基因分型,结果见图2。
2.3 基因型数据质控结果
应用PLINK v1.07[22]软件分别对F2/F3资源家系脐疝群体的 40个多态位点 (Sscrofa10.2版本 http: //www.ensembl.org/Sus_scrofa/Info/Index)及在远缘三联体核心家系群中需要验证的4个SNP位点进行质量控制。剔除个体基因型检出率小于75%的个体;剔除SNP位点检出率小于90%、哈迪温伯格平衡卡方检验P≤10-5和最小等位基因频率小于0.05的SNP位点。结果表明,F2/F3资源家系脐疝群体中有157个个体通过检验,质控后剩余30个SNP标记可用于后续研究。远缘群体中325个个体的4个SNP位点全部通过检验,可用于后续TDT重复验证分析。
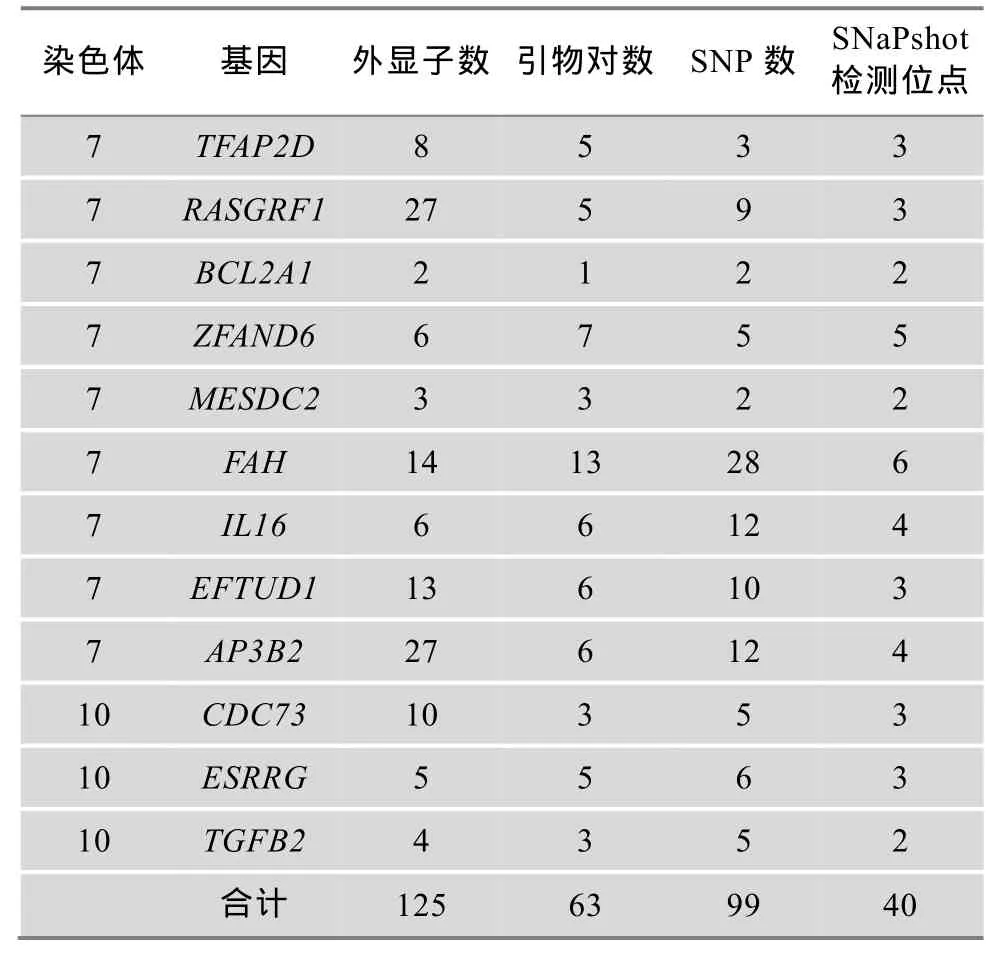
表4 12个位置候选基因中SNP搜寻结果汇总
2.4 资源家系脐疝群体多态位点与猪脐疝的关联性分析结果
2.4.1 白色杜洛克×二花脸资源家系脐疝群体的TDT分析
TDT分析结果显示,白色杜洛克×二花脸F2/F3资源家系脐疝群体中12个位置候选基因内的40个SNP位点有30个通过质量控制并用于TDT分析,与脐疝关联性最强的多态位点是位于SSC7上IL16基因内的g.708C>T位点(P<0.05) (表5)。另外,SSC10上CDC73基因内的 g.10664G>A位点与脐疝的关联性也达到显著水平(P<0.05)。
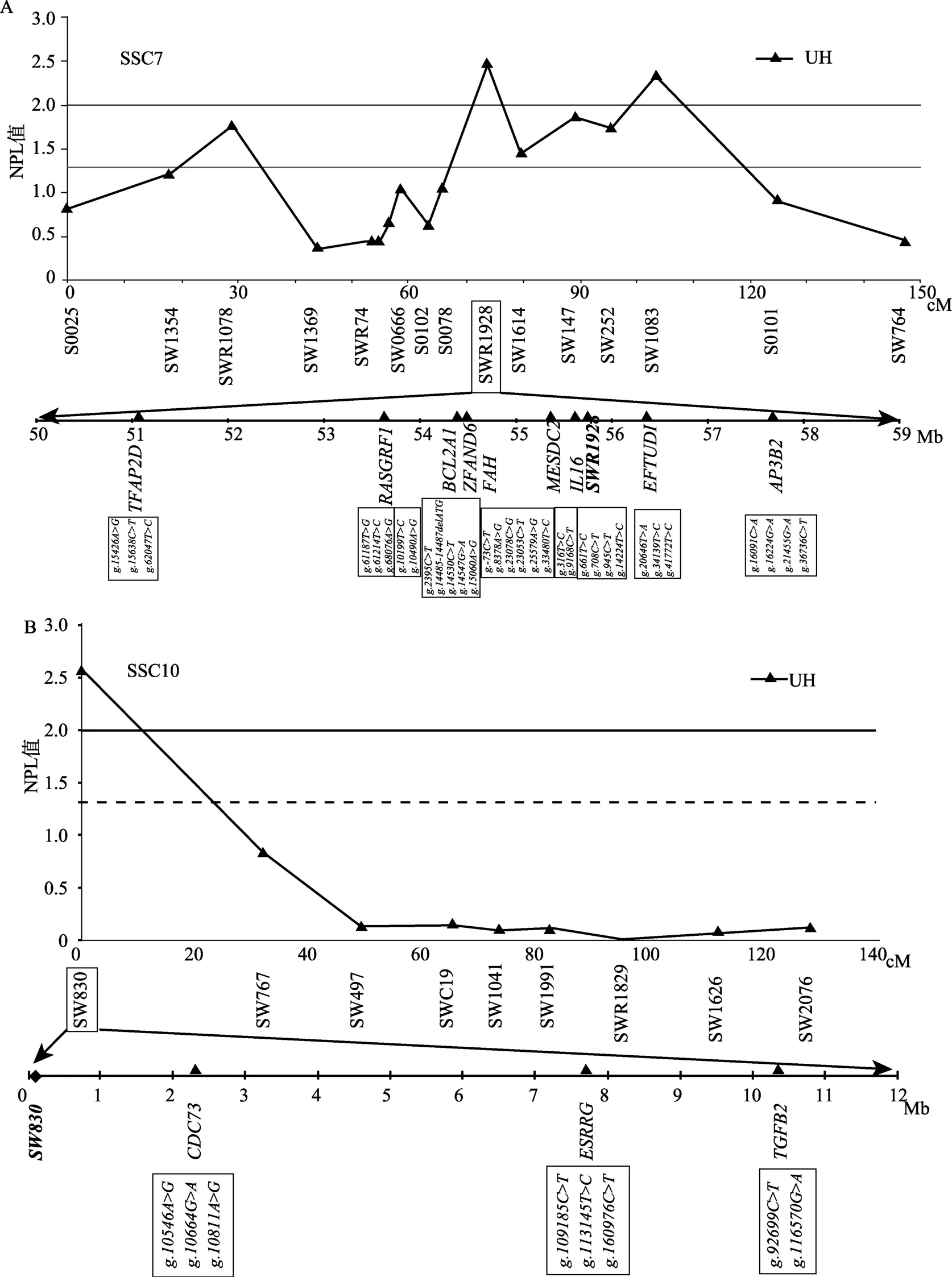
图1 SSC7(A)和SSC10(B)上脐疝易感区域的基因及所检测的SNP位点分布图A:利用微卫星标记在白色杜洛克×二花脸资源家系中采用NPL方法在SSC7上与脐疝连锁分析检测结果的折线图。其中横坐标表示染色体上的位置,用cM表示;纵坐标表示NPL值,下同。下方横线为SWR1928标记易感区域放大图,横线上黑色三角表示候选基因的物理位置位置,黑色菱形表示微卫星的物理位置,用Mb表示,下同。基因下方的黑框内是位于候选基因内所检测的SNP位点名称,其位置是从该基因起始密码子ATG开始算起的物理距离,以bp表示,下同。B:SSC10上与脐疝连锁分析检测结果的折线图。下方横线为SW830标记易感区域的放大图;基因下方的黑框内是位于候选基因内所检测的SNP位点名称。
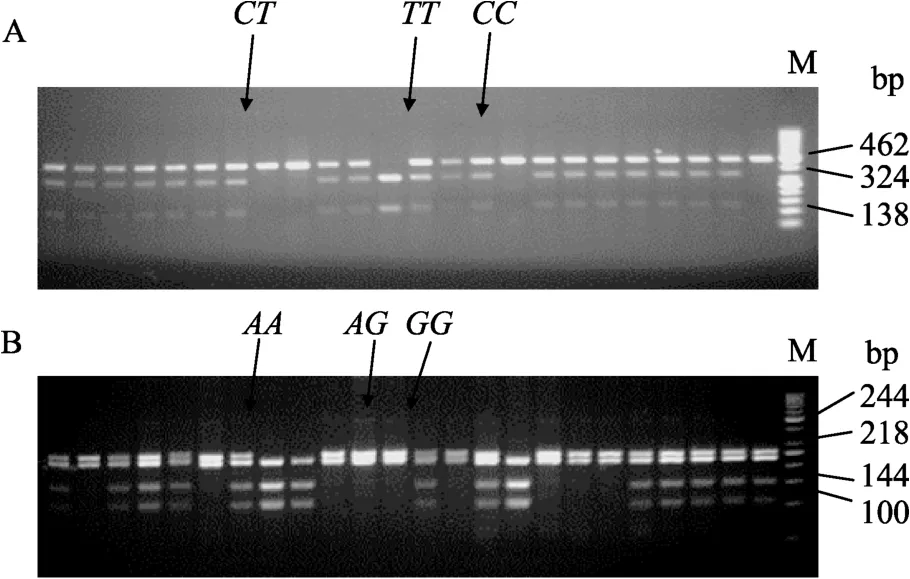
图2 ZFAND6基因两个SNPs的PCR-RFLP判型结果A:g.2395C>T位点酶切结果(两个条带为CC型,3个电泳条带为CT型,1个条带为TT型);B:g.15060A>G位点酶切结果(2个条带为AA型,4个条带为AG型,3个条带为GG型)。
2.4.2 IL16基因和CDC73基因中SNP位点在远缘群体中与脐疝的TDT分析
IL16基因内 g.708C>T位点和 CDC73基因内g.10664G>A位点在资源家系中与脐疝的发生呈显著关联,对该两位点在远缘群体中进行关联重复验证,TDT分析表明IL16基因内 g.708C>T位点在远缘群体中并没有表现出与脐疝发病的关联性,但CDC73基因中的 g.10664G>A位点与脐疝发生呈显著相关。鉴于这一结果,对CDC73基因中的另外两个在资源家系脐疝群体中呈弱相关(P<0.1)的位点 g.10546A>G和g.10811A>G(表 6)在远缘商业猪群 325个个体中进行基因分型及 TDT分析,结果表明:g.10546A>G和g.10811A>G位点与脐疝的发生呈极显著相关(P<0.01)。
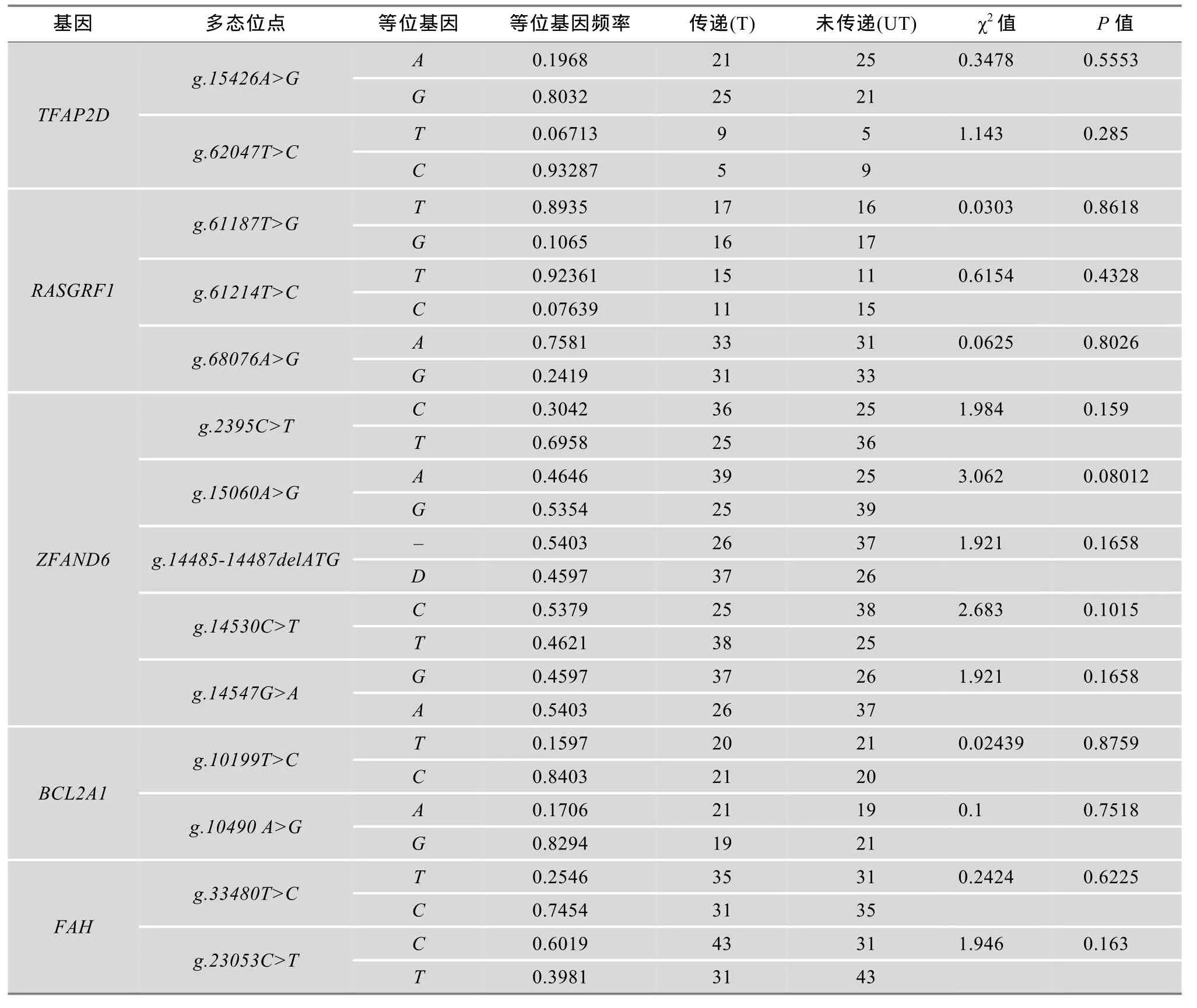
表5 12个位置候选基因内30个SNP 位点与白色杜洛克×二花脸资源家系中脐疝的关联性分析结果
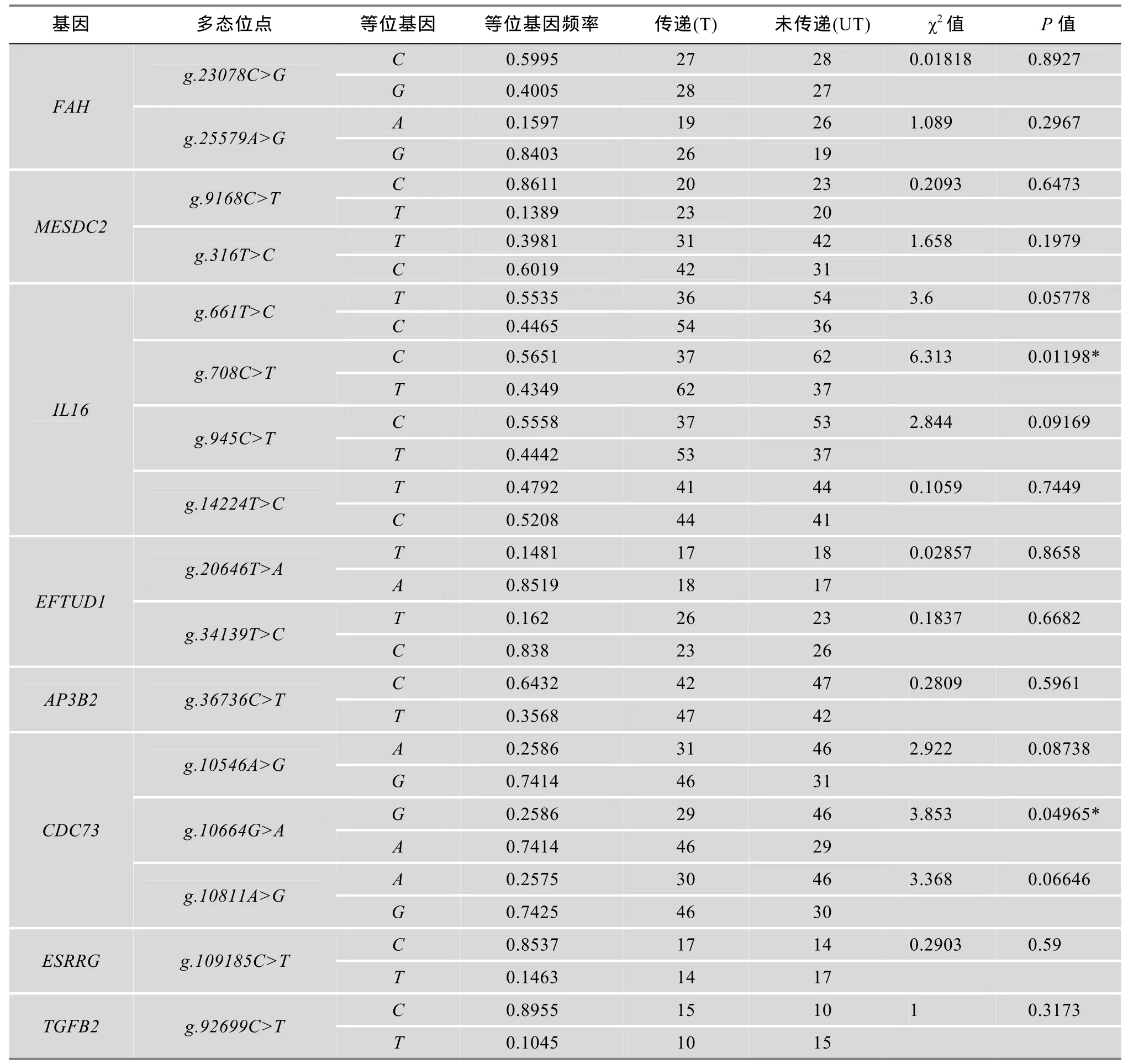
续表5

表6 IL16和CDC73基因中SNP位点在远缘群体中与脐疝的TDT分析结果
3 讨 论
脐疝作为一种复杂的遗传缺陷疾病,给养猪业带来较大经济损失且影响猪只福利。本研究通过对本实验室前期在白色杜洛克×二花脸F2资源家系脐疝群体中定位到的两个影响脐疝发生的QTL区域[15]内12个位置候选基因的研究发现:IL16基因中g.708C>T位点和CDC73基因中g.10664G>A位点与脐疝发生呈显著关联,且分别是突变型T和A传递给后代患脐疝的可能性更大(表5)。IL16是淋巴细胞趋化因子,其前体在胞质内由胱天蛋白酶3裂解成2个片段:N端片段和C端片段。N端片段包含2个PDZ结构域和一个有利于转移至核内的核定位序列,该片段转入核内参与细胞周期的调节(阻滞G0/G1转化)[25],高志涛等[26]证实 IL16对人白血病细胞系 MT2 细胞增殖受阻且出现细胞凋亡现象。胚胎发育早期,胚位于脐环部位的外胚层基板,处于外胚层与中胚层的连接过度区域,随着胚胎的发育,外胚层出现细胞程序性凋亡、基底膜分解,中胚层细胞则正常增殖并在中胚层囊腔中沉积,最终在脐环处闭锁形成腹壁[27],脐环闭锁失败则可能导致脐疝的发生。推测IL16基因的突变可能引起外胚层细胞凋亡或中胚层细胞增殖受阻而导致脐部闭合缺陷,从而导致脐疝的发生。CDC73基因则编码细胞分裂蛋白,亦是RNA聚合酶II复合物因子1(Paf1)的重要组成成分,CDC73基因通过Paf1来参与转录因子的激活、3'mRNA的加工剪切、补充和激活组蛋白及染色质的正常延伸等与细胞循环分裂、蛋白转录合成、核酸代谢等过程[28]。Rather等[29]研究表明CDC73下调可抑制细胞凋亡,上调抑制细胞增殖,且可通过PAF1C参与wnt通路,而wnt通路可调控上皮细胞-间叶细胞互作和表型转化(EMT)[30],EMT发生异常会导致腹壁闭合和发育异常,从而诱导脐疝的形成。
对这两个基因中的关联 SNP在远缘群体中进行重复验证研究,CDC73基因在两个群体中均表现出与脐疝发生不同程度的关联性,推测其可能在猪脐疝的发生中发挥重要作用。而IL16基因中候选SNP并未得到验证,其原因可能是:作为一种复杂的遗传缺陷疾病,脐疝具有复杂疾病所特有的致病因子多、遗传异质性强、遗传和环境因素存在紧密互作等特点,微效多基因模式可能是其主要的发生机制[31]。微效多基因模式提示,脐疝易感或风险基因有可能在群体中的发生频率都很低,每个基因作用微小,单独不足以致病,且可能对于疾病既非充分也非必要,但它们之间存在相互作用,通过多个基因的剂量效应,达到疾病发生的临界点,从而共同决定脐疝的遗传易感性。因此,IL16基因和CDC73基因内的SNP位点与脐疝的关联性仅仅达到显著水平,其可能只是众多易感基因中的一小部分。同时,IL16基因并未在远缘群体中得到验证可能是群体异质性造成的,即资源家系与远缘群体中的脐疝致病突变存在共性的同时也存在一定的特异性。
综上所述,本研究在白色杜洛克×二花脸资源家系中鉴别到两个脐疝易感基因IL16和CDC73中存在脐疝关联位点,经在远缘群体中的进一步TDT重复验证,发现CDC73基因极有可能是影响猪脐疝的候选易感基因。
[1] 施启顺, 李菊芬, 蒋武明, 谢新民. 猪遗传性疾患发生率的调查. 遗传, 1988, 10(4): 25-27.
[2] 金基燮, 徐海洪, 姜国伟, 段莹莹. 仔猪脐疝的防治.黑龙江畜牧兽医, 2005, (4): 43-43.
[3] Straw B, Bates R, May G. Anatomical abnormalities in a group of finishing pigs: prevalence and pig performance. J Swine Health Prod, 2009, 17(1): 28-31.
[4] Petersen HH, Nielsen EO, Hassing AG, Ersbøll AK, Nielsen JP. Prevalence of clinical signs of disease in Danish finisher pigs. Vet Rec, 2008, 162(12): 377-382.
[5] Searcy-Bernal R, Gardner IA, Hird DW. Effects of and factors associated with umbilical hernias in a swine herd. J Am Vet Med Assoc, 1994, 204(10): 1660-1664.
[6] Brewer S, Williams T. Finally, a sense of closure? Animal models of human ventral body wall defects. Bioessays, 2004, 26(12): 1307-1321.
[7] Brewer S, Williams T. Loss of AP-2α impacts multiple aspects of ventral body wall development and closure. Dev Biol, 2004, 267(2): 399-417.
[8] 胡烈榛, 夏焙, 林洲, 于红奎, 王娟. 儿童先天性肠旋转不良的超声诊断价值. 中华临床医师杂志(电子版), 2013, 7(6): 2429-2432.
[9] Acloque H, Adams MS, Fishwick K, Bronner-Fraser M, Nieto MA. Epithelial-mesenchymal transitions: the importance of changing cell state in development and disease. J Clin Invest, 2009, 119(6): 1438.
[10] Dünker N, Krieglstein K. Tgfβ2-/-Tgfβ3-/-double knockout mice display severe midline fusion defects and early embryonic lethality. Anat Embryol, 2002, 206(1-2): 73-83.
[11] Baum B, Settleman J, Quinlan MP. Transitions between epithelial and mesenchymal states in development and disease. Semin Cell Dev Biol, 2008, 19(3): 294-308.
[12] Bendavid R. The unified theory of hernia formation. Hernia, 2004, 8(3): 171-176.
[13] Klinge U, Zheng H, Si Z, Schumpelick V, Bhardwaj RS, Muys L, Klosterhalfen B. Expression of the extracellular matrix proteins collagen I, collagen III and fibronectin and matrix metalloproteinase-1 and -13 in the skin of patients with inguinal hernia. Eur Surg Res, 1999, 31(6): 480-490.
[14] Jansen PL, Mertens PR, Klinge U, Schumpelick V. The biology of hernia formation. Surgery, 2004, 136(1): 1-4.
[15] Ding NS, Mao HR, Guo YM, Ren J, Xiao SJ, Wu GZ, Shen HQ, Wu LH, Ruan GF, Brenig B et al. A genomewide scan reveals candidate susceptibility loci for pig hernias in an intercross between White Duroc and Erhualian. J Anim Sci, 2009, 87(8): 2469-2474.
[16] Ron M, Tager-Cohen I, Feldmesser E, Ezra E, Kalay D, Roe B, Seroussi E, Weller JI. Bovine umbilical hernia maps to the centromeric end of Bos taurus autosome 8. Anim Genet, 2004, 35(6): 431-437.
[17] Mesaeli N, Nakamura K, Zvaritch E, Dickie P, Dziak E, Krause K-H, Opas M, MacLennan DH, Michalak M. Calreticulin is essential for cardiac development. J Cell Biol, 1999, 144(5): 857-868.
[18] Schorle H, Meier P, Buchert M, Jaenisch R, Mitchell PJ. Transcription factor AP-2 essential for cranial closure and craniofacial development. Nature, 1996, 381(6579): 235-238.
[19] Zhang J, Hagopian-Donaldson S, Serbedzija G, Elsemore J, Plehn-Dujowich D, McMahon AP, Flavell RA, Williams T. Neural tube, skeletal and body wall defects in mice lacking transcription factor AP-2. Nature, 1996, 381(6579): 238-241.
[20] Ben-Avi L, Durst R, Shpitzen S, Leitersdorf E, Meiner V. Aplipoprotein E genotyping: accurate, simple, high throughput method using ABI Prism® SNaPshot™ Multiplex System. J Alzheimers Dis, 2004, 6(5): 497-501.
[21] Pati N, Schowinsky V, Kokanovic O, Magnuson V, Ghosh S. A comparison between SNaPshot, pyrosequencing, and biplex invader SNP genotyping methods: accuracy, cost, and throughput. J Biochem Biophys Methods, 2004, 60(1): 1-12.
[22] Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MAR, Bender D, Maller J, Sklar P, De Bakker PIW, Daly MJ, Sham PC. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet, 2007, 81(3): 559-575.
[23] Whittemore AS, Halpern J. A class of tests for linkage using affected pedigree members. Biometrics, 1994, 50(1): 118-127.
[24] Risch N, Merikangas K. The future of genetic studies of complex human diseases. Science, 1996, 273(5281): 1516-1517.
[25] Sciaky D, Brazer W, Center DM, Cruikshank WW, Smith TJ. Cultured human fibroblasts express constitutive IL-16 mRNA: cytokine induction of active IL-16 protein synthesis through a caspase-3-dependent mechanism. J Immunol, 2000, 164(7): 3806-3814.
[26] 高志涛, 朱小飞, 朱茉莉, 王辉. IL-16对人白血病细胞系 MT2 细胞增殖抑制和凋亡的影响. 细胞与分子免疫学杂志2010, (10): 1002-1004.
[27] Munger GT, Munger BL. Differentiation of the anterior body wall and truncal epidermis and associated co-migration of cutaneous nerves and mesenchyme. Anat Rec, 1991, 231(2): 261-274.
[28] Moniaux N, Nemos C, Deb S, Zhu B, Dornreiter I, Hollingsworth MA, Batra SK. The human RNA polymerase II-associated factor 1 (hPaf1): a new regulator of cell-cycle progression. PLoS ONE, 2009, 4(9): e7077.
[29] Rather MI, Nagashri MN, Swamy SS, Gopinath KS, Kumar A. Oncogenic microRNA-155 down-regulates tumor suppressor CDC73 and promotes oral squamous cell carcinoma cell proliferation: implications for cancer therapeutics. J Biol Chem, 2013, 288(1): 608-618.
[30] Jiang YG, Luo Y, He DL, Li X, Zhang LL, Peng T, Li MC, Lin YH. Role of Wnt/β-catenin signaling pathway in epithelial-mesenchymal transition of human prostate cancer induced by hypoxia-inducible factor-1α. Int J Urol, 2007, 14(11): 1034-1039.
[31] Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M. Genomewide scans of complex human diseases: true linkage is hard to find. Am J Hum Genet, 2001, 69(5): 936-950.
(责任编委: 李明洲)
Identification of susceptibility gene for pig umbilical hernia in different populations using transmission disequilibrium test
Ying Su1, Yi Long1, Guorong Ruan1,2, Lihua Wu1, Zhiyan Zhang1, Shijun Xiao1, Weiyun Deng1, Xianshan Lv1, Dou Hu1, Guozao Wu1, Huqun Shen1, Xinjun Liao1,3, Nengshui Ding1, Lusheng Huang1
1. Candidate of National Key Laboratory for Animal Biotechnology, Jiangxi Agricultural University, Nanchang, 330045, China;
2. Fujian Vocational College of Agriculture, Fuzhou 350119, China;
3. College of Life Science of Jinggangshan University, Ji’an 343009, China
A genome-wide scan for pig umbilical hernia (UH) was performed in a White Duroc × Erhualian resource population reported by our previously study, which detected two susceptibility microsatellite markers (SWR1928 on SSC7 and SW830 on SSC10) significantly affecting pig UH. Herein, fine mapping studies and identification of susceptibility genes for UH were performed in two different populations. A total of 40 SNPs in 12 positional candidate genes located on the two significant segments were genotyped in the F2/F3resource population. Quality control of the genotype data and transmission disequilibrium test (TDT) were conducted using Plink v1.07 software. The results showed that g.708G>A in IL16 (interleukin 16) gene and g.10664G>A in CDC73 (cell division cycle 73) gene were significantly associated with pig UH. These two prominent SNPs and another two weakly associated SNPs g.10546A>G and g.10811A>G in CDC73 were also undergone the replication TDT test in the outbred commercial populations. All SNPs in the CDC73 gene were confirmed to be significantly associated with pig UH (P<0.05), including g. 10546A>G and g.10811A>G with extreme significant level (P<0.01). Based on these results, CDC73 should be a susceptibility gene for pig UH according to its biological functions and the molecular pathogenesis of UH.
pig; umbilical hernia; positional and functional candidate gene; transmission disequilibrium test; CDC73
2014-03-16;
2014-08-11
国家自然科学基金项目(编号:31272422),教育部新世纪优秀人才计划项目(编号:教技函[2012]80),国家科技支撑项目(编号:2011BAD28B01)和江西省现代农业产业技术体系建设专项资金(编号:JXARS-03)资助。
宿英,硕士,专业方向:动物遗传育种与繁殖。E-mail:xy19880910@yeah.net;龙毅,博士,研究方向:动物遗传育种与繁殖。E-mail:longyilongyily@163.com宿英和龙毅同为第一作者。
丁能水,博士,研究员,博士生导师,研究方向:动物育种新技术研究。E-mail: dingyd2005@hotmail.com
致 谢: 本文脐疝样本的采集得到江西正邦集团辖属种猪场、广西农垦永新畜牧有限公司良圻原种猪场、广西杨翔农牧有限公司、广东华农温氏畜牧股份有限公司、河南正阳诸美原种猪场、海南罗牛山种猪育种有限公司、安徽长风农牧科技有限公司、福建省永诚华多种猪有限公司、山东省日照原种猪场等全国15个原种猪场的大力支持,在此一并感谢!
10.3724/SP.J.1005.2014.0995
时间: 2014-9-2 10:34:34
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140911.1049.004.html