猪microRNA组学研究进展
2014-05-25冉茂良陈斌尹杰杨岸奇李智蒋明
冉茂良,陈斌,尹杰,杨岸奇,李智,蒋明
1. 湖南农业大学动物科学技术学院,长沙 410128;
2. 畜禽遗传改良湖南省重点实验室,长沙 410128;
3. 中国科学院亚热带农业生态研究所,长沙 410125;
4. 中国科学院研究生院,北京 100049
猪microRNA组学研究进展
冉茂良1,2,陈斌1,2,尹杰3,4,杨岸奇1,2,李智1,2,蒋明1,2
1. 湖南农业大学动物科学技术学院,长沙 410128;
2. 畜禽遗传改良湖南省重点实验室,长沙 410128;
3. 中国科学院亚热带农业生态研究所,长沙 410125;
4. 中国科学院研究生院,北京 100049
MicroRNA(miRNA)是一类长约22 nt的非编码小RNA,广泛存在于各种生物中,调节生物体生长、发育和凋亡等过程。研究表明,miRNA在猪肌肉、脂肪、生殖系统以及免疫系统等的发育过程中发挥着重要的调控作用。此外,高通量的新一代测序技术在猪miRNA的挖掘和差异表达研究中发挥着巨大的作用。文章综述了高通量的新一代测序技术在挖掘猪miRNA中的应用以及一些miRNA在猪脂肪代谢、肌肉发育、卵母细胞成熟和B、T淋巴细胞发育中的调控作用,旨在为猪miRNA的研究提供参考,为利用miRNA调控和改善猪肉品质、生长性能、繁殖性能以及免疫机能提供理论基础和研究思路。
猪;microRNA;脂肪代谢;肌肉发育;卵母细胞成熟;免疫
Keywords:porcine; miRNA; fat metabolism; muscle development; oocyte maturation; immunity
猪不仅为人类提供日常肉质品还可以作为模式动物研究人类疾病[1],在畜牧业和科学研究中占据着十分重要的地位,因此关于猪的基因研究一直是科研领域的热点。自 1993年从线虫中首次发现miRNA(Lin-4)[2]且其在线虫发育过程中的时间控制作用同时也得到证实以来,动植物基因的表达调控研究中涌现出了一个崭新的领域——miRNA调控。miRNA 调控主要是通过碱基互补配对原则与靶mRNA结合后形成双链 RNA,从而诱导靶 mRNA降解或抑制其翻译过程[3,4],研究表明 miRNA在细胞增殖、分化和凋亡,生物的生长、发育以及疾病的发生和发展中均发挥着重要的调控作用[5,6]。但是,截止2014年4月15日,miRBase 20.0数据库(http: //www.mirbase.org/)收录猪miRNAs前体280条,成熟体326条;而人miRNAs前体1872条,成熟体2578条;小鼠miRNAs前体1186条,成熟体1908条;大鼠miRNAs前体449条,成熟体728条,相比于同等基因组大小的人、小鼠和大鼠等哺乳动物,猪miRNA的研究还需亟待加强。目前关于猪miRNA的研究方法主要集中于微阵列芯片、高通量测序、实时荧光定量PCR等技术以提高检测通量、灵敏度和特异性[7];其研究的主要内容多集中于miRNA对肌肉、脂肪和生殖系统生长发育以及疾病的发生和发展的调控作用。因此,本文对猪miRNA的研究进展,尤其是猪 miRNA的挖掘、部分重要 miRNAs对猪脂肪和肌肉发育、繁殖生理及免疫调控进行了综述,以期为猪miRNA的研究提供参考。
1 猪miRNA挖掘的高通量技术
挖掘和鉴定猪miRNA是研究猪miRNA的前提,随着实验方法和技术的不断改进,高通量技术在挖掘鉴定 miRNA中表现出巨大的优势,推动了猪miRNA的研究。目前,高通量技术主要包括新一代高通量测序技术、miRNA芯片技术和实时荧光定量PCR(qRT-PCR),集中于挖掘和鉴定猪已知的或新的miRNA,以及不同生理状态、品种或品系、发育阶段间差异表达的miRNA种类和丰度(表1),从而证实这些差异表达的miRNA在相应的生理状态、发育阶段以及不同的品种或品系之间潜在的生物学功能,但这些被挖掘和鉴定出来的大量miRNAs在猪生长发育和生理活动中的生物学功能、miRNAs互相之间的调控网络以及 miRNA的表达调控仍需进一步实验验证。
高通量的新一代测序技术,例如Illumina 公司开发的Illumina/Solexa Genome Analyzer、Roche公司开发的 Roche/454 FLX以及 ABI公司开发的SOLIDTMSystem等,在很大程度上加快了全基因组的从头测序和重测序[8]。自新一代高通量测序技术出现以来,利用其挖掘猪miRNA的研究开始涌现,推动了猪miRNA的研究进展(表1)。本文着重介绍几项在猪 miRNA挖掘鉴定中具有推重作用的研究供参考。Li等[9]采用Solexa测序技术从7、240日龄的荣昌猪背部皮下脂肪中检测出的miRNAs属于32个簇,包括 miR-143、miR-103、miR-148和 Let-7等簇,其中miR-378和miR-143分别在仔猪和成年猪脂肪组织中表达量最高,且 miR-143、miR-378、miR-103、miR-148a、miR-21、miR-10b、miR-30a-5p和 miR-199a-3p均高表达于两个发育阶段的背部皮下脂肪组织。Bai等[10]采用SOLIDTMSystem从阉割和非阉割的出生后 161日龄的公猪背部皮下脂肪组织中检测出177个差异表达的miRNAs,其中45个上调、132个下调,miR-143、miR-145、miR-199b、miR-103和miR-10a等高表达于阉割组,miR-320a、miR-21、miR-191和miR-143等高表达于非阉割组,表明 miRNA在不同生理状态下的表达不同。Hou等[11]采用Solexa测序技术从通城猪18个时间点(胚胎期:33、40、45、50、55、60、65、70、75、80、85、90、95、100、105 d;出生后:0、10、100、180 d)背最长肌混合 RNA文库中检测出 275个miRNAs,其中miR-1、miR-378和miR-206表达量最高,且miR-378在胚胎期第33 d低表达、第65~90 d逐渐增加,出生时达最高水平,随后维持在相对稳定的水平,这为全面揭示猪骨骼肌中 miRNA的表达起到了重要作用。Timoneda等[12]利用Roche/454 FLX高通量测序技术从欧洲原种猪(伊比利亚品种、欧洲野猪)、欧洲商品猪(长白猪、大白猪、皮特兰猪)、亚洲血统品种(梅山猪、越南猪(Vietnamese))肾组织中检测出42个miRNAs表达于每个品种,其中以miR-200b-3p、miR-125b和miR-23b的表达量最高,125个miRNAs在不同品种间呈差异表达,由于样品来源于血缘关系较远的品种,进一步说明不同品种的同一组织中miRNA的表达存在很大差异。
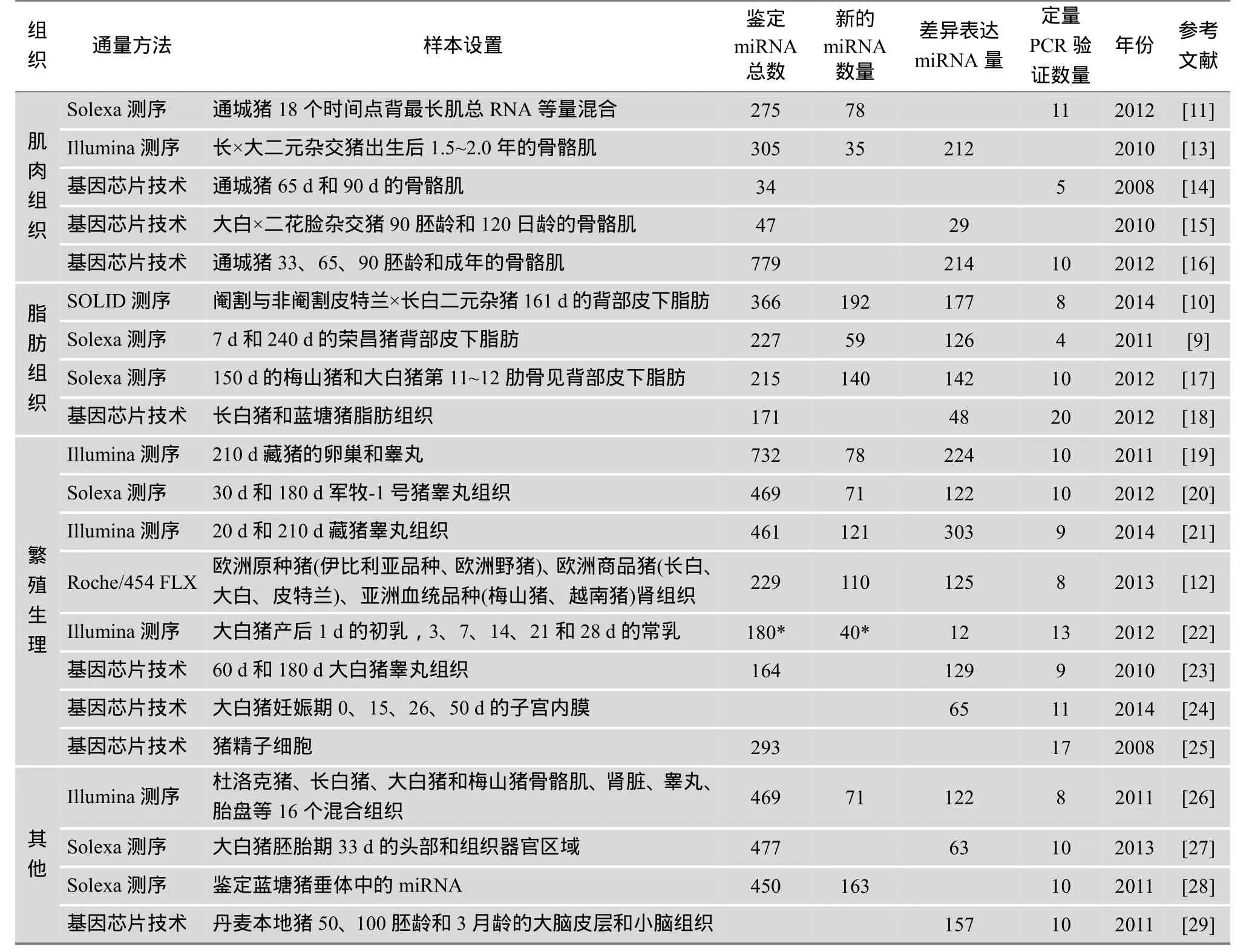
表1 高通量技术挖掘猪miRNA的部分结果
miRNA芯片技术可以在短时间内同时鉴定所有已知miRNA的表达谱,速度快、信息量大,但其仅限于检测已知的 miRNA。目前关于利用 miRNA芯片技术鉴定猪miRNA表达谱的研究也较多,主要研究对象有肌肉、脂肪和繁殖生理等。在肌肉发育方面,Chen等[14]从猪65和90胚龄的骨骼肌中检测出有强烈信号的miRNAs包括miR-206、miR-133a/b、miR-127、miR-106a、miR-27a和miR-1等,它们均是调控肌肉发育的典型 miRNAs,qRT-PCR证实miR-24和miR424在90胚龄的骨骼肌中显著下调,而 miR-133a则显著上调。相比之下,Zhou等[15]从猪90胚龄和120日龄的骨骼肌(背最长肌)中检测出的miRNAs稍多,其中miR-199b、let-7在90胚龄的骨骼肌中高表达,miR-1a、miR-133a在120日龄的骨骼肌中高表达,miR-1826、miR-26a则在两个样品中均高表达。Zhao等[16]从猪 33、65、90胚龄和成年的骨骼肌(背最长肌)中检测出20 201个基因(1947个差异表达)和779个miRNAs(214个差异表达)。在繁殖生理方面,Luo等[23]在60和180日龄猪睾丸中检测出129个差异表达miRNAs,51个在成熟睾丸组织中显著上调,78个显著下调,生物信息学分析发现它们存在15 919个靶位点,调控网络较为复杂。Su等[24]在大白猪妊娠期15和26 d的子宫内膜中检测出包括miR-181簇在内的51个差异表达的 miRNAs,生物信息学分析显示它们在胚胎植入和着床过程中发挥重要的调控作用,后续实验也证实miR-181a/c可能通过SPP1、ITGB3和ESR1基因调控猪胚胎植入和着床过程。而在脂肪和其他组织发育和生理方面,Li等[18]从大白猪和蓝塘猪脂肪组织中鉴定出48个差异表达的miRNAs,21个在蓝塘猪脂肪组织中显著上调,33个高表达于长白猪脂肪组织,从而证实不同品种的同一组织miRNA的表达有所不同。Podolska等[19]通过对丹麦本地猪 50、100胚龄和 3月龄的大脑皮层和小脑组织中鉴定出的157个差异表达的miRNAs进行生物信息学分析,显示它们在猪脑部发育过程中具有重要的调节发育和生理过程的作用。基于miRNA芯片技术通量高、速度快的特点,利用其鉴定猪各组织差异表达的miRNA,能快速、准确地筛选出差异表达的miRNA,有利于推动miRNA的功能研究。
荧光定量 PCR(qRT-PCR)技术是一种新型的核酸定量技术,与常规PCR相比,qRT-PCR具有检测灵敏、精确、特异性强、无污染、快速等特点。在新一代高通量测序技术和 miRNA芯片技术挖掘和鉴定miRNA过程中,qRT-PCR发挥着对这两种技术的准确性进行验证的作用。如表 1所示,qRT-PCR验证是每一项利用高通量技术挖掘和鉴定 miRNA的必要步骤,但目前利用qRT-PCR验证量均少于30个,验证量相比于高通量技术每次检测出的大量miRNAs则显得较少。qRT-PCR也可以单独作为一种技术研究miRNA表达,目前关于利用qRT-PCR技术构建特定 miRNA在不同猪种或不同组织中表达谱的研究报道较多。例如:qRT-PCR表达分析显示不同猪种的不同组织中 10个 miRNAs(Let-7a、miR-103、miR-17-3p、miR-25、miR-93、miR-106a、miR-191、miR-16、miR-26a和miR-17-5p)均具有较好的稳定性,可以作为参照miRNA[30];qRT-PCR证实了Let-7a、-7d、-7e和miR-22高表达于猪形态异常精子,而 Let-7a、-7e高表达于低活力精子[31]。qRT-PCR技术在猪miRNA研究中的用途广泛,且与其他技术联合使用能进一步提高实验结果的可靠性。
随着测序和基因芯片费用逐渐降低以及对实验结果可靠性的要求不断提高,将新一代高通量测序技术、基因芯片技术和qRT-PCR技术联合应用于挖掘和鉴定猪 miRNA中的研究报道也逐渐增多。Li等[18]联合使用这3种技术从长白猪和蓝塘猪骨骼肌和脂肪组织中发掘鉴定184个已知的miRNA(Solexa测序鉴定113个,基因芯片鉴定171个)、521个新的 miRNAs,并用 qRT-PCR验证了从 181个已知miRNAs中随机抽取的20个,最终分析表明在猪脂肪和肌肉发育中存在miRNAs的复杂调控网络。此外,本实验室马海明教授课题组联合使用这3 种技术挖掘和鉴定了某两个地方猪种骨骼肌中 miRNA的表达,并成功构建差异表达谱(未发表)。因此,在挖掘和鉴定猪miRNA过程中,将不同高通量技术根据实验需求进行联合使用有利于提高实验结果的准确性和可靠性。
2 miRNA与猪脂肪代谢
脂肪不仅是动物机体能量储存的一种形式,而且还参与调节包括食欲和能量消耗等多个机体功能。研究表明,脂肪还调控着多个复杂的生理过程,例如繁殖、炎症和免疫反应[32]。研究猪的脂肪代谢不仅有利于提高猪的胴体性能,还有助于推动人类肥胖和代谢性疾病的研究。研究表明,miRNA参与调节脂肪细胞的增殖和分化,因此研究已鉴定的miRNA在脂肪组织中的调控作用将显著提高人们对脂肪组织生长和发育的认识。如表 1所示,猪脂肪组织表达的miRNA在不同品种、发育时期和生理状态下主要表现为数量和种类上的差异,由此表明这些miRNAs在脂肪的生理过程中具有重要的作用,而差异表达的miRNAs则在某一特定的状态下对脂肪组织的生理过程发挥着重要的调控作用。
脂肪细胞的分化起始于多能干细胞或胚胎干细胞,经历脂肪母细胞、前体脂肪细胞和不成熟脂肪细胞,最后止于成熟脂肪细胞。在脂肪细胞的分化过程中,miRNA发挥着重要的调控作用,但它们的调控效应和靶基因则存在很大的差异。如表2所示,miR-103、miR-106b、miR-93、miR-26b、miR-143、miR-21和 miR-146b等促进脂肪细胞或前体脂肪细胞分化,而miR-191、miR-135a、miR-224-5p、miR-24、miR-27、miR-33b、miR-125a、miR-130和miR-133a等则在脂肪细胞的分化过程中发挥抑制作用。就前体脂肪细胞而言,miR-103、miR-143和 miR-146b等分别通过靶向RAI14、PTN和KLF7基因的表达实现促进前体脂肪细胞分化,而miR-135a、miR-125a和miR-133a等则通过靶向APC、ERRα和Prdm16基因抑制其分化。在miRNA调控脂肪细胞分化的过程中,各miRNA可通过靶向相同的基因而实现拮抗或协同作用,进而形成对脂肪细胞分化的调控网络;miR-21可通过抑制 AP-1的表达显著地促进脂肪细胞分化[37],而miR-24则可通过上调AP-1的表达而显著抑制3T3-L1脂肪细胞的分化和成熟[42];miR-191、miR-27、miR-33b和miR-130等均可通过抑制PPARγ的表达抑制脂肪细胞或前体脂肪细胞分化。miRNA也可以通过靶向多个基因以调控脂肪细胞的分化,miR-27在前体棕色脂肪细胞分化过程中通过负向调节棕色脂肪细胞转录本中的重要组成部分——Prdm16、Pparα、Creb和部分Pgc1β而抑制前体棕色脂肪细胞分化[43];在猪前体细胞中过表达miR-191可显著增加其转录本,且降低C/EBPβ、PPARγ以及Ap2基因的mRNA水平,抑制猪前体脂肪细胞分化,C/EBPβ的蛋白水平在48 h后降低55%,双荧光素酶报告基因检测证实miRNA-191可直接作用于C/EBPβ mRNA 3′-UTR,从而证实miR-191可能通过抑制脂肪细胞分化早期标志基因C/EBPβ的表达而抑制猪前体细胞分化[39];miR-33b通过抑制脂肪合成基因(从EBF1到C/EBPα和 PPARγ之间的级联)的表达而抑制猪皮下前体脂肪细胞的分化和发育[44]。
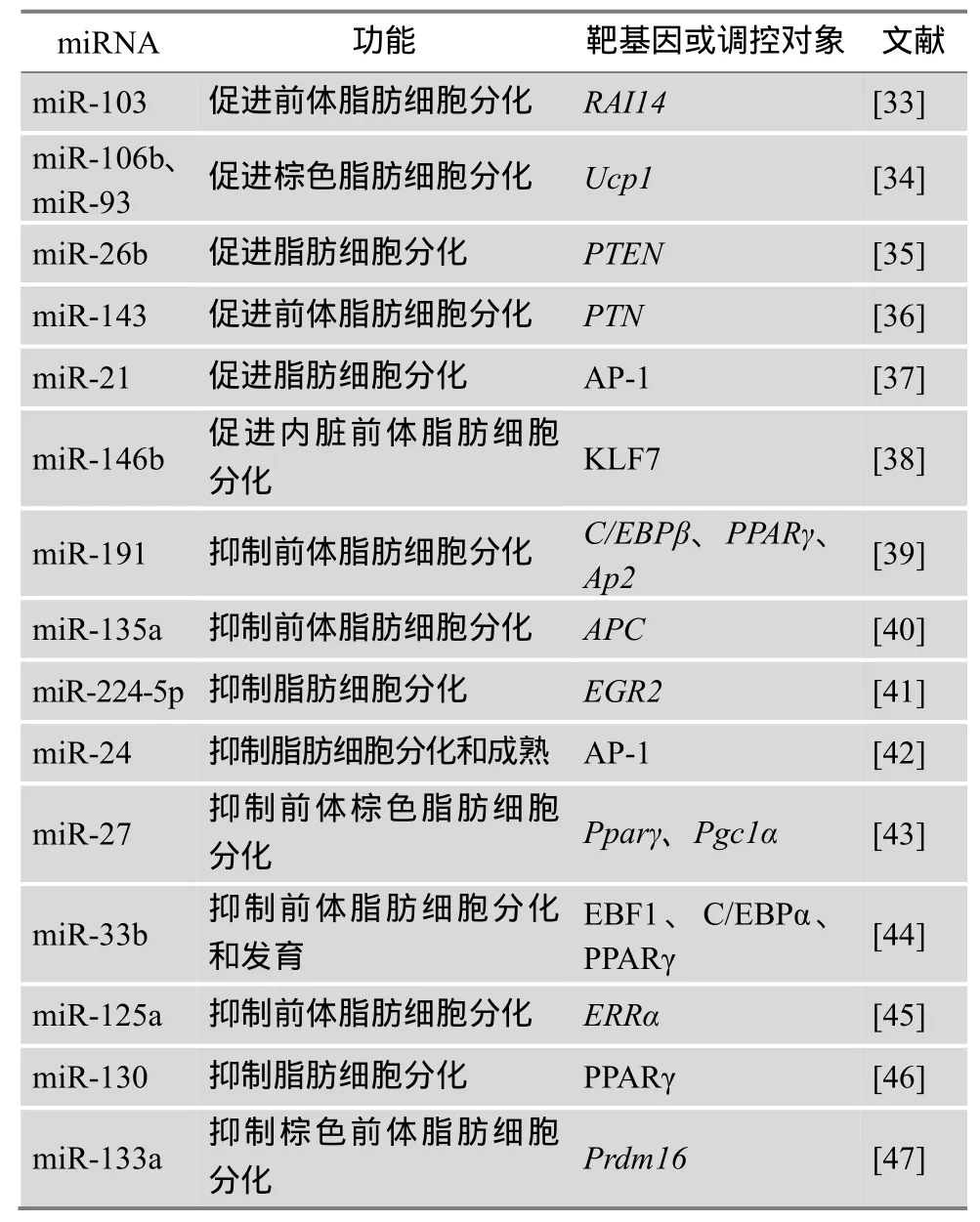
表2 对猪脂肪细胞分化具有调控作用的miRNA
此外,miRNA在脂肪细胞增殖、脂滴积聚以及脂肪形成中也发挥着重要的调控作用。在猪脂肪细胞中过表达 miR-27a能够显著加速脂肪水解以释放出更多的甘油和游离脂肪酸,而miR-143的过表达则能在脂肪细胞中积累更多的甘油三酯以促进脂肪形成[48];miR-181a靶向TNF-α mRNA 3′-UTR,并且miR-181a能够抑制Hela细胞中TNF-α蛋白水平,而在猪前体脂肪细胞中过表达 miR-181a以及抑制TNF-α基因的表达可明显增加猪前体脂肪细胞中脂滴积聚,从而证实 miR-181a可通过作用于 TNF-α基因以调节猪脂肪形成[49]。miR-145在猪去分化脂肪细胞中的表达量增加,且过表达miR-145可通过调节IRS1基因的mRNA水平以抑制脂肪细胞的两个标志基因C/EBPα和PPARγ2,进而抑制甘油三酯积聚和脂肪形成[50]。
miR-15a通过微调DLK1(Delta-like1homologue)基因促进3T3-L1前脂肪细胞的发育而抑制其增殖,而抑制miR-15a的表达则促进3T3-L1前脂肪细胞增殖而抑制其发育,证实miR-15a和DLK1基因在前脂肪细胞的生长信号通路中发挥重要作用[51]。
3 miRNA与猪肌肉发育
miR-1和 miR-133主要在骨骼肌和心肌特异性表达,并调节着细胞增殖和分化[52],二者均位于同一条染色体上,在发育过程中以组织特异性方式共同转录,但是miR-1和miR-133的表达水平和调节功能均存在着显著的差异。研究证实miR-1在成肌细胞发育的整个阶段均呈中度表达,在成熟肌细胞中表达量最高,而miR-133仅在成熟肌细胞中呈中度表达[53];miR-1通过与一种肌肉基因表达的转录抑制因子(HDAC4)结合,促进成肌细胞分化为成熟的肌肉细胞而抑制其增殖,但miR-133则通过抑制血清应答因子加速成肌细胞的增殖但抑制其分化[54];在氧化应激诱导的H9c2大鼠心肌细胞中,miR-1和miR-133对细胞凋亡的调控截然相反,miR-1通过降低 HSP60和 HSP70蛋白的表达促进细胞凋亡而miR-133则通过抑制caspase-9基因的转录表达和蛋白表达水平抑制细胞凋亡[52]。以 miR-1为中心的mTOR-miR-1-HDAC4-follostatin通路对骨骼肌再生和成肌细胞分化过程中的细胞融合具有重要的调节作用,即mTOR通过调控MyoD蛋白的稳定性调节miR-1的转录表达,miR-1抑制HDAC4的表达而导致卵泡抑素生成并与心肌细胞融合[55]。此外,miR-1在血管平滑肌细胞的分化过程中也具有重要作用,其通过KLF4的表达而促进血管平滑肌细胞的分化[56]。在猪体上的研究也表明,miR-1对骨骼肌的发育具有重要的调控作用,miR-1负向调节 CNN3基因mRNA的表达,CNN3基因的SNPs与仔猪初生重和21日龄断奶重显著相关,从而证实miR-1可通过靶向 CNN3基因而调节猪骨骼肌的发育[57];miR-1 SNPs与猪Ⅰ型和Ⅱa型肌纤维数量和分布显著相关,改变miR-1的表达水平可显著改变猪肌纤维成分,从而证实miR-1可能靶向猪肌纤维成分相关的一个候选基因而调控猪肌纤维的发育[58]。miR-133在肌肉发育过程中主要发挥抑制肌肉细胞分化而促进其增殖的作用,miR-133a和 miR-133b可在转录水平抑制参与ERK1/2信号转导的FGFR1和PP2AC基因表达和ERK1/2的磷酸化,且FGFR1和PP2AC可以抑制C2C12成肌细胞分化而促进其增殖,表明miR-133a和miR-133b分别通过调节ERK1/2信号通路中的FGFR1和PP2AC基因促进成肌细胞分化而抑制其增殖[59]。
miR-29和miR-206在肌肉发育过程中也具有重要的调控作用。在骨骼肌的发育过程中,miR-29具有重要的调控作用,miR-29可通过下调 Akt3(丝氨酸/苏氨酸蛋白激酶家族成员之一)的转录水平以抑制骨骼肌细胞增殖而促进其分化[60];miR-29还可以通过靶向YY1而促进骨骼肌细胞分化[61]。miR-29调节骨骼肌分化的作用也受到其他一些细胞因子或信号通路的调节,如TGF-β、Rybp和NF-KappaB-YY1等。在成肌细胞转分化为肌成纤维细胞过程中,miR-29的促分化作用受到 TGF-β的负向调节[62],TGF-β-Smad3信号通过抑制MyoD的结合作用和增强YY1的作用而抑制miR-29的促成肌细胞分化作用[63]。而miR-29又可以通过抑制TGF-β-Smad3信号介导的HDAC4表达上调效应而削弱TGF-β对成肌分化的抑制作用[64],从而形成一个调节成肌分化的反馈网络。Rybp(Ring1 and YY1-binding protein)作为骨骼肌分化和再生的反向调节因子,可以与YY1共同作用于miR-29,使其沉默,进而负向调节骨骼肌生成[65]。在成肌细胞中,NF-KappaB通过YY1抑制 miR-29的表达,但是在肌肉生成过程中下调NF-KappaB和YY1的表达则可以消除这种抑制作用,进而使得 miR-29通过靶向其本身的抑制因子 YY1而促进骨骼肌细胞分化[61]。miR-206高表达于新形成的骨骼肌纤维,具有促进骨骼肌纤维再生和成熟的作用[66];此外,还具有促进骨骼肌的分化,在骨骼肌发育过程中具有重要的调控作用。miR-206通过靶向多个基因调控骨骼肌发育,如Id1-3、MyoR、Pax7、BDNF、Notch-3、Hmyb3、RUNX1、ZNF238和Cx43等,除RUNX1和ZNF238以外,miR-206均是通过抑制这些靶基因的表达而促进骨骼肌细胞分化、发育和再生而抑制其增殖。同样,miR-206调控骨骼肌发育的作用也受到其他一些因子的调节,在成肌细胞转化为骨骼肌细胞过程中,MyoD通过直接激活miR-206的表达而抑制Fstl1和Utm的表达,从而证实miR-206的调控作用受到MyoD的调节[67]。Notch3本身即为抑制骨骼肌分化的调控基因,研究表明Mef2c可促进miR-206的表达而下调或完全抑制Notch3的表达以促进骨骼细胞分化[68]。此外,miR-208、miR-486、miR-499、miR-23a、miR-214和miR-155等在肌肉组织的发育过程中也具有重要的调控作用,但相关研究较少。miR-214表达于成肌细胞和肌管,在成肌细胞分化时被显著上调,抑制miR-214可通过显著下调肌细胞生成素和肌球蛋白的表达而抑制成肌细胞分化,此外还可以抑制成肌细胞增殖[69]。双荧光素酶报告和 RT-PCR证实miR-155可调节OLFML3基因的mRNA表达水平,而OLFML3基因能够影响胚胎期骨骼肌的发育,从而证实miR-155可通过靶向OLFML3基因而调节猪胚胎期骨骼肌的发育[70]。
4 miRNA与猪卵母细胞成熟
卵母细胞作为卵原细胞经历增殖分化、DNA复制、两次减数分裂后的产物,在卵细胞发育成熟过程中扮演着重要的中间体角色。因此,卵母细胞的生长、成熟和发育能力直接影响着卵细胞的成熟,进而影响雌性哺乳动物的产仔性状。目前,已有很多研究表明miRNA能够参与调节卵母细胞的成熟。Tesfaye等[71]研究证实在成熟和未成熟的卵母细胞分别有28和31种miRNA表达,其中有7个(miR-496、miR-292-3p、miR-99a、miR-410、miR-145 和 miR-515-5p)和2个(miR-512-5p、miR-214)差异表达于成熟和未成熟的卵母细胞。Wright等[72,73]证实,与GV(Germinal vesicle)期的卵母细胞相比,miR-21在MII(Second meiotic metaphase)期的表达被显著提高,而在GV期到MII期,Western blot证实PDCD4蛋白则显著降低,从而证实 miR-21的表达上调与PDCD4基因的转录后水平之间存在非常密切的关系。此外,当抑制miR-21的表达则会显著降低卵母细胞的成熟率,为了解miRNA与卵母细胞发育成熟的关系提供了直接证据。研究表明,颗粒细胞对卵母细胞具有营养作用与信号转导作用,这对卵母细胞的生长、成熟和发育能力具有重要的作用,因此,miRNA对颗粒细胞的调节作用可间接调节卵母细胞生长、成熟和发育能力。Lin等[74]在猪颗粒细胞中转染miR-26b模拟物和寡核苷酸(对照组)72 h后,转染miR-26b模拟物后颗粒细胞死亡,而转染寡核苷酸的颗粒细胞正常生长,证明miR-26b可促进颗粒细胞凋亡,而Real-time PCR和双荧光素酶报告显示miR-26b发挥功能是通过靶向ATM基因mRNA的5555位点以抑制ATM基因的表达,而ATM基因在DNA修复中发挥至关重要的作用,证实miR-26b可通过靶向 ATM基因而抑制卵母细胞发育成熟。Yan等[75]通过Real-time PCR、Western blot和双荧光素酶报告证实 miR-145通过靶向 ACVRIB基因mRNA 3′-UTR而抑制ACVRIB基因mRNA和蛋白表达水平,并激活诱导Smad2的磷酸化,从而抑制颗粒细胞增殖。Zhang等[76]证实在小鼠颗粒细胞中过表达miR-181a可通过靶向ACVR2a基因的3′-UTR而抑制其表达,从而下调细胞周期蛋白D2和增殖细胞细胞核抗原表达,最终抑制细胞增殖,而过表达ACVR2a基因则可削弱miR-181a对颗粒细胞增殖的这种抑制作用。Yin等[77]证实 miR-383主要表达于卵泡中的卵母细胞和颗粒细胞,过表达miR-383可通过靶向RBMS1基因(Single stranded interacting protein 1)的mRNA而促进颗粒细胞释放雌二醇,而miR-383在颗粒细胞中的表达和功能受到 SF-1(steroidogenic factor-1)的调节。Xu等[78]也证实miR-378时序性表达于猪颗粒细胞,且通过靶向芳香化酶 mRNA的 3′-UTR的两个位点以调节颗粒细胞释放雌二醇。尽管一些关于miRNA在卵母细胞生长、发育中的功能研究是以小鼠为模型,但小鼠作为研究哺乳动物生理和生殖的模式生物,对研究miRNA在猪卵细胞中的功能也有很好的参考作用。
5 miRNA与B、T免疫细胞发育
miR-155、miR-17-92、miR-150、miR-28、miR-34a和miR-21等在B细胞的发育过程中均具有重要的调控作用。miR-155是目前发现的靶基因最多的miRNA之一,可以作用于细胞因子、蛋白受体、转录因子和酶类等,在免疫系统中发挥重要的调控作用。miR-155通过抑制 GAB1(GRB2-associated binding protein 1)和FOXP1(Forkhead box P1)影响慢性淋巴细胞性白血病的B细胞受体信号[79],也可以通过抑制SHIP1基因的表达促进依赖TNFα的B细胞淋巴瘤的生长[80]。此外,c-MAF、PU1、AID和SHIP等也是miR-155的靶基因,从而在B细胞的成熟和产生高亲和力抗体中发挥重要的调控作用。miR-17-92包括miR-17、miR-18a、miR-19a、miR-19b、miR-20a和miR-92a,具有维持免疫细胞正常发育的作用[81]。Mu等[82]利用基因敲除证实miR-17-92簇、尤其是簇内的miR-19a和miR-19b在原癌基因诱导的B细胞淋巴瘤中具有抑制凋亡的作用;同时结合基因表达谱、体外筛选和靶标预测的结果证实 miR-19a和miR-19b在维持细胞活性中具有重要作用。miR-150作为B细胞发育的负向调控因子,主要表达与成熟的淋巴细胞,促使 B细胞滞留与 pro-B阶段[83],miR-150通过剂量依赖的方式调控 c-Myb(转录因子,在多个步骤控制淋巴细胞的发育)在体内的表达,进而影响B细胞的发育和应答能力[84]。
在T细胞的发育和活性维持过程中同样也受到miRNA的调控,如 miR-155、miR-223、miR-10a、miR-34a、miR-181a和miR-182等。miR-155直接作用于 IFN-gRα 3′非翻译区而抑制其表达,进而促进CD+ T细胞中Th1的分化以维持辅助性T细胞分化的平衡[85];miR-155也可以通过促进炎症T细胞的发育而促进动物机体的自身免疫性炎症[86];此外,miR-155在维持 T细胞内环境稳定和分化的平衡还可以通过靶向 c-MAF和 SOCS1等实现。miR-34a抑制DGKζ(具有抑制甘油二酯与T细胞受体结合的作用)的表达而促进 T细胞活性[87]。Li等[88]研究表明,免疫细胞中miR-181a的表达与T细胞的敏感性呈较强的正相关,从而证实在 T细胞发育过程中miR-181a是一种内在的抗原敏感性“变阻器”;但miR-181a通过抑制CD69和T细胞受体的表达使胸腺内成熟T细胞输出量和阳性选择后存活的T细胞数量减少,达到增强T细胞活性而抑制T细胞数量的作用[89]。此外,miR-350、miR-92、miR-669c、miR-297、miR-15b、miR-150、miR-24和 miR-27a也参与T细胞的发育和分化过程,但相关研究较少,有待进一步深入研究。
6 结 语
在猪miRNA后续的相关研究中,应当充分利用新一代的高通量测序和芯片技术挖掘更多的猪miRNA,此外猪 miRNA的表达分析和功能验证也有待进一步研究。对miRNA的表达谱分析应着眼于不同生理状态以及不同发育阶段特异性表达的miRNA,探讨猪 miRNA与生长性能、猪肉品质、繁殖性能和健康状况等的相关性,从而筛选出miRNA的分子标记。而对于猪miRNA的功能验证,则应深入研究特异性表达的miRNA的作用机理,以及环境因素、营养水平等对这些miRNA的调控机制。此外,关于怎样利用miRNA来改善猪生长性能、猪肉品质、繁殖性能和健康状况也非常值得研究。
目前,通过细胞培养验证miRNA的功能,利用转染、人工芯片抑制、基因敲除以及双荧光素酶报告检测等技术研究 miRNA在生理过程中的靶基因及其调控作用,此此基础上,在机体水平对miRNA进行研究,加强探讨猪miRNA抑制和过表达与猪生长性能、猪肉品质、繁殖性能和健康状况的影响也是非常有必要的。猪作为重要的畜牧经济动物和模式动物,探明miRNA调节其生长性能、肉品质、繁殖性能和健康状况的潜在机理,可为提升猪生产性能、改善猪肉品质以及分子育种提供新的实试验证据。此外,还可以为治疗人类代谢疾病、生殖疾病等提供新的思路。
[1] Bendixen E, Danielsen M, Larsen K, Bendixen C. Advances in porcine genomics and proteomics-a toolbox for developing the pig as a model organism for molecular biomedical research. Brief Funct Genomics, 2010, 9(3): 208-219.
[2] Lee RC, Feinbaum RL, Ambros V. The C. elegansheterochronicgene lin-4 encodes small RNAs with antisense complementarityto lin-14. Cell, 1993, 75(5): 843-854.
[3] 王天宇, 董园园, 李海燕, 李校堃. MicroRNAs的分子进化与调控机制. 遗传, 2010, 32(9): 874-880.
[4] 孟雅楠, 孟丽军, 宋亚娟, 刘美玲, 张秀军. 小 RNA分子与精子发生调控. 遗传, 2011, 33(1): 9-16.
[5] Wang Y, Stricker HM, Gou DM, LiuL. MicroRNA: past and present. Front Biosci, 2007, 12: 2316-2329.
[6] Bushati N, Cohen SM. MicroRNA functions. Annu Rev Cell DevBiol, 2007, 23: 175-205.
[7] 景花, 宋沁馨,周国华. MicroRNA定量检测方法的研究进展. 遗传, 2010, 32(1): 31-40.
[8] Huang WC, Marth G. EagleView: a genome assembly viewer for next-generation sequencing technologies. Genome Res, 2008, 18(9): 1538-1543.
[9] Li GX, Li YJ, Li XJ, Ning XM, Li MH, Yang GS. MicroRNA identity and abundance in developing swine adipose tissue as determined by solexa sequencing. J Cell Biochem, 2011, 112(5): 1318-1328.
[10] Bai Y, Huang JM, Liu G, Zhang JB, Wang JY, Liu CK, Fang MY. A comprehensive microRNA expression profile of the backfat tissue from castrated and intact full-sib pair male pigs. BMC Genomics, 2014, 15(1): 47.
[11] Hou XH, Tang ZL, Liu HL, Wang N, Ju HM, Li K. Discovery of microRNAs associated with myogenesis by deep sequencing of serial developmental skeletal muscles in pigs. PLoS ONE, 2012, 7(12): e52123.
[12] Timoneda O, Balcells I, Núñez JI, VeraG, Castelló A, Tomàs A, Sánchez A. MiRNA expression profile analysis in kidney of different porcine breeds. PLoS ONE, 2013, 8(1): e55402.
[13] Nielsen M, Hansen JH, Hedegaard J, Nielsen RO, Panitz F, Bendixen C, Thomsen B. MicroRNA identity and abundance in porcine skeletal muscles determined by deep sequencing. Anim Genet, 2010, 41(2): 159-168.
[14] Chen JH, Wei WJ, Xiao X, Zhu MJ, Fan B,, Zhao SH. Expression analysis of miRNAs in porcine fetal skeletal muscle on days 65 and 90 of gestation. Asian Austral J Anim Sci, 2008, 21(7): 954-960.
[15] Zhou B, Liu HL, Shi FX, Wang JY. MicroRNA expression profiles of porcine skeletal muscle. Animal Genetics, 2010, 41(5): 499-508.
[16] Zhao SH, Cao JH, Feng Y, Liu H, Li XY. Identification of novel regulators in porcine skeletal muscle growth by integrated analysis of miRNA and mRNA expression. In: Plant and Animal Genome XX Conference.2012, 14-18.
[17] Chen C, Deng B, Qiao M, Zheng R, Chai J, Ding Y, Peng J, Jiang SW. Solexa sequencing identification of conserved and novel microRNAs in backfat of large white and chi-nesemeishan pigs. PLoS ONE, 2012, 7(2): e31426.
[18] Li HY, Xi QY, Xiong YY, Liu XL, Cheng X, Shu G, Wang SB, Wang LN, Gao P, Zhu XT, Jiang QY, Yuan L, ZhangYL. Identification and comparison of microRNAs from skeletal muscle and adipose tissues from two porcine breeds. Anim Genet, 2012, 43(6): 704-713.
[19] Li MZ, Liu YK, Wang T, Guan JQ, LuoZG, ChenHS, WangX, ChenL, MaJD, MuZP, Jiang AA, Zhu L , LangQL, ZhouXC, Wang JY, Zeng WX, Li N, Li K, GaoXL, Li XW. Repertoire of porcine microRNAs in adult ovary and testis by deep sequencing. Int J Biol Sci, 2011, 7(7): 1045-1055.
[20] Lian CJ, Sun BX, Niu SL, Yang RJ, Liu BY, Lu CY, Meng JL, Qiu ZY, Zhang LY, Zhao ZH. A comparative profile of the microRNA transcriptome in immature and mature porcine testes using solexa deep sequencing. FEBS J, 2012, 279(6): 964-975.
[21] Liu YH, Ma JD, Chen L, Luo PB, Zhou J, Li MZ, Li XW. Comparison of microRNA transcriptomes between immature and mature porcine testes. J Anim Vet Adv, 2014, 13(3): 132-138.
[22] Gu YR, Li MZ, Wang T, Liang Y, Zhong ZJ, Wang XY, Zhou Q, Chen L, Lang QL, He ZP, Chen XH, Gong JJ, Gao XL, Li XW, Lv XB. Lactation-related microRNA expression profiles of porcine breast milk exosomes. PLoS ONE, 2012, 7(8): e43691.
[23] Luo LF, Ye LZ, Liu G, Shao GC, Zheng R, Ren ZQ, Zuo B, Xu DQ, Lei MG, Jiang SW, Deng CY, Xiong YZ, LiFG. Microarray-based approach identifies differentially expressed microRNAs in porcine sexually immature andmature testes. PLoS ONE, 2010, 5(8): e11744.
[24] Su LJ, Liu RZ, Cheng W, Zhu MJ, Li XP, Zhao SH, Yu M. Expression patterns of microRNAs in porcine endometrium and their potential roles in embryo implantation and placentation. PLoS ONE, 2014, 9(2): e87867.
[25] Curry E, Ellis SE, Pratt SL. Detection of porcine sperm microRNAs using a heterologous microRNA microarray and reverse transcriptase polymerase chain reaction. Mol Reprod Dev, 2009, 76(3): 218-219.
[26] Xie SS, Li XY, Liu T, Cao JH, Zhong Q, Zhao SH. Discovery of porcine microRNAs in multiple tissues by a solexa deep sequencing approach. PLoS ONE, 2011, 6(1): e16235.
[27] Zhou Y, Tang X, Song Q, Ji Y, Wang H, Wang H, Jiao H, Ouyang H, Pang D. Identification and characterization of pig embryo microRNAs by solexa sequencing. Reprod Domest Anim, 2013, 48(1): 112-120.
[28] Li HY, Xi QY, Xiong YY, Cheng X, Qi Q, Yang L, Shu G, Wang SB, Wang LN,Gao P, Zhu XT, Jiang QY, Zhang YL, Yuan L. A comprehensive expression profile of micro-RNAs in porcine pituitary. PLoS ONE, 2011, 6(9): e24883.
[29] Podolska A, Kaczkowski B, Busk PK, Søkilde R, Litman T, Fredholm M, Cirera S. MicroRNA expression profiling of the porcine developing brain. PLoS ONE, 2011, 6(1): e14494.
[30] Timoneda O, Balcells I, Córdoba S, CastellóA, Sánchez A. Determination of reference microRNAs for relative quantification in porcine tissues. PLoS ONE, 2012 7(9): e44413.
[31] Curry E, Safranski TJ,Pratt SL. Differential expression of porcine sperm microRNAs and their association with sperm morphology and motility. Theriogenology, 2011, 76(8): 1532-1539.
[32] Sauerwein H, Bendixen E, Restelli L, Ceciliani F. The adipose tissue in farm animals: a proteomic approach. Curr Protein Pept Sci, 2014, 15(2): 146-155.
[33] Li GX, Wu ZS, Li XJ, Ning XM, Li YJ, Yang GS. Biological role of microRNA-103 based on expression profile and target genes analysis in pigs. Mol Biol Rep, 2011, 38(7): 4777-4786.
[34] Wu Y, Zuo JR, Zhang YC, Xie Y, Hu F, Cheng LH, Liu BL, Liu F. Identification of miR-106b-93 as a negative regulator of brown adipocyte differentiation. Biochem Biophys Res Commun, 2013,438(4): 575-580.
[35] Song GX, Xu GF, Ji CB, Shi CM, Shen YH, Chen L, Zhu LJ, Yang L, Zhao YP, Guo XR. The role of microRNA-26b in human adipocyte differentiation and proliferation. Gene, 2014, 533(2): 481-487.
[36] Yi C, Xie WD, Li F, Lv Q, He J, Wu JB, Gu DY, Xu NH, Zhang YO. MiR-143 enhances adipogenic differentiation of 3T3-L1 cells through targeting the coding region of mouse pleiotrophin. FEBS Lett, 2011, 585(20): 3303-3309.
[37] Kang M, Yan LM, Zhang WY, Li YM, Tang AZ, Ou HS. Role of microRNA-21 in regulating 3T3-L1 adipocyte differentiation and adiponectin expression. Mol Biol Rep, 2013, 40(8): 5027-5034.
[38] Chen L, Dai YM, Ji CB, Yang L, Shi CM, Xu GF, Pang LX, Huang FY, Zhang CM, Guo XR. MiR-146b is a regulator of human visceral preadipocyte proliferation and differentiation and its expression is altered in human obesity. Mol Cell Endocrinol, 2014, 393(1-2): 65-74.
[39] 刘帅, 宁小敏, 李美航, 仇杨, 李艳杰, 董培越, 杨公社. miR-191通过调控C/EBPβ转录影响猪前体脂肪细胞分化. 生物化学与生物物理进展, 2013, 40(2): 165-176.
[40] 陈晨. miR-135a和miR-183对3T3-L1前脂肪细胞分化及脂肪形成的调控作用研究[学位论文]. 武汉: 华中农业大学, 2013.
[41] Peng YD, Xiang H, Chen C, Zheng R, Chai J, Peng J, Jiang SW. MiR-224 impairs adipocyte early differentiation and regulates fatty acid metabolism. Int J Biochem Cell Biol, 2013, 45(8): 1585-1593.
[42] Kang M, Yan LM, Li YM, Zhang WY, Wang H, Tang AZ, Ou HS. Inhibitory effect of microRNA-24 on fatty ac-id-binding protein expression on 3T3-L1 adipocyte differentiation. Genet Mol Res, 2013, 12(4): 5267-5277.
[43] Sun L, Trajkovski M. MiR-27 orchestrates the transcriptional regulation of brown adipogenesis. Metabolism, 2014, 63(2): 272-282.
[44] Taniguchi M, Nakajima I, Chikuni K, Kojima M, Awata T, Mikawa S. MicroRNA-33b downregulates the differentiation and development of porcine preadipocytes. Mol Biol Rep, 2014, 41(2): 1081-1090.
[45] Ji HL, Song CC, Li YF, He JJ, Li YL, Zheng XL, Yang GS. miR-125a inhibits porcine preadipocytes differentiation by targeting ERRα. Mol Cell Biochem, 2014, 395(1-2): 155-165.
[46] Lee EK, Lee MJ, Abdelmohsen K, Kim W, Kim MM, Srikantan S, Martindale JL, Hutchison ER, Kim HH, Marasa BS, Selimyan R, Egan JM, Smith SR, Fried SK, Gorospe M. miR-130 suppresses adipogenesis by inhibiting peroxisome proliferator-activated receptor γ expression. Mol Cell Biol, 2011, 31(4): 626-638.
[47] Liu WY, Bi PP, Shan TZ, Yang X, Yin H, Wang YX, Liu N, Rudnicki MA, Kuang S. miR-133a regulates adipocyte browning in vivo. PLoS Genet, 2013, 9(7): e1003626.
[48] Wang T, Li M, Guan J, Li P, Wang H, Guo Y, Shuai S, Li X. MicroRNAs miR-27a and miR-143 regulate porcine adipocyte lipid metabolism. Int J Mol Sci, 2011, 12(11): 7950-7959.
[49] Li HY, Chen X, Guan LZ, Qi Q, Shu G, Jiang QY, Li Y, Xi QY, Zhang YL. MiRNA-181a regulates adipogenesis by targeting tumor necrosis factor-α (TNF-α) in the porcine model. PLoS ONE, 2013, 8(10): e71568.
[50] Guo Y, Chen Y, Zhang Y, Zhang Y, Chen L, Mo D. Up-regulated miR-145 expression inhibits porcine preadipocytes differentiation by targeting IRS1. Int J Biol Sci, 2012, 8(10): 1408-1417.
[51] Andersen DC, Jensen CH, Schneider M, Nossent AY, Eskildsen T, HansenJL, TeisnerB, SheikhSP. Micro-RNA-15a fine-tunes the level of delta-like 1 homolog (DLK1) in proliferating 3T3-L1 preadipocytes. Exp Cell Res, 2010, 316(10): 1681-1691.
[52] Xu CQ, Lu YJ, Pan ZW, Chu WF, Luo XB, Lin HX, Xiao JN, Shan HL, Wang ZG, Yang BF. The muscle-specific microRNAs miR-1 and miR-133 produce opposing effects on apoptosis by targeting HSP60, HSP70 and caspase-9 in cardiomyocytes. J Cell Sci, 2007, 120(17): 3045-3052.
[53] McDaneld TG, Smith TPL, Doumit ME, Miles JR, Coutinho LL, Sonstegard TS, Matukumalli LK, Nonneman DJ,Wiedmann RT. MicroRNA transcriptome profiles during swine skeletal muscle development. BMC Genomics, 2009, 10(1): 77.
[54] Chen JF, Mandel EM, Thomson JM, Wu QL, Callis TE, Hammond SM, Conlon FL, Wang DZ. The role of microRNA-1 and microRNA-133 in skeletal muscle proliferation and differentiation. Nat Genet, 2006, 38(2): 228-233.
[55] Sun YT, Ge YJ, Drnevich J, Zhao Y, Band M, Chen J. Mammalian target of rapamycin regulates miRNA-1 and follistatin in skeletal myogenesis. J Cell Biol, 2010, 189(7): 1157-1169.
[56] Xie CQ, Huang HR, Sun X, Guo YH, Hamblin M, Ritchie RP, Garcia-Barrio MT, Zhang JF, Chen YE. MicroRNA-1 regulates smooth muscle cell differentiation by repressing Kruppel-like factor 4. Stem Cells Dev, 2011, 20(2): 205-210.
[57] Tang ZL, Liang RY, Zhao SP, Wang RQ, Huang RH, Li K. CNN3 Is regulated by microRNA-1 during muscle development in pigs. Int J Biol Sci, 2014, 10(4): 377-385.
[58] Hong JS, Noh SH, Lee JS, Kim JM, Hong KC, Lee YS. Effects of polymorphisms in the porcinemicroRNA-miR-1locus on muscle fiber type composition and miR-1 expression. Gene, 2012, 506(1): 211-216.
[59] Feng Y, Niu LL, Wei W, Zhang WY, Li XY, Cao JH, Zhao SH. A feedback circuit between miR-133 and the ERK1/2 pathway involving an exquisite mechanism for regulating myoblast proliferation and differentiation. Cell DeathDis, 2013, 4(11): e934.
[60] Wei W, He HB, Zhang WY, Zhang HX, Bai JB, Liu HZ, Cao JH, Chang KC, Li XY, Zhao SH. miR-29 targets Akt3 to reduce proliferation and facilitate differentiation of myoblasts in skeletal muscle development. Cell Death Dis, 2013, 4:e668.
[61] Wang HT, Garzon R, Sun H, Ladner KJ, Singh R, Dahlman J, Cheng A, Hall BM, Qualman SJ, Chandler DS, Croce CM, Guttridge DC. NF-κB-YY1-miR-29 regulatory circuitry in skeletal myogenesis and rhabdomyosarcoma. Cancer Cell, 2008, 14(5): 369-381.
[62] Wang LJ, Zhou L, Jiang PY, Lu LN, Chen XN, Lan HY, Guttridge DC, Sun H, Wang HT. Loss of miR-29 in myoblasts contributes to dystrophic muscle pathogenesis. Mol Ther, 2012, 20(6): 1222-1233.
[63] Zhou L, Wang LJ, Lu LN, Jiang PY, Sun H, Wang HT. Inhibition of miR-29 by TGF-beta-Smad3 signaling through dual mechanisms promotes transdifferentiation of mouse myoblasts into myofibroblasts. PLoS ONE, 2012, 7(3): e33766.
[64] Winbanks CE, Wang B, Beyer C, Koh P, White L, Kantharidis P, Gregorevic P. TGF-β regulates miR-206 and miR-29 to control myogenic differentiation through regulation of HDAC4. J Biol Chem, 2011, 286(16): 13805-13814.
[65] Zhou L, Wang LJ, Lu LN, Jiang PY, Sun H, Wang HT. A novel target of microRNA-29, Ring1 and YY1-binding protein (Rybp), negatively regulates skeletal myogenesis. J Biol Chem, 2012, 287(30): 25255-25265.
[66] Yuasa K, Hagiwara Y, Ando M, Nakamura A, Takeda S, Hijikata T. MicroRNA-206 is highly expressed in newlyformed muscle fibers: implications regarding potential for muscle regeneration and maturation in muscular dystrophy. Cell Struct Funct, 2008, 33(2): 163-169.
[67] Rosenberg MI, Georges SA, Asawachaicharn A, Analau E, Tapscott SJ. MyoD inhibits Fstl1 and Utrn expression by inducing transcription of miR-206. J Cell Biol, 2006, 175(1): 77-85.
[68] Gagan J, Dey BK, Layer R, Yan Z, Dutta A. Notch3 and Mef2c proteins are mutually antagonistic via Mkp1 protein and miR-1/206 microRNAs in differentiating myoblasts. J Biol Chem, 2012, 287(48): 40360-40370.
[69] Feng Y, Cao JH, Li XY, Zhao SH. Inhibition of miR-214 expression represses proliferation and differentiation of C2C12 myoblasts. Cell Biochem Funct, 2011, 29(5): 378-383.
[70] Zhao SP, Zhang J, Hou XH, Zan LS, Wang N, Tang ZL, Li K. OLFML3 expression is decreased during prenatal muscle development and regulated by microRNA-155 in pigs. Int J Biol Sci, 2012, 8(4): 459-469.
[71] TesfayeD, WorkuD, RingsF, PhatsaraC, TholenE, SchellanderK, HoelkerM. Identification and expression profiling of microRNAsduring bovine oocyte maturation using heterologous approach. Mol Reprod Dev, 2009, 76(7): 665- 677.
[72] Wright EC. MicroRNA expression and function during porcine oocyte maturation and early embryonic development. USA: Iowa State University, 2012.
[73] Wright EC, Yang CX, Scanlon R, Selman B, Ross J. MicroRNA-21 and PDCD4 expression during in vitro maturation of porcine oocytes. Biol Reprod, 2010, 83: 322.
[74] Lin F, Li R,Pan ZX, Zhou B, Yu DB, Wang XG, Ma XS, Han J, Shen M, Liu HL. MiR-26b promotes granulosa cell apoptosis by targeting ATM during follicular atresia in porcine ovary. PLoS ONE, 2012, 7(6): e38640.
[75] Yan GJ, Zhang LX, Fang T, Zhang Q, Wu SG, Jiang Y, Sun H, Hu YL. MicroRNA-145 suppresses mouse granulosa cell proliferation by targeting activin receptor IB. FEBS Lett, 2012, 586(19): 3263-3270.
[76] Zhang Q, Sun HX, Jiang Y, Ding LJ, Wu SG, Fang T, Yan GJ, Hu YL. MicroRNA-181a suppresses mouse granulosa cell proliferation by targeting activin receptor IIA. PLoS ONE, 2013, 8(3): e59667.
[77] Yin MM, Lü MR, Yao GD, Tian H, Lian J, Liu L, Liang M, Wang Y, Sun F. Transactivation of microRNA-383 by steroidogenic factor-1 promotes estradiol release from mouse ovarian granulosa cells by targeting RBMS1. Mol Endocrinol, 2012, 26(7): 1129-1143.
[78] Xu SY, Linher-Melville K, Yang BB, Wu D, Li J. Micro-RNA378 (miR-378) regulates ovarian estradiol production by targeting aromatase. Endocrinology, 2011, 152(10): 3941-3951.
[79] Mraz M, Chen LG, Rassenti LZ, Ghia EM, Li HY, Jepsen K, Smith EN, Messer K, Frazer KA, Kipps TJ. miR-150 influences B-cell receptor signaling in chronic lymphocytic leukemia by regulating expression of GAB1 and FOXP1. Blood, 2014, 124(1): 84-95.
[80] Pedersen IM, Otero D, Kao E, Miletic AV, Hother C, Ralfkiaer E, Rickert RC, Gronbaek K, David M. Onco-miR-155 targets SHIP1 to promote TNFα-dependent growth of B cell lymphomas. EMBO Mol Med, 2009, 1(5): 288-295.
[81] Sandhu SK, Fassan M, Volinia S, Lovat F, Balatti V, Pekarsky Y, Croce CM. B-cell malignancies in microRNA Eμ-miR-17~92 transgenic mice. Proc Natl Acad Sci USA, 2013, 110(45): 18208-18213.
[82] Mu P, Han YC, Betel D, Yao E, Squatrito M, Ogrodowski P, de Stanchina E, D'Andrea A, Sander C, Ventura A. Genetic dissection of the miR-17~92 cluster of microRNAs in Myc-induced B-cell lymphomas. Genes Dev, 2009, 23(24): 2806-2811.
[83] Zhou BY, Wang S, Mayr C, Bartel DP, Lodish HF. miR-150, a microRNA expressed in mature B and T cells, blocks early B cell development when expressed prematurely. Proc Natl Acad Sci USA, 2007, 104(17): 7080-7085.
[84] Xiao CC, Calado DP, Galler G, Thai TH, Patterson HC, Wang J, Rajewsky N, Bender TP, Rajewsky K. MiR-150 controls B cell differentiation by targeting the transcription factor c-Myb. Cell, 2007, 131(1): 146-159.
[85] Banerjee A, Schambach F, DeJong CS, Hammond SM, Reiner SL. Micro-RNA-155 inhibits IFN-gamma signaling in CD4+ T cells. Eur J Immunol, 2010, 40(1): 225-231.
[86] O'Connell RM, Kahn D, Gibson WSJ, Round JL, Scholz RL, Chaudhuri AA, Kahn ME, Rao DS, Baltimore D. MicroRNA-155 promotes autoimmune inflammation by enhancing inflammatory T celldevelopment. Immunity, 2010, 33(4): 607-619.
[87] Shin J, Xie DL, Zhong XP. MicroRNA-34a enhances T cell activation by targeting diacylglycerol kinase ζ. PLoS ONE, 2013, 8(10): e77983.
[88] Li QJ, Chau J, Ebert PJR, Sylvester G, Min H, Liu G, Braich R, Manoharan M, Soutschek J, Skare P, Klein LO, Davis MM, Chen CZ. miR-181a is an intrinsic modulator of T cell sensitivity and selection. Cell, 2007, 129(1): 147-161.
[89] Neilson JR, Zheng GX, Burge CB, Sharp PA. Dynamic regulation of miRNA expression in ordered stages of cellular development. Genes Dev, 2007, 21(5): 578-589.
(责任编委: 李明洲)
Advances in porcine miRNAome
Maoliang Ran1,2, Bin Chen1,2, Jie Yin3,4, Anqi Yang1,2, Zhi Li1,2, Ming Jiang1,2
1. College of Animal Science & Technology, Hunan Agricultural University, Changsha 410128, China;
2. Hunan Provincial Key Laboratory for Genetic Improvement of Domestic Animal, Changsha 410128, China;
3. Institute of Subtropical Agriculture, Chinese Academy of Sciences, Changsha 410125, China;
4. University of the Chinese Academy of Sciences, Beijing 100049, China
MicroRNA (miRNA), a class of non-coding RNA (about 22 nt), widely exists in different organisms and plays an important role in growth, development, and apoptosis. Recently, many studies have shown that miRNAs serve as a regulatory mechanism in mediating the development of pig muscle, fat, reproductive and immunity traits. The next-generation of high-throughput sequencing technology plays a critical role on finding new miRNAs and identifying their different expressions in pig. In this review, we summarize the application of the next-generation of high-throughput sequencing technology in the study of pig miRNAs as well as the regulatory roles of several important miRNAs in fat metabolism, muscle development, oocyte maturation and development of B cells and T cells. This review will provide insight into the research on pig miRNAs, and some ideas on how to improve pork quality, growth, reproductive and immunity performance.
2014-05-04;
2014-08-18
国家现代农业产业技术体系建设专项资金(CARS-36)资助
冉茂良,博士研究生,专业方向:猪的遗传育种。E-mail: ranmaoliang0903@126.com
陈斌,教授,博士生导师,研究方向:猪的遗传育种。E-mail: chenbin7586@126.com
10.3724/SP.J.1005.2014.0974
时间: 2014-9-17 17:24:14
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140917.1724.003.html