母源效应蛋白在基因组印记维持中的作用
2014-05-25马馨张胜杨树宝王晓晨朱屹然李子义栾维民
马馨,张胜,杨树宝,王晓晨,朱屹然,李子义,栾维民
1. 吉林农业大学动物科技学院,动物生产及产品质量安全教育部重点实验室,长春 130118;
2. 吉林大学动物医学学院,长春 130062
母源效应蛋白在基因组印记维持中的作用
马馨1,2,张胜2,杨树宝1,王晓晨1,朱屹然1,李子义2,栾维民1
1. 吉林农业大学动物科技学院,动物生产及产品质量安全教育部重点实验室,长春 130118;
2. 吉林大学动物医学学院,长春 130062
基因组印记是指生殖细胞发生过程中双亲基因组发生差异表观修饰,使带有亲代印记的等位基因出现父源或母源单等位基因表达。在配子发生和早期胚胎发育过程中,基因组印记甲基化经历一个去除、重建和维持的复杂过程。这个过程中的任何环节被干扰都将导致印记紊乱,造成胚胎发生、胎盘形成及出生后发育异常。近来研究表明,早期胚胎发育过程中一些母源效应蛋白在印记基因表观调控中起重要作用。为了更好地理解这些母源因子对印记基因建立及维持的作用与机制,文章综述了DPPA3、ZFP57、TRIM28和DNMT1等母源效应因子近年来的相关研究进展,并探讨了这些因子对基因组印记的表观调控机制。
基因组印记;母源调控基因;DNA甲基化;植入前胚胎
基因组印记源于双亲基因组差异表观修饰,通过这种差异表观修饰机制,使父源或母源等位基因出现特异的单等位基因表达。只含父源或母源二倍体基因组的胚胎不能正常发育以及多种重大疾病特别是癌症中都伴随着印记丢失的现象,让人们认识到印记基因的正确表达在正常生长发育过程中非常重要[1,2]。目前,在哺乳动物中这种非等位表达的印记基因已经超过100种[3],它们大多都在胚胎发生、胎盘形成及脑发育过程中扮演重要的角色。
印记调控区(Imprinting control regions, ICRs)是印记基因表达的关键调控序列,来源于双亲等位基因中的一个ICR会被DNA甲基化标记,这些区域的差异性甲基化(Differentially methylated regions, DMRs)使双亲等位基因出现差异表达[4]。这种DNA甲基化的建立分别发生于卵母细胞和精子发生过程中。例如,印记基因Igf2r在精子和卵母细胞中存在不同的DNA甲基化模式,发育过程中母源Igf2r等位基因一直都保持甲基化状态。因此,理解基因组印记,探讨配子发生过程和受精后这些区域的甲基化维持机制非常关键。如果这个发育过程被干扰,如繁殖生物技术的实施等,将导致严重的动物及人类疾病,如Beckwith-Wiedemann综合征、Angelman综合征等。近来已经鉴定出了一些与植入前胚胎印记基因调控相关的母源效应因子,本文将对这些因子在基因组印记调控中的作用进行综述。
1 基因组印记建立及其重编程
基因组印记是在雌、雄配子发生过程中建立的。雌、雄配子发生遵循着不同的途径,最后产生具有不同表观遗传特征的高度分化的配子。DNA甲基化是基因组印记的主要分子机制,配子发生及成熟过程中,基因组DNA甲基化重编程研究也最为清楚。在配子发生的起始阶段,早期基因组印记通过主动去甲基化过程被删除,最终形成了截然不同的精子、卵子DNA甲基化模式[5]。雄性配子印记甲基化的建立开始于胎儿期,而雌性配子开始于出生后的卵子发生期,这些标记会非常严格地遗传给受精卵及其子代细胞。
在胚胎时期,小鼠卵原细胞进入减数分裂第一期并停留在第一次减数分裂前期,成为初级卵母细胞,直到出生前一直保持静止状态。出生后,初级卵母细胞分布于原始卵泡中。随着卵泡激活,卵泡直径会逐渐增加,卵母细胞进入生长期,在初级卵泡向次级卵泡过渡阶段,卵母细胞区域性差异甲基化开始建立[6]。雄性生殖细胞区域性差异甲基化亦发生在精子发生前:在胚胎的雄性生殖腺睾丸形成时开始,至出生时结束[7]。
小鼠卵子受精后父源基因组迅速发生主动去甲基化,而母源基因组仍然保持甲基化状态。随着卵裂的进行,母源基因组发生被动去甲基化,直到桑葚胚阶段达到最低水平。但是与大多数基因组区域相比,印记调控区能抵御这种全基因组的广泛去甲基化,从而可以稳定地从生殖系向后代遗传[8]。早期胚胎发育阶段,维持生殖系来源特异性甲基化的机制对于基因组印记遗传非常重要。一旦这种印记甲基化维持机制受到破坏,将发生印记紊乱,导致各种发育缺陷及严重疾病。目前对于配子基因组印记建立及胚胎印记维持机制的认识还比较匮乏,主要进展也基本来自于小鼠和人。
2 母源转录及胚胎基因组激活
在卵母细胞成熟过程中,母源基因组转录本会不断累积。大多数转录本(90%)会被翻译成蛋白质,并且在卵母细胞成熟过程中发挥作用,待其发育成卵子/形成合子后,大多会消失。但是仍然还有一些母源转录本在合子中起作用,为早期胚胎正常发育所必需[9]。在胚胎基因激活前,植入前期的早期胚胎发育很大程度上依赖于母源转录因子、多能因子和染色质重塑因子等。因此,在受精卵向胚胎转化过程中,这些母源效应基因可能在基因组印记调控中发挥作用。目前,研究者先后鉴定出了4个主要的在植入前早期胚胎保护印记基因甲基化位点的母源效应蛋白,分别为 DPPA3(Developmental pluripotency-associated 3或称STELLA或PGC7)[10]、ZFP57 (Zinc finger protein 57)[11]、TRIM28(Tripartite motif-containing 28,或称KAP1或TIF1b)[12]和DNMT1 (DNA methyltransferase 1)[13]。
3 母源效应基因和印记基因调控
3.1 DPPA3(PGC7)
起初,DPPA3作为卵巢和睾丸中的原始生殖细胞标记被报道[14]。虽然DPPA3对于小鼠性腺及生殖细胞发育并非必要,但如果缺乏母源DPPA3,则大部分受精卵在 4-细胞之前就会停育,很难发育至囊胚阶段[15,16]。显然,母源DPPA3为植入前早期胚胎(卵裂阶段)发育所必需。在胚胎发育过程中,父源基因的激活始于2细胞阶段,而在2细胞激活的父源DPPA3等位基因并不能拯救由于母源DPPA3敲除导致的停育。这一结果表明,DPPA3行使功能的关键时期是受精后、2-细胞之前[10]。在这期间,合子经历了父源基因组的主动去甲基化,TET3(Ten-eleven translocation 3)将5-甲基胞嘧啶(5-methylcytosine, 5mc)氧化成5-羟甲基胞嘧啶(5-hydroxylmethylcytosine, 5hmc)[10,17],本实验室前期研究也得出了一致的结论[18]。在缺乏母源DPPA3的合子中,父源和母源基因组都发生主动去甲基化[10],说明母源DPPA3在合子中有保护母源基因组免受主动去甲基化的作用。这种保护作用主要通过DPPA3与组蛋白H3第9位赖氨酸残基二甲基化(Dimethylated histone H3 lysine 9, H3K9me2)的结合实现:DPPA3结合到含H3K9me2的母源染色质DNA上,使染色质结构发生变化,降低了TET3等与染色质的亲和性,阻止5mc向5hmc转化[17]。DPPA3对包含H3K9me2的印记基因甲基化也具有保护作用,缺乏DPPA3会导致印记位点甲基化丢失。这些基因包括母源印记基因 Peg1、Peg3和Peg10,父源印记基因H19和Rasgrf1等。因此,DPPA3是一个母源效应蛋白,具有保护母源和父源印记基因免受主动去甲基化的作用。
3.2 ZFP57
ZFP57被认为是一种未分化的小鼠胚胎干细胞标记[19],为植入前胚胎发育所必需。ZFP57通过结合甲基化DNA的六聚核苷酸序列TGCCGC,抑制基因表达[20]。小鼠和人的印记基因DMRs均存在这种序列。植入前,母源ZFP57可能是这个蛋白的核心来源。根据表达谱,胚胎ZFP57在囊胚之前并不转录[21],囊胚阶段是胚胎ZFP57的检出时间[11]。为了检测ZFP57在早期胚胎发育过程中的出现时间,同时为区分母源和胚胎来源的ZFP57对早期胚胎发育所产生的影响,研究者通过特异基因敲除方案和制定交配策略获得母源及胚胎ZFP57缺失的效应。母源及胚胎的ZFP57同时删除导致胚胎致死;只删除胚胎 ZFP57导致部分新生小鼠致死;只删除母源ZFP57,由于胚胎ZFP57的拯救,并不致死。对ZFP57在基因组印记调控中的作用进行研究表明,同时删除母源及胚胎ZFP57的胚胎,其多个母源印记基因(Snrpn、Peg1、Peg3和 Peg5)及父源印记基因(Gtl2)DMRs甲基化都不能正常维持[11]。只删除母源ZFP57时,3.5 d小鼠早期胚胎的Snrpn出现低甲基化,但是甲基化水平在13.5 d被胚胎ZFP57所补偿。大量实验证明,ZFP57变异体胚胎经历了一个DNA甲基化部分丢失的过程,而这个过程在不同胚胎之间存在差异。人类ZFP57变异体胚胎也影响一些印记区域的DNA甲基化,如Plagl1、Grb10和Peg3[22]。ZFP57结合于这些印记基因甲基化的TGCCGC识别位点,维持 DMR甲基化状态,抑制基因表达。印记基因调控可能还需要一些其他蛋白质参与。近来研究发现,卵泡颗粒细胞和卵母细胞复合物中,外源HCG刺激调低了几个关键母源效应基因 (Zfp57、Dnmt1、H1foo、Zar1、Npm2和 Nlrp5)的转录[23]。这种下调将导致植入前胚胎所需的母源效应因子的转录储备降低,导致个别印记基因部分区域甲基化的随机丢失。
3.3 TRIM28
TRIM28(又称TIF1β或KAP-1)是一种转录中介因子,定位于细胞核内,能和染色质的特定区域发生作用,是许多基因转录调控复合体中的桥梁分子,最近才发现它能够保护印记区域免受去甲基化。TRIM28作为一个桥梁分子,主要还是以共抑制因子形式发挥转录抑制作用。TRIM28通过KRAB类型的锌指蛋白与特异的靶基因结合,同时可募集多种转录抑制效应的因子,如异染色质蛋白 1(Heterochromatin protein 1, HP1)、组蛋白脱乙酰基转移酶(Histone deacetylase, HDACs)、H3K9me3组蛋白甲基转移酶(Histone H3 lysine 9-specific methyltransferase, Setdb1)、DMNT1等,形成一种较大的转录抑制复合体,通过对染色质构象的调节导致附近基因表达沉默,作为转录因子或转录调控因子发挥特异的转录抑制功能(图2)[24,25]。
TRIM28是正常发育和分化过程中的关键调控因子,其在小鼠卵巢和早期胚胎中高表达,在正常分化细胞中几乎检测不到,在肿瘤细胞中表达量增加。我们实验室在对牛卵母细胞及胚胎的研究中也发现其在卵母细胞及早期胚胎中具有较高的表达水平。Messerschmidt等[12]研究表明,TRIM28在小鼠受精后重编程过程中发挥着关键作用。研究发现,在敲除母源Trim28的小鼠胚胎,1-细胞、2-细胞阶段均未检测到Trim28基因的RNA及蛋白质;随着早期合子基因激活,父源等位基因在 4细胞阶段开始转录,可检测到父源TRIM28蛋白表达。与此同时,印记基因H19、Snrpn和Gtl2的ICR存在甲基化丢失。可见母源Trim28基因的删除将广泛地影响早期胚胎基因组印记甲基化的维持,导致部分胚胎致死。令人关注的是,随后表达的父本Trim28基因并不足以在发育后期拯救这些胚胎,暗示母源TRIM28缺失造成的早期胚胎损伤会在发育过程中积累下来,印记缺陷一旦发生就不能修复,影响胚胎发育的后继阶段,展现出母源TRIM28在保护印记基因甲基化中的重要作用。
3.4 DNMT1
DNMT1属于DNA甲基化转移酶家族,既能催化重头甲基化又能维持甲基化DNA。在雌性和雄性生殖细胞中,未甲基化的胞嘧啶要获得甲基化修饰,需要DNA甲基化转移酶3A(DNMT3A)和它的从属蛋白DNMT3L[26~28]。比较而言,DNMT1优先识别和向半甲基化的双链DNA导入甲基,并在每个复制周期对这种甲基化进行维持,当然也包括印记区域甲基化的维持。DNMT1的2个亚型,即卵母细胞特异型(DNMT1o)和体细胞特异型(DNMT1s)。DNMT1o出现在成熟的卵母细胞和植入前胚胎。卵母细胞成熟过程中DNMT1o会累积到很高的浓度,并在卵母细胞和植入前胚胎中维持很高的表达量。DNMT1o在植入前胚胎主要分布在细胞质中,在 8-细胞期短暂分布于细胞核中[29]。这个入核现象是否真实仍然存在争议:有文献报道DNMT1o在植入前任何阶段都不会出现在细胞核中[13,30]。母源和胚胎来源的DNMT1s也在植入前胚胎中存在,但是比DMNT1o浓度低[30,31]。
DNMT1的两个亚型都在阻止印记基因被动去甲基化过程中起作用。作为一个母源效应蛋白,DNMT1o缺乏并不影响卵母细胞甲基化的获得,但是引起来源于DNMT1o敲除的卵母细胞变异体胚胎的一系列印记甲基化的丢失[13,31]。对于Igf2r,甲基化丢失发生在8-细胞、桑葚胚、囊胚,但是4-细胞变异体胚胎没有发生甲基化丢失。通过显微注射抗 DNMT1s抗体,删除母源DNMT1s,同样在桑葚胚阶段出现了H19印记甲基化的丢失[30]。同时删除母源的DNMT1o和 DNMT1s也导致囊胚阶段 H19、Rasgrf1、Peg3和Snrpn的ICR的部分甲基化丢失。因此,缺失母源DNMT1o及DNMT1s,将导致印记基因甲基化的部分丢失。相反,同时缺乏母源和胚胎的 DNMT1将导致H19、Rasgrf1、Peg3和Snrpn的ICR的完全去甲基化[13]。因此,母源和胚胎DNMT1提供保护,使印记基因抵御植入前早期胚胎被动去甲基化的影响,这个作用得以实现,可能是通过ZFP57识别并结合ICR中甲基化的TGCCGC序列,TRIM28与ZFP57结合,这样ZFP57/KAP1复合物再招募与其相关的染色质修饰因子如DNMT1、SETDB1、HP1及NP95等(图1),从而阻止了ICR的完全去甲基化,但是确切的机制仍然不清楚[20]。
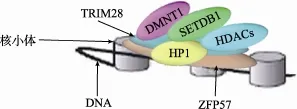
图1 TRIM28、ZFP57及相关蛋白形成转录抑制复合体
4 结语与展望
随着研究的不断深入,母源效应因子在胚胎发育中的作用也逐渐清晰,包括在卵母细胞发育及卵母细胞向胚胎转化过程中全基因组重编程中的作用。母源蛋白 DPPA3可以保护印记基因免受主动去甲基化的影响,而其他 3个蛋白ZFP57、TRIM28及DNMT1,目前研究认为可以保护印记基因在植入前早期胚胎免受被动去甲基化的影响。除以上 4个基因,还有一些母源效应基因也在保护印记基因免受去甲基化影响的过程起作用,如HIfoo[32]、Npm2[33]和Nlrp5[34]也是母源效应因子,参与卵母细胞向胚胎转化过程中的表观遗传调控。但是,这几个基因并不直接与基因组印记维持相关联。SETDB1也是母源效应因子,催化附加的甲基群从 H3K9me2到H3K9me3的转化。但是,它是否在印记维持过程中起作用还需进一步阐明。虽然 DPPA3、ZFP57和TRIM28是印记基因的ICR甲基化保护者,但是他们并不能保护所有印记基因的 ICR甲基化。如DPPA3删除并不影响Snrpn及Peg5的印记甲基化[17],母源及胚胎的ZFP57删除并不能导致H19的甲基化丢失[11],TRIM28删除并不影响Peg3印记甲基化[12]。最后,由于这些基因异常表达导致的印记丢失具有随机性,所以甲基化错误可能不被检测到,这就需要分析大量的胚胎才能出结论。
总之,母源效应基因的发现,给基因组印记调控研究带来了新的机遇。植入前早期胚胎表观遗传重编程,使特异性维持复合物募集于印记区域,以保证表观遗传机构的完整性,这个过程起始于受精后,并且一直持续整个卵裂阶段。大量研究表明,繁殖生物技术影响这一过程中的重编程事件,导致印记紊乱。因此,理解这些母源因子对印记基因维持的作用与机制,理解繁殖生物技术对这些母源效应因子分布及功能的干扰,对提高繁殖生物技术的效率及最大化其安全性,保证胚胎正常发育具有重要意义。
[1] Kono T, Obata Y, Wu Q, Niwa K, Ono Y, Yamamoto Y, Park ES, Seo JS, Ogawa H. Birth of parthenogenetic mice that can develop to adulthood. Nature, 2004, 428(6985): 860-864.
[2] Ferguson NM, Donnelly CA, Anderson RM. The footand-mouth epidemic in Great Britain: pattern of spread and impact of interventions. Science, 2001, 292(5519): 1155-1160.
[3] Williamson CM, Blake A, Thomas S, Beechey CV, Hancock J, Cattanach BM, Peters J. World Wide Web Site-Mouse Imprinting Data and References. MRC Harwell, Oxfordshire, 2013, http://www.har.mrc.ac.uk/research/genomic_ imprinting/.
[4] Spahn L, Barlow DP. An ICE pattern crystallizes. Nat Genet, 2003, 35(1): 11-12.
[5] Kelsey G, Feil R. New insights into establishment and maintenance of DNA methylation imprints in mammals. Philos Trans R Soc Lond B Biol Sci, 2013, 368(1609): 20110336.
[6] Denomme MM, Mann MRW. Genomic imprints as a model for the analysis of epigenetic stability during assisted reproductive technologies. Reproduction, 2012, 144(4): 393-409.
[7] Henckel A, Chebli K, Kota SK, Arnaud P, Feil R. Transcription and histone methylation changes correlate with imprint acquisition in male germ cells. EMBO J, 2012, 31(3): 606-615.
[8] Morgan HD, Santos F, Green K, Dean W, Reik W. Epigenetic reprogramming in mammals. Hum Mol Genet, 2005, 14(Suppl 1): R47-R58.
[9] Sirard MA. Factors affecting oocyte and embryo transcriptomes. Reprod Domest Anim, 2012, 47(Suppl. 4): 148-155.
[10] Nakamura T, Arai Y, Umehara H, Masuhara M, Kimura T, Taniguchi H, Sekimoto T, Ikawa M, Yoneda Y, Okabe M, Tanaka S, Shiota K, Nakano T. PGC7/Stella protects against DNA demethylation in early embryogenesis. Nat Cell Biol, 2007, 9(1): 64-71.
[11] Li XJ, Ito M, Zhou F, Youngson N, Zuo XP, Leder P, Ferguson-Smith AC. A maternal-zygotic effect gene, Zfp57, maintains both maternal and paternal imprints. Dev Cel, 2008, 15(4): 547-557.
[12] Messerschmidt DM, De V W, Ito M, Solter D, Ferguson-Smith A, Knowles BB. Trim28 is required for epigenetic stability during mouse oocyte to embryo transition. Science, 2012, 335(6075): 1499-1502.
[13] Hirasawa R, Chiba H, Kaneda M, Tajima S, Li E, Jaenisch R, Sasaki H. Maternal and zygotic Dnmt1 are necessary and sufficient for the maintenance of DNA methylation imprints during preimplantation development. Genes Dev, 2008, 22(12): 1607-1616.
[14] Goto T, Jones GM, Lolatgis N, Pera MF, Trounson AO, Monk M. Identification and characterisation of known and novel transcripts expressed during the final stages of human oocyte maturation. Mol Reprod Dev, 2002, 62(1): 13-28.
[15] Payer B, Saitou M, Barton SC, Thresher R, Dixon JPC, Zahn D, Colledge WH, Carlton MBL, Nakano T, Surani MA. Stella is a maternal effect gene required for normal early development in mice. Curr Biol, 2003, 13(23): 2110-2117.
[16] Bortvin A, Goodheart M, Liao M, Page DC. Dppa3 / Pgc7 /stella is a maternal factor and is not required for germ cell specification in mice. BMC Dev Biol, 2004, 4: 2.
[17] Nakamura T, Liu YJ, Nakashima H, Umehara H, Inoue K, Matoba S, Tachibana M, Ogura A, Shinkai Y, Nakano T. PGC7 binds histone H3K9me2 to protect against conversion of 5mC to 5hmC in early embryos. Nature, 2012, 486(7403): 415-419.
[18] Zhang P, Su L, Wang ZW, Zhang S, Guan JY, Chen Y, Yin YP, Gao F, Tang B, L ZY. The involvement of 5-hydroxymethylcytosine in active DNA demethylation in Mice. Biol Reprod, 2012, 86(4): 104.
[19] Okazaki S, Tanase S, Choudhury BK, Setoyama K, Miura R, Ogawa M, Setoyama C. A novel nuclear protein with zinc fingers down-regulated during early mammalian cell differentiation. J Biol Chem, 1994, 269(9): 6900-6907.
[20] Quenneville S, Verde G, Corsinotti A, Kapopoulou A, Jakobsson J, Offner S, Baglivo I, Pedone PV, Grimaldi G, Riccio A, Trono D. In embryonic stem cells, ZFP57/KAP1 recognize a methylated hexanucleotide to affect chromatin and DNA methylation of imprinting control regions. Mol Cell, 2011, 44(3): 361-372.
[21] Zeng FY, Baldwin DA, Schultz RM. Transcript profiling during preimplantation mouse development. Dev Biol, 2004, 272(2): 483-496.
[22] Mackay DJG, Callaway JLA, Marks SM, White HE, Acerini CL, Boonen SE, Dayanikli P, Firth HV, Goodship JA, Haemers AP, Hahnemann JMD, Kordonouri O, Masoud AF, Oestergaard E, Storr J, Ellard S, Hattersley AT, Robinson DO, Temple IK. Hypomethylation of multiple imprinted loci in individuals with transient neonatal diabetes is associated with mutations in ZFP57. Nat Genet, 2008, 40(8): 949-951.
[23] Agca C, Yakan A, Agca Y. Estrus synchronization and ovarian hyper-stimulation treatments have negligible effects on cumulus oocyte complex gene expression whereas induction of ovulation causes major expression changes. Mol Reprod Dev, 2013, 80(2): 102-117.
[24] Ryan RF, Schultz DC, Ayyanathan K, Singh PB, Friedman JR, Fredericks WJ, Rauscher FJ. KAP-1 corepressor protein interacts and colocalizes with heterochromatic and euchromatic HP1 proteins: a potential role for Kruppel-associated box-zinc finger proteins in heterochromatin-mediated gene silencing. Mol Cell Biol, 1999, 19(6): 4366-4378.
[25] Iyengar S, Ivanov AV, Jin VX, Rauscher FJ, Farnham PJ. Functional analysis of KAP1 genomic recruitment. Mol Cell Biol, 2011, 31(9): 1833-1847.
[26] Bourc'his D, Xu GL, Lin CS, Bollman B, Bestor TH. Dnmt3L and the establishment of maternal genomic imprints. Science, 2001, 294(5551): 2536-2539.
[27] Kaneda M, Okano M, Hata K, Sado T, Tsujimoto N, Li E, Sasaki H. Essential role for de novo DNA methyltransferase Dnmt3a in paternal and maternal imprinting. Nature, 2004, 429(6994): 900-903.
[28] Hara S, Takano T, Fujikawa T, Yamada M, Wakai T, Kono T, Obata Y. Forced expression of DNA methyltransferases during oocyte growth accelerates the establishment of methylation imprints but not functional genomic imprinting. Hum Mol Genet, 2014, 23(14): 3853-3864.
[29] Howell CY, Bestor TH, Ding F, Latham KE, Mertineit C, Trasler JM, Chaillet JR. Genomic imprinting disrupted by a maternal effect mutation in the Dnmt1 gene. Cell, 2001, 104(6): 829-838.
[30] Kurihara Y, Kawamura Y, Uchijima Y, Amamo T, Kobayashi H, Asano T, Kurihara H. Maintenance of genomic methylation patterns during preimplantation development requires the somatic form of DNA methyltransferase 1. Dev Biol, 2008, 313(1): 335-346.
[31] Cirio MC, Martel J, Mann M, Toppings M, Bartolomei M, Trasler J, Chaillet JR. DNA methyltransferase 1o functions during preimplantation development to preclude a profound level of epigenetic variation. Dev Biol, 2008, 324(1): 139-150.
[32] Maeda C, Sato S, Hattori N, Tanaka S, Yagi S, Shiota K. DNA hypomethylation circuit of the mouse oocyte-specific histone H1foo gene in female germ cell lineage. Biol Reprod, 2008, 78(5): 816-821.
[33] Burns KH, Viveiros MM, Ren YS, Wang P, DeMayo FJ, Frail DE, Eppig JJ, Matzuk MM. Roles of NPM2 in chromatin and nucleolar organization in oocytes and embryos. Science, 2003, 300(5619): 633-636.
[34] Fernandes R, Tsuda C, Perumalsamy AL, Naranian T, Chong J, Acton BM, Tong ZB, Nelson LM, Jurisicova A. NLRP5 mediates mitochondrial function in mouse oocytes and embryos. Biol Reprod, 2012, 86(5): 138, 1-10.
(责任编委: 高绍荣)
The roles of maternal-effect proteins in the maintenance of genomic imprints
Xin Ma1,2, Sheng Zhang2, Shubao Yang1, Xiaochen Wang1, Yiran Zhu1, Ziyi Li2, Weimin Luan1
1. College of Animal Science and Technology, Key Laboratory of Animal Production and Production Quality and Security, Ministry of Education, Jilin Agricultural University, Changchun 130118, China;
2. College of Veterinary Medicine, Jilin University, Changchun 130062, China
Genomic imprinting is a mechanism of differentially epigenetic modification that restricts monoallelic expression to either the maternally or paternally inherited copy of the gene during gametogenesis. Imprinted methylation undergoes a process of erasure, acquisition, and maintenance during gametogenesis and early embryogenesis. Disruptions in any of these steps may lead to imprinting disorders, resulting in the aberrant development of embryogenesis, placentation and postnatal growth. Recent studies have shown that maternal-effect proteins are important for the regulation of imprinted gene during the development of preimplantation embryos. In order to obtain a better understanding for the mechanism of maternal-effect proteins in the maintenance of genomic imprints, the recent study progress of maternal-effect proteins, such as DPPA3, ZFP57, TRIM28 and DNMT1, are summarized, and the regula-tion mechanism of these maternal-effect proteins for genomic imprints are discussed.
genomic imprinting; maternal-effect genes; DNA methylation; preimplantation embryos
2014-06-20;
2014-08-11
国家自然科学基金青年基金项目(编号:31302047)资助
马馨,博士,副教授,研究方向:动物克隆与转基因动物。E-mail: maxin3202@163.com
栾维民,博士,教授,研究方向:免疫器官发生发育。E-mail: luanweimin@aliyun.com
李子义,博士,教授,研究方向:动物克隆与转基因动物。E-mail: ziyi@jlu.edu.cn
10.3724/SP.J.1005.2014.0959
时间: 2014-9-17 17:22:14
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140917.1722.001.html