基于内容检索的硅藻细胞自动分类
2014-05-25牟琦,周平
牟 琦,周 平
(浙江理工大学信息学院,杭州310018)
基于内容检索的硅藻细胞自动分类
牟 琦,周 平
(浙江理工大学信息学院,杭州310018)
对硅藻细胞分类提出了一种基于内容的分类方法。该方法首先获取带外接圆的目标;然后,对其应用纹理主特征直方图,纹理变化度,纹理角特征3种方法提取出目标的特征向量;最后使用欧氏距离进行相似性度量。实验结果表明,该方法具有较好的分类准确率及召回率。
硅藻细胞分类;基于内容的图像检索;图像特征提取
0 引 言
硅藻是一种大型的,对环境十分重要的单细胞藻类群体,它基本被发现于所有的水生栖息地中。位于海洋生物链的底层,是其他海洋生物的重要食物来源,所以对整个海洋生物链起着地基的作用。同时,也对海洋的生态平衡起着至关重要的作用。此外,硅藻有着广泛的应用[1-2],包括水质量评价、天气估测、考古和医学制药等各个领域。但是,硅藻的每个种类的作用又各不相同,有的可以用来检测水体质量,但其本身却不具有药用价值。所以对硅藻的鉴定和识别变得尤为重要。
大多数分类使用分类关键点或者通过电脑和书本上的样本图片进行对比[3]。这不是一件简单的工作,因为考虑到生物学家可能要评估20万个不同种类的硅藻,而且许多基于形态非常难以区别。所以通过算法和软件对硅藻进行自动识别鉴定成为了一种必要的,同时也是一种非常方便、高效的检测手段。
大多数藻类细胞的分类思路是先分割再通过提取颜色、形状、纹理等特征得到特征向量[4-10],最后通过分类器得到结果。但是,如果分类的种类过多时,工程量非常巨大,最后所要使用的判断标准和判断数据也会极其复杂。所以,本文通过基于内容的图像检索找到与待检索图像最像的种类,以达到快速分类的目的。
1 获取外接圆
1.1 图片预处理
首先对图像进行灰度化和Sobel边缘检测,得到灰度边缘检测图像,再对结果图像进行Otsu分割。Otsu法是一种使类间方差最大的阈值确定方法,所以也称为最大类间方差法。该方法具有简单、处理速度快等特点,是一种常用的阈值选择方法。其基本思想是把图像中的像素按灰度值用阈值T分为两类C1和C2,C1由灰度值在[0,T]之间的像素组成,C2由灰度值在[T+1,L-1]之间的像素组成,按照下式计算两类之间的类间方差:

式(1)中ω1(t)为C1中所包含的像素数,ω2(t)为C2中所包含的像素数,μ1(t)为C1中所有像素数的平均值,μ2(t)为C2中所有像素数的平均值。让T在[0,L-1]范围依次取值,使σ(t)2最大的T值即为Otsu法的最佳阈值。
之后为所得图像进行膨胀和去除小面积。去除小面积使用轮廓跟踪、孔洞填充和面积过滤的方法。先使用轮廓跟踪找到图像各个连通域的轮廓并储存,之后对轮廓进行孔洞填充,并将小于总轮廓面积5%的小面积过滤掉。这样做不仅可以将图片的杂点去除,还可以将置于图片上的比例尺去除,方便后续的操作。
1.2 外接圆算法
通常使用的外接圆算法是将图像二值化后,通过求取形心和半径求取外接圆。但是这种方法易受到目标外干扰点及比例尺的影响导致外接圆提取过大。针对这种情况,本文提出一种外接矩收缩法获得外接圆。图1为本文所用外接圆算法和重心半径法的效果比较图,图2列出了外接圆的整个获取过程。其中外接矩收缩法的详细步骤如下:

图1 外接圆算法比较

图2 外接圆的获取过程
步骤1:求得左上方最小横纵坐标x1、y1,右下方最大横纵坐标x2、y2;
步骤2:按此二点(x1,y1)、(x2,y2)画出目标外接矩;
步骤3:初始化四个坐标为矩形中心的点,检测外接矩四条边上与目标的交点,若有交点则更新四点中对应点,四点从上边以顺时针方向命名为p1,p2,p3,p4;
步骤4:移动矩形右下角坐标,每次往八邻域中的左上角移动一个像素;移动后检测p2,p3是否与目标相交,若相交则停止;
步骤5:移动矩形左上角,每次往八邻域中的右下角移动一个像素;移动后检测p1,p4是否与目标相交,若相交则停止;
步骤6:外接圆半径,圆心以最终的矩形左上角和右下角点连线为直径计算。
2 特征提取
2.1 纹理主特征直方图
本文对灰度图像采用5位位屏蔽用于简化数据计算量,屏蔽后图像的纹理特征并未有根本性的变化,但是亮度值从256缩减到了8种。将屏蔽后的图像的外接圆从内到外按距离均分10等份,从而得到10个圆环。之后统计每个圆环上8种亮度的数目,就此得到一个8×10的特征向量,向量的从低到高排列顺序为按照圆环由内而外,亮度由低到高排列。实验划分图和数据直方图分别如图3和图4所示。由图4(a)和(c)、(e)和(g)比较可知,相似的图片通过上述方法可以获得相似的直方图数据,同时对目标具有旋转和绽放不变性。
计算相似度时,按照同色、同环对比,每个圆环所取得的数据都要乘以各自圆环的加权值,加权值由内而外为0.02、0.06、0.1、0.11、0.12、0.15、0.2、0.15、0.1、0.04。加权后,按照同色、同环的方式使用欧氏距离计算相似度。在欧式平面上,任意两点(i1,j1)和(i2,j2)的距离表示为
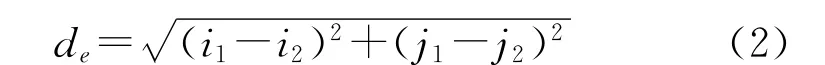

图3 实验同心圆划分示意
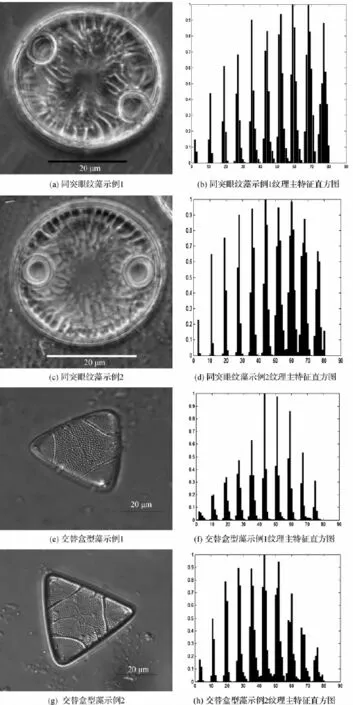
图4 同突眼纹藻、交替盒型藻纹理主特征直方图示例
2.2 纹理变化度
不同的纹理必然存在的不同的亮度变化,此处,本文正是利用这一点采用除去最外环后的其他9个圆环上的亮度变化次数,从而得到9维的目标特征向量。计算相似度时,按照同色、同环对比,每个圆环所取得的数据都要乘以各自圆环的加权值,加权值由内而外为0.04、0.09、0.12、0.13、0.13、0.13、0.13、0.12、0.1。加权后,按照同色、同环的方式使用欧氏距离计算相似度。表1为图4(a)、图4(e)的纹理变化度数据。
2.3 纹理角特征
目标纹理是由许多小结构组成的,而这些结构在图像上亮度相似,由于之前已经过5位位屏蔽处理使得亮度相近的归为了一类,所以在此处,本文采用统计圆环上同亮度点的夹角角度作为数据依据。通过实验观察(将角度1~180°划分为180份,每份1°),统计每个圆环上同亮°间角°数据时,角度越大圆环上存在的这种角度数目越少,3°以下的统计值过高且各个种类3°以下数值相差不大,这是由于噪声干扰和亮度不均造成的,故此处将角度区间以2为基的倍增方式划分,具体划分为(3°,10°]、(10°,20°]、(20°,40°]、(40°,80°]、(80°,160°]、(160°,180°]。由此可以得到一个8×6的特征向量(由于只有8个内圆环可以保证位于目标内)。计算相似度时,按照同色、同环对比,每个圆环所取得的数据都要乘以各自圆环的加权值,加权值由内而外为0.01、0.09、0.15、0.15、0.15、0.15、0.15、0.15。加权后,按照同色、同环的方式使用欧氏距离计算相似度。图5(a)、图5(b)分别为图4(a)、图4(e)的纹理角特征数据。
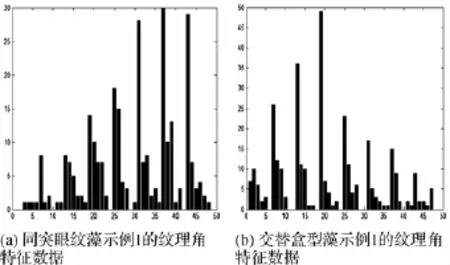
图5 同突眼纹藻及交替盒型的藻纹理角特征数据
3 实验结果
基于内容的图像检索的检索效果评价有多种方法,比较常用的方法有准确率与召回率、准确率-召回率曲线以及排序评价方法等[11-12]。本文采用准确率和召回率对基于内容的硅藻自动鉴别系统-DRS(diatom retrieval system)的检索性能进行评价,并对实验结果进行了分析。图6为准确率、召回率示意图,其中A表示检索到的相关文件,B表示检索到的不相关文件,C表示未检索到的相关文件,D表示未检索到的不相关文件。测试图像数据库含有396幅硅藻图像。随机抽取图像数据库中的20幅图像作为测试图像,分别按照纹理主特征直方图,纹理变化度,纹理角特征3种方法进行单特征和综合特征检索,构成20次检索。
召回率(recall rate):是检索出的相关文档数A和文档库中所有的相关文件数A+C的比率,衡量的是检索系统的查全比例。即R=A/(A+C)。
准确率(precision rate):是检索出的相关文档数A与检索出的文件总数A+B的比率,衡量的是检索系统的精度。即P=A/(A+B),本实验中A最大为3,A+B为40,所以理论最高值为7.5%。
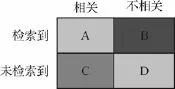
图6 准确率、召回率示意图
对于每次的检索结果,通过准确率和召回率计算当前的检索数据结果。综合10次检索数据结果,得到了相应的平均准确率和召回率(表2),其中精确率满值为7.5%,召回率满值为100%。由实验结果可知,综合特征比单一特征的检索效果要好,本文方法的检索性能明显优于SEH[13],SHE表示结构元素直方图(structure elements'histogram),结构元素的具体定义详见参考文献[13]。
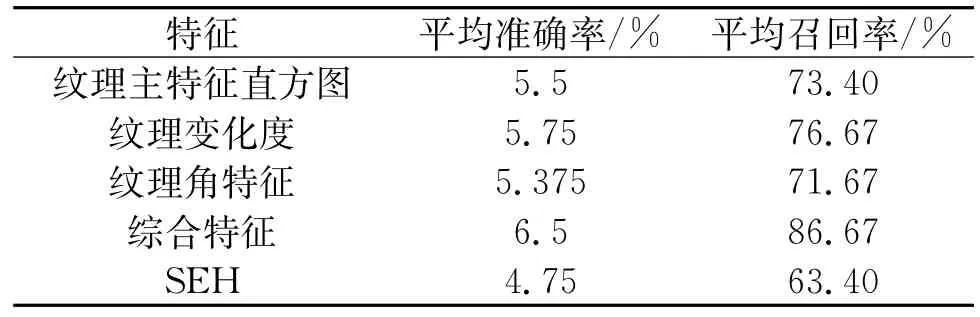
表2 不同方法的P-R比较
4 结 论
本文通过基于内容的图像检索技术,实施硅藻的自动鉴定识别,并研发了硅藻图像检索系统-DRS。DRS以硅藻为搜索对象,使用纹理主特征直方图,纹理变化度,纹理角特征3种方法提取目标的特征向量,最后通过相似性对比得到最有可能的分类类别,降低了因疲劳和主观知识范围的束缚所造成的分类失误以及因失误而引起的后续试验和生产的误差和错误。同时为快速准确地鉴定硅藻种类提供了技术支持。根据对系统进行的测试和分析可知,基于综合特征的内容检索相较单一特征准确率和召回率更高,SEH对目标和背景颜色差异较大的检索会有较好的效果,同时SEH方法较为依赖颜色特征所以对于有着复杂纹理特征和一些颜色不一样但是纹理相似的图片有着较差的准确率和召回率,但是本文所提出的3个特征对于旋转不变性、复杂纹理都有着较好的效果。
[1]Stoermer E F,Kreis Jr R G,Andresen N A.Checklist of diatoms from the Laurentian Great lakes.II[J].Journal of Great Lakes Research,1999,25(3):515-566.
[2]Jones V.Diatom Introduction[M].Elias S A.Encyclopedia of Quaternary Science.Amsterdam:Elsevier Inc,2007:476-484.
[3]Smol JP,Stoermer E F.The Diatoms:Applications for the Environmental and Earth Sciences[M].Cambridge:Cambridge University Press,2004:21-25.
[4]Songn Q,Wang G,Wang C.Automatic recommendation of classification algorithms based on dataset characteristics[J]. Pattern Recognition,2012(45):2672-2689.
[5]Dimitrovski I,Kocev D,Loskovska S,et al.Hierarchical classification of diatom images using ensembles of predictive clustering trees[J].Ecological Informatics,2012,7(1):19-29.
[6]Peng B,Zhang L,Zhang D.A survey of graph theoretical approaches to image segmentation[J].Pattern Recognition,2013(46):1020-1038.
[7]Xie F,Bovik A C.Automatic segmentation of dermoscopy images using self-generating neural networks seeded by genetic algorithm[J].Pattern Recognition,2013(46):1012-1019.
[8]Zhao F,Lin F,Seah H S.Binary SIPPER plankton image classification using random subspace[J].Neurocomputing,2010(73):1853-1860.
[9]Chang L,Duarte M M,Sucar L E,et al.A Bayesian approach for object classification based on clusters of SIFT local features[J].Expert Systems with Applications,2012,39(2):1679-1686.
[10]Ranzato M,Taylor P E,House J M,et al.Automatic recognition of biological particles in microscopic images[J].Pattern Recognition Letters,2007,28(1):31-39.
[11]韦 娜,耿国华,周明全.基于内容的图像检索系统性能评价[J].中国图象图形学报,2004,9(11):1271-1275.
[12]茹立云,彭 潇,苏 中,等.基于内容图像检索中的特征性能评价[J].计算机研究与发展,2003,40(11):1566-1570.
[13]Wang X,Wang Z.A novel method for image retrieval based on structure elements'descriptor[J].Journal of Visual Communication and Image Representation,2013,24(1):63-74.
Automatic CIassification of Diatom CeIIs Based on Content RetrievaI
MU Qi,ZHOU Ping
(The School of Information Science and Technology,Zhejiang Sci-Tech University,Hangzhou 310018,China)
A content-based classification method is proposed for sorting diatom cells.This method first gets the goal with a circumscribed circle and then extracts the feature vector of the target by applying the histogram of main features of the texture,texture change degree,texture angle characteristics.Finally,Euclidean distance is used for similarity measure.Experimental results show that the method has good classification precision rate and recall rate.
diatom cell classification;content-based image retrieval;image feature extraction
TP391.4
A
(责任编辑:陈和榜)
1673-3851(2014)02-0211-05
2013-09-06
牟 琦(1987-),男,山东青岛人,硕士研究生,主要从事图像处理方面的研究。
周 平,E-mail:zp@zstu.edu.cn
