植物乳杆菌中胆盐水解酶的研究现状及展望
2014-05-17董自星李炳薰堵国成李华钟
董自星 ,张 娟 ,*,李炳薰 ,堵国成 ,陈 坚 ,李华钟 ,*
(1.江南大学生物工程学院,江苏无锡 214122;2.江南大学工业生物技术教育部重点实验室,江苏无锡 214122;3.江南大学粮食发酵工艺与技术国家工程实验室,江苏无锡 214122;4.江南大学糖化学与生物技术教育部重点实验室,江苏无锡 214122)
根据流行病学和临床研究的结果,血清胆固醇过多具有诱发冠心病、动脉粥样硬化等众多心血管疾病的风险,而在许多国家心血管疾病是引起残疾和死亡的重要因素之一[1]。虽然有许多手段可以治疗血胆固醇过多症,比如药物,但是高昂的价格和产生的副作用限制了它们的广泛使用。近年来国内外许多研究者证实,口服益生菌及其相关制品可以显著降低人[2-3]、猪[4]、狗[5]和老鼠[6]等哺乳动物体内的血清胆固醇水平。益生菌的降胆固醇作用与它们产生的胆盐水解酶(bile salt hydrolase, BSH,EC 3.5.1.24)密切相关。该酶能将结合胆盐水解成游离胆酸和氨基酸。由于游离胆酸的溶解度低于结合胆盐,它便随粪便排出体外,这就促进了体内的胆固醇从头合成胆盐以弥补损失,从而使胆固醇水平降低[7]。目前,商业化的罗伊氏乳杆菌(加拿大,蒙特利尔,Micropharma公司)已经上市。这为治疗血胆固醇过多症提供了一种比较天然的方法,也使胆盐水解酶活性成为益生菌筛选时的一个必要特征。但也有一些临床实验表明益生菌对人体没有显著的降胆固醇作用[8-9],因此引起了一些争议。但这些益生菌是否产胆盐水解酶,目前还没有报道。
目前已在许多微生物中检测或纯化到胆盐水解酶,包括双歧杆菌,乳杆菌,梭状芽孢杆菌,类杆菌和粪肠球菌等[7]。关于植物乳杆菌(Lactobacillus plantarum)来源的胆盐水解酶也有一些报道,主要集中在高产菌株的筛选、克隆表达、生理生化特性、降胆固醇作用等方面,但对其降胆固醇的机理、可溶性表达、食品级表达以及底物结合机制等方面研究少。本文综述了植物乳杆菌胆盐水解酶的降胆固醇作用、底物结合机制以及异源表达等方面的研究,对揭示其降胆固醇的机制和开发相关产品具有重要意义。
1 植物乳杆菌来源的胆盐水解酶的研究现状
1.1 植物乳杆菌的降胆固醇作用
近年来,国内外许多研究者已经对植物乳杆菌的降胆固醇作用做了体内和体外的研究。它们都证明了植物乳杆菌具有显著的降胆固醇作用。表1列举了一些体内研究的结果,从中可以看出,口服植物乳杆菌能明显降低动物或人体的总胆固醇、低密度脂蛋白胆固醇、高密度脂蛋白胆固醇、甘油三酯等的水平,特别是降低低密度脂蛋白胆固醇和高密度脂蛋白胆固醇的比例(LDL-C/HDL-C,冠心病一个重要的风险因素[10])。其中,不同来源的菌株使哺乳动物体内的总胆固醇和不利于人体健康的低密度脂蛋白胆固醇分别降低了 2.5%~33%和7.9%~42%。而口服L. plantarum K25使昆明雄性老鼠体内的LDL-C/HDL-C降低了43.8%。但是,也有报道表明一些益生菌对人体没有明显的降胆固醇作用,这可能跟菌体的有效剂量和益生元的使用有关[11]。
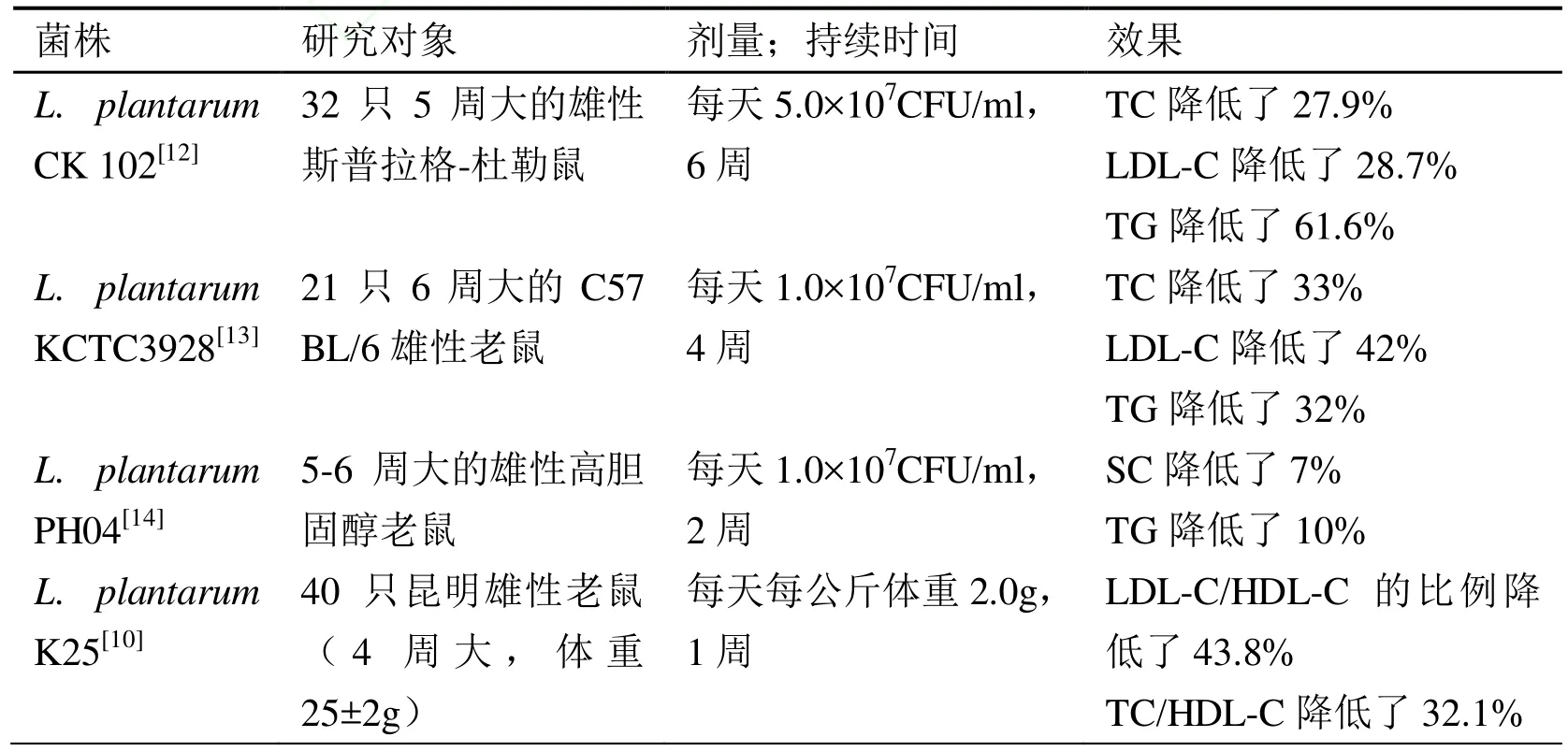
表1 植物乳杆菌在体内的降胆固醇作用Table 1 Cholesterol-lowering effect of different strains of L. plantarum in vivo
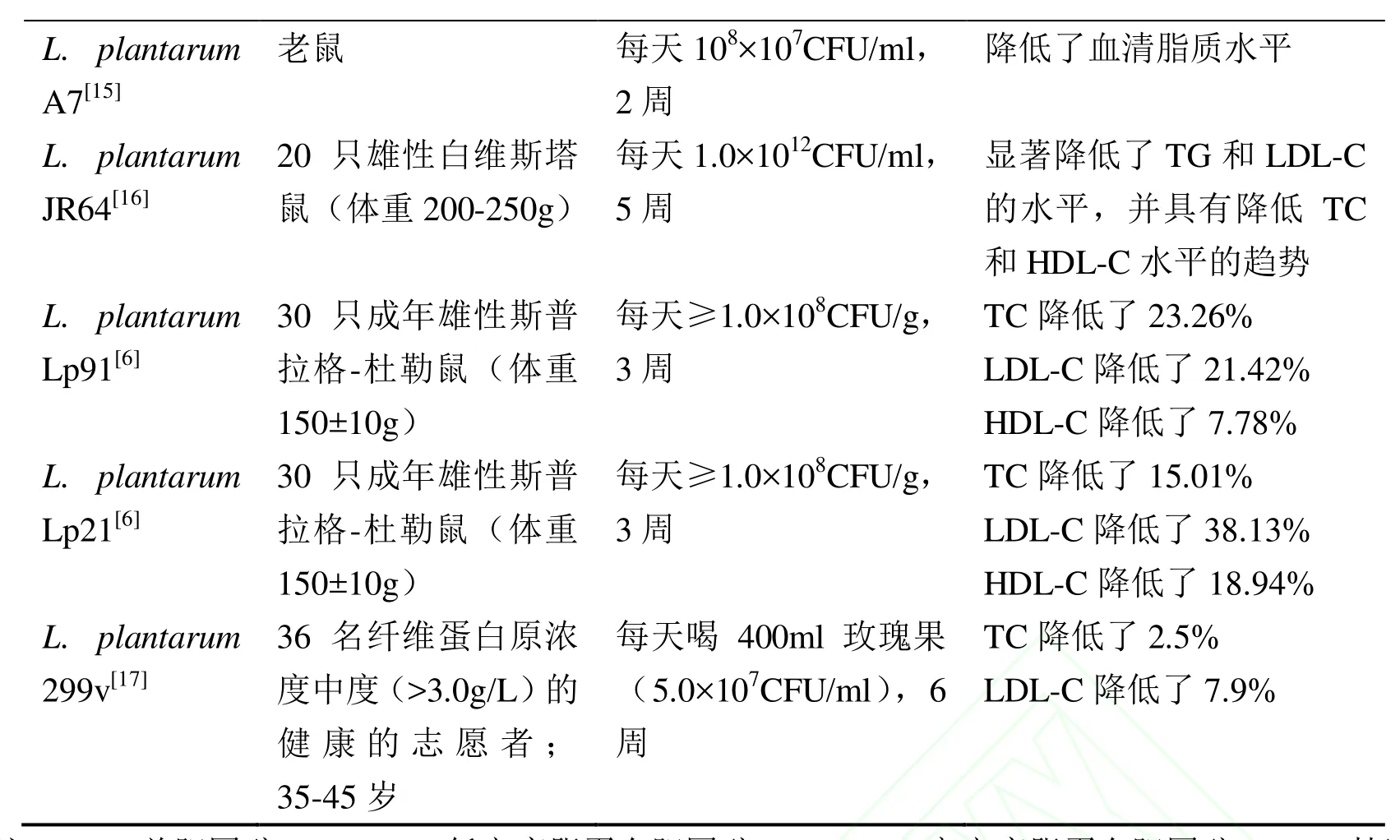
注:TC,总胆固醇;LDL-C,低密度脂蛋白胆固醇;HDL-C,高密度脂蛋白胆固醇;TG,甘油三酯;SC,血清胆固醇。
关于乳酸菌降解胆固醇的机制,目前还没有定论。主要存在以下几种观点:(1)同化吸收,即菌体细胞同化吸收介质中的胆固醇,从而降低了胆固醇的含量[18]。(2)共沉淀,胆盐水解酶使胆盐脱结合,脱结合的胆盐的溶解度低于结合胆盐,它在酸性条件下易与胆固醇形成复合物沉淀下来,导致介质中胆固醇含量下降[19]。(3)其他理论。尽管关于确实的机理尚无定论,国内外大部分学者认为是同化吸收和共沉淀的共同作用,而且乳酸菌的这种降胆固醇作用与它们产生的胆盐水解酶密切相关。除了共沉淀,胆盐水解酶降胆固醇的另一种可能的途径是:胆盐水解酶作用后的脱结合胆盐在大肠内不被吸收而随粪便排出体外,这就促使了胆盐由胆固醇的从头合成,从而降低体内的胆固醇浓度。
1.2 胆盐水解酶基因的克隆表达
Christiaens等[20]于1992年第一次报道了植物乳杆菌来源的胆盐水解酶基因的克隆。其它来源于植物乳杆菌的胆盐水解酶基因陆续被报道。从表2中可以看出,这些植物乳杆菌可以被分为两大类:一类是只产一种胆盐水解酶的菌,比如 L. plantarum 80、L. plantarum MBUL90和L. plantarum BBE7等,它们的胆盐水解酶基因的大小一般为975 bp;另一类是产四种不同大小的同工酶的菌,例如L. plantarum ST-III和 L. plantarum WCFS1等,这可能是基因水平转移的结果[7]。这四种胆盐水解酶基因(bsh1,bsh2,bsh3和bsh4)的大小分别为975 bp,1017 bp,987 bp和954 bp,且位于染色体的不同位置,氨基酸相似度从21%到39%。Lambert等[21]敲除L. plantarum WCFS1中胆盐水解酶基因以后发现bsh1对胆盐水解酶的酶活起到主要作用,bsh2,bsh3和bsh4在各菌株间相对保守,具有重要的生理学意义。
黄茜、战媛媛和我们都发现利用大肠杆菌对植物乳杆菌来源的胆盐水解酶进行异源表达时,它们很容易形成包涵体[22-24]。而其它来源的胆盐水解酶基因的异源表达则很少有产生包涵体的报道,这可能跟它们之间的基因差异有关。为了提高重组蛋白的可溶性,黄茜等利用MBP融合标签对BSH进行了融合表达,大幅提高了它的可溶性[25];战媛媛等则利用毕赤酵母表达系统实现了它的可溶性表达[26];我们利用双精氨酸途径的信号肽实现了它的胞外分泌表达,但表达量不高而且胞内还有包涵体[24]。
此外,为了使表达的BSH能作为一种治疗血清胆固醇过多症的药物或保健品使用,需对它进行食品级的表达,这意味着用于构建重组菌的所有基因工具(包括质粒载体,选择标记和转化方案等)应完全来自食品微生物。尽管Christiaens等[20]和Lambert等[21]将植物乳杆菌来源的bsh基因分别在野生菌L. plantarum 80和乳酸乳球菌中进行了克隆和表达。但他们所用的质粒含有抗生素抗性,因此不是完全食品级的表达。如何实现其食品级过量表达,也是目前亟需解决的问题。

表2 不同植物乳杆菌中胆盐水解酶基因的遗传信息Table 2 Genetic information of bsh genes from various strains of L. plantarum
1.3 胆盐水解酶的酶学性质研究
胆盐水解酶属于N-末端亲核水解酶家族,它们的N端都有半胱氨酸残基。在蛋白自动水解酶过程中,蛋氨酸脱除后,这个半胱氨酸就成为活性位点,这是 N-末端亲核水解酶家族的一个共同特征[7]。胆盐水解酶通常为胞内酶,最适反应pH在5和6之间,对氧气有些敏感。氧化巯基的试剂如高碘酸钠、碘乙酸、Hg2+和 Cu2+等能够强烈抑制胆盐水解酶的活性[24,33]。而DTT等还原剂能保护酶不被氧化[24]。这些都说明,胆盐水解酶N-末端半胱氨酸的巯基是催化反应的中心。
胆盐水解酶差异最大的特性是其底物特异性。胆盐水解酶的底物是甘氨结合胆盐和牛磺结合胆盐,它主要由类固醇核心和甘氨酸/牛磺酸脱水缩合形成的(图1)。这两类胆盐在人体内的比例为3:1[31]。Moser等和McAuliffe等的研究表明,胆盐水解酶先识别底物的类固醇核部分[34-35]。但是酶反应动力学的结果则显示BSH优先识别底物的氨基酸部位,并且大多数胆盐水解酶水解甘氨结合胆盐的能力大于水解牛磺结合胆盐的能力[7]。因此,关于胆盐水解对底物的识别位点还存在争议,它可能对类固醇核心和氨基酸基团都有识别作用。

图1 结合胆盐的化学结构Fig. 1 Chemical structure of conjugated bile salts
目前的研究也发现,植物乳杆菌来源的胆盐水解酶主要偏向于水解酶甘氨结合胆盐(表3)。而且大多数产胆盐水解酶的植物乳杆菌水解甘氨结合胆盐的能力也明显高于水解牛磺结合胆盐的能力。植物乳杆菌L. plantarum Lp-onlly对两种底物的水解能力相同,但这是由四种胆盐水解酶共同作用的结果。此外,进化拓宽了胆盐水解酶的底物特异性[31],使它们可以水解不同的底物。为了确定L. plantarum Lp-onlly中每一种胆盐水解酶的底物特异性,应该分别将它们进行异源表达,然后纯化。
胆盐水解酶的底物特异性差异跟它们的氨基酸序列以及空间结构有关。因此,可以从它们的氨基酸序列和蛋白结构出发来揭示它们结合底物的机制。目前信息理论法(information-theoretic approach)已广泛应用于寻找蛋白序列中决定底物特异性的氨基酸残基[36]。因为只用到蛋白的氨基酸序列,对于那些没有结构报道的蛋白来说,比如植物来源的胆盐水解酶,这种方法是研究它们的底物结合机制的不错选择。
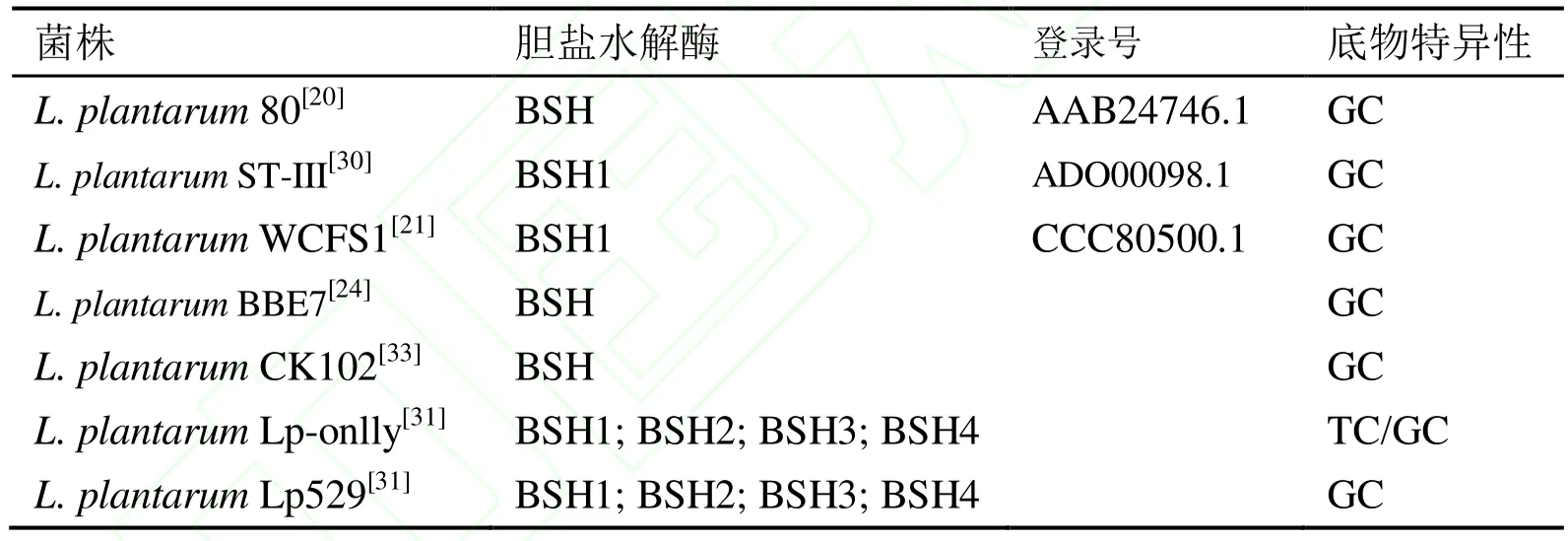
表3 不同植物乳杆菌或其胆盐水解酶的底物特异性Table 3 Substrate specificities of BSH proteins or different strains of L. plantarum
2 展望
综上所述,口服产胆盐水解酶的菌株或纯酶能显著降低哺乳动物体内的血清胆固醇水平,因此,可以用于治疗和预防血清胆固醇过多症。然而植物乳杆菌来源的胆盐水解酶的降胆固醇的机制、异源表达以及底物结合机制等方面还存在争议。以下几个方面的研究亟待加强:(1)确定益生菌的降胆固醇的机制,特别是BSH在其中所起到的作用。此外,使益生菌具有降胆固醇作用的有效剂量,以及益生元和益生菌的协同作用也待于进一步研究;(2)通过共表达分子伴侣,使用低拷贝的质粒以及与可溶性的标签融合表达等方法促进BSH的可溶性表达。而且还可以通过降低诱导温度等发酵工程的手段减少包涵体的形成;(3)利用食品级的表达元件,包括食品级的质粒、宿主、筛选标记、诱导剂以及转化方案等,实现BSH的食品级过量表达,为其应用奠定基础;(4)通过信息理论法等揭示BSH对底物的结合机制,以利于研究其水解产物的最终去向以及血清中次级胆盐含量的增高是否由它所引起的,从而确定BSH的确切功能;(5)开发高效降胆固醇的菌株或相关产品,并用它们来控制人体的血清胆固醇水平,这具有很大的应用潜力。
[1]Pereira D I A, Gibson G R. Cholesterol assimilation by lactic acid bacteria and bifidobacteria isolated from the human gut[J]. Appl Environ Microbiol, 2002, 68 (9): 4689.
[2]Jones M, Martoni C, Parent M, et al. Cholesterol-lowering efficacy of a microencapsulated bile salt hydrolase-active Lactobacillus reuteri NCIMB 30242 yoghurt formulation in hypercholesterolaemic adults[J]. Br J Nutr, 2012, 107 (10): 1505-1513.
[3]Agerbaek M, Gerdes L, Richelsen B. Hypocholesterolaemic effect of a new fermented milk product in healthy middle-aged men[J]. Eur J Clin Nutr, 1995, 49 (5): 346-352.
[4]Park Y H, Kim J G, Shin Y W, et al. Effects of Lactobacillus acidophilus 43121 and a mixture of Lactobacillus casei and Bifidobacterium longum on the serum cholesterol level and fecal sterol excretion in hypercholesterolemia-induced pigs[J]. Biosci Biotech Bioch, 2008, 72 (2): 595-600.
[5]Strompfová V, Marciňáková M, Simonová M, et al. Application of potential probiotic Lactobacillus fermentum AD1 strain in healthy dogs[J]. Anaerobe, 2006, 12 (2): 75-79.
[6]Kumar R, Grover S, Batish V K. Hypocholesterolaemic effect of dietary inclusion of two putative probiotic bile salt hydrolase-producing Lactobacillus plantarum strains in Sprague-Dawley rats[J]. Br J Nutr, 2011, 105 (4): 561-573.
[7]Begley M, Hill C, Gahan C G. Bile salt hydrolase activity in probiotics[J]. Appl Environ Microbiol, 2006, 72 (3): 1729-1738.
[8]Lewis S, Burmeister S. A double-blind placebo-controlled study of the effects of Lactobacillus acidophilus on plasma lipids[J]. Eur J Clin Nutr, 2005, 59 (6): 776-780.
[9]Moroti C, Magri L F S, De Rezende Costa M, et al. Effect of the consumption of a new symbiotic shake on glycemia and cholesterol levels in elderly people with type 2 diabetes mellitus[J]. Lipids Health Dis, 2012, 11 (1): 1-8.
[10]Zhang L, Zhang X, Liu C, et al. Manufacture of Cheddar cheese using probiotic Lactobacillus plantarum K25 and its cholesterol-lowering effects in a mice model[J]. World J Microb Biot, 2013, 29(1): 127-135.
[11]Ooi L-G, Liong M-T. Cholesterol-lowering effects of probiotics and prebiotics: a review of in vivo and in vitro findings[J]. Int J Mol Sci, 2010, 11 (6): 2499-2522.
[12]Ha C-G, Cho J-K, Lee C-H, et al. Cholesterol lowering effect of Lactobacillus plantarum isolated from human feces[J]. J Microbiol Biotechnol, 2006, 16 (8): 1201-1209.
[13]Jeun J, Kim S, Cho S-Y, et al. Hypocholesterolemic effects of Lactobacillus plantarum KCTC3928 by increased bile acid excretion in C57BL/6 mice[J]. Nutrition, 2010, 26 (3): 321-330.
[14]Nguyen T, Kang J, Lee M. Characterization of Lactobacillus plantarum PH04, a potential probiotic bacterium with cholesterol-lowering effects[J]. Int J Food Microbiol, 2007, 113 (3): 358-361.
[15]Fazeli H, Moshtaghian J, Mirlohi M, et al. Reduction in serum lipid parameters by incorporation of a native strain of Lactobacillus Plantarum A7 in Mice[J]. Iran J Diabetes Lipid Disord, 2010, 9: 1-7.
[16]Lanjar S, Djumali M, Anas M F, et al. Ability of Lactobacillus plantarum JR64 isolated from noni juice in lowering cholesterol in vivo[J]. Int J Sci Eng, 2011, 2 (1): 17-21.
[17]Naruszewicz M, Johansson M-L, Zapolska-Downar D, et al. Effect of Lactobacillus plantarum 299v on cardiovascular disease risk factors in smokers[J]. Am J Clin Nutr, 2002, 76 (6): 1249-1255.
[18]Ra I J L, Vuji I I F, Krinjar M, et al. Assimilation of cholesterol by some cultures of lactic acid bacteria and bifidobacteria[J]. Biotechnol Lett, 1992, 14 (1): 39-44.
[19]Tahri K, Grille J P, Schneider F. Bifidobacteria strain behavior toward cholesterol: coprecipitation with bile salts and assimilation[J]. Curr Microbiol, 1996, 33 (3): 187-193.
[20]Christiaens H, Leer R J, Pouwels P H, et al. Cloning and expression of a conjugated bile acid hydrolase gene from Lactobacillus plantarum by using a direct plate assay[J]. Appl Environ Microbiol,1992, 58 (12): 3792-3798.
[21]Lambert J M, Bongers R S, De Vos W M, et al. Functional analysis of four bile salt hydrolase and penicillin acylase family members in Lactobacillus plantarum WCFS1[J]. Appl Environ Microbiol,2008, 74 (15): 4719-4726.
[22]黄茜, 黄璐, 潘道东, 等. 植物乳杆菌 Lactobacillu plantarum Y1菌株胆盐水解酶基因(bsh)的克隆及重组表达[J]. 南京师大学报: 自然科学版, 2010, 33 (3): 91-96.
[23]战媛媛, 王长远, 于长青. 植物乳杆菌中胆盐水解酶基因的原核表达及纯化[J]. 中国生物制品学杂志, 2009, 22 (9): 876-879.
[24]Dong Z, Zhang J, Lee B H, et al. Secretory expression and characterization of a bile salt hydrolase from Lactobacillus plantarum in Escherichia coli[J]. J Mol Catal B-enzym, 2013, 93 (0): 57-64.
[25]黄茜, 黄璐, 潘道东, 等. 重组融合蛋白MBP-BSH在大肠杆菌中的表达及其纯化, 功能鉴定[J]. 食品科学, 2012, 33 (7): 198-203.
[26]于长青, 战媛媛, 王长远. 胆盐水解酶基因在毕赤酵母中的表达[J]. 中国生物制品学杂志,2010, 23 (9): 949-952.
[27]Kumar R, Grover S, Mohanty A K, et al. Molecular cloning and sequence analysis of bile salt hydrolase gene (bsh)from Lactobacillus plantarum MBUL90 strain of human origin[J]. Food Biotechnol, 2010, 24 (3): 215-226.
[28]Dong Z, Zhang J, Lee B, et al. A bile salt hydrolase gene of Lactobacillus plantarum BBE7 with high cholesterol-removing activity[J]. Eur Food Res Technol, 2012, 235 (3): 419-427.
[29]李蓉, 于长青. 植物乳杆菌中胆盐水解酶结构基因的克隆与序列分析[J]. 黑龙江八一农垦大学学报, 2011, 23 (3): 55-57.
[30]Ren J, Sun K, Wu Z, et al. All 4 bile salt hydrolase proteins are responsible for the hydrolysis activity in Lactobacillus plantarum ST-III[J]. J Food Sci, 2011, 76 (9): M622-628.
[31]Jiang J, Hang X, Zhang M, et al. Diversity of bile salt hydrolase activities in different Lactobacilli toward human bile salts[J]. Ann Microbiol, 2010, 60 (1): 81-88.
[32]赵时玮, 任婧, 王荫榆, 等. 植物乳杆菌JPP2胆盐水解酶相关基因克隆和表达[J]. 工业微生物, 2012, 42 (1): 34-38.
[33]Ha C G, Cho J K, Chai Y G, et al. Purification and characterization of bile salt hydrolase from Lactobacillus plantarum CK 102[J]. J Microbiol Biotechnol, 2006, 16 (7): 1047-1052.
[34]Moser S A, Savage D C. Bile salt hydrolase activity and resistance to toxicity of conjugated bile salts are unrelated properties in lactobacilli[J]. Appl Environ Microbiol, 2001, 67 (8): 3476-3480.
[35]Mcauliffe O, Cano R J, Klaenhammer T R. Genetic analysis of two bile salt hydrolase activities in Lactobacillus acidophilus NCFM[J]. Appl Environ Microbiol, 2005, 71 (8): 4925-4929.
[36]Yip K Y, Patel P, Kim P M, et al. An integrated system for studying residue coevolution in proteins[J]. Bioinformatics, 2008, 24 (2): 290-292.
