两品系小鼠食物过敏模型的比较
2014-05-10赵新凤曾本华谭毅魏泓程茜
赵新凤,曾本华,谭毅,魏泓,程茜
(1.重庆医科大学附属儿童医院,儿童发育疾病研究省部共建教育部重点实验室,儿科学重庆市重点实验室,重庆市儿童发育重大疾病诊治与预防国际科技合作基地,重庆 400014; 2.第三军医大学基础部实验动物学教研室,重庆 400038;3.重庆医科大学实验动物学中心,重庆 400016)
研究报告
两品系小鼠食物过敏模型的比较
赵新凤1,曾本华2,谭毅3,魏泓2,程茜1
(1.重庆医科大学附属儿童医院,儿童发育疾病研究省部共建教育部重点实验室,儿科学重庆市重点实验室,重庆市儿童发育重大疾病诊治与预防国际科技合作基地,重庆 400014; 2.第三军医大学基础部实验动物学教研室,重庆 400038;3.重庆医科大学实验动物学中心,重庆 400016)
目的比较两种不同品系小鼠食物过敏模型的敏感性和肠道菌群变化的差异,旨在为食物过敏模型的建立提供依据。方法分别对30只4~5周龄BALB/c和KM雌鼠用卵清蛋白(ovalbumin,OVA)致敏建立食物过敏模型,ELISA法检测小鼠血清OVA特异性IgE水平;HE染色观察空肠组织形态;采用DGGE技术检测粪便菌群的变化。结果(1)30只致敏的BALB/c小鼠中有27只血清OVA特异性IgE水平明显升高(P<0.001),而30只致敏的KM小鼠中有21只,且BALB/c小鼠空肠绒毛炎症细胞浸润、上皮脱落及坏死比KM小鼠明显;(2)食物过敏造模后,BALB/c小鼠肠道菌群的改变明显(P<0.001),而KM小鼠中仅有均匀度改变显著(P<0.05);(3) BALB/c小鼠和KM小鼠对照组肠道菌群的丰富度、Shannon指数及均匀度都有差异。结论BALB/c小鼠对OVA的敏感性高于KM小鼠,不同品系小鼠肠道菌群结构不同,OVA处理后,BALB/c小鼠菌群的改变比KM小鼠更明显。
小鼠;食物过敏;模型;卵蛋白;敏感性;肠道菌群
食物过敏已成为威胁人类健康的主要问题,尤其在发达国家,其发病率在成人为3%、儿童为6%[1,2]。然而,食物过敏的发病机制仍不明确,需要合适的食物过敏模型以便于更深入的研究。目前已有的BALB/c小鼠、C57小鼠模型敏感性太高,不符合人群的发病规律,且近交系个体之间相同的遗传组成与人类个体之间的遗传异质性差异很大。因此本实验建立两种不同品系小鼠的食物过敏模型,以评价封闭群KM小鼠和近交系BALB/c小鼠在食物过敏模型中的敏感性和了解肠道菌群变化的差异性,旨在建立与人发病规律更接近的过敏模型,为食物过敏与肠道菌群关系的研究提供参考。
1 材料和方法
1.1 实验动物
SPF级雌性BALB/c和KM小鼠各40只,4~5周龄,体重15.5~17 g,由第三军医大学实验动物中心提供【SCXK(渝)2007-0002】,实验地点在第三军医大学实验动物中心【SYXK(渝)2012-0002】。喂饮条件为:温度20~25℃,12 h/12 h昼夜规律照明,自由饮水与进食。以特殊饲料(不含鸡蛋)饲养,饲料基本成分均按规定的实验动物营养需要量配制,具体如下:
动物饲料配方(每50 kg含量):玉米12.5 kg、麦麸5 kg、豆粨7.5 kg、次粉10 kg、鱼粉7 kg、骨粉1 kg、酵母粉2.5 kg、植物油0.75 kg、动物油0.25 kg、盐0.5 kg、鱼肝油0.5 kg、维生素E 0.25 kg、复合维生素80 g、水4 L。
1.2 方法
1.2.1 动物分组
每品系40只雌鼠,随机分成实验组和对照组,实验组30只、对照组10只。实验组小鼠经肠道激发致敏,对照组给予等量生理盐水,于实验第28天末次激发2 h内处死小鼠,留取标本。
1.2.2 动物模型的建立
[3]方法。基础致敏:实验的第1天经腹腔注射给予含10μg OVA(V级,Sigma公司)和1 mg氢氧化铝(Sigma公司)的生理盐水,每只0.5 mL。强化致敏:于实验第15天经腹腔注射含10μg OVA (V级,Sigma公司)的生理盐水,每只0.5 mL。肠道激发:于实验第20天起至第28天,用500 mg/L OVA (II级,Sigma公司)溶液隔日进行灌胃,每只0.2 mL。1.2.3 血清OVA特异性IgE水平的检测
采用ELISA法。血清OVA特异性IgE水平检测:末次激发后2 h内经眼球取血,分离血清,以10 μg/mL OVA包板,每孔100μL,1%牛血清白蛋白封闭,每孔200μL;加入血清样品(1∶1)稀释,每孔100 μL;加入HRP标记的羊抗小鼠IgE(1∶1000稀释) (Southern Biotechnology Associates),每孔100μL; TMB(凯基生物)显色后终止反应,使用全自动酶标仪(BIO-RAD,USA)在波长450 nm处检测A值。
1.2.4 肠道组织学观察
处死小鼠,取0.5 cm空肠固定,苏木素-伊红染色,镜下观察空肠绒毛组织形态。
1.2.5 粪便菌群检测
于末次灌胃前收集小鼠粪便,进行denaturing gradient gel electrophoresis(DGGE)分析。
(1)粪便菌群总DNA的提取:样品置于2 mL螺口管(Biospec)中,加入700μL裂解液、250μL酚/氯仿/异戊醇(25∶24∶1)和0.3 g锆珠,混匀,用Mini-Beadbeater-16(Biospec,USA)研磨 2 min, 12 000 r/min离心 5 min,取上清,加入 250μL 10 mol/L乙酸铵,然后使用2次酚/氯仿/异戊醇和氯仿抽提,异丙醇沉淀,70%的乙醇洗涤,风干后,加入50μL去离子水和2μL DNA free-RNAase(生工生物),37℃孵育15 min。取2μL的DNA溶液于NanoDrop ND-1000核酸定量仪中测定其DNA含量和纯度。
(2)PCR扩增:扩增16S rRNA基因V6-V8区引 物 为 U968F(5’-CGCCCGCCGCGCGCGGCGGGCGGGCGGGGCGGGGGCACGGGGGGAACGC GAAGAACCTTA-3’),L1401R(5’-CGGTGTGTACAAGACCC-3’)。扩增体系:25μL,包括12.5μL 2 PCR MasterMix,0.5μL 10 mol/L的引物,1μL 100 ng/μL的DNA模板,10.5μL的ddH2O。反应条件:94℃5 min,94℃30 s,56℃30 s,72℃1 min,30个循环;72℃7 min。
(3)DGGE分析:PCR产物用DGGE进行分析,采用Bio-Rad公司DcodeDGGE系统,做法参照Rowan等方法。DGGE使用8%聚丙烯酰胺凝胶,采用45%~58%变形梯度。首先在220 V电压下预电泳10 min,随后85 V电泳16 h。电泳结束后,进行硝酸银染色。DGGE凝胶采用Quantity One进行分析。
1.3 统计学处理
应用SPSS 17.0统计软件进行数据处理。实验数据以均数±标准差(±s)表示,组间比较用One-way ANOVA方差分析,Student-Newman-Keuls检验,以P<0.05为差异有统计学意义。
2 实验结果
2.1 血清OVA特异性IgE水平
ELISA法测定血清OVA特异性IgE水平,以X +2SD为标准,大于对照组X+2SD为过敏组,小于对照组X+2SD为无反应组。结果显示,KM对照组OVA-sIgE(0.45±0.12)和BALB/c对照组(0.69± 0.17)差异无显著性(P>0.05),实验组血清中的OVA-sIgE与对照组相比,30只BALB/c中有27只(2.10±0.64)大于对照组(0.69±0.17)两倍的SD (P<0.001),而30只KM中有21只(2.00±0.67)大于对照组(0.45±0.12)两倍的SD(P<0.001)。
2.2 肠道组织学变化
空肠组织切片HE染色(见图1,彩插4)可见BALB/c小鼠和KM小鼠实验组的空肠绒毛上皮细胞局灶坏死、脱落,炎性细胞浸润,而BALB/c小鼠实验组更严重。
2.3 肠道菌群结构的变化
2.3.1 DGGE图谱
由图2可得,BALB/c和KM小鼠各组之间既有共有条带又有差异条带,即使是同一位置的条带,明暗程度也各有不同。如条带2和5在各组中是共有的,而条带1只在BALB/c过敏组明显可见,条带7在BALB/c对照组中可见,可见食物过敏造模使肠道菌群的结构发生了改变。条带6存在于KM小鼠中,而在BALB/c小鼠中不明显,说明不同品系的小鼠肠道菌群的结构也不同。
2.3.2 16SrRNA基因V6-V8区PCR-DGGE指纹图谱的条带分析及相似性UPGMA聚类分析
(1)UPGMA聚类分析:对V3-DGGE指纹图谱进行多样性指数分析(见表1),结果表明:BALB/c小鼠的过敏组和对照组比较,丰富度、Shannon指数和均匀度均有明显差异(P<0.001);而KM小鼠的过敏组和对照组相比,只有均匀度差异有显著性(P<0.05)。BALB/c小鼠的对照组和KM小鼠的对照组比较,丰富度(P<0.001)、Shannon指数(P<0.001)和均匀度(P<0.05)差异有显著性。

注:泳道上方数字为泳道编号。1~6号泳道为BALB/c过敏组,7~12号泳道为BALB/c对照组,13~18号泳道为KM对照组组, 19~24号泳道为KM过敏组。图2 BALB/c和KM各组小鼠肠道菌群V6-V8区DGGE图谱Note:1-6:BALB/c food allergy groups,7-12:BALB/c control groups,13-18:KM control groups,19~24:KM food allergy groupsFig.2 V6-V8 DGGE profiles of the intestinal flora between the BALB/c and KM mice
(2)多样性分析:DGGE图谱的聚类分析(图3)显示各组之间的总相似度为13%。BALB/c过敏组1-6聚成一簇,其余几组也大部分分别独成一簇,说明各组之间的肠道菌群结构存在差异。结果提 示,小鼠的聚类与品系和食物过敏造模明显相关。

表1 BALB/c和KM小鼠对照组与过敏组V6-DGGE指纹图谱的多样性指数分析Tab.1 Diversity indexes of the V6-DGGE profile between control and FA groups of BALB/c and KM mice

图3 BALB/c和KM小鼠对照组和过敏组粪便菌群PCR-DGGE图谱聚类分析Fig.3 Dendrogram of PCR-DGGE profiles of the microbiota in the control and FA groups of BALB/c and KM mice
2.3.3 DGGE指纹图谱的PCA分析
对各组DGGE指纹图谱进行PCA分析,根据各样品在第一和第二主成分的分值绘制PCA图(图4)。X坐标表示第一主成分(48.551%),Y坐标表示第二主成分(27.306%)。图中可以看出,各组绝大部分在PCA分析图中分离,其中BALB/c和KM过敏组有所聚拢。
3 讨论
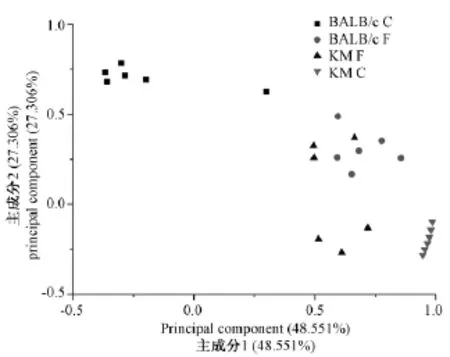
图4 BALB/c和KM小鼠对照组和过敏组粪便菌群PCR-DGGE图谱PCA分析Fig.4 PCA analysis of the PCR-DGGE profiles in the control and FA groups of BALB/c and KM mice
国际上目前仍未建立标准的食物过敏模型,常用的动物种类和品种有很多,但国内最常用的是BALB/c小鼠。然而BALB/c小鼠作为食物过敏动物模型的可行性一直存在争议[4,5]。Deman等[5]认为BALB/c是合适的动物模型,吕相征[6]等研究发现BALB/c小鼠对无致敏史的食物也产生过敏反应,与人体过敏反应的情况差异较大,指出BALB/c小鼠不适合作为食物过敏动物模型。鸡蛋蛋白是最常见食物过敏原,而OVA是鸡蛋蛋白的主要成分,因此本实验食物过敏模型采用 OVA激发致敏。OVA致敏小鼠的食物过敏以IgE介导的速发型超敏反应为主,致敏小鼠出现特异性的IgE水平升高,空肠绒毛破损、坏死和炎性细胞浸润,证实IgE介导的食物过敏动物模型建立成功。本研究中致敏的30只BALB/c小鼠血清OVA特异性IgE的水平有27只明显高于对照组,而KM小鼠中仅有21只和对照组有显著差异;并且BALB/c小鼠小肠的组织形态学改变要明显严重于KM小鼠,说明了BALB/c小鼠对OVA的敏感性高于KM小鼠。这可能与BALB/c小鼠是近交系小鼠,其基因背景一致有关。正是因为BALB/c小鼠的敏感性高,所以常被用于食物过敏的造模。而KM小鼠基因背景可能与人类更相似,各个小鼠间遗传背景差异大,敏感性不可能完全一致,可以更直实地反映OVA致敏时所造成的机体过敏反应状态。故我们认为,在建立食物过敏模型时,应根据不同的实验目的选用不同的小鼠品系或品种。人类在接触相同的食物时一小部分人会出现过敏现象,而大部分人正常,这可能是遗传和环境共同作用的结果。通过OVA致敏后过敏小鼠和无反应小鼠之间的差异来研究食物过敏与肠道菌群作用,相对敏感性低的小鼠模型比较适合。
通过比较BALB/c和KM小鼠对照组的肠道菌群结构,可以看出不同品系的小鼠其肠道菌群结构不同。Campbell等[7]研究了不同品系小鼠的肠道菌群发现,小鼠的品系与肠道菌群显著相关,与我们的研究结果相似。张晓婧等[8]的研究也指出不同遗传背景的小鼠对肠道菌群具有选择性的定植效果。BALB/c小鼠过敏组和对照组肠道菌群的丰富度、Shannon指数和均匀度均有显著差异,而KM小鼠过敏组和对照组肠道菌群也有不同,但仅有均匀度的差异有统计学意义。这说明OVA致敏后两种品系小鼠的肠道菌群有所改变,但改变的程度不同, BALB/c小鼠菌群改变的幅度更大,PCA分析发现虽然KM和BALB/c差异很大,但过敏发生时,菌群聚拢,提示菌群的改变相似。食物过敏的发病机制还不明确,许多动物实验都支持食物过敏与肠道菌群相关[9,10],人群研究也显示食物过敏儿童和正常儿童相比,其肠道菌群不同[11,12],肠道菌群在食物过敏中的重要性正在被揭示。KM与BALB/c小鼠在食物过敏状态下所表现的发病差异,以及不同品系间小鼠食物过敏后肠道菌群的不同,将可能对研究食物过敏的机制提供新的思路和动物模型。
(本文图1见彩插4。)
参考文献
[1] Eigenmann PA,Beyer K,Wesley Burks A,et al.New visions for food allergy:an iPAC summary and future trends[J].Pediatr Allergy Immunol,2008,19(19):26-39.
[2] Sicherer SH,Sampson HA.Food allergy[J].JAllergy Clin Immunol,2010,125:116-125.
[3] 白枫,黎海芪.黄褐毛忍冬皂苷对卵清蛋白致敏小鼠的抗过敏作用[J].第四军医大学学报,2008,29(15):1395-1398.
[4] Akiyama H,Teshima R,Sakushima JI,et al.Examination of oral sensitization with ovalbumin in Brown Norway rats and three strains ofmice[J].Immunol Lett,2001,78(1):1-5.
[5] Dearman RJ,Kimber I.Determination of protein allergenicity: studies in mice[J].Toxicol Lett,2001,120(1-3):181-186.
[6] 吕相征,刘秀梅,郭云昌,等.BALB/c小鼠食物过敏动物模型的实验研究[J].卫生研究,2005,34(2):211-213.
[7] Campbell JH,Foster CM,Vishnivetskaya T,et al.Host genetic and environmental effects on mouse intestinal microbiota[J]. ISME J,2012,6(11):2033-2044.
[8] 张晓婧,曾本华,刘智伟.两种不同品系小鼠的人源菌群模型的建立与肠道菌群的比较[J].中国微生态学杂志,2013, 25(4):376-380.
[9] Sonoyama K,Ogasawara T,Goto H,et al.Comparison of gut microbiota and allergic reactions in BALB/c mice fed different cultivars of rice[J].Br JNutr,2010,103(2):218-226.
[10] Inoue R,Tsukahara T,Ueno K,et al.Possible link of a compositional change in intestinalmicrobiotawith the anti-allergic effect of fructo-oligosaccharides in NC/jic mice[J].Biosci Biotechnol Biochem,2010,74(9):1947-1950.
[11] Watanabe S,Narisawa Y,Arase S,et al.Differences in fecal microflora between patients with atopic dermatitis and healthy control subjects[J].J Allergy Clin Immunol,2003,111(3): 587-591.
[12] Bjorksten B,Sepp E,Julge K,et al.Allergy development and the intestinalmicroflora during the first year of life[J].JAllergy Clin Immunol,2001,108(4):516-520.
Com parison of the BALB/c and Kunm ing mousemodels of food allergy
ZHAO Xin-feng1,ZENG Ben-hua2,TAN Yi3,WEIHong2,CHENG Qian1
(1.Key Laboratory of Child Development and Disorders of Ministry of Education,Chongqing Key Laboratory of Pediatrics,Chongqing International Science and Technology Cooperation Center for Child Development and Disorders,Children’s Hospital of Chongqing Medical University,Chongqing 400014,China; 2.Department of Laboratory Animal Science,College of Basic Medical Sciences,Third Military Medical University, Chongqing 400038;3.Laboratory Animal Center,Chongqing Medical University,Chongqing 400016)
ObjectiveIn order to provide the basis for establishment of food allergy models,we compared the differences of sensitivity and alterations of intestinal flora of food allergy models in two strains ofmice.M ethodsForty 4-5-week old female BALB/c and Kunmimgmice were divided into control group(n=10)and food allergy goup(n= 30),respectively.Ovalbumin(OVA)was injected to themice to establish food allergymodels.Serum OVA-specific IgE of themice was assayed by ELISA.The jejunum tissue was examined by pathology with HE staining.The changes of fecal flora were detected by denaturing gradient gel electrophoresis(DGGE).Results(1)Among the sensitized 60 mice, OVA-sIgE levels were significantly increased in 27/30 BALB/c mice and 21/30 KM mice compared with those of control groups(P<0.001).Moreover,thereweremore evident inflammatory cell infiltration,epithelial cell shedding and cytolysis in the jejunal villiof BALB/cmice than those of KM mice.(2)After food allergymodeling,therewere significant changes of intestinal flora in the BALB/c mice(P<0.001),while only significant change of evenness was found in the KM mice(P<0.05).(3)There were changes of abundance,Shannon index and evenness of intestinal flora in themodel groups of BALB/c and KM mice.ConclusionsBALB/cmice aremore sensitive to OVA allergy than KM mice.The composition of intestinal flora is different among different strains ofmice.The changes of intestinal flora after OVA challenge in BALB/c mice aremore obvious than those in KM mice.
Mice;Food allergy;Models;Ovalbumin;Sensitivity;Intestinal flora
Q95-33,R378
A
1005-4847(2014)03-0035-05
10.3969/j.issn.1005-4847.2014.03.007
2013-11-19
国家自然科学基金(81370906);“973”计划(2013CB531406)。
赵新凤(1987-),女,重庆医科大学儿童医院儿童保健科硕士研究生,从事食物过敏与肠道菌群的研究。
程茜。E-mail: chqq5@126.com
