新德里金属β内酰胺酶-1序列和抗药性的生物信息学分析
2014-04-23程琪温权胡小林徐真昊廖智宇赵一雷
程琪,温权,胡小林,徐真昊,廖智宇,赵一雷,
(1. 上海交通大学生命科学技术学院,微生物代谢国家重点实验室,上海,200240;2. 上海中学,上海,200231)
近年来,β-内酰胺类抗生素(beta-lactam antibiotics)在临床及农业上的广泛应用,是细菌的耐药性不断增强的原因之一,其中产生β-内酰胺酶(beta-lactamase)水解该类抗生素是细菌耐药的重要机制之一[1]。根据Ambler分类法,β-内酰胺酶分为A/B/C/D四类,其中,A/C/D三类通过活性位点的丝氨酸残基来催化水解;而B类β-内酰胺酶为金属β-内酰胺酶(MBL, metallo-beta-lactamase),其活性位点需要一个或两个金属离子协助催化水解[2]。MBL可以催化水解除单环外的所有β-内酰胺酶,且对常规的β-内酰胺酶抑制剂有抵制作用。金属酶编码的耐药基因位于细菌染色体、质粒或者转座子上,并以基因盒的形式存在于整合子中。整合子属于可移动基因元件,可借助整合子的移动、基因盒的插入、切除而导致细菌间耐药性呈水平传播,同时,基因盒与整合子随着基因环境的改变也将不断进化,产生新的耐药方式,给临床抗菌治疗带来严峻的挑战[3]。
新德里金属β-内酰胺酶-1(NDM-1, New Delhi metallo-betalactamase)最早于2009年从肺炎克雷伯菌的瑞典患者中分离得到[4],其催化活性需要一个或两个锌离子的协助,属于Bush分类中B1类MBL[5,6]。通过对其三维结构的研究发现,其骨架具有α/β/β/α折叠特征[7]。除替加环素和多粘菌素,NDM-1能够水解几乎所有抗生素,药物动力学实验表明,NDM-1相比其他大部分NDM-1具有更强的水解活性。NDM-1编码基因主要携带在不同质粒上,能够在敏感菌株中自由转移[8]。
本文先利用生物信息学的方法,从NDM-1与其它金属β-内酰胺酶同源性较低入手,对NDM-1序列进行分析,为NDM-1的低同源性和广谱耐药性由于基因水平转移过程中造成基因特性改变的猜想提供佐证。然后利用计算机分子模拟手段,对NDM-1与替加环素进行对接,分析替加环素抑制NDM-1的分子机制,为NDM-1抑制剂的合理设计奠定基础。
2 材料与方法
2.1 实验材料
金属-β-内酰胺酶氨基酸和编码基因序列信息来自于生物信息数据库NCBI。首先将NDM-1的氨基酸序列进行BLAST比对,结合文献[9]中MBL的种类和来源,在BLAST打分较高的序列中筛选氨基酸序列进行后续研究。在NCBI数据库中,下载所有待研究MBL的编码基因序列,用于基因序列分子进化树的构件分析。
NDM-1 [Klebsiella pneumoniae]和YP_458405.1[Erythrobacter litoralis HTCC2594] 编码基因及其上下游1000bp基因信息均来自生物信息数据库NCBI。分别下载Klebsiella pneumoniae strain 601 plasmid pNDM-OM(NCBI编号:NC_019889.1)和Erythrobacter litoralis HTCC2594 chromosome(NCBI编号:NC_007722.1)的基因序列,找到NDM-1和bl2编码基因的位置,选取其上下游1000bp的基因序列,进行GC含量分析。
用于与药物分子替加环素对接的蛋白Apo-NDM-1的立体结构3S0Z-A来自蛋白质三维结构PDB数据库。替加环素药物分子由Sybyl-x软件绘制得到。
2.2 实验方法
2.2.1多序列比对与分子进化树构建
本文构建氨基酸和基因序列进化树的方法是序列相似性比较,利用Mega软件[10]采用NJ邻接法对上述所得金属-β-内酰胺酶的氨基酸和基因序列进行多序列比对,生成分子进化树文件,并修改注释信息,生成环状系统树图像。
2.2.2GC含量分析
在R软件的seqinr程序包[11]中,计算NDM-1 [Klebsiella pneumoniae]和YP_458405.1 [Erythrobacter litoralis HTCC2594]编码基因及其上下游1000bp基因的GC含量,滑动窗口为100bp,每次向右滑动1bp。以编码基因开始的位置为横轴0点,得到GC含量随基因位置变化图。
2.2.3替加环素与NDM-1分子对接
选用3S0Z-A蛋白的结构文件,使用pymol软件包,去掉蛋白质中的小分子和水,再利用Sybyl-x软件绘制替加环素分子。结合文献报导以及CASTp网站工具[12]预测得到的3S0Z-A的活性位点,将替加环素分子移动到3S0Z-A预测作用位点。将对接文件读入Autodock软件中,确定小分子构象变化的范围,进行vina批处理,生成最优结合方式的文件以及其结合能。最后用DS软件读入对接好的文件,选定小分子为中性,显示与其相距5.00Å的蛋白的部分,此范围为可能形成氢键的范围,统计与替加环素分子相距5.00Å的氨基酸和该位置出现潜在氢键的构象个数。
3 结果与讨论
3.1 氨基酸序列的分子进化树构建
使用Mega软件对金属β-内酰胺酶族的氨基酸序列进行多序列比对,构建分子进化树并查看环状无根树结构图像。分子进化树共分为3个主枝,分别对应为Bush分类中的三大类。与NDM系列分子进化关系最近的MBL为3个分类并不明确的bl2,分别为YP_003061440.1[Hirschia baltica ATCC 49814],YP_003270512.1[Haliangium ochraceum DSM 14365],YP_458405.1[Erythrobacter litoralis HTCC2594],Hirschia baltica属于红细菌属,Erythrobacter litoralis属于赤杆菌属,Haliangium ochraceum是来源于海洋的一类粘细菌,三种细菌均来自于海洋。
蛋白质的序列差异关系所构建的分子进化树,给出分支层次或拓扑图形,是产生新的基因复制或享有共同祖先的生物体的歧异点的一种反映,通过蛋白质的分子进化树分析,为从分子水平研究蛋白质进化关系。金属-β-内酰胺酶族的氨基酸序列的分子进化树显示NDM-1与来自海洋细菌的MBL具有在功能上具有较近的距离。因此,从分子进化的角度来看,NDM-1与YP_003061440.1, YP_003270512.1, YP_458405.1具有较高的同源性,而与其他MBL同源性较低。这个现象启示我们NDM-1可能在遗传上与来自海洋细菌的MBL有着密不可分的关系。
3.2 基因序列的分子进化树构建
使用Mega软件对金属-β-内酰胺酶族的基因序列进行多序列比对,构建分子进化树并查看无根树结构图像,发现MBL的基因序列分子进化树与氨基酸序列的分子进化树并不完全一致,改变最为明显的为NDM系列与三种分类不明确的bl2。与NDM系列分子进化关系最近的MBL基因,其编码蛋白是YP_458405.1[Erythrobacter litoralis HTCC2594],另两个与NDM系列功能进化关系较近的M B L,即YP_003061440.1[Hirschia baltica ATCC 49814],YP_003270512.1[Haliangium ochraceum DSM 14365],其基因序列的进化关系较远。
在亲缘关系较远的物种间某个特定基因序列有较高相似性,一般可以作为基因水平转移的初始证据或怀疑对象。虽然进化树也有其本身的缺陷,用于构建进化树的序列信息并不能准确的反映所有物种之间的进化关系,但进化树法仍是检测基因水平转移行之有效的方法之一。根据我们所得到的基因序列分子进化树,与NDM编码基因分子进化关系最近的MBL编码基因为YP_458405.1[Erythrobacter litoralis HTCC2594]编码基因,来源物种为海洋细菌,在物种进化关系上与肺炎克雷伯菌和大肠杆菌等细菌关系较远,这一结果为NDM-1与基因水平转移相关提供佐证。
3.3 GC含量分析
计算目标基因序列及其上下游1000bp序列的GC含量,取编码基因起始的地方为x轴的0值,发现NDM-1上游100~350bp的GC含量异常,出现较大波动,由60%左右下滑至越35%。与YP_458405.1对比来看,NDM-1与其在编码区段GC含量具有较高的吻合度,平均在63%附近,并且波动正常,而在非编码区段的上下游1000bp内GC含量差别较大,并且没有直观的联系。
原核生物不同细菌物种之间基因的GC含量不同,而每个细菌物种基因组GC含量相对来说是较稳定的,不受外界因素影响,某段特定DNA序列的GC含量明显高于或低于其基因组的其他部分或者出现较为异常的波动可以作为推断其是否发生基因水平转移的依据。NDM-1上游GC含量的剧烈波动以及其与YP_458405.1编码基因区段GC含量的高度吻合,在一定程度上证明了基因水平转移的可能性。
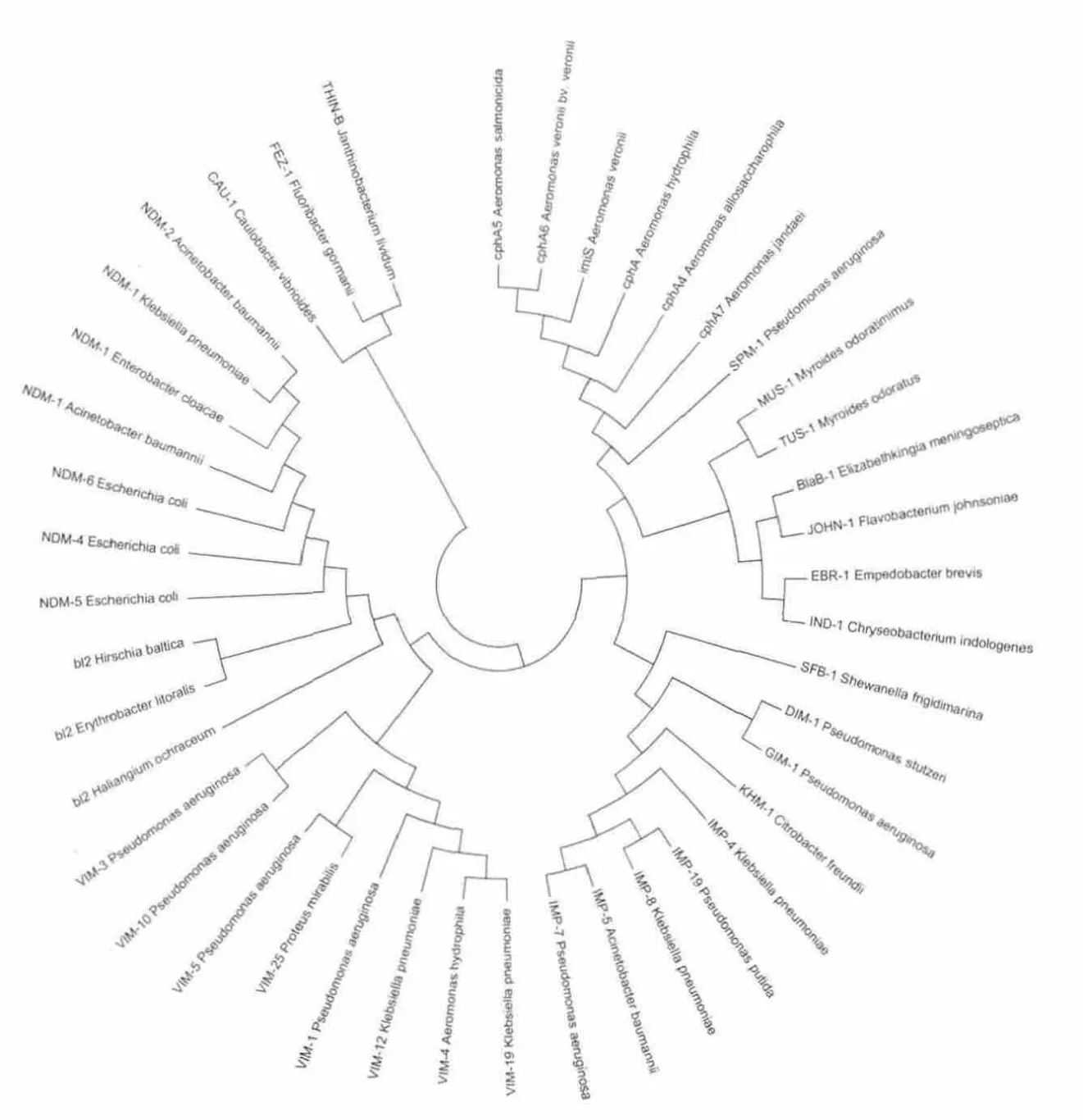
图1金属-β-内酰胺酶氨基酸序列的分子进化树Fig.1Phylogenetic tree of metallo-β-lactamase amino acid sequence
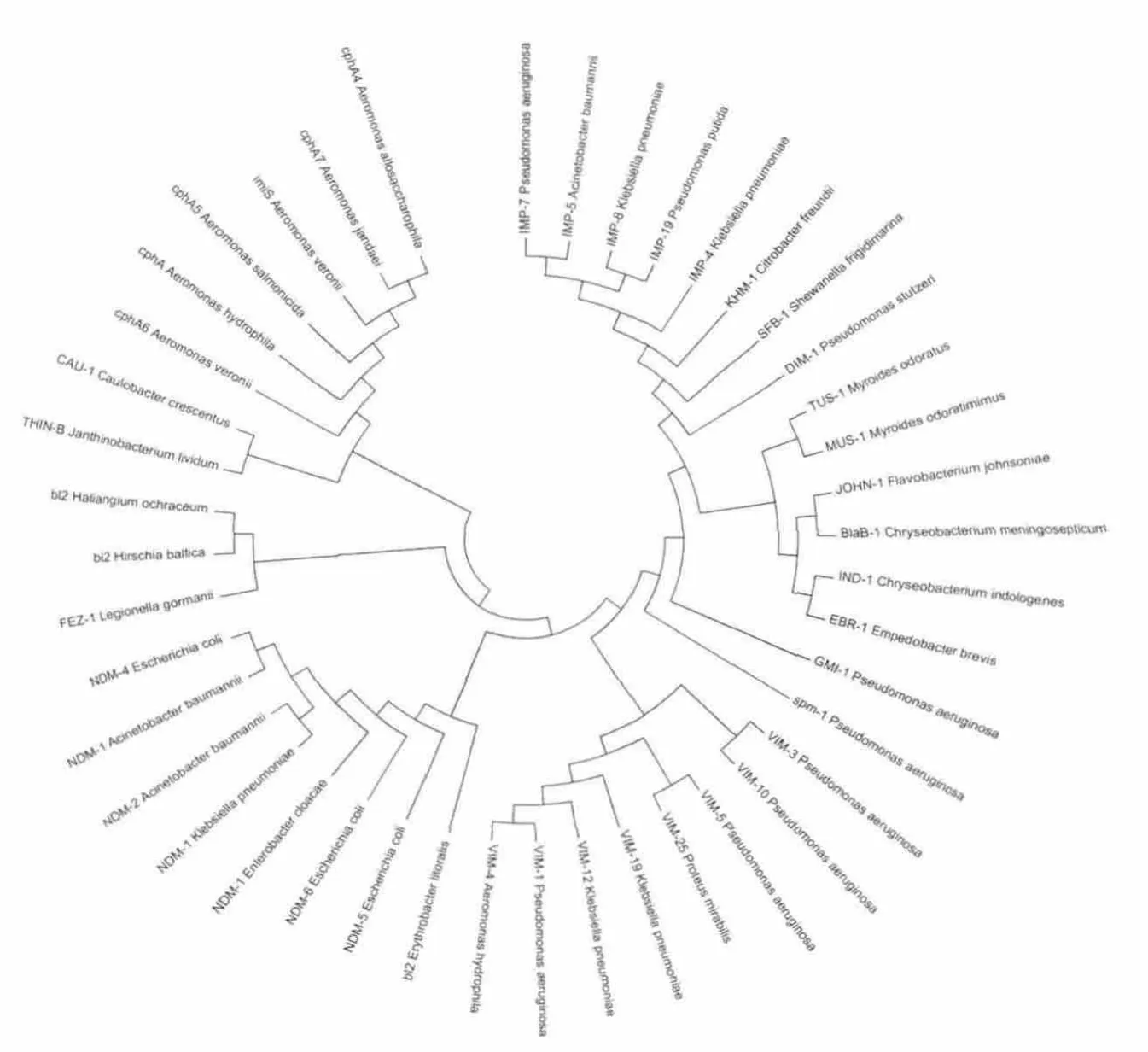
图2金属β-内酰胺酶基因序列的分子进化树Fig.2Phylogenetic tree of metallo-β-lactamase gene sequence
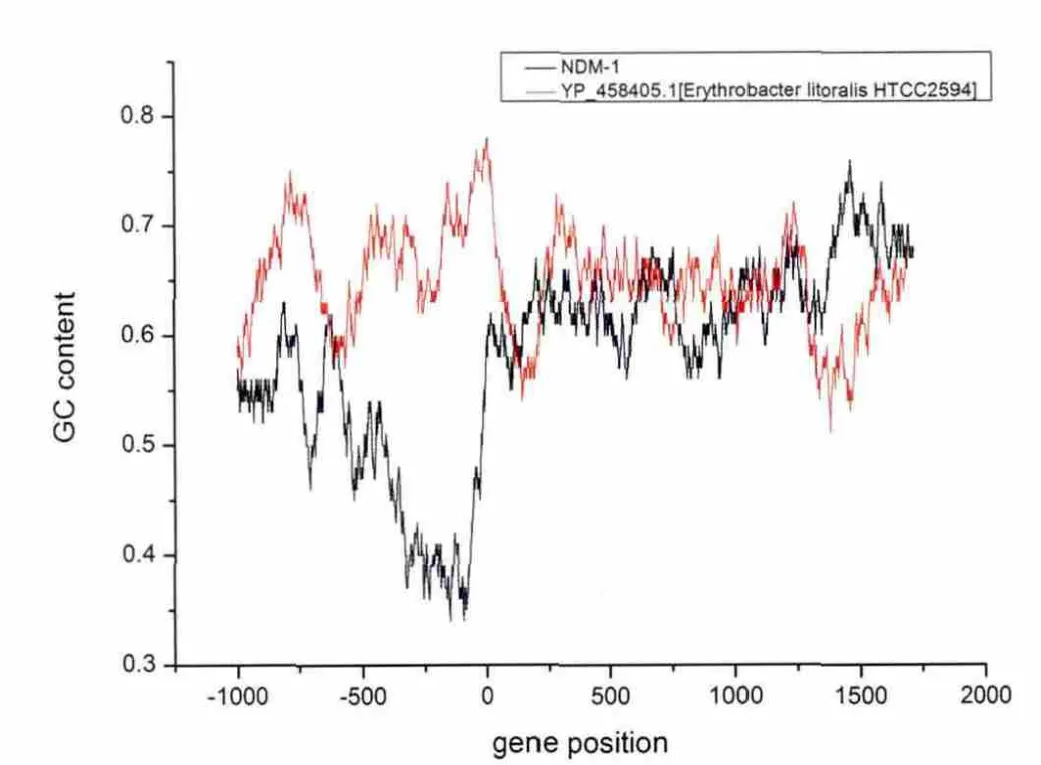
图3NDM-1与YP_458405.1编码基因的GC含量Fig.3GC content of NDM-1 and YP_458405.1 encoding gene
3.4 NDM-1与替加环素对接分析
利用Autodock软件得到9种最优的结合方式(表1),其中最优构象如图4所示。在Discovery studio软件中显示与替加环素分子相距5.00Å的蛋白部分,记录对应的氨基酸和该位置出现潜在氢键的构象个数(表2)。
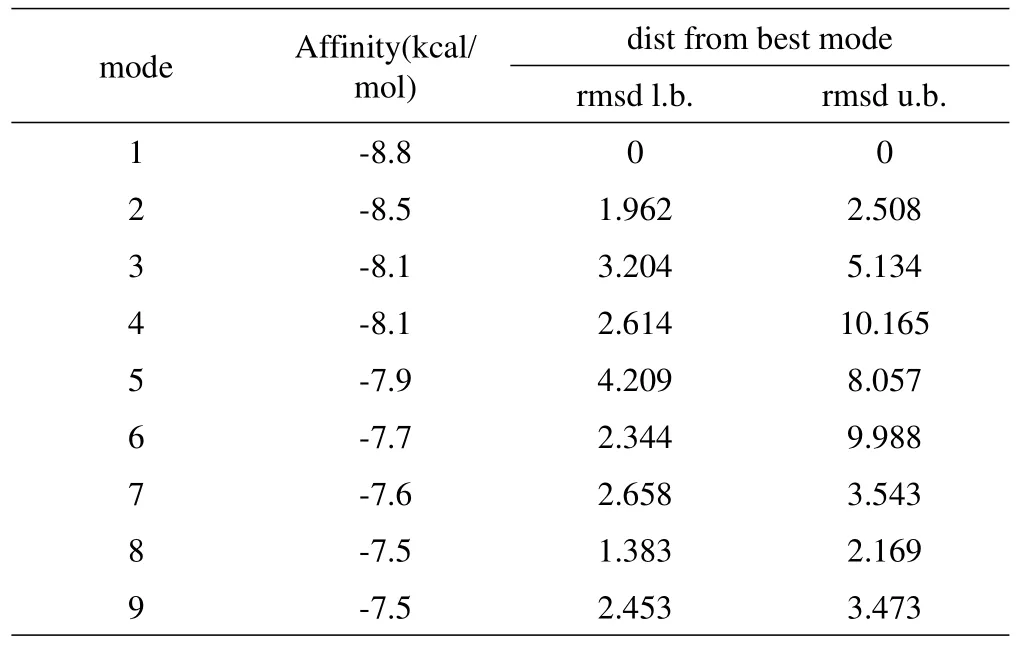
表1替加环素与3S0Z-A对接的最优结合方式Table 1the optimal combination ways of tigecycline docking with 3S0Z-A

表2与替加环素分子相距5.00Å的氨基酸和该位置出现潜在氢键的构象个数Table 2The amino acid and number of potential hydrogen bonds within 5.00Å apart
NDM-1对除了多粘菌素(colistin)和替加环素(图5)(tigecyline)外的几乎所有类型的抗生素均具有耐药性[13]。多粘菌素的反应机理是与原核生物的核糖体S30小亚基相互结合抑制细菌,而且现已出现抗药型菌株。替加环素是2005年FDA认证的抗生素药物[14],可以竞争性抑制NDM-1蛋白,然而替加环素并不能始终如一的抑制NDM-1蛋白,所以基于替加环素的新药研发成为一个治疗超级细菌感染的突破点。
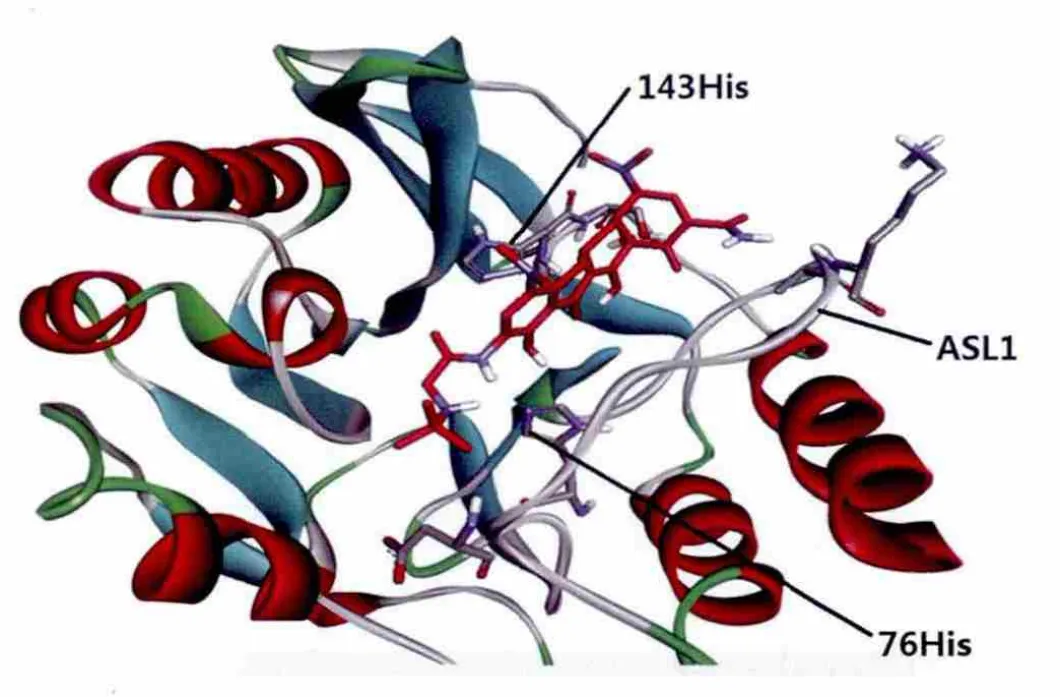
图4替加环素与3S0Z-A对接的最优构象Fig 4the optimal conformation of tigecycline docking with 3S0Z-A
文献中报导了Ceftriaxone、Cefepime、Cefoperazone、Imepenum和Meropenum这5种抗生素与3S0Z-A对接形成的氢键方式(表3)[15],结合本文潜在氢键分析统计(表2),其中204H和165K是其它抗生素的氢键区,而替加环素的9种结合方式都普遍与氢键143H和76H形成氢键,即这两个氢键很可能是替加环素独特的结合方式。
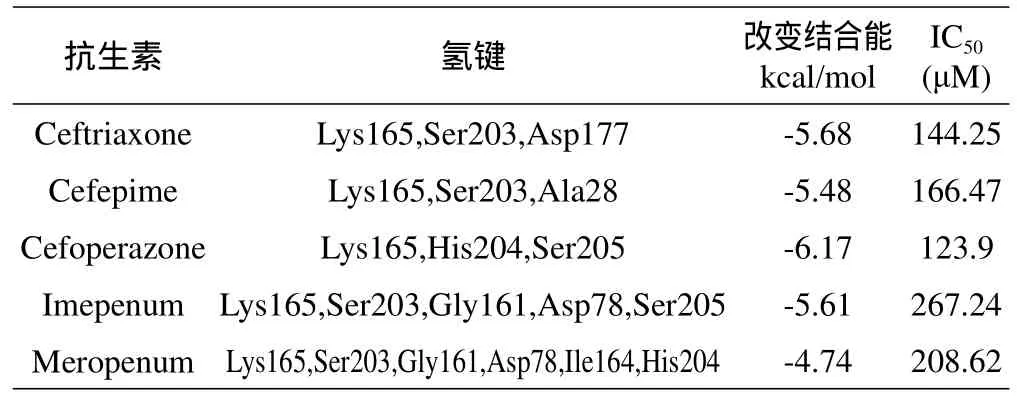
表35种抗生素与3S0Z-A对接形成的氢键方式Table 3 hydrogen bond of 5 antibiotics docking with 3S0Z-A
分析对接的结合能改变,最优结构为结合能为-8.8kcal/mol,最差结构为-7.5kcal/mol,均比5种抗生素的结合能低,所以推断出替加环素的IC50可能更小,说明灭菌一半时所需抗生素药物浓度更少,所以在同样浓度抗生素与蛋白质作用时替加环素比其它β-内酰胺类抗生素更加有竞争性抑制优势。
结合NDM-1和替加环素结合的三维结构,ASL1环位于反应中心上,像一个盖子一样盖住反应中心。当有底物结合时ASL1环上的76H可能与替加环素上的-HN-CH2-CO-NH-的2个N原子形成氢键。另外143H可能与替加环素八氢并四苯环上的-NH3基团形成氢键。此外替加环素上的羰基、氨基等都是可能形成氢键的基团。这些基团都可能通过改变取代基的方式来降低结合能,使得更加有竞争性优势。
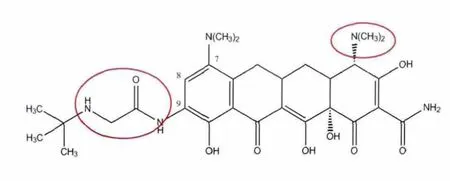
图5替加环素分子结构Fig 5Molecular structure of tigecycline
4 结论
本文通过对金属-β-内酰胺酶氨基酸序列的系统进化分析,发现NDM-1与YP_003061440.1[Hirschia baltica ATCC 49814],YP_003270512.1[Haliangium ochraceum DSM 14365],YP_458405.1[Erythrobacter litoralis HTCC2594]三种来自海洋细菌的MBL在功能上关系较近。而通过对MBL的基因序列进化分析,发现NDM-1仅与YP_458405.1[Erythrobacter litoralis HTCC2594]具有较近的进化关系。无论从氨基酸序列还是从基因序列,NDM-1与其它MBL进化距离均较远。分子进化树与物种进化树之间巨大的差异有很大可能是由于基因水平转移导致的。
接着,通过对NDM-1的GC含量进行分析,发现其编码基因上游100~350bp处具有较大的波动,而YP_458405.1编码基因波动平缓,NDM-1与YP_458405.1在编码基因区段GC含量相似度较高,基于我们对基因GC含量异常可能的原因认识,认为有可能是基因进行水平转移而导致的GC含量异常。
最后,通过半柔性分子对接,并对NDM-1与替加环素的优势结合构象的潜在氢键进行分析,与另外5种抗生素与NDM-1的对接结果进行比较,初步确定替加环素对于NDM-1蛋白存在竞争抑制优势。
因此,本文通过NDM-1的功能进化关系和遗传进化关系,以及NDM-1的GC含量变化,提出NDM-1可能与YP_458405.1[Erythrobacter litoralis HTCC2594]进行基因水平转移相关的猜想。通过替加环素与NDM-1分子对接及与其他药物分子对比,提出替加环素对于NDM-1蛋白存在竞争抑制优势,从而为NDM-1抑制剂的设计提供有价值的信息。
[1] Walsh T. The emergence and implications of metallo-β-lactamases in Gram-negative bacteria. Clinical Microbiology and Infection,2005, 11(s6): 2-9.
[2] Ambler R. The Structure of $ beta $-Lactamases. Philosophical Transactions of the Royal Society of London B. Biological Sciences,1980, 289(1036): 321-331.
[3] 李志江, 李海权, 刁现民. 基因水平转移的评判方法和转移方式研究进展. 遗传, 2008, 30(9): 1108-1114.
[4] Yong D, Toleman M A, Giske C G,et al.Characterization of a new metallo-beta-lactamase gene, bla(NDM-1), and a novel erythromycin esterase gene carried on a unique genetic structure in Klebsiella pneumoniae sequence type 14 from India. Antimicrob Agents Chemother, 2009, 53(12): 5046-5054.
[5] Zhang H, Hao Q. Crystal structure of NDM-1 reveals a common beta-lactam hydrolysis mechanism. FASEB J, 2011, 25(8): 2574-2582.
[6] Branko J, Zorica L, Vesna S. Emergence of NDM-1 metallo-betalactamase in Pseudomonas aeruginosa clinical isolates from Serbia.Antimicrob Agents Chemother, 2011, 55(8): 3929-3931.
[7] Green V L, Verma A, Owens R J. Structure of New Delhi metalloβ-lactamase 1 (NDM-1). Acta Crystallogr Sect F Struct Biol Cryst Commun, 2011, 67(Pt 10): 1160-1164.
[8] Wachino J, Arakawa Y. Exogenously acquired 16S rRNA methyltransferases found in aminoglycoside-resistant pathogenic Gram-negative bacteria: an update. Drug Resist Updat, 2012, 15(3):133-148.
[9] Bebrone C. Metallo-beta-lactamases (classification, activity, genetic organization, structure, zinc coordination) and their superfamily.Biochem Pharmacol, 2007, 74(12): 1686-1701.
[10] Tamura K, Dudley J, Nei M. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0. Molecular biology and evolution, 2007, 24(8): 1596-1599.
[11] Charif D. Lobry J R. SeqinR 1.0-2: a contributed package to the R project for statistical computing devoted to biological sequences retrieval and analysis. Structural approaches to sequence evolution Biological and Medical Physics, Biomedical Engineering. Springer,2007: 207-232.
[12] Dundas J, Ouyang Z, Tseng J ,et al.CASTp: computed atlas of surface topography of proteins with structural and topographical mapping of functionally annotated residues. Nucleic acids research,2006, 34(suppl 2): 116-118.
[13] Kumarasamy K K, Toleman M A, Walsh T R,et al.Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK:a molecular, biological, and epidemiological study. Lancet Infect Dis, 2010, 10(9): 597-602.
[14] Livermore D M. Tigecycline: what is it, and where should it be used?Journal of Antimicrobial Chemotherapy, 2005, 56(4): 611-614.
[15] Sowmiya M, Umashankar V, Muthukumaran S,et al.Studies on New Delhi Metallo-Beta-Lactamse-1 producing Acinetobacter baumannii isolated from donor swab in a tertiary eye care centre,India and structural analysis of its antibiotic binding interactions.Bioinformation, 2012, 8(10): 445-452.
