狮棘球绦虫(Echinococcus felidis)生物学特性研究进展
2014-04-02刘聪暖娄忠子范彦雷闫鸿斌贾万忠
刘聪暖,娄忠子,李 立,范彦雷,闫鸿斌,贾万忠
细粒棘球绦虫(Echinococcusgranulosus)是重要人兽共患寄生虫病——棘球蚴病的病原,其种内变异现象广泛存在,因此细粒棘球绦虫有狭义种(sensu stricto)和广义种(sensu lato)之分。 前者包括G1-G3基因型复合群,后者包括G4-G10基因型和狮株(Lion strain),其中G4基因型(马株)被重新确立为马棘球绦虫(E.equinus),G5基因型(牛株)被重新确立为奥氏棘球绦虫(E.ortleppi),G6-G10被重新确立为加拿大棘球绦虫(E.canadensis)。细粒棘球绦虫狮株最早由Ortlepp在1937年命名为狮棘球绦虫,病原体来自于南非的虎狮。将其作为一个新种进行描述是基于其吻钩皱褶明显和以猫科动物而非犬科动物作为终末宿主,这在细粒棘球绦虫复合群中是唯一的或独特的[1]。由于狮株具有吻钩皱褶明显的形态学特点,且其线粒体基因组序列与其它基因型之间差异较大,因而一些专家和学者据此建议也将其独立设种,称之为狮棘球绦虫(E.felidis)[1-2]。不同的虫种、虫株(基因型)在幼虫(包囊)形态、对人的致病性、宿主范围、流行病学意义等方面存在差异,这些特性或者特征对疫苗、诊断试剂及抗虫药物的研制和开发具有重要意义[3-4]。本文特简要概述狮棘球绦虫(物)种生物学特性研究取得的进展。
1 基因分子遗传标记特征
1.1线粒体cox1和nad1基因片段序列 细粒棘球绦虫G1-G3基因型复合群拥有广阔和交叉的地理分布及宿主范围[5-8],因此细粒棘球绦虫G1-G3基因型可以被当做一个独立单一的分类单元,即细粒棘球绦虫狭义种[3,8-9]。近年来,在Hüttner等人的研究中,狮棘球绦虫的分类地位与G1-G3基因型复合群相近,但最新的分子生物学研究结果认为,狮棘球绦虫与G1-G3基因型复合群是姊妹种[1]。考虑到最近从乌干达狮粪便中提取的虫卵DNA序列与在南非已保存40年的狮棘球绦虫成虫的DNA序列一致,因而狮棘球绦虫可以成为一个独立的基因型,并选用新近测定的源自乌干达的狮棘球绦虫样品基因序列作为其DNA分子信息分析的依据。
起初对细粒棘球绦虫的基因分型和虫株内遗传变异性分析是通过对线粒体cox1和nad1基因片段的序列比对来实现的[6,9-13]。狮棘球绦虫(源自乌干达样品)线粒体基因组序列(登录号:AB732958)中cox1(366 bp:cox1基因全长序列中第745~1110区段)和nad1(471 bp:nad1基因全长序列中第136~606区段)基因片段序列与细粒棘球绦虫G1基因型序列(登录号:AB786664)相比较,发现其自身的一些分子遗传标记特征。
通过对狮棘球绦虫和细粒棘球绦虫G1基因型序列的比较表明,cox1的碱基突变有27个,主要有A突变为G(共16个),其它为A、G和C突变为T(11个),两个序列间的核苷酸差异性为7.8%;nad1的碱基突变共有64个,其碱基突变主要为A突变为G(共33个),其次为C突变为T(共21个),少数为A、G突变为T(10个)或者G突变为C(1个);两个序列间的核苷酸差异性为15.8%。狮棘球绦虫cox1和nad1基因序列与细粒棘球绦虫G1-G3复合群以外其它棘球绦虫的序列差异相近或者更大,见表1。
表1狮棘球绦虫和其它棘球绦虫之间线粒体基因和核基因核苷酸序列差异的比较[1]
Tab.1Comparisonsofpairwisedivergence(%)ofmitochondrialandnuclearDNAsequencesbetweenE.felidisandotherE.granulosussensulato
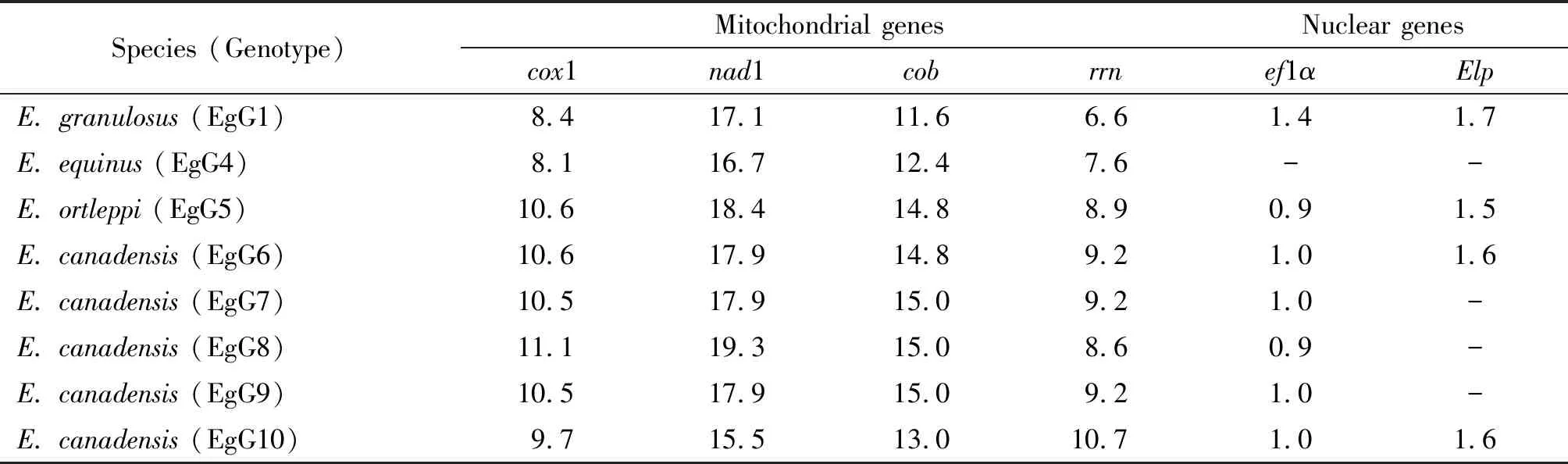
Species (Genotype)Mitochondrial genesNuclear genes cox1nad1cobrrnef1αElpE. granulosus (EgG1)8.417.111.66.61.41.7E. equinus (EgG4)8.116.712.47.6--E. ortleppi (EgG5)10.618.414.88.90.91.5E. canadensis (EgG6)10.617.914.89.21.01.6E. canadensis (EgG7)10.517.915.09.21.0-E. canadensis (EgG8)11.119.315.08.60.9-E. canadensis (EgG9)10.517.915.09.21.0-E. canadensis (EgG10)9.715.513.010.71.01.6
Note: The complete sequences of mitochondrialcox1,nad1,cobandrrngenes are used in analysis of pair nucleotide divergence,while the partial nuclear genes ofefla andelpare used. G9 found in the Poland patient is probably a variant of genotype G7[15,18,19],and Saarma et al[8]report that genotypes G6,G7,and G9 are located in the same position of the same phylogenetic branch,which is constructed based on mitochondrial genomes and nuclear DNA sequences. Therefore,we consider the sequences divergence betweenE.felidisandE.canadensisG9 to be equal with that betweenE.felidisandE.canadensisG7.
1.2核DNA序列
1.2.1核基因编码序列 仅用线粒体DNA序列构建进化树存在误差风险,这是由于线粒体DNA的母性遗传不一定与真实的物种进化过程相符合,从而对进化过程造成误判[14-15]。因此,除了用线粒体DNA作为进化分子标记特征外,多种核DNA也逐渐被作为分子标记而广泛应用[15]。如:elp(ezrin-radixin-moesin- like protein,埃兹-根蛋白-膜突蛋白样蛋白 )、ef1a(elongation factor 1 alpha,延伸因子1α)、pepck(磷酸烯醇式丙酮酸羧激酶)、pold(DNA polymerase delta,DNA 聚合酶δ)基因或基因片段序列。
通过比较狮棘球绦虫和细粒棘球绦虫狭义种的序列发现,elp基因(狮棘球绦虫的登录号为EF558360,细粒棘球绦虫G1基因型的登录号为EU834886)共有15个碱基突变,其碱基突变主要为G突变为A(共5个),其次为A突变为G(3个),T突变为C(4个),少数为G突变为T(1个),G突变为C(1个),C突变为T (1个);两个序列间的核苷酸差异性为1.7%。
ef1a基因(狮棘球绦虫的登录号为FN568379,细粒棘球绦虫的登录号为FN568380)共有13个碱基突变,其碱基突变主要为T突变为C(共5个),其次为C突变为T(3个),少数为T突变为G(1个),A突变为G(1个),G突变为A(1个),C突变为A(1个),G突变为T(1个);两个序列间的核苷酸差异性为1%。
pepck基因(狮棘球绦虫的登录号为FN567989,细粒棘球绦虫狭义种的登录号为FN567990)共有10个碱基突变,其碱基突变主要为A突变为G(共4个),C突变为T(共4个),其次为G突变为A(1个),T突变为C(1个);两个序列间的核苷酸差异性为0.7%。
pold基因(狮棘球绦虫的登录号为FN568360,细粒棘球绦虫狭义种的登录号为FN568361)共有15个碱基突变,其碱基突变主要为A突变为G(共6个),其次为C突变为T(4个),G突变为A (3个),少数为C突变为G(1个),G突变为T(1个);两个序列间的核苷酸差异性为1.4%,见表2。
表2狮棘球绦虫和细粒棘球绦虫狭义种之间核基因核苷酸序列差异的比较
Tab.2Comparisonsofpairwisedivergencevalues(%)ofnuclearDNAsequencesbetweenE.felidisandE.granulosussensustricto
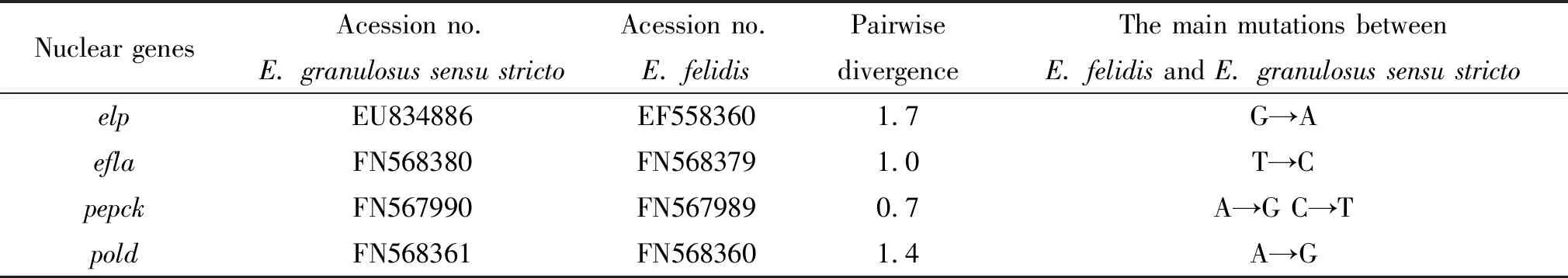
Nuclear genesAcession no. E. granulosus sensu strictoAcession no. E. felidisPairwise divergenceThe main mutations between E. felidis and E. granulosus sensu stricto elpEU834886EF5583601.7G→A eflaFN568380FN5683791.0T→CpepckFN567990FN5679890.7A→G C→TpoldFN568361FN5683601.4A→G
1.2.2核糖体rRNA基因序列 内转录间隔区序列1(ITS1):对狮棘球绦虫分离株(NCBI登录号:FJ426641-FJ426646)和细粒棘球绦虫G1-G3基因型(NCBI登录号:AY969043)的ITS1序列进行比对分析发现,狮棘球绦虫分离株有2~3个序列缺失位点,其大小分别为18 bp,3 bp和54 bp,其中54 bp的缺失位点只在个别分离株中出现(例如:NCBI登录号:FJ426643和FJ426646)。此外,狮棘球绦虫分离株与细粒棘球绦虫G1-G3基因型还有12个可能的突变位点,这些零散的突变位点只是在部分狮棘球绦虫分离株中出现,并不适用于设计特异性引物对狮棘球绦虫分离株进行区别鉴定。狮棘球绦虫分离株ITS1的缺失位点使通过种间特异性聚合酶链反应区别狮棘球绦虫分离株和细粒棘球绦虫G1-G3基因型成为可能,在18 bp和54 bp两个序列缺失位点设计引物可以完成细粒棘球绦虫G1-G3基因型的PCR扩增,而在狮棘球绦虫分离株中则无扩增结果。
18S rRNA基因序列(18S rDNA):对狮棘球绦虫和细粒棘球绦虫的18S rDNA 序列进行比对分析发现,狮棘球绦虫有2个序列插入位点,其大小分别为10 bp和5 bp;此外,狮棘球绦虫(狮棘球绦虫的登录号为AB731638)和细粒棘球绦虫(细粒棘球绦虫的登录号为GQ260092)有35个碱基突变,其碱基突变主要为A突变为G(10个),G突变为A(8个),T突变为C(6个),其次为C突变为G(4个),C突变为T(3个),少数为T突变为G(2个),G突变为T(1个),C突变为A(1个);两个序列间的核苷酸差异性为1.6%。
2 与细粒棘球绦虫其它虫株(基因型)的亲缘关系
最近的研究表明,狮棘球绦虫与细粒棘球绦虫G1-G3基因型复合群分类单位亲缘关系相近,二者的分类地位为姊妹种关系。在此基础上,以目前所有可用的数据为基础,可以得出狮棘球绦虫和细粒棘球绦虫G1-G3复合群基因型在具有共同祖先的结论,但目前前者已经演化成一个独立的系统发育实体,甚至极有可能也演化成为一个独立的种[1,8]。
对狮棘球绦虫线粒体基因DNA全长序列(cox1、nad1、cob和rrn),用最大似然法和贝叶斯推论,确定狮棘球绦虫的分类地位及与其它棘球绦虫的系统发育关系。细粒棘球绦虫狭义种和细粒棘球绦虫狮株一对新的姊妹种关系就是通过最大似然法和贝叶斯推论被确认,并通过引导比例和贝叶斯后验概率的高度准确性给这些姊妹种关系提供了后验支持。通过贝叶斯推论得到的进化树显示细粒棘球绦虫狭义种和狮棘球绦虫在已经确定的分类中形成了最基础的谱系,并展示了它们亲缘关系的程度。此后,细粒棘球绦虫进化树也通过由cox1、nad1 和cob翻译而来的线粒体蛋白质序列而被重新构建。虽然最大似然法和运用蛋白质数据进行贝叶斯推论产生的一个进化树,在关于马棘球绦虫(E.equinus)的分类地位和福氏棘球绦虫(E.vogeli)的分类地位以及涉及少节棘球绦虫(E.oligarthra)的方面存在一些不同,但细粒棘球绦虫狭义种和狮棘球绦虫的姊妹种关系仍然被这两种分析方法所高度支持[1]。
狮棘球绦虫和其它种类棘球绦虫的线粒体DNA和核DNA序列对比所产生的差异度见表1,其中狮棘球绦虫与其它棘球绦虫线粒体DNA的差异性在6.6%~19.3%之间,且即使是与与其有姊妹种关系的细粒棘球绦虫狭义种的线粒体DNA的差异性也在6.6%~17.1%之间。狮棘球绦虫与其它棘球绦虫核DNA的差异性在0.9%~1.7%之间,但是狮棘球绦虫与与其亲缘关系最近的细粒棘球绦虫狭义种核DNA的差异性在1.4%~1.7%之间,差异也很明显[1]。通过对乌干达狮株和细粒棘球绦虫G1型(登录号:NC_008075)线粒体基因组的部分基因进行比较分析,发现狮株与G1基因型的cox1序列相似性为92.6%,nad1序列相似性为87.2%,atp6的序列相似性为86.8%,cox2的相似性为89.6%,cox3的相似性为90.4%,nad2的序列相似性为88.6%,nad3的序列相似性为89.7%,nad4的序列相似性为87.6%,nad4L的序列相似性为93.6%,nad5的序列相似性为85.4%,nad6的序列相似性为88.1%,cob序列相似性为90.5%。诸多比较发现,棘球绦虫狮株和细粒棘球绦虫G1型线粒体多个基因的相似度较高,均在85%以上,这也是棘球绦虫狮株与细粒棘球绦虫G1-G3基因型能够作为姊妹种的分子基础。
线粒体DNA和核DNA在进化分析上也具有明显的差异,当用线粒体DNA进行进化分析时,细粒棘球绦虫G1-G10基因型和狮棘球绦虫属于并系关系,少节棘球绦虫与其它种类的棘球绦虫属于姊妹种关系[1,9,16-17],而用核蛋白编码基因进行进化分析的结果显示,细粒棘球绦虫是一个单一居群,多房棘球绦虫(E.multilocularis)与细粒棘球绦虫属于姊妹种关系。然而,尽管线粒体DNA和核DNA在进化分析上具有明显的差异,但是二者的进化分析均表明狮棘球绦虫和细粒棘球绦虫狭义种属于姊妹种关系,图1。[8,17]
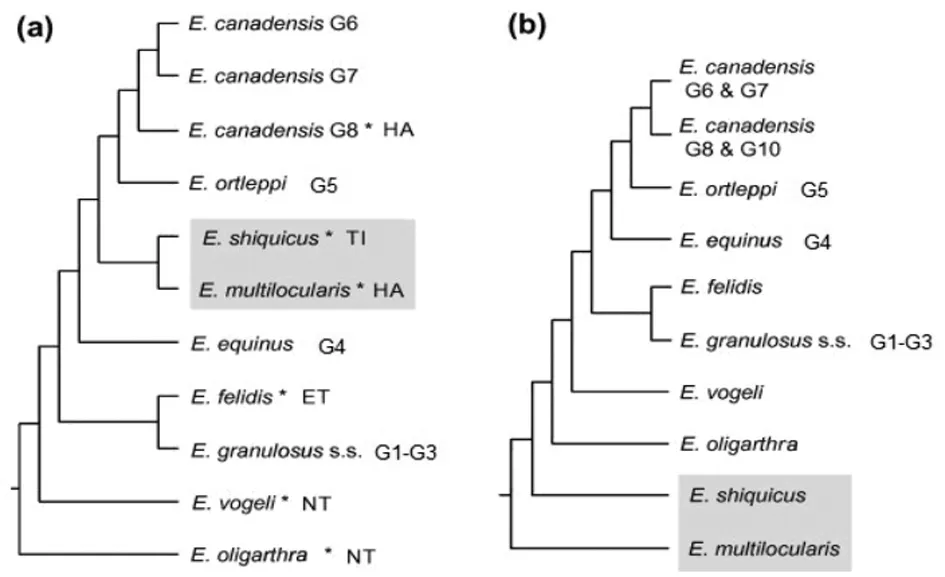
图1对于最近发表的关于棘球绦虫分子系统发育关系的总结[17]
Fig.1Summaryofnewly-publishedmolecularphylogenetictreesforthegenusEchinococcus
(a) The phylogenetic tree was constructed based on mitochondrial DNA sequences[1,9].
(b) The phylogenetic tree was constructed based on nuclear protein-coding genes sequences[8]. The shadow area indicates the category shift based on mitochondrial DNA sequences. The starred parasites show that their hosts are the wild mammals,meanwhile,HA stands for Holarctic region,TI for Tibet,ET for Ethiopia and NT for Neotropical region,and the other unmarked species use the mammals from Japan as the host distributing all over the world[17].
3 独立种地位与物种形成(进化史)
狮棘球绦虫与细粒棘球绦虫狭义种DNA序列差异性同多房棘球绦虫与石渠棘球绦虫(E.shiquicus)之间的差异相当,但是要远远高于奥氏棘球绦虫与加拿大棘球绦虫之间的差异,同时也远远高于公认的带属(Taenia)绦虫中的亚洲带绦虫(T.asiatica)和牛带绦虫(T.saginata)间的差异[9,20]。此外,广泛地理分布上的各分离株即新近从乌干达采集的分离株和保存于20世纪60年代南非分离株在遗传学上保持一致,同时狮棘球绦虫具有明显不同的形态学特征和以独特的狮类为终末宿主[21]。这些证据都充分支持了狮棘球绦虫具有独立种地位。
根据线粒体DNA序列[1]和部分核基因DNA序列[17]认为,狮棘球绦虫很明显是细粒棘球绦虫狭义种的姊妹种,推测它们有一个共同的亚洲祖先,这是基于现代猫科动物起源于亚洲[22]这一观点得出的结论。非洲狮(Pantheraleo)是从上新世晚期生活在亚洲一带的祖先世系进化来的[22],然后在早更新世期间侵入非洲领地栖息。假定在亚洲一带就出现了狮棘球绦虫和细粒棘球绦虫狭义种的分化,则前者便在早更新世之后随非洲狮一起被带到了非洲。狮子在有史以来分布广泛,包括印度、中东和非洲等地,但是现在仅散布于非洲及印度部分区域[23]。根据Hüttner等人的调查,狮棘球绦虫是乌干达伊丽莎白女王国家公园狮子一种普通的寄生虫[15]。
4 鉴定方法
对ITS1基因进行RFLP-PCR(限制性片段长度多态性-聚合酶链反应)和对12S rRNA基因进行种间特异性聚合酶链反应都不能区别狮棘球绦虫和细粒棘球绦虫狭义种,细粒棘球绦虫和狮棘球绦虫12S rRNA仅有2个和3个核苷酸的差异,不能防止狮棘球绦虫12S rRNA的非特异性扩增。而对nad1基因进行限制性片段长度多态性-聚合酶链式反应检测,经过Hph1限制性内切酶消化的nad1 PCR产物的带型与所有已知的棘球绦虫的带型之间存在差异,在细粒棘球绦虫狭义种和狮棘球绦虫之间存在明显的差异,但是在细粒棘球绦虫狭义种G1、G2和G3之间并无差异。通过对nad1 PCR产物进行酶切消化后,可以观察到较短的条带,但这种条带在普通的琼脂糖凝胶电泳图上无法观察。现有数据库中有大量可以参考的cob基因序列,因此可以对cob基因进行限制性片段长度多态性-聚合酶链式反应,从而确定样品来自于何种宿主。cob基因的片段较短,但可以选用Hph1限制性内切酶,它可以将来自于狮子,豹和黑斑鬣犬的样品清楚的区分开[24]。当然,随着愈来愈多DNA序列的积累,可为设计狮棘球绦虫特异性PCR等分子生物学方法提供重要依据,以期建立一系列以DNA-PCR为基础的分子流行病学调查和虫种鉴定方法。
5 流行病学特征
对狮棘球绦虫进行准确鉴定,其目的在于对病原体的传播途径、人和动物易感性等流行病学数据与信息进行准确分析,为疾病的有效控制提供重要依据。据文献报道,狮棘球绦虫不仅存在于狮体,还可寄生于其它食肉动物。人或动物的囊型包虫病,可由G1-G10基因型细粒棘球绦虫和狮棘球绦虫引起,除狮棘球绦虫外,所有其它细粒棘球绦虫都能以家犬作为中间宿主。在乌干达西部的一个国家公园进行的调查显示,大部分在此饲养的狮子都被狮棘球绦虫所感染,在同一地理位置的疣猪体内,也能发现含有狮棘球蚴的囊泡[25]。狮棘球绦虫的中间宿主主要为斑马、牛羚、疣猪、薮猪、水牛和各种羚羊,但是否以长颈鹿和河马为中间宿主,目前还有待于进一步证实,但其终末宿主,现在的发现仅限于狮子[3,26]。由于没有确定的诊断标准,诊断狮棘球绦虫感染的难度大,因而对其宿主范围和地理分布的了解尚少,且有关其中间宿主范围的资料也很少。同时,没有确实的证据证明狮子是唯一的终末宿主,其它猫科动物、犬科动物、鬣狗科动物是否参与到其生活史也尚未可知[1,27],其对人是否具有致病性也有待进一步研究[1,3-4,26,28]。
狮棘球绦虫主要分布于埃塞俄比亚、南非、乌干达和东非保护区[3-4,24-25,27]。由于狮棘球绦虫与细粒棘球绦虫狭义种的亲缘关系很近,而细粒棘球绦虫狭义种是世界范围内人类感染棘球绦虫的主要种类,因此狮棘球绦虫有引起人类感染的潜在可能性。但狮子大多数被限制在很少有人类活动的国家公园和狩猎保护区,它可能对东非一些仍与野生动物共存的牧民有一定的影响,例如肯尼亚和坦桑尼亚的马赛人,但它对这些区域以外公众健康的危害性可能很小[1,26]。
6 研究中存在的问题
狮棘球绦虫的研究目前尚少,其主要原因可能是相关研究材料获取困难所致。就目前的研究结果表明,狮棘球绦虫的终末宿主仅为狮子,中间宿主种类虽然较多,但主要是非洲热带草原上野生动物,病原材料的获取非常困难。仅仅是从野外获取狮子粪便,就需要严格的安全防护措施以防范狮子等野生动物对人类的攻击,并需要对狮子的生活习性和粪便特征极为了解的人员陪同收集,其获取难度可想而知。对狮棘球绦虫的鉴定,跟其它寄生虫的鉴定一样,也是从形态学特征和分子遗传标记特征两方面入手,但狮棘球绦虫与细粒棘球绦虫G1-G10基因型的形态特征极为相似,只有以吻钩皱褶作为区分标准,无其它明显的眼观特征,因此从形态学上很难区分。狮棘球绦虫的主要分子遗传标记与其它棘球绦虫的差异度在0.9%~22.9%之间,多数分子标志的差异度在10% 左右,因而是鉴定狮棘球绦虫较为理想的方法,但目前以之为基础的分子生物学鉴定方法因方法复杂,尚有诸多技术性问题需要解决。
7 未来研究方向
目前用于鉴定的狮棘球绦虫分离株数目有限,鉴定的分子靶标主要集中于线粒体cox1、nad1、cob、rrnS和rrnL基因,以及核ITS1、elp、ef1a、pepck、pold基因或基因片段序列,序列信息量有限,因此一方面应增加分离株的数目,以期获取更多群体的核苷酸变异信息,另一方面应在使用原来分子标记的同时应分离与鉴定新的有价值的分子遗传学标记,以便有助于精确鉴定狮棘球绦虫虫种(虫株)和分析其群体遗传变异规律。
参考文献:
[1]Huttner M,Nakao M,Wassermann T,et al. Genetic characterization and phylogenetic position ofEchinococcusfelidis(Cestoda: Taeniidae) from the African lion[J]. Int J Parasitol,2008,38(7): 861-868. DOI: 10.1016/j.ijpara.2007.10.013
[2]Huttner M,Romig T.Echinococcusspecies in African wildlife[J]. Parasitology,2009,136(10): 1089-1095. DOI: 10.1017/S0031182009990461
[3]Thompson RC. The taxonomy,phylogeny and transmission ofEchinococcus[J]. Exp Parasitol,2008,119(4): 439-446. DOI: 10.1016/j.exppara.2008.04.016
[4]Nakao M,Yanagida T,Okamoto M,et al. State-of-the-artEchinococcusandTaenia: phylogenetic taxonomy of human-pathogenic tapeworms and its application to molecular diagnosis[J]. Infect Genet Evol,2010,10(4): 444-452. DOI: 10.1016/j.meegid.2010.01. 011
[5]Busi M,Snabel V,Varcasia A,et al. Genetic variation within and between G1 and G3 genotypes ofEchinococcusgranulosusin Italy revealed by multilocus DNA sequencing[J]. Vet Parasitol,2007,150(1/2): 75-83. DOI: 10.1016/j.vetpar.2007.09.003
[6]Varcasia A,Canu S,Kogkos A,et al. Molecular characterization ofEchinococcusgranulosusin sheep and goats of Peloponnesus,Greece[J]. Parasitol Res,2007,101(4): 1135-1139. DOI: 10.1007/s00436-007-0568-x
[7]Rinaldi L,Maurelli MP,Capuano F,et al. Molecular update on cystic echinococcosis in cattle and water buffaloes of southern Italy[J]. Zoonoses Public Hlth,2008,55: 119-123. DOI: 10.1111/j.1863-2378.2007.01101.x
[8]Saarma U,Jogisalu I,Moks E,et al. A novel phylogeny for the genusEchinococcus,based on nuclear data,challenges relationships based on mitochondrial evidence[J]. Parasitology,2009,136(3): 317-328. DOI: 10.1017/S0031182008005453
[9]Nakao M, McManus DP, Schantz PM,et al. A molecular phylogeny of the genusEchinococcusinferred from complete mitochondrial genomes[J]. Parasitology,2007,134(Pt 5): 713-722. DOI: 10.1017/S0031182006001934
[10]Bowles J,McManus DP. NADH dehydrogenase 1 gene sequences compared for species and strains of the genusEchinococcus[J]. Int J Parasitol,1993,23: 969-972. DOI: 10.1016/0020-7519(93)90065-7
[11]Bowles J,McManus DP. Molecular variation inEchinococcus[J]. Acta Trop,1993,53: 291-305. DOI: 10.1016/0001-706X(93)90035-A
[12]Bowles J,Blair D,McManus DP. Genetic variants within the genusEchinococcusidentified by mitochondrial DNA sequencing[J]. Mol Biochem Parasitol,1992,54: 165-174. DOI: 10.1016/0166-6851(92)90109-W
[13]Obwaller A,Schneider R,Walochnik J,et al.Echinococcusgranulosusstrain differentiation based on sequence heterogeneity in mitochondrial genes of cytochrome coxidase-1 and NADH dehydrogenase-1[J]. Parasitology,2004,128(Pt 5): 569-575. DOI: 10.1017/S0031182004004871
[14]Ballard JW,Whitlock MC. The incomplete natural history of mitochondria[J]. Mol Ecol,2004,13(4): 729-744. DOI: 10.1046/j.1365-294X.2003.02063.x
[15]Nakao M,Lavikainen A,Yanagida T,et al. Phylogenetic systematics of the genusEchinococcus(Cestoda: Taeniidae)[J]. Int J Parasitol,2013,43(12-13): 1017-1029. DOI: 10.1016/j.ijpara.2013.06.002
[16]Moks E,Jogisalu I,Valdmann H,et al. First report ofEchinococcusgranulosusG8 in Eurasia and a reappraisal of the phylogenetic relationships of ‘genotypes’ G5-G10[J]. Parasitology,2008,135(5): 647-654. DOI: 10.1017/S0031182008004198
[17]Knapp J, Nakao M, Yanagida T,et al. Phylogenetic relationships withinEchinococcusandTaeniatapeworms (Cestoda: Taeniidae): An inference from nuclear protein-coding genes[J]. Mol Phylogenet Evol,2011,61(3): 628-638. DOI: 10.1016/j.ympev.2011.07.022
[18]Scott JC,Stefaniak J,Pawlowski ZS,et al. Molecular genetic analysis of human cystic hydatid cases from Poland: identi?cation of a new genotypic group (G9) ofEchinococcusgranulosus[J]. Parasitology,1997,114: 37-43.
[19]Kedra AH,Swiderski Z,Tkach VV,et al. Genetic analysis ofEchinococcusgranulosusfrom humans and pigs in Poland,Slovakia and Ukraine. A multicenter study[J]. Acta Parasitol,1999,44: 248-254.
[20]Jeon HK,Eom KS.TaeniaasiaticaandTaeniasaginata: genetic divergence estimated from their mitochondrial genomes[J]. Exp Parasitol,2006,113(1): 58-61. DOI: 10.1016/j.exppara.2005.11.018
[21]Verster AJ. Review ofEchinococcusspecies in South Africa[J]. Onderstepoort J Vet Res,1965,32(1): 7-118.
[22]Johnson WE,Eizirik E,Pecon-Slattery J,et al. The late Miocene radiation of modern Felidae: a genetic assessment[J]. Science,2006,311: 73-77. DOI: 10.1126/science.1122277
[23]Bertola LD,van Hooft WF,Vrieling K,et al. Genetic diversity,evolutionary history and implications for conservation of the lion (Pantheraleo) in West and Central Africa[J]. J Biogeogr,2011,38: 1356-1367. DOI: 10.1111/j.1365-2699.2011.02500.x
[24]Huttner M,Siefert L,Mackenstedt U, et al. A survey ofEchinococcusspecies in wild carnivores and livestock in East Africa[J]. Int J Parasitol, 2009,39(11): 1269-1276. DOI: 10.1016/j.ijpara.2009.02.015
[25]Wahlers K, Menezes CN, Wong ML ,et al. Cystic echinococcosis in sub-Saharan Africa[J]. Lancet Infect Dis, 2012,12(11): 871-880. DOI: 10.1016/S1473-3099(12)70155-X
[26]Thompson RC, McManus DP. Towards a taxonomic revision of the genusEchinococcus[J]. Trends Parasitol,2002,18(10): 452-457. DOI: 10.1016/S1471-4922(02)02358-9
[27]Moro P, Schantz PM. Echinococcosis: a review[J]. Int J Infect Dis, 2009,13(2): 125-133. DOI: 10.1016/j.ijid.2008.03.037
[28]Schantz PM. Progress in diagnosis,treatment and elimination of echinococcosis and cysticercosis[J]. Parasitol Int, 2006,55 Suppl: S7-S13. DOI: 10.1016/j.parint.2005.11.050
