棘球属绦虫分子种系发生研究进展
2014-04-02娄忠子闫鸿斌范彦雷李建秋贾万忠
李 立,娄忠子,闫鸿斌,范彦雷,李建秋,殷 宏,贾万忠
棘球蚴病(包虫病)是由隶属于扁形动物门(Platyhelminthes)绦虫纲(Cestoda)带科(Taeniidae)棘球属(Echinococcus)绦虫的中绦期或幼虫寄生于动物和人的肝、肺等脏器所引起,是一种严重危害人和动物健康的人兽共患寄生虫病,呈全球性分布,而我国是该病的高发区[1-2]。尽管早在公元前古希腊医生希波克拉底(Hippocrates)、阿雷提乌斯(Aretaeus)和伽林(Galen)等就认识到人的这种疾病,但棘球蚴病的病原体,即棘球属绦虫的确定及分类是近几个世纪完成的,研究人员对棘球属绦虫内种的成员已经基本澄清,对其研究历经了早期对虫体的描述即形态学方面的观察和分类,之后虫种和虫株的识别以及近期现代分子生物学的研究[4-6]。
近年来,以自动化脱氧核糖核酸(Deoxyribonucleic acid, DNA)测序技术和统计计算机算法为基础的生物信息学取得的飞速发展,从根本上改变了对生物体如何进行分类,影响着如何能够重建较高级分类单元到种和居群的进化关系[7-8]。随着对棘球属绦虫分子生物学研究的深入,逐渐加深了人们对其种群多样性的认识和理解,最终取得了棘球属绦虫种阶元及种系发生关系的一致性认识。鉴于棘球属内含有形态学上不易区分的隐藏种的存在,分子生物学技术对鉴定虫种必不可少。因此,本文作者将从系统发生种概念这一角度入手,对棘球属绦虫9个有效种的确立及其分子种系发生研究方面所取得的重要进展进行综述。
1 种系发生概念
种系发生也被称作系统发生,是指在地球历史发展过程中生物种系的发生和发展。通过形态学、解剖学、生理学特征以及DNA序列等数据特征,为生物建立一棵种系发生树(演化树),以描述各个物种之间可能有的亲缘关系。其中以分子数据为基础的种系发生称为分子种系发生(Molecular phylogeny)。由于种系发生的进化过程是不能被直接通过观察和实验所证实的,所以需要将各个方面的证据都综合起来分析。此外,由于针对这些证据的侧重点不同,经常会产生不同的演化(进化)树版本。物种简称“种”,是生物分类的基本单位,关于物种的定义有多种,其中较为常用的物种概念有两种——系统发生(物)种概念(Phylogenetic species concept, PSC)和生物种概念(Biological species concept)。系统发生种(Phylogenetic species,系统发育种,又译为分支物种概念或者进化物种概念)被定义为最小集物种,是不可再分的(基本的)一群生物,与其他这样的生物群可明确鉴别,群内成员来自共同祖先。而与此相对应的另一物种概念——生物学种(Biological species)概念由迈尔[9]提出,他认为物种实际上是彼此已进行杂交或彼此可能杂交的一组自然群体,它们跟其它这类群体在生殖上是隔离的。这个定义假设只适用于进行有性生殖的物种,定义生物学种的关键在于两个种群间没有显著的基因物质交流。以单系群(Monophyly)为基础的系统发生种概念要比生物学种概念灵活,这个观念不需要考虑到生物的性别,而是以找寻个体的共同祖先为原则,允许亲缘关系近的种之间可能杂交[10]。鉴于棘球属绦虫独特的繁殖方式,即成虫阶段行自体交配和幼虫阶段行无性繁殖,因而本文将主要针对种系发生物种概念来对棘球属绦虫分类学进行论述。
2 棘球属的一般特征与种的鉴定
2.1棘球属绦虫一般特征 棘球绦虫生活史中虫体发育要经历两个重要阶段:成虫期和中绦期(幼虫期、蚴期)。棘球绦虫成虫为小型虫体,一般由头节、颈节和3~5个体节组成,体长不超过1 cm,这在带科绦虫很特别,而其它带科绦虫多为大型绦虫,体节数成百上千,体长可超过1 mm。棘球绦虫中绦期为包囊形式,包囊有3种类型包括单囊型(如细粒棘球绦虫)、多囊型(如伏氏棘球绦虫)和多房型(泡球型,只有多房棘球绦虫)[11-12],虽然这些包囊形态不同,但均具有相同的包囊结构。食肉动物和被猎食的哺乳动物是维持棘球绦虫生活史所不可或缺的两类宿主,成虫寄生于食肉动物而中绦期寄生于草食动物、杂食动物包括人等。
2.2棘球属绦虫虫株及基因型 由于棘球属绦虫尤其是细粒棘球绦虫种内变异现象非常普遍,因此同种异名或者不同种同名现象时有发生,报道中常引入亚种、虫株或者基因型等概念。但是,这些报道中的不少亚种其独立性不能成立。虽然虫株概念不是寄生虫分类学上的正式阶元,但是它在人们的实践活动中被广泛地接受和使用。后来的研究也表明,细粒棘球绦虫虫株在不同的地理环境和宿主中,存在着实质性的基因遗传多样性差异,其基因型与原来各虫株间保持着相互对应的关系,且两者之间极为一致。不同虫株(基因型)在幼虫(包囊)形态、对人和动物的致病性、宿主范围、流行病学意义等方面存在差异(见表1),对疫苗、诊断试剂及抗虫药物的研制和开发具有重要意义[13]。目前,业已发现的基因型或者虫株(Strains)至少有10个(G1-G10)[14-18],这些虫株以它们所寄生的中间宿主的名称进行命名。此外,广义的细粒棘球绦虫还包括狮株(Lion strain)[19-20],但它是以终末宿主的名字进行命名的。在细粒棘球绦虫广义种内,其中的一部分虫株(基因型)可以升格或者重新修正为单一种。因此,根据最新研究结果认为,棘球属绦虫至少包括9个有效种,每一个种都有着不同的宿主—寄生虫相互关系(见表1)。
2.3种的识别和鉴定
直到现在,关于棘球属的分类仍有争议,这主要是由于虫体的形态具有可塑性。其次,当虫体寄生于异常宿主时使得人们对病原体的鉴定更为困难。历史上,曾经将棘球属模式种——细粒棘球绦虫认为是囊性棘球蚴病和泡型棘球蚴病的病原体。虽然每一个种都有着不尽相同的宿主和寄生虫关系,但是它们之间无论在地理分布还是形态学等方面有交叉和重叠现象,这给种的准确识别造成了困难。因而,种的确切鉴定还需要其它方法和手段。
二十世纪九十年代,随着澳大利亚一研究小组应用分子分类学分析方法才使人们对棘球绦虫的分类和鉴定有了大的突破。研究人员通过对分离株样品线粒体细胞色素c氧化酶亚基1(Cytochromecoxidase subunit 1,cox1)和NADH脱氢酶亚基1(NADH dehydrogenase subunit 1,nad1)的基因片段的序列测定和分析,利用分子遗传标记特征成功将细粒棘球绦虫不同虫株进行了基因分型,澄清了不同虫株与基因型之间的确切关系,并且确定了棘球绦虫不同种之间的差异[35]。随后,更多的基因序列被用于棘球绦虫虫种鉴定与分子进化关系分析,使种的识别成为现实,种水平上的分类成为可能。通过这些基因片段序列数据可成功将细粒棘球绦虫G1-G3型定名为狭义种,G4定名为马棘球绦虫,G5定名为奥氏棘球绦虫,G6-G10定名为加拿大棘球绦虫。大约在同一时期,科学家们在我国青藏高原发现了石渠棘球绦虫,狮株被定名为狮棘球绦虫,加之之前已定名的3种棘球绦虫,共确立了棘球属绦虫9个种的分类地位。
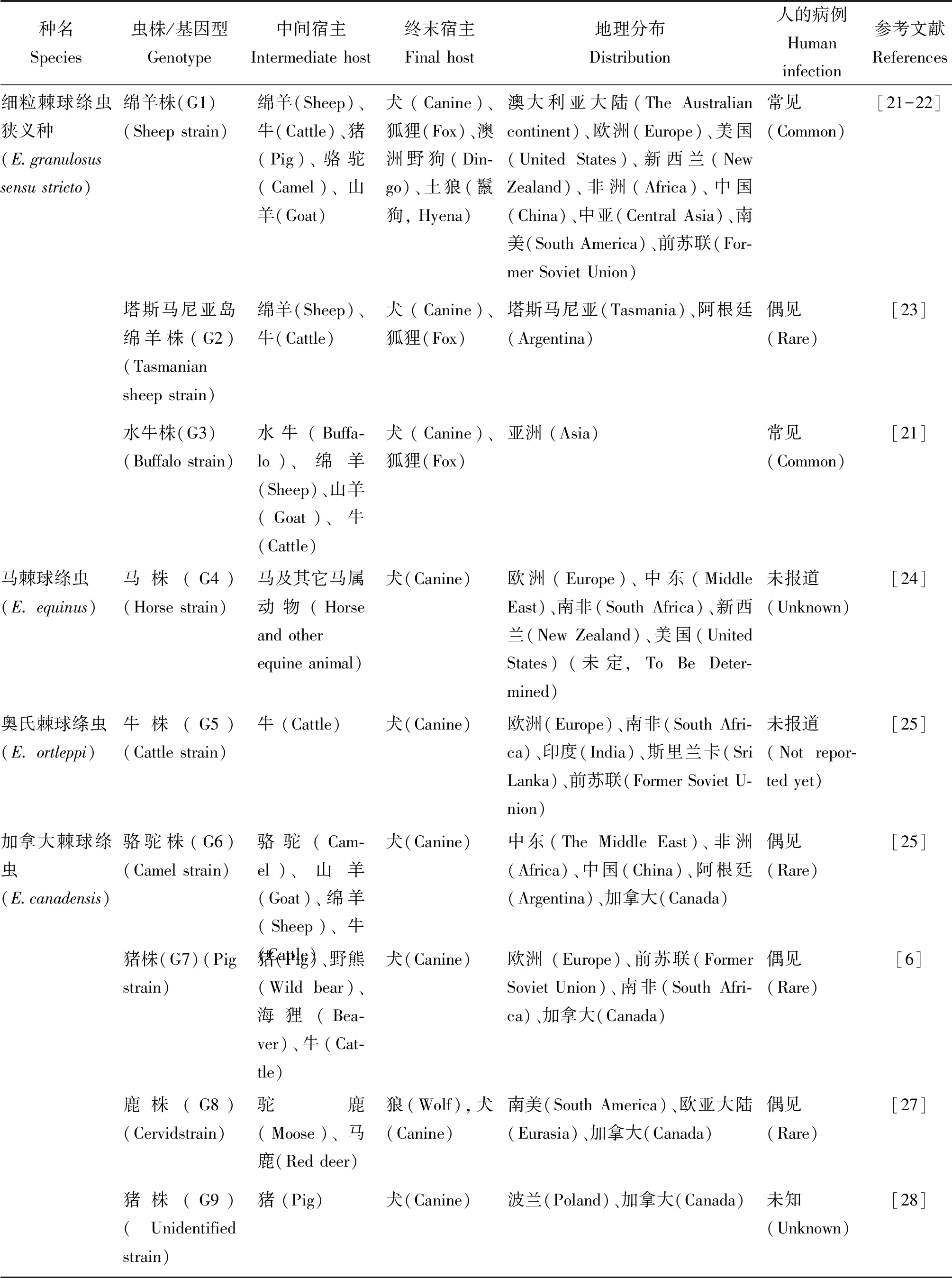
表1 棘球绦虫9个有效种流行病学主要特征之间的比较
表1续
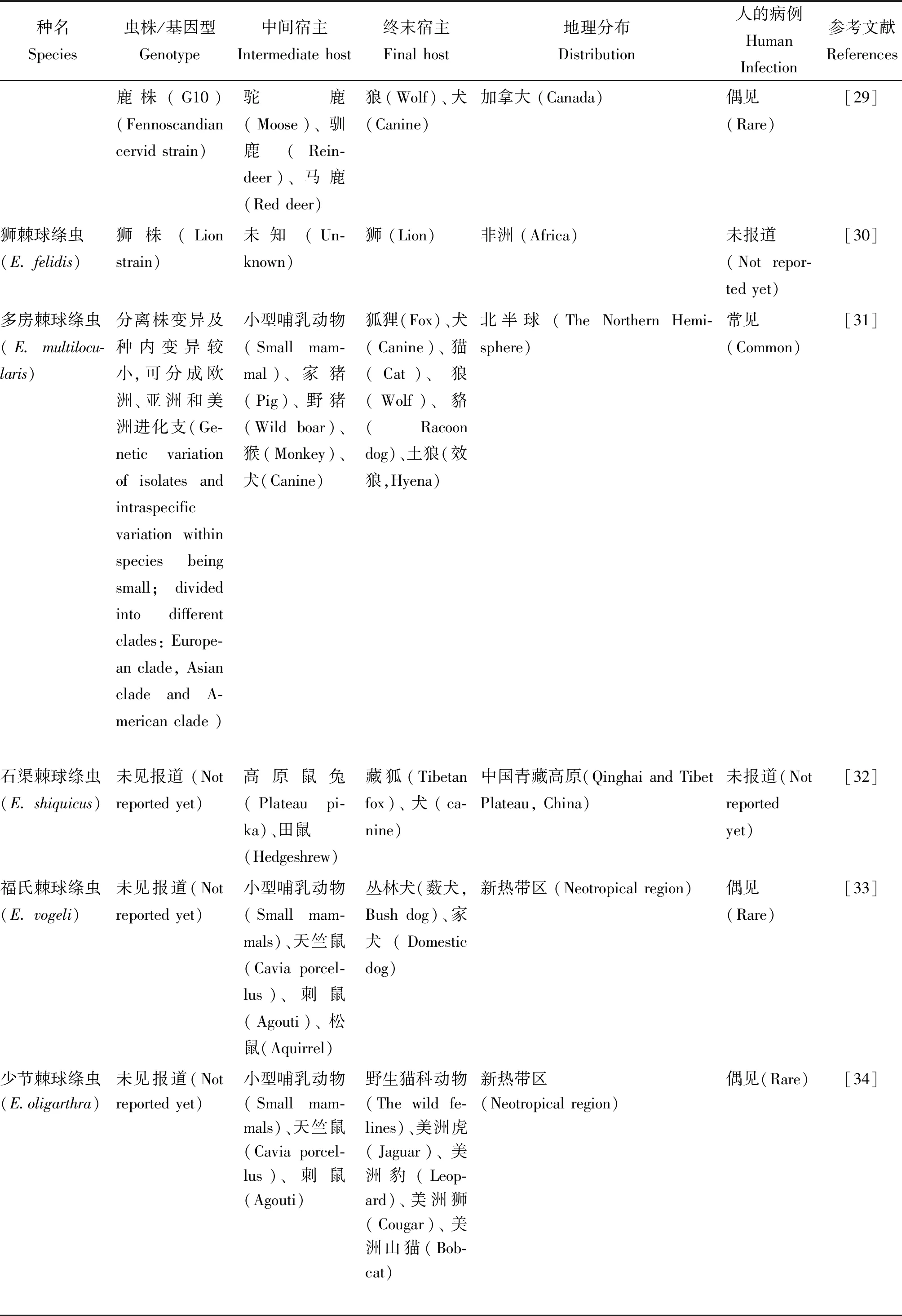
种名Species虫株/基因型Genotype中间宿主Intermediate host终末宿主Final host地理分布Distribution人的病例Human Infection参考文献References鹿株(G10)(Fennoscandian cervid strain)驼鹿(Moose)、驯鹿(Rein-deer)、马鹿(Red deer)狼(Wolf)、犬(Canine)加拿大 (Canada)偶见(Rare)[29]狮棘球绦虫(E. felidis)狮株(Lion strain)未知 (Un-known)狮 (Lion)非洲 (Africa)未报道(Not repor-ted yet)[30]多房棘球绦虫(E.multilocu-laris)分离株变异及种内变异较小,可分成欧洲、亚洲和美洲进化支(Ge-netic variation of isolates and intraspecific variation within species being small; divided into different clades: Europe-an clade, Asian clade and A-merican clade )小型哺乳动物(Small mam-mal)、家猪(Pig)、野猪(Wild boar)、猴(Monkey)、犬(Canine)狐狸(Fox)、犬(Canine)、猫(Cat)、狼(Wolf)、貉(Racoon dog)、土狼(效狼,Hyena)北半球 (The Northern Hemi-sphere)常见(Common)[31]石渠棘球绦虫(E. shiquicus)未见报道 (Not reported yet)高原鼠兔(Plateau pi-ka)、田鼠(Hedgeshrew)藏狐(Tibetan fox)、犬(ca-nine) 中国青藏高原(Qinghai and Tibet Plateau, China)未报道(Not reported yet)[32]福氏棘球绦虫(E. vogeli)未见报道(Not reported yet)小型哺乳动物(Small mam-mals)、天竺鼠(Cavia porcel-lus)、刺鼠(Agouti)、松鼠(Aquirrel)丛林犬(薮犬,Bush dog)、家犬(Domestic dog)新热带区 (Neotropical region)偶见(Rare)[33]少节棘球绦虫(E.oligarthra)未见报道(Not reported yet)小型哺乳动物(Small mam-mals)、天竺鼠 (Cavia porcel-lus)、刺鼠(Agouti)野生猫科动物(The wild fe-lines)、美洲虎(Jaguar)、美洲豹(Leop-ard)、美洲狮(Cougar)、美洲山猫(Bob-cat)新热带区(Neotropical region)偶见(Rare)[34]
3 种系发生关系
DNA序列信息已成为种系发生分析的先决条件,其中线粒体DNA序列由于受到线粒体内氧化环境的影响而容易发生突变,因此其进化速率快,在构建亲缘关系相近物种的进化树时比使用核DNA序列更有优势[36]。大量线粒体基因(组)序列信息已经为提高种系进化树的分辨率积累了重要信息(见表2)。棘球属绦虫线粒体基因组核苷酸序列差异性比较结果见表3。

表2 棘球属绦虫线粒体基因组信息汇总
注:*cox1基因3′末端注释有疑问。
Note: *The annotation of 3′ end ofcox1 gene is in doubt.
4 种系进化史
棘球绦虫具有独特的繁殖方式:成虫为雌雄同体,以自体受精为主;中绦期(幼虫或者蚴)行无性繁殖。棘球绦虫这种繁殖形式造就了它的生物多样性,这种双向发育和繁殖模式加速了遗传一致性种群的形成[6]。一个突变体变为一克隆种群的趋势将
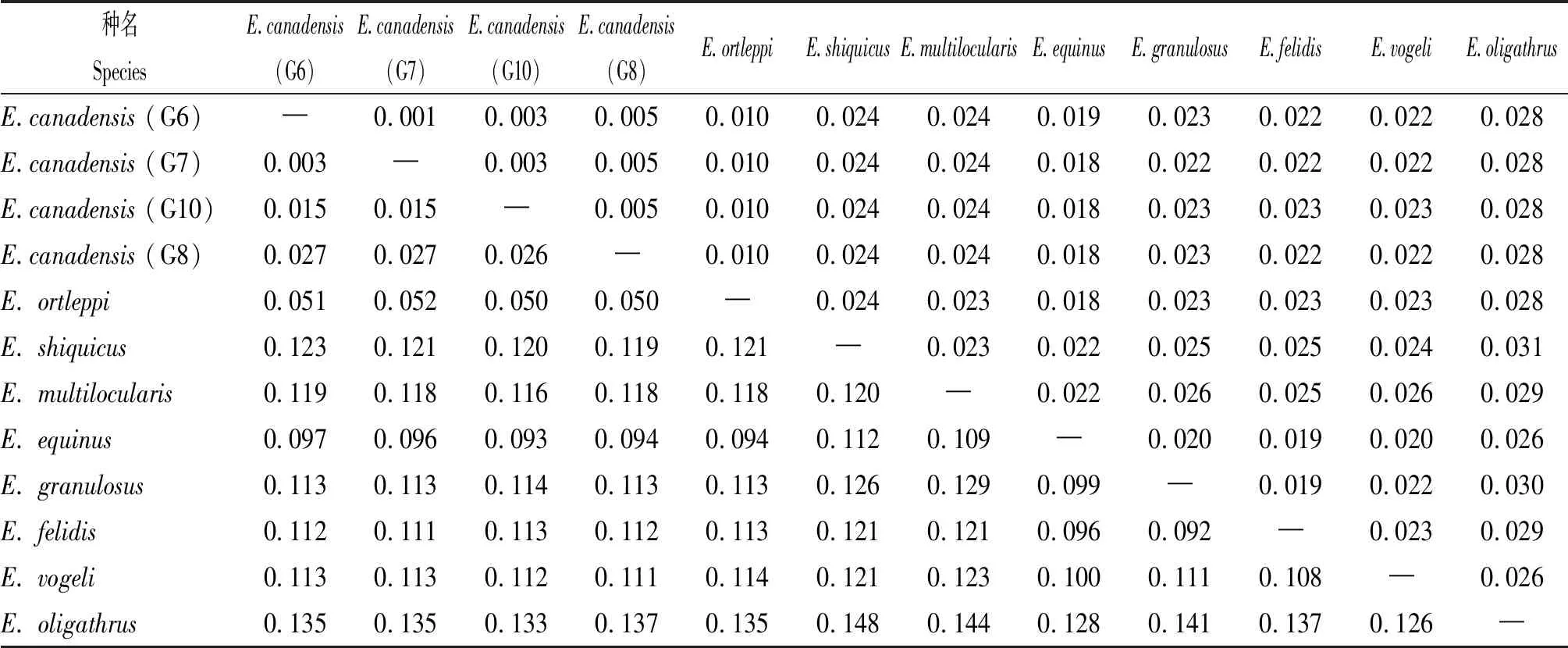
表3 棘球属绦虫线粒体蛋白编码基因核苷酸序列差异比较
影响棘球绦虫种的形成。因此,棘球绦虫成功自适应突变体所需宿主在两个发育阶段——成虫和幼虫间的切换可能会出现,而宿主可能对变异体的成功形成起到适应性选择作用。
根据线粒体基因组12个蛋白编码基因核苷酸序列第1位和第2位碱基构建的棘球属绦虫分子进化树见图1(根据线粒体12个蛋白编码基因核苷酸序列用MEGA5软件的邻接法绘制),其拓扑结构与Nakao等人构建的系统发生图相似[6]。
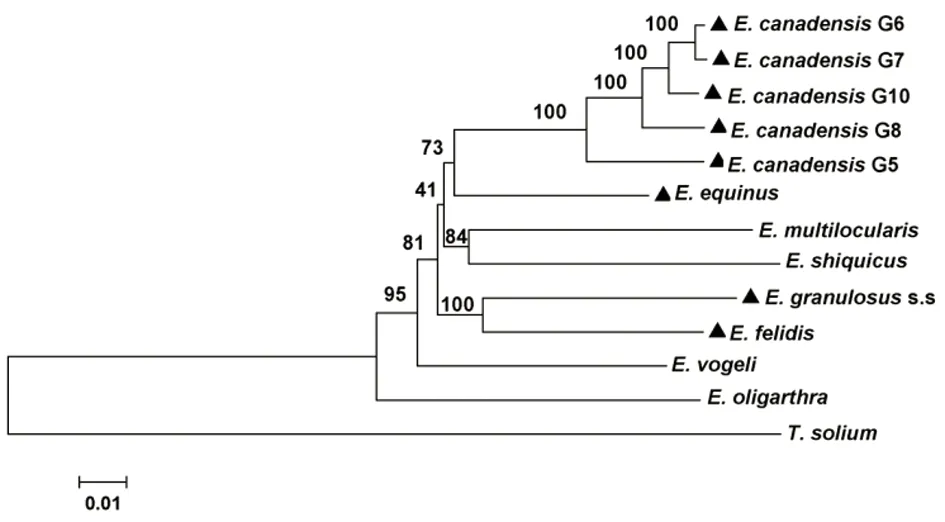
图1 棘球属绦虫分子种系发生树
分子进化树显示:(1)E.oligarthra和E.vogeli位于进化树基部,这两种棘球绦虫分布于新热带区(Neotropical region);(2)少节棘球绦虫与棘球属其它成员呈姊妹关系;(3)多房棘球绦虫与石渠棘球绦虫之间,细粒棘球绦虫狭义种和狮棘球绦虫之间,奥氏绦虫和加拿大绦虫之间分别呈姊妹关系;(4)马棘球绦虫与加拿大棘球绦虫、奥氏棘球绦虫、多房棘球绦虫及石渠棘球绦虫形成一进化支;(5)加拿大棘球绦虫G6-G10基因型之间亲缘关系密切;(6)多房棘球绦虫和石渠棘球绦虫组成的节点与细粒棘球绦虫G4-G10基因型组成的节点之间的自展检验值比较低。
当然,只用线粒体基因组数据构建分子进化树也存在对真正进化史误解的风险,这是因为行母性遗传的线粒体DNA并不与物种真实的进化史相吻合或者相匹配[37]。为此,除用线粒体DNA作为进化的分子标记外,研究者还用多个核基因DNA序列分子标记串联后构建了棘球绦虫的分子进化树[43]。这些核基因DNA分子标记包括elp(Ezrin-radixin-moesin-like protein,埃兹-根蛋白-膜突蛋白样蛋白)、ef1α(Elongation factor 1 alpha,延伸因子1α)、pepck(Phosphoenolpyruvate carboxykinase,磷酸烯醇式丙酮酸羧激酶)、polδ(DNA polymerase delta,DNA 聚合酶δ)等[20]。结果表明,核基因DNA分子进化拓扑结构总体上与线粒体基因组序列构建的进化树的拓扑结构相似,但是核基因DNA分子进化树的最显著的特征是少节棘球绦虫和伏氏棘球绦虫不仅位于树的基部,而且分布于新热带的这两个种之间呈现姊妹关系。这种分子种系发生分析结果似乎可以得出一可信的假设:几种猫科动物作为少节棘球绦虫的终末宿主,而薮犬(Bush dog,学名Speothosvenaticus)作为伏氏棘球绦虫的终末宿主,这些食肉动物作为外来“移民”源从北美洲,棘球绦虫的祖先种显然是随着这些猫科动物或者犬科动物宿主侵入南美洲。化石记录表明,除了浣熊外再没有其它有胚盘的食肉动物在南美洲一带扩张过。北美洲食肉动物大扩散好像发生在早更新世,它们进化为南美洲本地食肉动物种群。但是,最新的分子进化研究表明,美洲本地新热带区猫科动物的一支——虎猫属(Leopardus)是更早时候从其它主要猫科谱系衍化来的,这意味着这些小型猫科动物很可能在巴拿马地峡出现前就侵入了南美洲。只不过,巴拿马大陆桥的形成可能使得虎猫属动物物种的进一步分化变得更为便利。正如这一例子所揭示的那样,食肉动物入侵新热带区的时间节点以及接下来发生的动物区系变化对分析棘球绦虫种系进化至关重要。假设棘球绦虫新热带区种的分化和食肉动物的扩张与巴拿马大陆桥介入动物迁徙相一致,将发生在约三百万年前的美洲动物大交换作为棘球绦虫核基因DNA分子进化的关键时间校准点,以新热带区的两种棘球绦虫最近的共同祖先存在时间定于三百万年前构建分子钟,那么这些推测会表明棘球绦虫在第三纪中新世结束时(五百八十万年前)已经开始分化。
棘球属绦虫为单系群,在形态学、发育和遗传学组成方面极其相似,关于它的祖先以及带科绦虫中棘球属绦虫和带属(Taenia)中的内鼬带绦虫(T.mustela)比较生态学、生理学和遗传学关系的科学揭示将对严重危害人和动物健康的棘球属绦虫的进化史的认识至关重要。
5 小 结
棘球属绦虫分子种系发生、分子分类学和分子进化研究尚处于初始阶段,研究方法和手段还不成熟,但是这方面工作的开展无疑会对揭示寄生虫和宿主相互关系框架的创立起始动和推动作用,对棘球蚴病和其它带科绦虫蚴病的防控提供理论指导和奠定基础。
参考文献:
[1]Brunetti E, Kern P, Vuitton DA. Expert consensus for the diagnosis and treatment of cystic and alveolar echinococcosis in humans[J]. Acta Trop, 2010, 114: 1-16. DOI: 10.1016/j.actatropica.2009.11.001
[2]Craig PS. Epidemiology of human alveolar echinococcosis in China[J]. Parasitol Int, 2006, 55: S221-S225. DOI: 10.1016/j.parint.2005.11.034
[3]Beaver PC, Jung RC, Cupp EW. Clinical parasitology[M]. 9th ed. Philadelphia: Lea & Febiger, 1984: 544-547.
[4]McManus DP, Rishi AK. Genetic heterogeneity withinEchinococcusgranulosus: isolates from different hosts and geographical areas characterized with DNA probes[J]. Parasitology, 1989, 99: 17-29.
[5]Bowles J, McManus DP. NADH dehydrogenase 1 gene sequences compared for species and strains of the genusEchinococcus[J]. Int J Parasitol, 1993, 23: 969-972. DOI: 10.1016/0020-7519(93)90065-7
[6]Nalao M, Lavikainen A, Yanagida T, et al. Phylogenetic systematics of the genusEchinococcus(Cestoda: Taeniidae)[J]. Int J Parasitol, 2013, 43: 1017-1029. DOI: 10.1016/j.ijpara.2013.06.002
[7]Lemey P, Salemi M, Vandamme AM. The phylogenetic handbook: A practical approach to phylogenetic analysis and hypothesis testing[M]. Cambridge: Cambridge University Press, 2009.
[8]Templeton AR. Population genetics and microevolutionary theory[M]. New Jersey: John Wiley & Sons, Inc., 2006.
[9]De Queiroz K. Ernst Mayr and the modern concept of species[J]. Proc Natl Acad Sci U S A, 2005, 102(Suppl 1): 6600-6607. DOI: 10.1073/pnas.0502030102
[10]Nixon KC, Wheeler QD. An amplification of the phylogenetic species concept[J]. Cladistics, 1990, 6: 211-223. DOI: 10.1111/j.1096-0031.1990.tb00541.x
[11]Eckert J, Gemmell MA, Meslin FX, et al. WHO/OIE manual on echinococcosis in humans and animals: A public health problem of global concern[M]. Paris: World Organization for Animal Health, 2001.
[12]D’alessandro A, Rausch RL. New aspects of neotropical polycystic (Echinococcusvogeli) and unicystic (Echinococcusoligarthrus) echinococcosis[J]. Clin Microbiol Rev, 2008, 21: 380-401. DOI: 10.1128/CMR.00050-07
[13]Huttner M, Romig T.Echinococcusspecies in African wildlife[J]. Parasitology, 2009, 136(10): 1089-1095. DOI: 10.1017/S0031182009990461
[14]Thompson RC. The taxonomy, phylogeny and transmission ofEchinococcus[J]. Exp Parasitol, 2008, 119(4): 439-446. DOI: 10.1016/j.exppara.2008.04.016
[15]Nakao M, Yanagida T, Okamoto M, et al. State-of-the-artEchinococcusandTaenia: phylogenetic taxonomy of human-pathogenic tapeworms and its application to molecular diagnosis[J]. Infect Genet Evol, 2010, 10(4): 444-452. DOI: 10.1016/j.meegid.2010.01.011
[16]Busi M, Snabel V, Varcasia A, et al. Genetic variation within and between G1 and G3 genotypes ofEchinococcusgranulosusin Italy revealed by multilocus DNA sequencing[J]. Vet Parasitol, 2007, 150(1-2): 75-83. DOI: 10.1016/j.vetpar.2007.09.003
[17]Varcasia A, Canu S, Kogkos A, et al. Molecular characterization ofEchinococcusgranulosusin sheep and goats of Peloponnesus, Greece[J]. Parasitol Res, 2007, 101(4): 1135-1139. DOI: 10.1007/s00436-007-0568-x
[18]Rinaldi L, Maurelli MP, Capuano F, et al. Molecular update on cystic echinococcosis in cattle and water buffaloes of southern Italy[J]. Zoonoses Public Health, 2008, 55: 119-123. DOI: 10.1111/j.1863-2378.2007.01101.x
[19]Nakao M, McManus DP, Schantz PM, et al. A molecular phylogeny of the genusEchinococcusinferred from complete mitochondrial genomes[J]. Parasitology, 2007, 134(Pt 5): 713-722. DOI: 0.1017/S0031182006001934
[20]Saarma U, Jogisalu I, Moks E, et al. A novel phylogeny for the genusEchinococcus, based on nuclear data, challenges relationships based on mitochondrial evidence[J]. Parasitology, 2009, 136(3): 317-328. DOI: 10.1017/S0031182008005453
[21]Vural G, Baca AU, Gauci CG, et al. Variability in theEchinococcusgranulosuscytochrome Coxidase 1 mitochondrial gene sequence from livestock in Turkey and a reappraisal of the G1-3 genotype cluster[J]. Vet Parasitol, 2008, 154: 347-350. DOI: 10.1016/j.vetpar.2008.03.020
[22]Abushhewa MH, Abushhiwa MH, Nolan MJ, et al. Genetic classification ofEchinococcusgranulosuscysts from humans, cattle and camels in Libya using mutation scanning-based analysis of mitochondrial loci[J]. Mol Cell Probes, 2010, 24: 346-351. DOI: 10.1016/j.mcp.2010.07.005
[23]Casulli A, Manfredi MT, La Rosa G, et al.EchinococcusortleppiandE.granulosusG1, G2 and G3 genotypes in Italianbovines[J]. Vet Parasitol, 2008, 155: 168-172. DOI: 10.1016/j.vetpar.2008.04.004
[24]Le TH, Pearson MS, Blair D, et al. Complete mitochondrial genomes confirm the distinctiveness of the horse-dog and sheep-dog strains ofEchinococcusgranulosus[J]. Parasitology, 2002, 124: 97-112. DOI: 10.1017/0031182001008976
[25]Thompson RC, McManus DP. Towards a taxonomic revision of the genusEchinococcus[J]. Trends Parasitol, 2002, 18: 452-457. DOI: 10.1016/S1471-4922(02)02358-9
[26]Schneider R, Gollackner B, Schindl M, et al.EchinococcuscanadensisG7 (pig strain): an underestimated cause of cystic echinococcosis in Austria[J]. Am J Trop Med Hyg, 2010, 82: 871-874. DOI: 10.4269/ajtmh.2010.09-0639
[27]Thompson RC, Boxell AC, Ralston BJ, et al. Molecular and morphological characterization ofEchinococcusin cervids from North America[J]. Parasitology, 2006, 132: 439-447. DOI: 10.1017/S0031182005009170
[28]Scott JC, Stefaniak J, Pawlowski ZS, et al. Molecular genetic analysis of human cystic hydatid cases from Poland: identification of a new genotypic group (G9) ofEchinococcusgranulosus[J]. Parasitology, 1997, 114: 37-43. DOI:10.1017/S0031182096008062
[29]Schurer J, Shury T, Leighton F, et al. Surveillance forEchinococcuscanadensisgenotypes in Canadian ungulates[J]. Int J Parasitol Parasit Wildl, 2013, 2: 97-101. DOI: 10.1016/j.ijppaw.2013.02.004
[30]Huttner M, Nakao M, Wassermann T, et al. Genetic characterization and phylogenetic position ofEchinococcusfelidis(Cestoda: Taeniidae) from the African lion[J]. Int J Parasitol, 2008, 38: 861-868. DOI: 10.1016/j.ijpara.2007.10.013
[31]Davidson RK, Romig T, Jenkins E, et al. The impact of globalisation on the distribution ofEchinococcusmultilocularis[J]. Trends Parasitol, 2012, 28: 239-247. DOI: 10.1016/j.pt.2012.03.004
[32]Xiao N, Qiu J, Nakao M, et al.Echinococcusshiquicusn. sp., a taeniid cestode from Tibetan fox and plateau pika in China[J]. Int J Parasitol, 2005, 35: 693-701. DOI: 10.1016/j.ijpara.2005.01.00
[33]Santos GB, Soares Mdo C, de F Brito EM, et al. Mitochondrial and nuclear sequence polymorphisms reveal geographic structuring in Amazonian populations ofEchinococcusvogeli(Cestoda: Taeniidae)[J]. Int J Parasitol, 2012, 42: 1115-1118. DOI: 10.1016/j.ijpara.2012.10.010
[34]D’Alessandro A, Rauschr L. Erroneous reports of the neotropicalEchinococcusoligarthrusas a cause of echinococcosis in India[J]. J Parasitol, 2004, 90: 202-203. DOI: 10.1645/GE-3207CC
[35]Nakao M, Lacikainen A, Iwaki T, et al. Molecular phylogeny of the genusTaenia(Cestoda: Taeniidae): proposals for the resurrection of Hydatigera Lamarck, and the creation of a new genus Versteria[J]. Int J Parasitol, 2013, 43: 427-437. DOI: 10.1016/j.ijpara.2012.11.014
[36]Brown WM, George M Jr, Wilson AC. Rapid evolution of animal mitochondrial DNA[J]. Proc Natl Acad Sci U S A, 1979, 76: 1967-1971.
[37]Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J].Mol Biol Evol, 2011, 28: 2731-2739. DOI: 10.1093/molbev/msr121
