基于mtDNA COⅠ和Cytb基因序列对南中国海不同海域波纹唇鱼群体遗传多样性的研究
2014-03-29静侯新远尹绍武祝斐贾一何王小军
胡 静侯新远尹绍武祝 斐贾一何王小军
(1. 南京师范大学生命科学学院, 南京 210023; 2. 中国水产科学院南海水产研究所热带水产研究中心, 三亚 572018)
基于mtDNA COⅠ和Cytb基因序列对南中国海不同海域波纹唇鱼群体遗传多样性的研究
胡 静1,2侯新远1尹绍武1祝 斐1贾一何1王小军1
(1. 南京师范大学生命科学学院, 南京 210023; 2. 中国水产科学院南海水产研究所热带水产研究中心, 三亚 572018)
研究以海南陵水、马来西亚、西沙、南沙4个海域共101尾波纹唇鱼作为研究对象, 通过线粒体DNA的COⅠ和Cytb基因序列分析方法对波纹唇鱼进行了遗传多样性研究。经PCR扩增、克隆与序列测定, 分别获得1560 bp COⅠ基因和1141 bp Cytb基因序列。两者多态性遗传参数统计显示, 101尾个体分别存在23 (COⅠ)和30 (Cytb)个变异位点, 分别检测出20 (COⅠ)和27 (Cytb)个单倍型, 总群体单倍型多样性(Hd)分别为0.629 (COⅠ)和0.755 (Cytb), 平均核苷酸差异数(K)分别为1.195 (COⅠ)和1.424 (Cytb), 核苷酸多样性指数(Pi)分别为0.00077 (COⅠ)和0.00126 (Cytb)。分子方差分析(AMOVA)结果分别为26.26% (COⅠ)和4.22% (Cytb)的变异来自群体间, 73.74% (COⅠ)和95.78% (Cytb)的变异来自群体内。同时, 两个基因的单倍型网络图呈星状放射结构, 不同地理来源的单倍型无明显分支, 呈交错分布, 没有体现地理差异性。研究初步认为,波纹唇鱼的遗传多样性处于较低水平, 遗传分化存在但不显著, 该结果可为今后波纹唇鱼的种质资源保护工作提供必要的科学依据。
波纹唇鱼; CO ; CytⅠ b; 遗传多样性; 遗传分化
波纹唇鱼(Cheilinus undulatus)在分类学上隶属鲈形目(Perciformes)、隆头鱼科(Labridae)、唇鱼属(Cheilinus),是大型的珊瑚礁鱼类之一。在我国主要分布在海南的万宁、陵水、三亚等海域, 以及香港、台湾诸岛近海域; 世界范围内, 红海、非洲东岸以及印度洋至太平洋中心较为多见。波纹唇鱼肉质好营养高, 市场需求量大。但由于滥捕滥杀、环境污染等因素, 自然海域波纹唇鱼越来越少, 甚至濒临灭绝。因此, 有关波纹唇鱼的资源保护、遗传资源调查和人工繁殖等工作亟待加强。
本研究以海南陵水、马来西亚、西沙以及南沙4个不同地理群体波纹唇鱼共 101尾个体为研究对象, 对其COⅠ和Cytb基因序列进行扩增、克隆及测序, 目的在于分析波纹唇鱼群体的 COⅠ和 Cytb基因序列变异和序列多态性情况, 研究其遗传多样性以及群体的遗传关系, 为该物种保护学、遗传学研究提供科学资料。
1 材料与方法
1.1 样品采集
野生群体波纹唇鱼分别取自海南陵水(LC)、马来西亚(MC)、西沙(XC)以及南沙(NC)(表1), 各31、20、14、36尾,共101尾个体, 对其编号, 并用95 %乙醇保存备用。

表1 波纹唇鱼样品的基本信息Tab. 1 Basic information of Cheilinus undulatus samples
1.2 DNA提取、扩增、克隆及测序
实验材料为各个体0.1 g尾鳍组织。基因组DNA的提取方法为常规的“酚–氯仿”法[16]。DNA先经冰乙醇沉淀, 后由 70%(体积分数)乙醇洗涤并干燥,最后 用50 μL TE溶解, –20℃保存备用。
用于扩增 COⅠ、Cytb基因的引物设计及合成步骤: 导出 Genbank中波纹唇鱼的 mtDNA全序列(序列号: GU296101)、利用软件Primer primer 5.0进行设计、上海捷瑞生物工程有限公司合成。COⅠ基因序列的两条引物分别为 F1 (5′CAGGATTTC AACCAGGAC 3′), R1 (5′TTTTGGACGTTAAGCTA CTG 3′); Cytb基因序列的两条引物分别为 F2(5′GCCGACTACTTTGGATTT 3′), R2 (5′AGTTGTGAT GGATGTGCC 3′)。
两个 PCR反应的总体积均为 25 μL: 2.5 μL 10×PCR Buffer, 2 μL dNTPs (2 mmol/L), 2 μL镁离子(25 mmol/L), 0.3 μL TaqDNA 聚合酶 (5 U/μL), 正、反向引物各1 μL (10 μmol/L), 模板2 μL, 14.2 μL ddH2O。且PCR程序均为: 94℃预变性4min后进行30个热循环(94℃ 30s, 51℃ 30s, 72℃ 2min), 72℃延伸10min; 10℃保温。
1%琼脂糖凝胶电泳检测纯化后回收扩增产物,将其与pMDTM18-T Vector*1 (宝生物工程有限公司)连接、与 DH5α感受态细胞充分混匀。液体培养基培养单克隆菌落, 后菌液 PCR, 产物送至上海华大基因公司测序。
(1)High speed in the analysis and design(reducing design time and cost).
1.3 数据处理与分析
双向测序结果在BLAST上比对分析、人工完成完整序列拼接; ClustalX 软件上对比文件输入MEGA5.0软件[17]分析依据Kimura 2-parameter计算群体内及群体间的平均遗传距离; 遗传多样性参数,包括核苷酸多样性 Pi以及单倍型多样性 Hd由Dnasp5.0软件[18]计算得出; 由软件 Areliquin3.5[19]进行群体的AMOVA分析; 生成单倍型数目、类型及分布以及pairwise difference模型下的遗传分化参数。应用NETWORKv 4.6软件对波纹唇鱼的2个线粒体基因单倍型进行分析。
2 结果
2.1 COⅠ基因序列的分析
对4个波纹唇鱼群体共101尾个体的COⅠ序列进行测定, 经ClustalX软件编辑和比对后, 获得了长度为1560 bp的同源序列。各群体COⅠ基因序列的A、T、G、C平均含量分别为24.05%、28.37%、18.54%和29.04%, 即A+T(52.42%)>G+C(47.58%)。遗传多样性参数统计显示 101尾个体共有 23个变异位点,占分析位点总数的1.5%, 共检出20个单倍型, 总群体单倍型多样性指数(Hd)为 0.629, 平均核苷酸差异数(K)为 1.195, 核苷酸多样性指数(Pi)为 0.00077(表2), 显示出波纹唇鱼群体的遗传多样性水平偏低。
由AMOVA分析得出的直观结论为群体间存在遗传分化不明显、群体内部分化为变异主导的结果(表 4), 仅26.26%的变异来自群体间而73.74%的来自群体内。群体内及群体间的遗传距离分析结果见表 3, 群体内遗传距离大小次序为: 海南陵水(0.00086)>南沙(0.00079)>西沙(0.00016)>马来西亚(0.00000), 而群体间最大遗传距离为陵水和南沙(0.00101), 略高于群体内的遗传距离。最小为马来西亚和西沙(0.00008)。各群体间的遗传分化指数(Fst)均较低, 支持了AMOVA分析关于群体间遗传分化不明显的结论, 由Nm= [(1/Fst)-1]/2计算得出的基因流均大于4[21], 同样证实了以上结果。
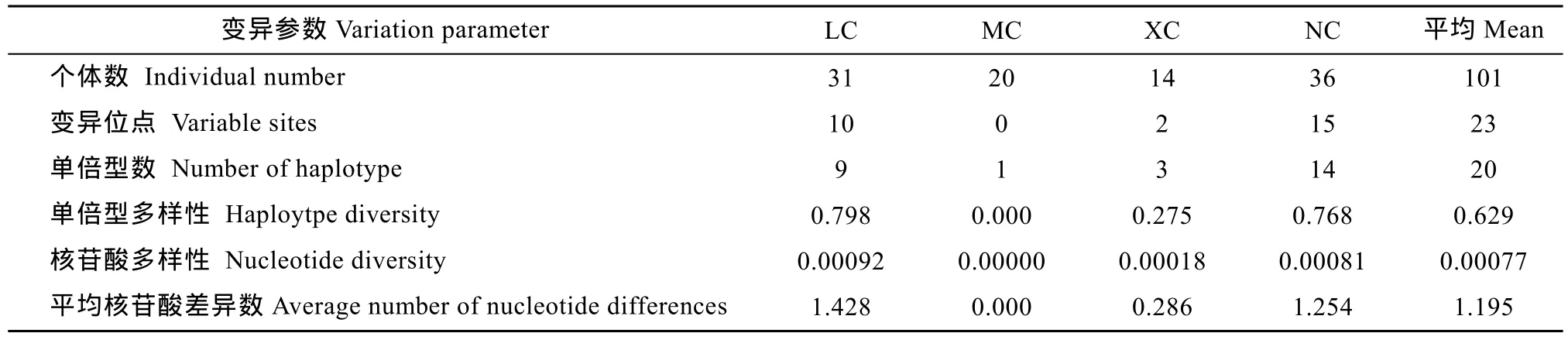
表2 波纹唇鱼4个群体COⅠ基因的遗传多样性参数Tab. 2 Genetic diversity of four Cheilinus undulatus populations inferred from mtDNA COⅠ

表 3 波纹唇鱼4个群体COⅠ基因群体间/群体内的Kimura 2-parameter遗传距离及遗传分化Tab. 3 The average Kimura 2-parameter distance within and between populations and Fstof pairwise difference in CO Ⅰ sequences from four Cheilinus undulatus populations
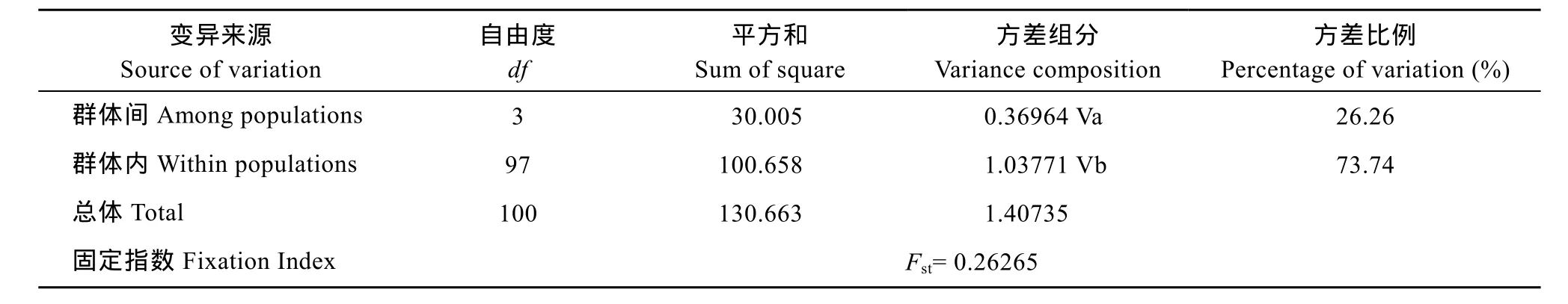
表4 波纹唇鱼4个群体COⅠ基因的AMOVA分析Tab. 4 AMOVA analysis of all sequences from four Cheilinus undulatus populations of mt DNA CO geneⅠ
基于 COⅠ基因序列对波纹唇鱼群体进行单倍型分析, 共得出 20个单倍型, 其中共享单倍型有 4个(HAP1、HAP2、HAP16和HAP20), 所占比例达20.00% (4/20), 其中HAP1分布最广泛, 包括海南陵水10尾个体、马来西亚全部20尾个体、西沙12尾个体以及南沙17尾个体, 比例高达58.42% (59/101), HAP1甚至为马来西亚的唯一单倍型, 除此之外, 其他共享单倍型均为 2种群共享。单倍型分布及频率见表 5。由NETWORKv 4.6软件生成的4个群体COⅠ基因的单倍型网络图呈星状放射结构(图 1), 不同地理来源的单倍型无明显分支, 呈交错分布, 没有体现地理差异性, 其中单倍型 HAP1 位于网络图的中心。
2.2 Cytb基因序列的分析
扩增得到Cytb基因片段长度为1141 bp。经软件Dnasp4.10分析显示101尾个体共有30个变异位点, 27个单倍型, 总群体单倍型多样性指数(Hd)为0.755, 平均核苷酸差异数(K)为 1.424, 核苷酸多样性指数(Pi)为 0.00126 (表6), 显示出波纹唇鱼群体遗传多样性水平不高。
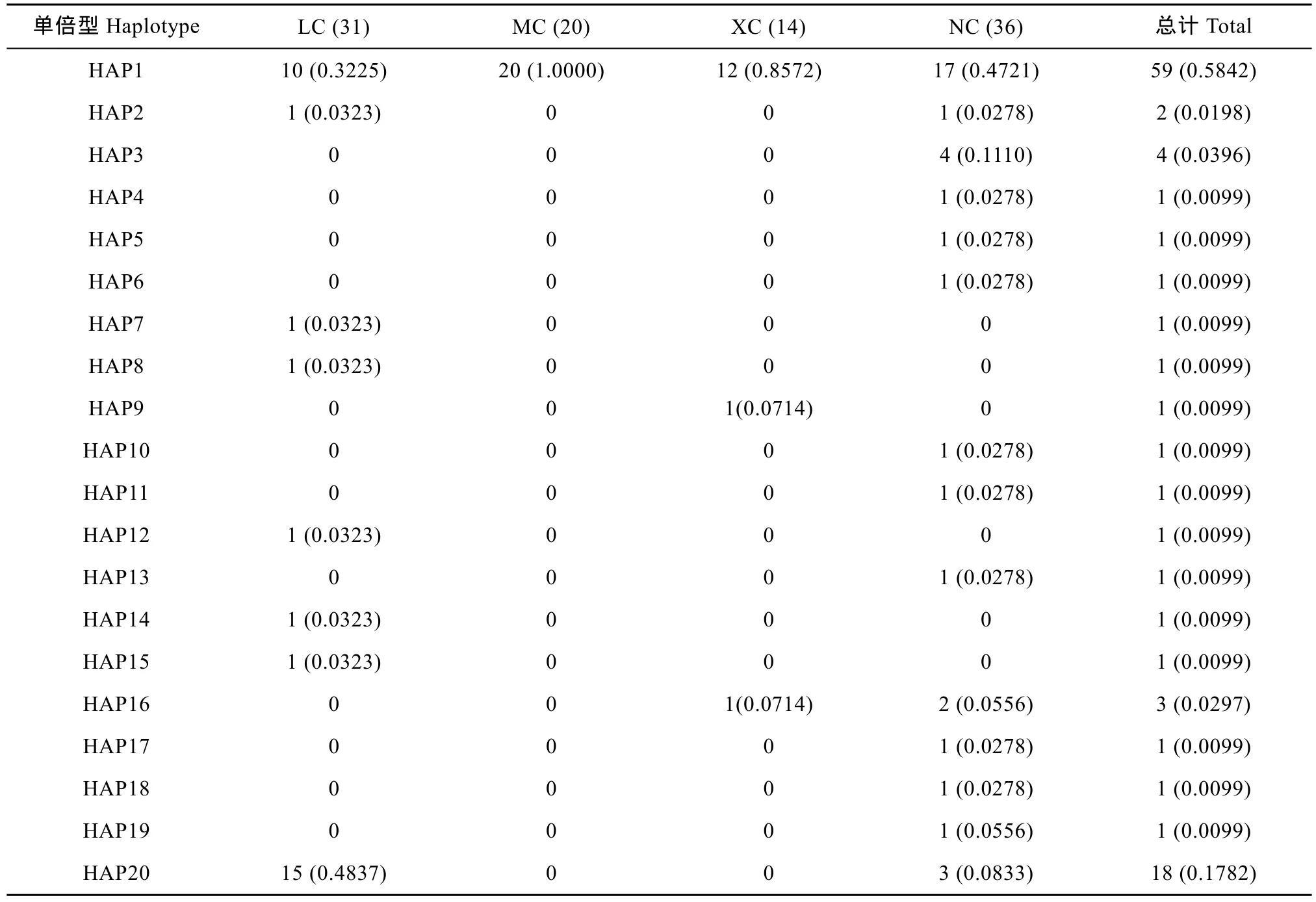
表5 波纹唇鱼4个群体COⅠ基因的单倍型数目类型、频率及分布Tab.5 Haplotype numbers and frequencies in four Cheilinus undulatus populations inferred from mtDNA COⅠ

图1 波纹唇鱼4个群体COⅠ基因单倍型网络图Fig. 1 Haplotype network of Cheilinus undulatus from four populations inferred from mtDNA COⅠ
由AMOVA分析得出的直观结论为群体间存在遗传分化不明显、群体内部分化为变异主导的结果(表8), 仅4.22%的变异来自群体间而95.78%的来自群体内。群体内及群体间的遗传距离分析结果见表7, 群体内遗传距离大小次序为: 南沙(0.00127)>海南 陵 水 (0.00125) >西 沙 (0.00089) >马 来 西 亚(0.00082), 而群体间最大遗传距离为陵水和南沙(0.00128), 略高于群体内的遗传距离。最小为马来西亚和西沙(0.00089)。各群体间的遗传分化指数(Fst)均较低, 支持了AMOVA分析关于群体间遗传分化不明显的结论, 虽然西沙和南沙间的 Fst(1.33236)处于0.05—015以上, 但其P值>0.05 (P=0.61798),即表明两者之间存在遗传分化但不显著。
基于 Cytb 基因序列对波纹唇鱼群体进行单倍型分析, 共得出 27个单倍型, 其中共享单倍型有4个(HAP1、HAP6、HAP9和 HAP19), 所占比例达14.81% (4/27), 其中HAP19分布最广泛, 包括海南陵水13尾个体、马来西亚9尾个体、西沙8尾个体以及南沙14尾个体, 比例高达43.56% (44/101)。其他23个单倍型均为独享单倍型, 单倍型分布及频率见表 9。Cytb基因单倍型网络图结果(图 2)与基于COⅠ基因片段构建的单倍型网络图一致, 没有体现地理差异性。
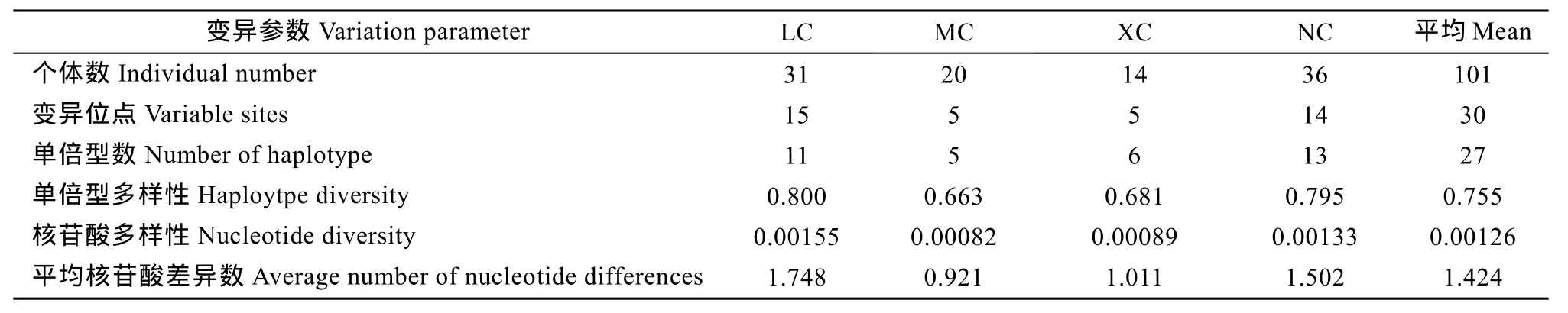
表6 波纹唇鱼4个群体Cytb基因的遗传多样性参数Tab. 6 Genetic diversity of four Cheilinus undulatus populations inferred from mtDNA Cytb
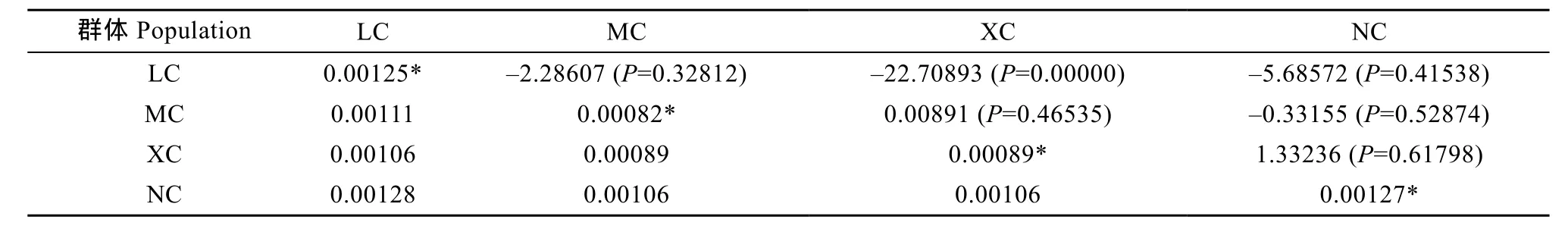
表7 波纹唇鱼4个群体Cytb基因群体间/群体内的Kimura 2-parameter遗传距离及遗传分化Tab.7 The average Kimura 2-parameter distance within and between populations and Fstof pairwise difference in Cytb sequences from four Cheilinus undulatus populations
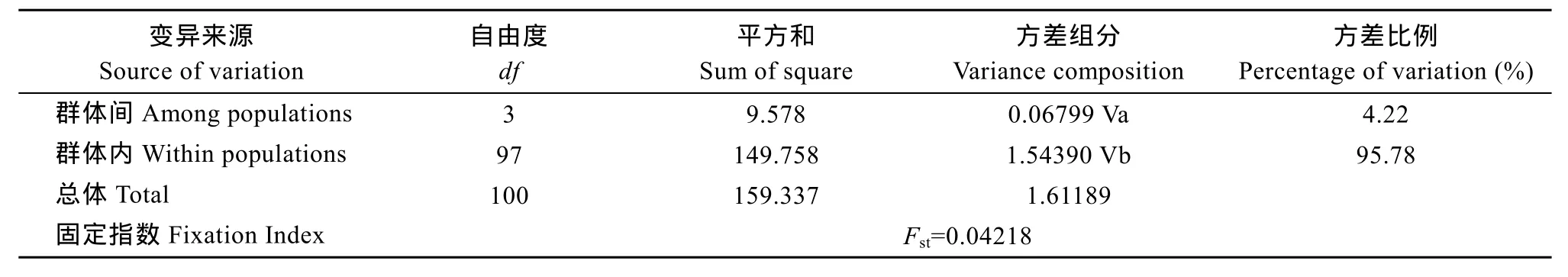
表8 波纹唇鱼4个群体Cytb基因的AMOVA分析Tab. 8 AMOVA analysis of all sequences from four Cheilinus undulatus populations of mt DNA Cytb gene
3 讨论
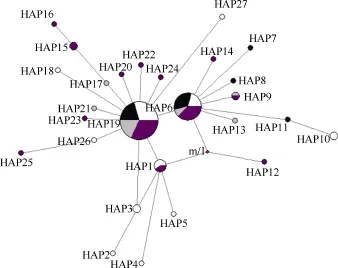
图2 波纹唇鱼4个群体Cytb基因单倍型网络图Fig. 2 Haplotype network of Cheilinus undulatus from four populations inferred from mtDNA Cytb
3.1 二个线粒体基因序列特征分析
2个线粒体基因分析得出的序列碱基含量均为A+T>G+C, Folmer等[22]认为A+T 含量的增加也增加了密码子第三位点上 A与 T颠换的可能性, 即A+T的含量越高, 线粒体DNA的进化地位也高, 具有明显进化优势。动物线粒体基因组4种核苷酸的特性为不均一分布, 该研究结果体现了这一点。同时各群体 COⅠ基因序列的 A+T (52.42%)>G+C (47.58%), 与吴宁等[24]对6种鳗鲡COⅠ片段的研究结果相似[24]。各群体 Cytb基因序列的碱基含量结果A+T (50.82%)>G+C (49.18%), 与怒江濒危鱼类缺须盆唇鱼[25]的结果相似, 而与闽浙地区香鱼的研究结果[26]相反。可见, 不同的物种A+T与G+C的含量是不对等的。
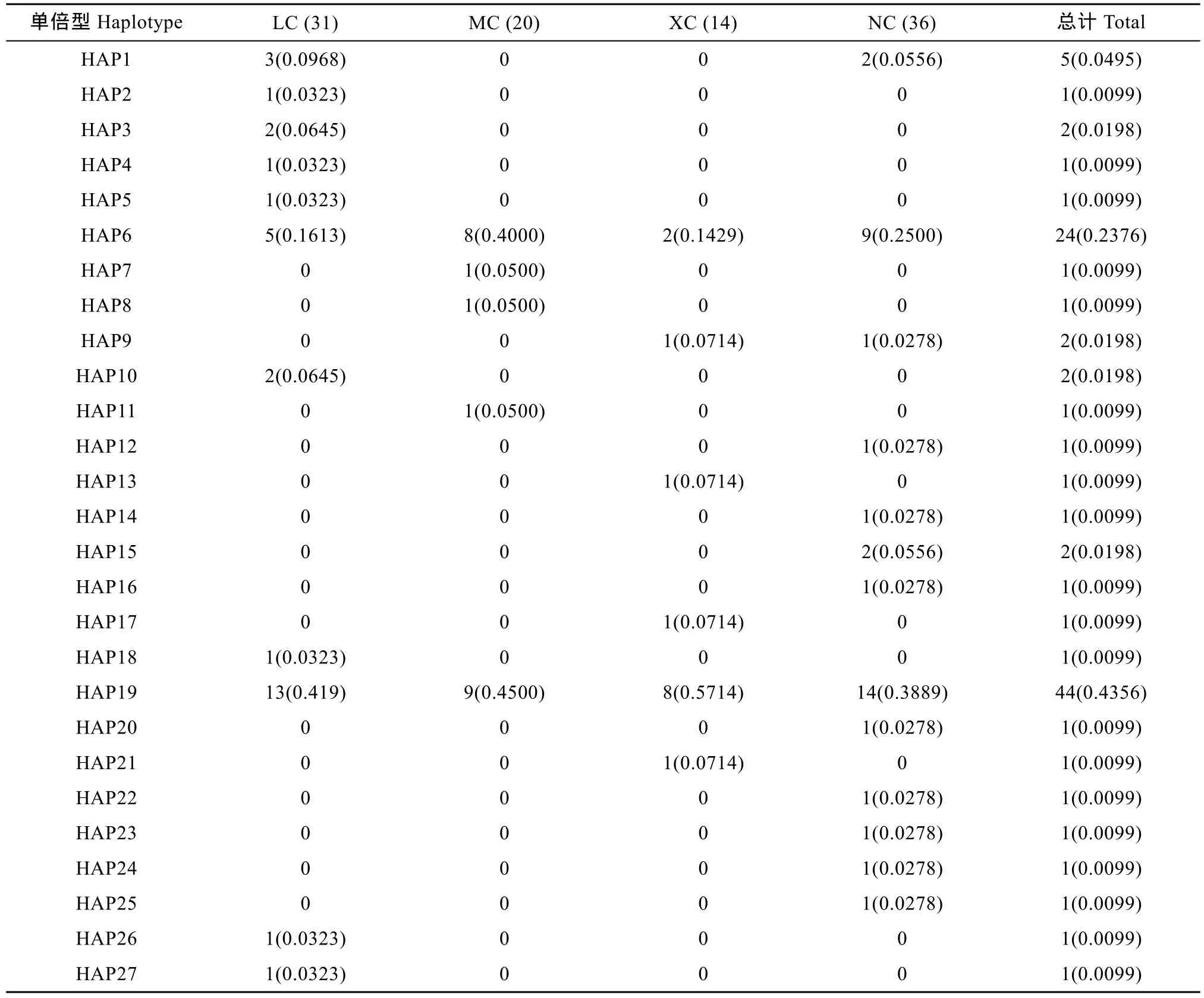
表9 波纹唇鱼4个群体Cytb基因的单倍型数目类型、频率及分布Tab. 9 Haplotype numbers and frequencies in four Cheilinus undulatus populations inferred from mtDNA Cytb
3.2 遗传多样性以及群体间的遗传关系分析
本研究选取的2个mtDNA基因COⅠ和Cytb序列, 通过软件计算得出的整体水平的遗传多样性参数S、Hd、K以及Pi, 结果表明COⅠ和Cytb的这些参数几乎在同一水平上, COⅠ略小于Cytb, 证实了Meyer等[13]基于13个蛋白编码mtDNA所含系统发育信息丰度的划分, 即均为系统发育信息良好的一组, 其中包括Cytb、ND2、ND4、ND5和COⅠ。并且这 2个基因分析所得的遗传分化及单倍型分布、网络图等参数基本在同一水平上, 都大同小异,即由 Fst及其 P值提示群体间存在分化, 但不显著;单倍型在个体间均匀分布, 无明显分支, 存在占有较大比例的单倍型共享型。因此, Cytb和COⅠ2个基因均较适于分析群体的遗传结构及系统发育。
遗传多样性即基因多样性, 存在于生物个体内、单个物种内以及物种间, 是适应性和进化的基础, 种内的遗传多样性与该物种对环境的适应能力呈正向线性关系。单倍型间的平均遗传距离(K)和核苷酸多样性(Pi)是衡量遗传多样性2个重要指标, 而由于Pi值考虑各种mtDNA单倍型在群体中所占的比例的特性, 故其更能精确的揭示一个群体的mtDNA的多态程度[27]。Pi值越大表示群体多态程度越高, 反之亦然, 是衡量群体多态程度和群体遗传分化的重要指标之一。当Pi值在0.0015—0.0047[28]时, 则表明遗传多样性处于较低水平。本研究中由COⅠ、Cytb序列得出的整体水平 Pi值分别为0.00077和 0.00126, 均低于此范围, 表明波纹唇鱼的遗传多样性较低。首先, 波纹唇鱼自身的建群效应(Founder Effect)或种类差异可能导致上述现象,而人为因素更不可忽视。如人为的栖息地破坏, 尤其是对其栖息地珊瑚礁的破坏。作为一种大型珊瑚礁鱼类, 其栖息地珊瑚礁的大面积的污染, 对该物种产生了致命的杀伤力。同时越来越先进的捕捞技术和高杀伤力渔具也导致了其不可恢复的损伤, 甚至濒临消失。本文的研究对象仅为南中国海域的野生波纹唇鱼, 由于珊瑚礁的破坏导致波纹唇鱼的生活环境受到限制, 在一定程度上起到了生殖隔离的作用, 有一定的参考价值。但并不能忽略生殖交流对本结果的影响, 因此有必要进一步对其他海域如非洲东海、红海等的波纹唇鱼进行遗传多样性分析。
3.3 单倍型分布分析
溯祖理论[29]认为分布最广泛的单倍型为祖先类型, 而从COⅠ基因单倍型分析结果可看出HAP1为这4个野生波纹唇鱼群体的主导单倍型。因此推测HAP1可能为这4个群体波纹唇鱼的起源, 而HAP1又为马来西亚群体全部个体单倍型, 由此可预测波纹唇鱼的起源地可能为马来西亚海域。同时, 在COⅠ基因单倍型构建的网络图中HAP1位于网络图的中心, 可推测这个单倍型为较为原始的单倍型, 其他单倍型由其衍生而来。而由COⅠ和Cytb基因序列对这4个群体的单倍型分析结果中均存在独享单倍型, 暗示着群体间一定存在程度的遗传分化。
结合COⅠ和Cytb基因序列分析得出的波纹唇鱼101尾个体单倍型网络图可以看出: 4个地理群体的个体交错在一起, 没有体现明显的地理差异性,单倍型分布 无明显分支, 在个体间呈均匀交错分布, 这也暗示了群体间遗传分化水平低, 进行了广泛的基因交流。
利用线粒体 COⅠ和 Cytb基因作为分析手段,本研究对海南陵水、马来西亚、西沙和南沙4个不同地理群体的波纹唇鱼进行了遗传多样性和遗传分化研究。结果表明, 波纹唇鱼的遗传分化存在但不显著, 且遗传多样性处于较低水平。受制于取材, 本研究不能完全反应该物种群体分化程度和遗传多样性水平, 也不能衡量人为因素对其遗传多样性的影响, 但一定程度上为波纹唇鱼的种质资源保护工作提供了科学依据, 也为后续的研究提供了参考资料。
[1] Shen S J. Fishes of Taiwan [M]. Taipei: Department of Animal Science at Taiwan University. 1993, 143—144 [沈世杰. 台湾鱼类志. 台北: 台湾大学动物学系. 1993, 143—144]
[2] Jie X Y, Li C H, Xu Z N, et al. Industrial aquaculture of famous and expensive marine fishes [J]. Hunan Agricultural Sciences, 2011, (11): 141—143 [颉晓勇, 李纯厚, 许忠能,等. 名贵海水鱼类工厂化养殖试验研究. 湖南农业科学, 2011, (11): 141—143]
[3] Qi X Z, Yin S W, Luo J, et al. Complete mitochondrial genome sequence of the humphead wrasse, Cheilinus undulatus [J]. Genetics and Molecular Research, 2013, 12(2): 1095—1105
[4] Qu Y J, Li J E. Analysis and evaluation of nutrition composition of double-headed parrotfish Cheilinus undulatus [J]. Journal of Tropical Oceanography, 2010, 29(3): 97—102 [区又君, 李加儿. 野生波纹唇鱼 Cheilinus undulatus 营养成分分析与评价. 热带海洋学报, 2010, 29(3): 97—102]
[5] Donaldson T J, Sadovy Y. Threatened fishes of the world: Cheilinus undulatus RÜppell, 1835 (Labridae) [J]. Environmental Biology of Fishes, 2001, 62: 428
[6] Sadovy Y, Kulbicki M, Labrosse P, et al. The humphead wrasse, Cheilinus undulatus: synopsis of a threatened and poorly known giant coral reef fish [J]. Reviews in Fish Biology and Fisheries, 2003, 13: 327—364
[7] Hu J, Qi X Z, Yin S W, et al. The variation analysis of mtDNA D-loop sequence of Cheilinus undulatus [J]. Marine Sciences, 2012, 35(4): 50—56 [胡静, 齐兴柱, 尹绍武, 等.波纹唇鱼mtDNA D-loop序列变异分析. 海洋科学, 2012, 35(4): 50—56]
[8] Hu J, Zhu X P, Yin S W, et al. Development and characterization of microsatellite loci in a threatened marine fish, Cheilinus undulatus (humphead wrasse) [J]. Genetics and Molecular Research, 2013, 12(3): 2633—2636
[9] Guo X H, Liu S J, Liu Q, et al. New progresses on Mitochondrial DNA in fish [J]. Acta Genetica Sinica, 2004, 31(9): 983—1000 [郭新红, 刘少军, 刘巧, 等. 鱼类线粒体DNA研究新进展. 遗传学报, 2004, 31(9): 983—1000]
[10] Liu S F, Chen L L, Dai F Q, et al. Applicaction of DNA Barcoding gene COⅠ for classifying family Sciaenidae [J]. Oceanologia et Limnologia Sinica, 2010, 41(2): 223—232 [柳淑芳, 陈亮亮, 戴芳群, 等. 基于线粒体 COⅠ基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用. 海洋与湖沼, 2010, 41(2): 223—232]
[11] Peng J L, Wang X Z, Wang D, et al. Application of DNA barcoding based on the Mitochondrial CO Ⅰ gene sequences in classification of Culter (Pisces: Cyprinidae) [J]. Acta Hydrobiologica Sinica, 2009, 33(2): 271—276 [彭居俐, 王绪祯, 王丁, 等. 基于线粒体COⅠ基因序列的DNA条形码在鲤科 鲌属鱼类物种鉴定中的应用. 水生生物学报, 2009, 33(2): 271—276]
[12] Irwim D, Kocher T, Wilson A. Evolution of the cytochrome b gene of mammals [J]. Journal of Molecular Evolution, 1991, 32(2): 128—144
[13] Meyer A, Kocher T D, Basasibwaki P, et al. Monophyletic origin of Lake Vietoria cichlid fishes suggested by mitochondrial DNA sequences [J]. Nature, 1990, 347: 550—553
[14] Gao T X, Bi X X, Zhao L L, et al. Population genetic structure of Roughskin sculpin Trachidermus fasciatus based on the mitochondrial Cytb sequence [J]. Acta Hydrobiologica Sinica, 2013, 37(2): 199—207 [高天翔, 毕潇潇, 赵林林, 等. 基于线粒体Cytb基因全序列的松江鲈群体遗传结构分析. 水生生物学报, 2013, 37(2): 199—207]
[15] Lybeard C, Roe K J. The phylogenetic utility of the mitochondrial cytoehrome b gene for inferring relationships among Actinopterygian fishes: In: Koeher T D, Stepien C A (Eds.), Molecular Systematies of Fishes [M]. San Diego: Academic Press. 1997, 285—303
[16] Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning: a Laboratory Manual [M]. Beijing: Science Press. 1995, 27—29 [萨姆布鲁克J, 弗里奇 E F, 曼尼阿蒂斯T. 分子克隆实验指南. 北京: 科学出版社. 1995, 27—29]
[17] Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evolutionary analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Molecular Biology and Evolution, 2011, 28: 2731—2739
[18] Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data [J]. Bioinfomatics, 2009, 25: 1451—1452
[19] Excoffier L, Laval G, Schneider S. Arlequin Ver.3.0: an integrated software package for population genetics data analysis [J]. Evolution Bioinformation, 2005: 47—50
[20] Wright S. The genetical structure of population [J]. Annals of Eugenics, 1951, 15: 323—334
[21] Millar C L, Libby W J. Strategies for Conserving Clinal, Ecotypic, and Disjunct Population Diversity in Widespread Species. In: Fald D A, Holsinger K E (Eds.), Genetics and Conservation of Rare Plants [M]. New York: Oxford University Press. 1991, 149—170
[22] Folmer O, Black M, Hoeh W, et al. DNA primer for amplification of mitochondrial cytochrome coxidase subunitⅠfrom diverse metazoan invertebrates [J].Molecular Marine Biology Biotechnology, 1994, 3(5): 294—299
[23] Brown W M. The Mitochondrial Genome of Animals [M]. New York: Plenum Press. 1985, 102—110
[24] Wu N, Li Z B, Lin X Y, et al. Comparative study on MtDNA CO Ⅰgene fragment of sixAnguilla [J]. Oceanologia et Limnologia Sinica, 2010, 11(41): 930—934 [吴宁, 黎中宝,林小云, 等. 6种鳗鲡(Anguilla) 线粒体 DNA COⅠ序列的比较研究. 海洋与湖沼, 2010, 11(41): 930—934]
[25] Zhang D Y, Wang D Q, Zhang S P, et al. Populationgenetic structure analysis of endangered species Placocheilus cryptonemus in the Nujiang River based on Cytb sequences of mtDNA [J]. Journal of Fishery Sciences of China, 2009, 7(16): 477—485 [张东亚, 汪登强, 张绍平, 等. 怒江濒危鱼类缺须盆唇鱼基于 Cytb序列的群体遗传结构分析. 中国水产科学, 2009, 7(16): 477—485]
[26] Li N, Chen S B, Xie Q L, et al. Polymorphisms of mitochondrial Cytb gene and D-loop region in sweetfish (Plecoglossus altivelis Temminck et Schlegel) from Zhejiang and Fujian Provinces [J]. Hereditas, 2008, 30(7): 919—925 [李娜, 陈少波, 谢起浪, 等. 闽浙地区香鱼线粒体Cytb基因和 D-loop区序列多态性分析. 遗传, 2008, 30(7): 919—925]
[27] Xiao M S, Cui F, Kang J, et al. Analysis on sequence polymorphism of the mitochondrial DNA control region and population genetic diversity of the cultivated and natural Chinese longsnout catfish (Leiocassis longirostris) [J]. Acta Hydrobiologica Sinica, 2013, 37(1): 90—99 [肖明松, 崔峰,康健, 等. 长吻(鱼危)养殖群体与野生群体遗传多样性分析. 水生生物学报, 2013, 37(1): 90—99]
[28] Lan H, Shi L M. The origin and genetic differentiation of native breeds of pigs in south west China: An approach from mitochondrial DNA polymorphism [J]. Biochemistry Genetics, 1993, 31: 51—60
[29] Crandall K A, Templeton A R. Empirical tests of some prediction from coalescent theory with application to intraspecific phylogeny reconstruction [J]. Genetics, 1993, 134(3): 959—969
GENETIC DIVERSITY AND DIVERGENCE OF CHEILINUS UNDULATUS OF DIFFERENT GEOGRAPHIC POPULATIONS OF THE SOUTH CHINA SEA REVEALED BY COⅠ AND CYTB GENE ANALYSES
HU Jing1,2, HOU Xin-Yuan1, YIN Shao-Wu1, ZHU Fei1, JIA Yi-He1and WANG Xiao-Jun1
(1. College of Life Sciences, Nanjing Normal University, Nanjing 210023, China; 2. Tropical Fishes Research and Development Center, South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Sanya 572018, China)
The genetic diversity of Cheilinus undulatus was studied based on the sequence analysis of mtDNA (CO Ⅰand Cytb) of 101 individuals from four geographical populations including Lingshui (n=31), Malaysia (n=20), Xisha (n=14), and Nansha (n=36). Using PCR amplification and sequencing, 1560 bp of CO Ⅰand 1141 bp of Cytb sequences were obtained. For COⅠsequences, the number of haplotype is 20, haplotype diversity was 0.629, the nucleotide diversity was 0.00077, the average nucleotide difference was 1.195, and the number of polymorphic site was 23. For Cytb sequences, the number of haplotype was 27, the haplotype diversity was 0.755, the nucleotide diversity was 0.00126, the average nucleotide difference was 1.424, and the number of polymorphic site was 30. AMOVA showed a higher percentage of 73.74% with-population variation than that of 26.26% among-population variation in CO Ⅰsequences.Similarly, the percentage of 95.78% within-population variations was higher than that of 4.22% the among-population variation in Cytb sequences. The haplotype network of CO Ⅰ and Cytb genes with stellate radiation structure suggested that haplotypes distribution did not have the characteristics of clear geographical differentiation. Genetic diversity was low and the genetic divergence was inconspicuous between populations of C. undulatus. These results may be very useful to protect this species.
Cheilinus undulatus; CO ; CytⅠ b; Genetic diversity; Genetic divergence
Q346+.5
A
1000-3207(2014)06-1008-09
10.7541/2014.149
2013-09-02;
2014-04-06
国家自然科学基金项目(40966003); 海南省自然科学基金(809009); “十一五” 国家科技支撑计划重点项目(2007BAD29B03);江苏省高校优势学科建设工程项目资助
胡静 (1987—), 女, 湖北武汉人; 硕士; 主要从事鱼类种质资源与遗传育种研究。E-mail: hijklnm123@sina.com
尹绍武(1969—), 男, 博士, 教授; 主要从事鱼类种质资源与遗传育种研究。E-mail: yinshaowu@163.com
