基于COⅠ基因的西南大西洋部分经济鱼类DNA条形码鉴定
2014-11-05张馨月张秀梅高天翔
张馨月 刘 岩 张秀梅 高天翔
(1. 中国海洋大学水产学院, 青岛 266003; 2. 中国水产科学研究院 南海水产研究所, 广州 510300)
目前, 国际上广泛使用 FishBase (http://fishbase.org)等鱼类分类检索系统进行鱼类形态学鉴定, 但由于部分地区鱼类开发和研究水平不一, 因此单一的形态学鉴定无法完全满足物种鉴定的需求。DNA序列辅助分类作为一个理想的辅助形态学分类手段应运而生, 迄今已有 30多年历史。2003年, Hebert等[1,2]明确提出了DNA条形码(DNA barcoding)的概念, 即通过使用短的、标准化的基因片段来进行物种鉴定, 以提高真核生物识别的效率, 同时使得DNA序列辅助分类方法得到一定程度的统一, 加快进程发展。目前, DNA条形码技术已经成为一个被广泛接受的物种鉴定工具, 它重点强调了标准化和数据验证[3], 亦被称为 DNA分类学[4]。目前, 线粒体细胞色素 C氧化酶亚基Ⅰ(COⅠ)基因是动物 DNA条形码研究中使用最为广泛的标准基因[5], 因为它基本满足DNA条形码理想序列的3个基本判断标准: (1)序列变异水平适宜; (2)变异区域两端的序列高度保守; (3)序列长度适宜[6]。这一结论在许多鱼类[6,7]、昆虫[8,9]和鸟类[10]等动物中均得到很好验证。
西南大西洋公海渔场涵盖巴西北部至阿根廷南部的南美洲东部沿海, 巴西暖流和福克兰寒流在此交汇产生涡流,为多种海洋渔业资源生物提供产卵场和索饵场。因此, 西南大西洋公海渔场渔业资源丰富, 许多国家、地区的拖网及鱿钓船只在此作业。我国在西南大西洋公海渔场拖网作业始于 2008年, 起步较晚, 因此我国对于该海域的相关研究相对较少, 尤其关于该海域常见渔获种类的研究报道在国内仍属空白。同时, 拖网作业对于物种的选择度不高, 所得渔获物种类十分丰富, 为了更好地认识渔获物的生态习性和经济价值, 正确鉴定所得渔获的种类显得尤为重要。本文旨在针对西南大西洋公海部分拖网渔获物进行DNA条形码研究, 探索其在鱼类辅助物种鉴定中的适用性, 以期填补我国对该海域鱼类DNA条形码相关研究的空白, 为我国远洋拖网渔业发展提供更多的基础资料。
1 材料与方法
1.1 实验材料与形态学鉴定
本研究样本来自于山东荣成某渔业集团有限公司的双拖网生产船在西南大西洋(44.8°—47.0°S, 59.5°—61.0°W)巴塔哥尼亚海域探捕作业生产时随机采集的渔获物, 采集时间为2011年1—4月。
参照文献[11—14]、FishBase(http://fishbase. org/)等进行形态学鉴定, 样品初步判定为 14个物种, 参考综合分类学信息系统(Integrated Taxonomic Information System,ITIS) 和《拉汉世界鱼类名典》[15]对物种的有效名以及分类地位进行确定(表 1)。对每个样品拍照记录, 取背部肌肉组织, 用95%乙醇固定, –20℃保存备用。

1.2 DNA提取、PCR扩增及测序
取约 100 mg的肌肉组织经蛋白酶 K消化后, 参考《分子克隆实验指南》的酚/氯仿抽提法[16], 提取基因组DNA, 于–20℃存放。COⅠ基因序列扩增引物为[6]: F1:5′-TCAACCAACCACAAAGACATTGGCAC-3′ F2: 5′-TC GACTAATCATAAAGATATCGGCAC-3′; R1: 5′-TAGAC TTCTGGGTGGCCA AAGAATCA-3′; R2: 5′-ACTTCAGG GTGACCGAAGAATCAGAA-3′。
将F1与F2等体积混匀标记为引物F, R1与R2等体积混匀标记为引物 R, –20℃保存备用。PCR反应总体积为 25 μL, 其中包含: 超纯水 17.25 μL, 10×Buffer 2.5 μL,dNTPs 2 μL, 5 μmol/ L 浓度的引物各 1 μL, Taq聚合酶0.25 μL和DNA模板 1 μL。PCR反应条件为: 95℃预变性2min; 94℃变性0.5min, 52℃退火0.5min和72℃延伸1min,35个循环; 72℃延伸 10min; 最后保持在 4℃[6]。所有的PCR反应均在Biometra热循环仪上完成。每组PCR反应均设置阴性对照用来检测是否存在污染。扩增产物用1.5%琼脂糖凝胶电泳检测, 目的产物经小量胶回收试剂盒(上海华舜)纯化后, 送上海桑尼生物科技有限公司进行双向测序, 以确保序列的可靠性。
1.3 物种核对与序列分析
利用Dnastar软件包(DNASTAR.Inc., Madison.USA)进行序列比对、拼接, 并辅以手工调整。利用DNASP[17]软件计算序列的变异位点数、单倍型数和单倍型多样性等。用MEGA4.0软件[18]分析核苷酸组成, 基于Kimura-2-parameter双参数模型(K2P)计算遗传距离, 利用邻接法(Neighbor joining)构建分子系统进化树, 经1000次重复抽样(Bootstraps)检测其置信度。
同时将所测得序列与NCBI (National Center for Biotechnology Information)网站以及BOLD参考条码库(Reference Barcode Database)中的同源片段序列进行比对,辅助评估形态学分类的准确性。
2 结果
2.1 形态学鉴定及有效种名的确定
经形态学鉴定, 最终确认的鱼类有效种名及其分类地位如表1 所示。71个样本分属于2纲6目10科14种。通过综合比较, 发现文献[11—14]中相关鱼类的物种信息存在一定差异。因此, 样本的物种鉴定又辅以 FishBase网站, 采用三种资料中一致性良好的物种描述信息进行种类鉴定。并最终参考分类学信息系统(Integrated Taxonomic Information System, ITIS)和《拉汉世界鱼类名典》[15]确定所研究样本的有效种名(图1)。
2.2 COⅠ基因片段序列分析
所测定的71条序列长度均约为652 bp, 没有插入和缺失。辐鳍鱼纲的58条COⅠ序列的平均碱基组成为T:29.7%、C: 28.1%、A: 23.5%、G: 18.8%, 其中, A+T含量(53.2%)高于G+C含量(46.9%); 软骨鱼纲的13条COI序列的平均碱基组成为T: 31.8%、C: 26.2%、A: 25.2%、G:16.9%, 其中A+T含量(57%)明显高于G+C含量(43.1%)。同时, 硬骨鱼纲的GC含量高于软骨鱼纲鳐类的GC含量,这与Ward等[6]的研究结果相一致。各密码子的碱基含量结果显示, 第1密码子位点GC含量(54.3%)显著高于第2和第3密码子位点(44.5%和38.6%), 碱基组成表现出明显偏倚性。
各分类阶元遗传距离(K2P)如表 2所示, 种内遗传距离平均为0.0028, 而同属物种间的遗传距离为0.0335, 约为种内遗传距离的12倍, 这与Hebert等[8]提出的“10×规则”相一致。同样, 科内属间遗传距离平均为 0.1866, 同目的科间平均为 0.2361, 同纲内平均为 0.2369以及不同纲间平均值为 0.3184。由此可见, 遗传距离(K2P)随着分类阶元的提高而增大, 且种以上分类阶元随着其等级的升高, 其遗传距离(K2P)的增长明显减缓。
2.3 DNA条形码序列比对
将本研究获得的COⅠ基因序列与GenBank数据库进行序列比对分析, 结果显示, 除眼点平 鲉(Sebastes oculatus)外, 所有物种均具有一致性大于98%的同源DNA条形码序列, 其中 13个鱼种形态学鉴定结果与一致性最高的比对结果相对应。数据库中与形态鉴定为眼点平鲉(Sebastes oculatus)一致性最高的序列为南太平洋密星平鲉(Sebastes constellatus)(100%), 但后者形态特征与分布情况同该鱼种差距甚大, 因此将该样本确定为比对结果中一致性为99% 的眼点平 鲉。
将所有序列再导入BOLD系统v3中进行检索, 以进一步核实物种的分类地位以及有效的物种名。BOLD中检索结果与 NCBI网站比对结果基本一致。但鞍斑杜父鲈䲢(Cottoperca gobio)在 BOLD系统中比对结果为似杜父鲈䲢(Cottoperca trigloides)(99%), 二者均为牛鱼科杜父鲈 䲢属(Cottoperca), 似杜父鲈 䲢(Cottoperca trigloides)在综合分类学信息系统(Integrated Taxonomic Information System, ITIS)和《拉汉世界鱼类名典》中均无记录, 且FishBase等数据库中也仅对其分布区域有所描述, 为南极洲海域底栖性鱼类, 其形态学描述和图片处于缺少状态。由于 FishBase 所描述的物种似杜父鲈 䲢(Cottoperca trigloides)分布区域与本实验样品采集地点不一致, 故此鱼种种名最终根据形态鉴定和 GenBank比对的结果确定为鞍斑杜父鲈(Cottoperca gobio)。
2.4 分子系统学分析
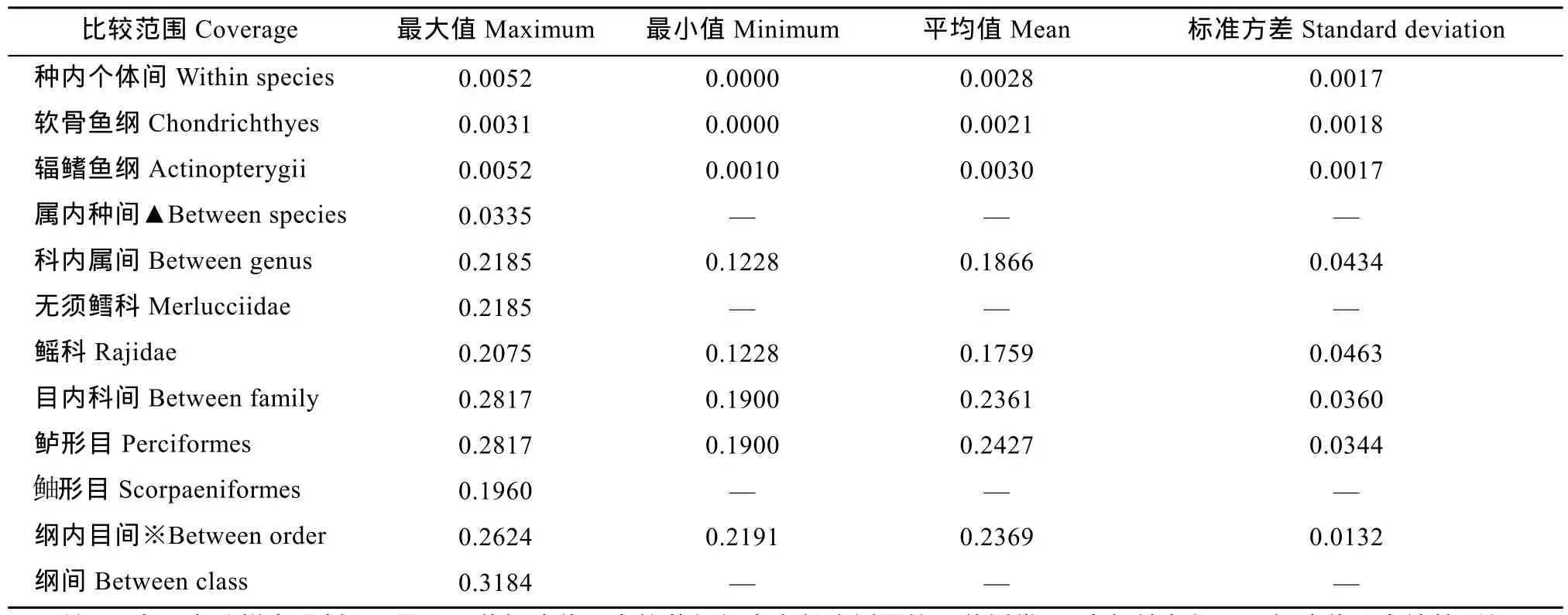
表2 各分类阶元遗传距离(K2P)统计表Tab. 2 Summary of genetic divergences (K2P) within various taxonomic levels
本文涉及14个物种的71条COⅠ序列共有28个单倍型, 基于28个单倍型构建系统树(图2)。由图可以看出,各物种的个体均各自聚为独立的一支, 且不同物种界限清晰。各分类阶元内的聚类结果基本与形态学分类相一致。例如: 鳐科(Rajidae)的长吻鳐属(Dipturus)鱼类先聚为一支, 然后与鳐科的其他属聚为一支; 无须鳕科(Merlucciidae)的无须鳕属(Merluccius)和尖尾无须鳕属(Macruronus)也聚为独立的一支; 鲉形目(Scorpaeniformes)的两种鱼独立聚为一支; 鲈形目(Perciformes )的鞍斑杜父鲈 䲢与拉氏南美南极鱼也独立聚为一支。但目内属间的聚类出现一些异常, 鲈形目(Perciformes )乌鲂、腹翼鲭分别与其他目的鱼类先聚在一起。
3 讨论
本文运用COⅠ基因DNA序列标记对我国西南大西洋远洋渔业的部分渔获鱼种的形态学鉴定结果进行进一步探讨与分析。资料收集阶段发现我国目前仅针对本国近海海域进行鱼类学相关研究, 但有关其他外海海域的鱼类学相关资料极度匮乏, 鱼类形态学分类系统亟待补充与修订。因此, 本研究中形态学鉴定主要依据文献[11—14]等国外研究报道。但由于不同材料编纂角度有所区别,导致部分内容出现分歧, 为避免形态学分类结果的不确定性, 本文从分子辅助分类的角度进行一定程度的修正,保证了物种鉴定的准确性。
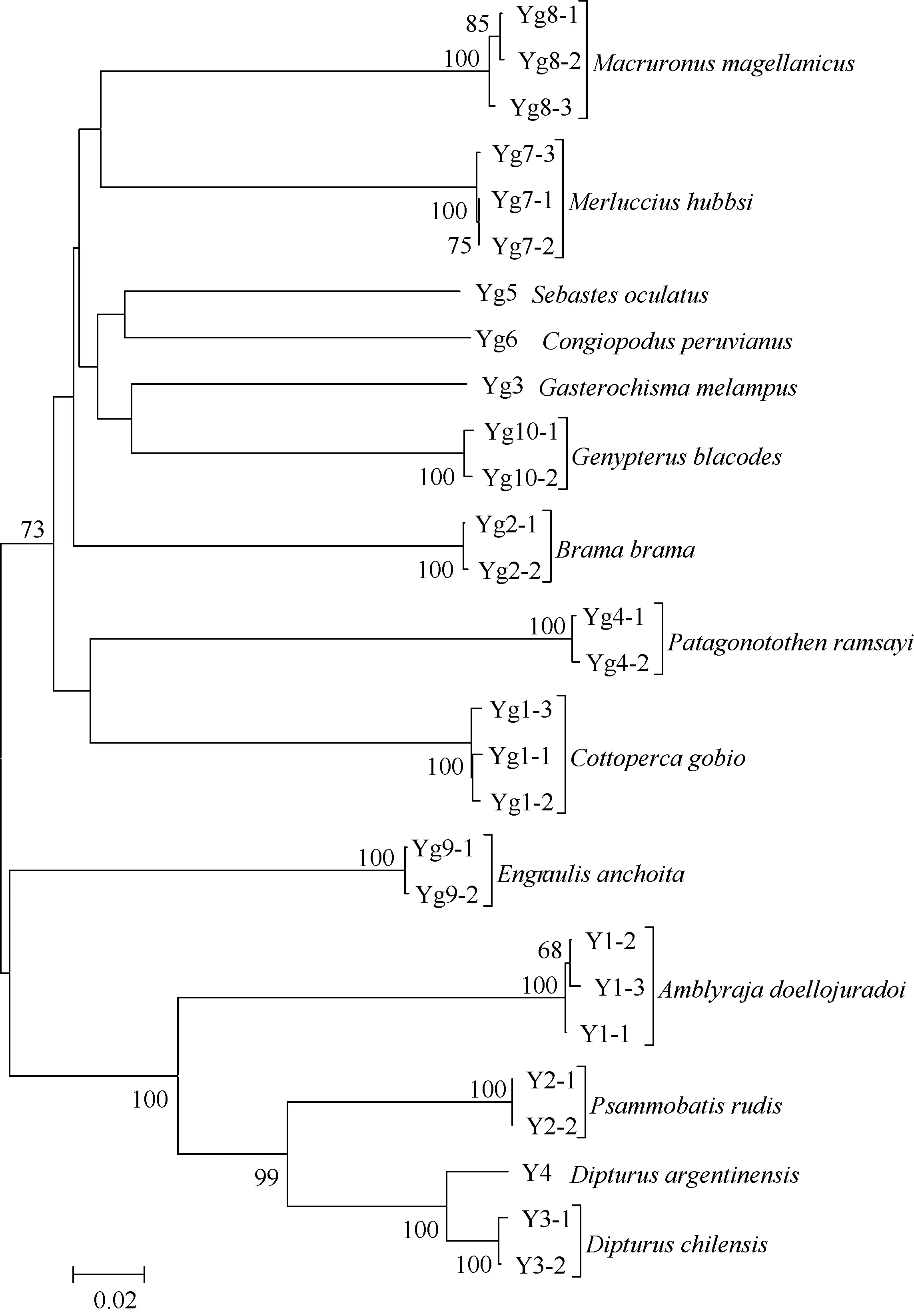
图2 基于14个物种的COⅠ序列单倍型构建的邻接关系树Fig. 2 The neighbor-joining tree of 14 species resulted from haplotypes of CO Ⅰsequences
3.1 COⅠ条形码鉴定存在的问题
自Hebert等[1]提出以COⅠ基因作为DNA条形码以来,该基因在不同生物类群中应用的有效性得到了越来越多的验证[6—10,19], 由此可见COⅠ基因在动物条形码应用中具有一定的优势。其中COⅠ通用引物[6]的广泛适用性在本研究中得到良好证明。本研究采用的通用引物对辐鳍鱼纲10种及软骨鱼纲鳐科4种进行扩增, 均顺利获得相应的序列(表1), 表明该通用引物在鱼类物种中具有较普遍的适用性。
然而, COⅠ条形码在鉴定物种过程中仍存在一定局限性。在确定物种的有效种名时, ITIS分类系统与FishBase网站中关于编号Y3的记载出现分歧。根据样本COⅠ序列与NCBI网站和BOLD系统的比对结果, 此物种的拉丁文名为Dipturus chilensis, 而FishBase网站中认为此种与Zearaja chilensis为同种异名。比较发现, Dipturus chilensis属于长吻鳐属(Dipturus), 而Zearaja chilensis为壳鳐属(Zearaja)物种。为确定准确的种名, 本研究根据种间遗传距离进行分析, Y3与本研究中阿根廷长吻鳐间的遗传距离仅为0.0335, 且在N-J系统树中, 两者首先聚为一支, 因此判断Y3为长吻鳐属的一种。故选择Dipturus chilensis为其有效种名。分析产生此类同种异名情况的原因: (1)缺乏足够的样本资料, 导致形态学鉴定出现偏差,命名错误; (2)外形相似且物种间遗传分化水平不高,COⅠ条形码序列难于分辨[20]; (3)研究时间久远, 鉴定方法不够完善, 且后期研究结果未及时更新至数据库。
3.2 COⅠ条形码序列在系统进化分析中的适用性
根据本研究COⅠ条形码序列得到属内种间的遗传距离为0.0335, 约为种内遗传距离(0.0028)的12倍, 这满足了 Hebert等提出的“10×规则”, 即利用 COⅠ序列进行有效物种鉴定的关键是种间遗传距离必须大于种内遗传距离, 且距离差异大于10倍[8], 这也表明了COⅠ条形码序列适用于物种鉴定的内在特征。同时, 通过本研究所得NJ系统进化树(图2)分析得出, COⅠ条形码序列不但可以进行物种水平分类鉴定, 还可以用来进行一定程度的系统进化分析。但是, 由于COⅠ序列长度仅为650 bp左右,信息位点数目有限, 且科间变异较大, 碱基的转换颠换比趋于饱和, 因此对于深入的系统进化分析来讲其参考价值不高。正如本文结果所示, 基于 COⅠ基因所建 NJ树仅对属内阶元具有较为准确的辨识力, 而对于其他高级阶元的准确性明显降低, 这与随着其分类阶元的升高,种间遗传距离(K2P)的增值明显减缓的趋势相一致。
4 结论
研究认为, 在形态学分类资料缺乏的情况下, COⅠ基因序列可以作为鱼种鉴定的一种便捷有效的方式, 同时可以用来对鱼类形态学分类系统进行补充和修订。但是,COⅠ条形码序列对种以上阶元的物种鉴定存在其局限性。由于COⅠ基因的突变速率相对线粒体DNA控制区的要慢些[21], 因此若针对分化程度低的物种,选择 Cytb或控制区可能更为有效。目前, 由于 COⅠ序列数据仍然有所欠缺, 本研究的拖网渔获物中仍有一些物种未能进行序列比对鉴定。因此, 为了更好地发挥 COⅠ基因序列在物种鉴定中的桥梁作用, COⅠ条形码数据库亟需完善,以求达到高质量高库容要求。与此同时, 为突破 COⅠ条形码序列的一些固有限制, 一方面要加强标本的形态学数据以及标准图片的收集和整理, 另一方面应及时补充低分化物种的其他相关数据(如线粒体DNA控制区等)进行更进一步的分析。
