褶皱臂尾轮虫同胞物种复合类群分类的研究进展
2014-03-26蔡志龙
王 珊, 蔡志龙, 赵 文
(大连海洋大学 辽宁省水生生物学重点实验室,辽宁 大连,116023)
人们最初对物种进行界定时使用了“形态种”的概念(morphospecies),这一概念一直盛行于整个19世纪和20世纪早期。然而,由于有些物种很难用形态学来区分, Mayr[1]首先使用了同胞物种(sibling species)的概念。同胞物种普遍存在于水生无脊椎动物中,人们常使用化学物质来鉴定这些相似的物种[2]。对同胞物种认识的匮乏常常导致生态调查者们在阐述生态系统时会出现概念上的混淆,因而对同胞物种的分类研究十分必要[2-3]。
1 轮虫分类手段的发展
轮虫的主要分类手段之一,是对轮虫进行形态学研究与观察。例如,对褶皱臂尾轮虫复合类群(Brachionusplicatiliscomplex)的分类早在20世纪80年代就已有学者提出,并依据轮虫的被甲背棘刺的形状特点(尖锐或钝圆、较宽或狭窄)和被甲腹褶片的形状特点(倾斜方向、突出物)等将该类群中的一些种类进行了初步命名[4]。后来,一些学者根据轮虫背甲的长、宽及各个背棘刺的距离等数量性状对复合类群中的部分轮虫进行了分类和命名[5-6]。但到目前为止,传统的形态学分类方法仍然是研究和实际生产中的主要分类方法。
随着分子生物学的发展,分子生物技术逐渐成为轮虫分类主要手段之一。早在20世纪80年代初就有学者指出对轮虫不同物种及物种复合体的分类和起源等问题[7],不少学者将等位酶技术应用于轮虫的分类上,尽管各个研究使用的酶系有所不同,但大多数的研究结果都表明,等位酶分析可以用来区分属内及属内复合类群的不同物种[8]。Gómez & King[9]建立了一套轮虫相关酶的测试方法,用于鉴别轮虫品系间的区别。尽管同工酶技术被广泛应用于轮虫的种类鉴定,但是能够应用于种类鉴定的同工酶种类却十分有限。例如,Fu等[10]使用同工酶对L-型和S-型轮虫进行分析时发现,在所使用的18种同工酶中只有6种(GPI、LDH、MDH、6PGD、PGM、SOD)表现出了种间差异。然而,在进行室外浮游生物种群调查时,采集的生物样本通常为混合样本,这一情况就会造成同一谱带上会混合各种不同生物的等位酶,影响了分析的结果。而由于这种方法的局限性,该方法并没有被普遍接受[11],因此,将DNA分析引入轮虫的分类研究就显得尤为重要,各学者研究的结果也表明,同属轮虫之间的差异在DNA水平上可以有效地被检测出[12-13]。
早期对轮虫分类的研究中,提取每个DNA样本的轮虫用量约为2~3 g,这就意味着需要耗费大量人力和时间来培养轮虫,以达到样本需要的量。然而,目前许多种类轮虫的培养方法还不成熟,很难达到高密度培养的水平,而且在培养过程中也很难做到单一物种培养,无法保证DNA样本的用量及纯度,因此限制了分子生物学在轮虫分类方面研究的发展。随着PCR技术的不断完善,对模板DNA的需求量大大降低,轮虫分类分子水平方面的研究才逐渐发展起来。2000年,Gómez等[12]用Chelexl00树脂进行单个褶皱臂尾轮虫的DNA提取,扩增了653 bp轮虫休眠卵的线粒体COI序列片段,分析了轮虫种群和休眠卵库遗传结构变化。Leutbecher[14]曾用RAPD分析了3种轮虫单只个体的基因组DNA,该分析中需要至少50个样本。RAPD-PCR技术在轮虫分类方面的运用,减少了基因组DNA的需要量,加快了分析速度,也不需要对目的序列有预先的了解,推动了轮虫分类的发展。国内的轮虫分类研究较晚,董云伟等[15]利用WizardTM基因组DNA纯化试剂盒提取了单只轮虫的DNA,扩增并测定了萼花臂尾轮虫(Brachionuscalyciflorus)线粒体COI部分基因的序列,经过比较后确定为轮虫的COI基因片段,从而证明使用该方法提取单只轮虫痕量DNA的可靠性。Dingmann[16]利用0.10 g褶皱臂尾轮虫的RNA建立了cDNA文库。
2 轮虫同胞物种复合类群的研究
长期以来研究者都认为轮虫可借助外界力量进行不同地域间的转移,因此曾被普遍认为是一种广泛分布的物种。也有学者指出一些大陆上的轮虫表现出了一定的地域差异。然而,Segers[17]指出,轮虫相对于其他动物群体来说,表现出了很广的分布性,地理隔离位于次要地位。Dumont & Segers[7, 17]都指出在广泛分布的轮虫物种中,包含了一些隐匿的同胞物种。褶皱臂尾轮虫是很多重要经济鱼类的开口饵料,因此确定它的种类是很必要的。而且,同胞物种的存在可能会导致研究者们对水体的生态容纳量和生殖类型的估计出现极大的误差。研究者对未知水体样品的采集和对隐匿物种的发现和描述,都大大促进了人们对轮虫生态多样性的了解。然而,对很多种轮虫来说,除非这种轮虫“真的”是这种轮虫,生态学研究结果并没有什么实际的意义。例如,同一地理品系的相同物种间的形态变化可以定义为周期形变,或者是中间的生态型改变,这使得之前提出的轮虫可以随着季节的改变而发生形变的模式与真实的轮虫种群组成情况有所偏差。就像King[18]对很多湖泊进行的“种群”调查一样,其中的结果大多数只是科学家的主观意愿,与真实的种群个体相关性不大。没有判断不同物种之间的遗传差异或者没有研究有没有混交现象的存在,就会造成大多数物种都被定义为多态性和广泛分布的。除了更多地了解轮虫的生物多样性之外,对轮虫种群中存在的隐匿物种的进一步认知,能促进轮虫研究者更深入地了解不同轮虫种群的生态环境、种群动力学和解决其他许多的生态和进化问题。而对不同种类轮虫复合类群的研究中,以褶皱臂尾轮虫为研究对象的较多。
3 褶皱臂尾轮虫复合类群的分类研究
在早期研究中,很多学者认为褶皱臂尾轮虫只是包括S和L两种类型的单一的物种,但经过长期的研究发现,S和L型轮虫在外形、生理等方面均有明显的不同[19],但仍旧在相当长的一段时间内认为这是同种轮虫不同品系之间的差异或季节变异。直到分子生物学的方法出现,对相似物种的研究才有了新的进展。Fu等对褶皱臂尾轮虫的等位酶进行了研究,发现两种类型的等位酶之间的差别较大,已经超出了同一种轮虫的不同地理品系之间的差异,而且从系统发生树可以看出两种类型的轮虫属于两个不同的物种[10],Runmenga等对褶皱臂尾轮虫两种类型的染色体组型研究的结果再次验证了Fu等观点[20-21]。Segers[22]根据前人的研究成果最终将褶皱臂尾轮虫的S-型和L-型分为两个不同的物种,即B.plicatilis和B.rotundiformis。然而,随着研究的进一步深入,人们又发现这个复合类群中还包括更多的种类。Gómez等[23]对该种轮虫群体的等位酶研究进行了研究,将该种轮虫的群体分为L(B.plicatilis)、SS(B.rotundiformisSS)和SM(B.rotundiformisSM)3个大的类群;同时,由于这3大类群之间存在杂合体缺失,所以L、SS和SM应属于不同的物种,而褶皱臂尾轮虫应属于一个物种的复合类群。Hagiwara等[6]在研究SS-型轮虫时指出,这种轮虫从形态、交配行为和同工酶分析中都没有表现出与B.rotundiformis有差异,因此SS-型轮虫实际上就应该是B.rotundiformis。对于这3类轮虫更近一步的区分由学者Ciros-Pérez等[5]提出(见图1),并对它们进行了重新命名:B.plicatilissensu stricto、B.ibericus和B.rotundiformis,其中B.plicatilissensu stricto属于L型,B.ibericus属于SM-型,B.rotundiformis属于SS-型,平均体长分别为(299±1.5),(193.4±1.4)和(148.7±1.3) μm,平均体宽分别为(225.4±0.9),(144.3±0.6)和(120±0.9) μm;这3种轮虫前棘刺的形状也有所差别,B.rotundiformis的前棘刺更尖并且突出,B.plicatilissensu stricto和B.ibericus的前棘刺则相对较钝;除此之外,3种轮虫休眠卵和咀嚼器都存在差异。
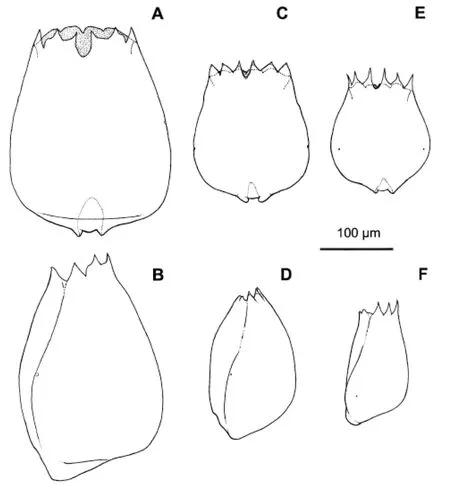
图1 3种臂尾轮虫同胞物种
A和B为Brachionusplicatilissensu stricto,C和D为Brachionusrotundiformis,E和F为Brachionusibericus.
Gómez等对该种轮虫复合类群的生殖行为及同工酶电泳分析进行了研究,并提出在褶皱臂尾轮虫复合类群中,除了L、SS和SM 3种类群以外,还可能存在大量的其他种类轮虫,它们与L、SS和SM 3种类群之间在生理和生态等方面都存在很大的差别[8]。Gómez等[13]对采集的57个轮虫样本(包括实验室培养的已知种类)和从伊比利亚盐湖、北非、北美、欧洲和澳大利亚采集的休眠卵进行了COⅠ和ITS1序列分析,并结合这些轮虫休眠卵孵化出的个体形态特征构建出了9个一致的遗传族系系统发生树(见图2),其中6种分布于伊比利亚半岛。结果是:3个主要的系统发生分枝与3个已经被命名的褶皱臂尾轮虫复合类群型L,M和S型极为一致(见图2)。Stelzer等利用流式细胞技术测定了褶皱臂尾轮虫复合类群中33个样本的基因组大小,结果发现其基因组大小差异可达7倍,同时比对了该复合类群的COI和ITS1序列,发现有32%的轮虫种类无法确定其分支,并且这些种类之间仍可进行混交配对[24]。伊朗科学家最近利用DNA条形码技术鉴定了其国内6个内陆湖中的15个无性繁殖品系,分离到4种轮虫同胞物种[25]。2012年,墨西哥淡水湖中也发现了褶皱臂尾轮虫,并鉴定为新的地理种群B. sp. ‘Mexico’[26]。国内关于褶皱臂尾轮虫分类研究中,殷旭旺等[27]通过调查大连沿岸水体中的褶皱臂尾轮虫复合类群分离出了5种同胞物种,B.plicatilisYamamoto、B.plicatilisMtiller、B.rotundiformisTschugunof、B.ibericusDalian和B.hepatotomusDalian。

图2 通过COⅠ和ITS1序列分析建立树褶皱臂尾轮虫复合类群系统发生树
4 结论与展望
目前对褶皱臂尾轮虫复合类群同胞物种分类的研究仍在继续。对该种复合类群中隐匿物种的深入研究,有助于提高生态调查的准确性,使生态学研究结果更为可靠,同时可以更清楚地了解一些轮虫种类中出现的季节性周期形变产生的机理和同属无脊椎动物的种间差异产生的原因等。虽然目前人们已从褶皱臂尾轮虫复合类群中鉴定出部分轮虫,但是由于复合类群中的同胞物种大多有同域分布状况的存在,因而确定鉴定出的各个同胞物种之间是否存在基因流动的现象就尤为重要。因此,尽管目前DNA技术在轮虫分类中的应用已经十分广泛,但是只依靠单一的研究方法很难做到准确分类,只有将生态、行为、分子技术和传统的形态学分类方法相结合,才可以最大限度的促进轮虫系统分类学的研究。
参考文献:
[1]Mayr E. Systematics and the origin of species[M]. New York: Columbia University Press, 1942, 334.
[2]Knowlton N. Sibling species in the sea[J]. Annual Review of Ecology and Systematics, 1993, 24: 189-216.
[3]Paterson H E H. The recognition of cryptic species among economically important insects. ed. P.e. In Zalucki Heliothis: Research Methods and Prospects[M]. Springer Nerlag. New York: Springer Nerlag, 1991:1-10.
[4]Sudzuki M. Intraspecific variability ofBrachionusplicatilis[J]. Hydrobiologia, 1987, 147: 45-47.
[5]Ciros P J, Gómez A, Serra M. On the taxonomy of three sympatric sibling species of theBrachionusplicatilis(Rotifera) complex from Spain, with the desciption ofB.ibericusn.sp[J]. Plankton Research, 2001, 23:1311-1328.
[6]Hagiwara A, Kotani T, Snell T W, et al. Morphology, reproduction, genetics, and mating behavior of small, tropical marineBrachonusstrains (Rotifera)[J]. Journal of Experimental Marine Biology and Ecology, 1995, 194: 25-37.
[7]Dumont H. Biogeography of rotifers[J]. Hydrobiologia, 1983:104.
[8]Gómez A, Snell T W. Sibling species and cryptic speciation in theBrachionusplicatilisspecies complex (Rotifera)[J]. Evolutionary Biology, 1996, 9:953-964.
[9]Gómez A. Allozyme electrophoresis: its application to rotifers[J]. Hydrobiologia, 1998(387/388): 385-393.
[10]Fu Y, Hirayama K, Natsukari Y. Genetic divergences between S and L type strains of the rotiferBrachonusplicatilisO. F. M ller[J]. Journal of Experimental Marine Biology and Ecology, 1991b, 151: 43-56.
[11]Gómez A. Molecular ecology of rotifers: from population differentiation to speciation[J]. Hydrobiologia, 2005, 546: 83-99.
[12]Gómez A, Carvalho G R, Lunt D H. Phylogeography and regional endemism of a passively dispersing zooplankter: mitochondrial DNA variation in rotifer resting egg banks[J]. The Royal Society, 2000, 267: 2189-2197.
[13]Gómez A, Adcock G J, Lunt D H, et al. The interplay between colonization history and gene flow in passively dispersing zooplankton: microsatellite analysis of rotifer resting egg banks[J]. Evolutionary Biology, 2002, 15:158-171.
[14]Leutbecher C. A routine method of DNA-extraction from extremely small metazoans, e.g. single rotifer specimens for RAPD-PCR analyses[J]. Hydrobiologia, 2000, 437: 133-137.
[15]董云伟, 牛翠娟. 单只轮虫DNA提取及其细胞色素C氧化酶Ⅰ亚基部分序列测定[J]. 动物学研究, 2002, 23: 81-83.
[16]Dingmann B J. Studies of a mate recognition gene and its product from the rotiferBrachionusplicatilis[J]. Georgia Institute of Technology, 2000, 1-69.
[17]Segers H. The biogeography of littoralLecaneRotifera[J]. Hydrobiologia, 1996, 323: 169-197.
[18]King C E. The genetic structure of zooplankton communities[M]. Evolution and Ecology of Zooplankton Communities. Oxford:New England University Press, 1980, 315-329.
[19]Fu Y, Hirayama K, Natsukari Y. Morphological differences between the two types of the rotiferBrachionusplicatilisO.F. Muller[J]. Journal of Experimental Marine Biology and Ecology, 1991a, 151: 29-41.
[20]Runmenga I F M, Kayano H, Hirayama K. Karyotypes of S and L type rotifersBrachionusplicatilisO.F. M ller[J]. Journal of Experimental Marine Biology and Ecology, 1991, 154: 171-176.
[21]Runmenga I F M. Chromosomes and isozymes of hypotriploid strains of the rotiferBrachionusplicatilis[J]. Hydrobiologia, 1993, (255/256): 213-217.
[22]Segers H. Nomenclatural consequences of some recent studies onBrachionusplicatilis(Rotifera, Brachionidae)[J]. Hydrobiologia, 1995, (313/314): 121-122.
[23]Gómez A, Temprano M, Serra M. Ecological genetics of a cyclical parthenogen in temporary habitats[J]. Evolutionary Biology, 1995, 8:601-622.
[24]Stelzer C, Riss S, Stadler P. Genome size evolution at the speciation level: the cryptic species complexBrachionusplicatilis(Rotifera) [J]. BMC Evolutionary Biology, 2011, 11:90.
[25]Malekzadeh-Viayeh R, Pak-Tarmani R, Rostamkhani N, et al. Diversity of the rotiferBrachionusplicatilisspecies complex (Rotifera: Monogononta) in Iran through integrative taxonomy[J]. Zoological Journal of the Linnean Society, 2014, 170: 233-244.
[26]Alc ntara-rodreguez J A, Ciros-perez J, Ortega-mayagoitia E, et al. Local adaptation in populations of aBrachionusgroupplicatiliscryptic species inhabiting three deep crater lakes in Central Mexico [J]. Freshwater Biology, 2012, 57: 728-740.
[27]殷旭旺, 赵 文. 沿海混盐水体褶皱臂尾轮虫的复合类群及其种群动态[J].大连水产学院学报, 2005, 20: 1-10.
