云南中缅树鼩线粒体细胞色素b和D-loop区遗传多样性研究
2014-03-26朱万龙蔡金红王政昆
朱万龙, 贾 婷, 蔡金红, 王政昆
(云南师范大学 生命科学学院, 昆明 650500)
中缅树鼩(Tupaiabelangeri)是东洋界小型哺乳动物,分布在热带和亚热带,是树鼩属中分布最北的一个种,中国南部及西南部可能是该种分布的北限[1]。在中国,中缅树鼩主要分布于广西、海南、贵州、云南、四川、西藏等地,主要生活于热带和亚热带森林、灌丛、村落附近。先前的研究表明横断山区的中缅树鼩在形态及生理适应特征上表现出某些过渡特征[2-5]。
遗传多样性的高低和气候变化、环境改变密切相关,其受到很多因素的影响,如经纬度、分布区域等[6]。线粒体DNA(mtDNA)是核外遗传物质,具有进化速度快、非重组变异和母系遗传等特点,是一种广泛使用的分子标记[7]。mtDNA多态性的研究主要解决种群的遗传多样性和探讨形成该遗传结构的原因[8]。目前,树鼩属的遗传多样性研究在国内研究较少[9]。本文通过测定中缅树鼩线粒体Cytb基因全序列和线粒体控制区D-loop基因部分序列,对其遗传多样性进行分析研究,为树鼩的保护提供实验依据。
1 研究方法
1.1 实验动物
中缅树鼩样本捕自云南省昆明市禄劝县屏山镇(海拔1679 m,N25°25′~26°22′,E102°13′~102°57′)灌丛、剑川县石龙村(海拔2500 m,N26°15′~26°45′,E99°40′~99°55′)果园、丽江地区(海拔2750 m,N26°40′23.5″,E99°48′41.3″)灌丛中。其中cytb基因的样本数为49个,分别为禄劝31只(12♀∶19♂),石龙村10只(3♀∶7♂)和丽江8只(4♀∶4♂);D-loop基因的样本数为33个,分别为禄劝21只(9♀∶12♂),石龙村4只(1♀∶3♂)和丽江8只(4♀∶4♂)。
1.2 基因组DNA的提取
中缅树鼩基因组DNA采用基因组DNA小量快速提取试剂盒 (博大泰克生物基因技术有限责任公司)提取。
1.3 细胞色素b基因的获取
Cytb基因根据已报道中缅树鼩Cytb基因的扩增方法获取[9]。
1.4 D-loop基因的获取
D-loop基因的引物利用Primer Premier 5.0软件来完成。引物覆盖长度为1250 bp。引物序列为:XK_F:5′-acagtcaacacccaaagctgacatgc-3′;XK _R:5′-gtgctatgtcattatcgtggtctca-3′。扩增体系为50 μL,含有5 μL的10×Buffer、1 μL的10 mmol/L dNTPs、20 μmol/ L引物各1 μL、25 mmol/ L MgCl22 μL、2 U Taq酶和50 ng左右的DNA模板。扩增条件:94℃预变性5 min,94℃变性1 min,55℃退火1 min,72℃延伸1 min,35个循环,72℃延伸10 min,4℃终止反应。
1.5 PCR产物纯化和测序
扩增产物采用2%琼脂糖电泳检测,用Glassmilk DNA 纯化回收试剂盒(博大泰克生物基因技术有限责任公司)进行割胶回收、纯化,回收产物送大连宝生物工程有限公司测序。
1.6 序列分析
所用序列均以Clustal X软件[10]进行排序。采用MEGA 4[11]计算碱基变异位点、简约信息位点。基于Kimura两参数[12],采用邻接法(neighbor-joining method,NJ)[13]构建邻接树,在MEGA 4软件中完成。系统树各分支的自举检验值(Bootstrap)由1 000次重复检验所得。采用DnaSP5.0完成多样性分析,采用TCS软件完成单倍型间的进化网络关系图。
2 结果
2.1 细胞色素b序列分析
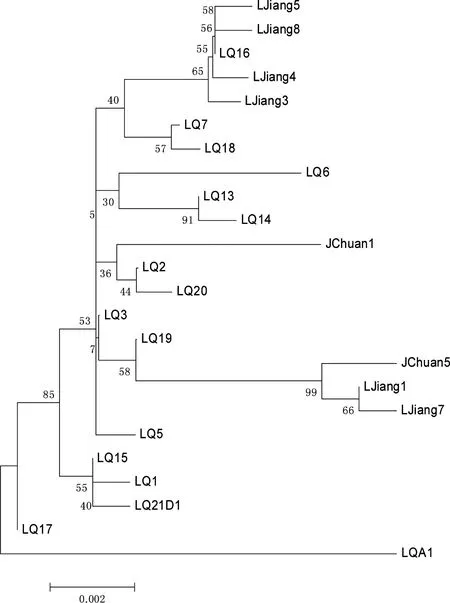
图1 基于线粒体Cyt b基因全序列构建的单倍型NJ树
3个地方中缅树鼩样本中扩增出cyt b全序列为1140 bp(n=49)。分析表明:多态性位点有47个,其中单变异位点23个,简约信息位点24个;禄劝种群内中缅树鼩的遗传距离为0%~1.7%,丽江种群内遗传距离为0%~1.1%,剑川种群内遗传距离为0%~1.2%,禄劝-丽江种群间的遗传距离为0.1%~1.9%,剑川-丽江种群间的遗传距离为0%~1.2%,禄劝-剑川种群间的遗传距离为0.1%~1.9%,种间的遗传距离略大于种内的遗传距离,且剑川与丽江种群间的遗传距离更近。本研究共定义了24个单倍型(占总样本数的48.98%),其中种群间的共享单倍型有2个(8.33%),为丽江-剑川种群所共享, 其余均为某个种群所特有单倍型多样度范围为0.80952 (剑川种群)~0.91532(禄劝种群),核苷酸多样度指数介于0.00326(禄劝种群)~0.00635(剑川种群)之间。利用24个单倍型构建系统树(图1),结果显示3个地区的中缅树鼩之间没有明显的分支,看不出3个地方种群之间的差异。
利用TCS软件构建单倍型间的进化网络关系(图2),结果显示:除LQ6和LQA1经过的突变步骤较多以外,大多数禄劝种群的单倍型之间仅有一到两步突变,而横断山种群(剑川、丽江种群)位于分支的末端,其需要更多地突变步骤才得以出现,说明这一突变方向可能是由禄劝种群到横断山种群,禄劝种群可能更接近祖先种。
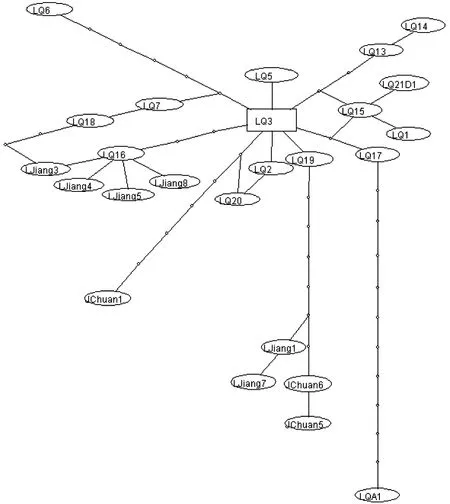
图2 基于Cyt b基因全序列构建单倍型间的进化网络关系
2.2 D-loop区序列分析
3个地方中缅树鼩样本中扩增的D-loop部分序列为745 bp(n=33),分析表明:多态性位点有21个,其中单变异位点10个,简约信息位点11个;禄劝种群内的遗传距离为0%~1.1%,丽江种群内的遗传距离为0%~1.1%,禄劝-丽江种群间的遗传距离为0.3%~1.4%,剑川-丽江种群间的遗传距离为0.1%~1.1%,禄劝-剑川种群间的遗传距离为0.5%~1.2%,种间的遗传距离略大于种内的遗传距离,且剑川与丽江种群间的遗传距离更近。
本研究共定义了16个单倍型(占总样本数的48.48%),无种群间的共享单倍型,单倍型多样度范围为0.76615(禄劝种群)~0.93333(丽江种群),核苷酸多样度指数介于0.00269(禄劝种群)~0.00583(丽江种群)之间。利用16个单倍型构建系统树(图3),结果显示3个地区的中缅树鼩明显分为两支,一支全为禄劝种群,另一支除有1个禄劝单倍型(LQL0D5)外,全为剑川-丽江种群。
利用TCS软件构建单倍型间的进化网络关系(图4)结果显示:除LQ L0D2经过的突变步骤较多以外,大多数禄劝种群的单倍型之间仅有一到两步突变,而横断山种群(剑川、丽江种群)位于分支的末端,其需要更多地突变步骤才得以出现,说明这一突变方向可能是由禄劝种群到横断山种群,禄劝种群可能更接近祖先种。
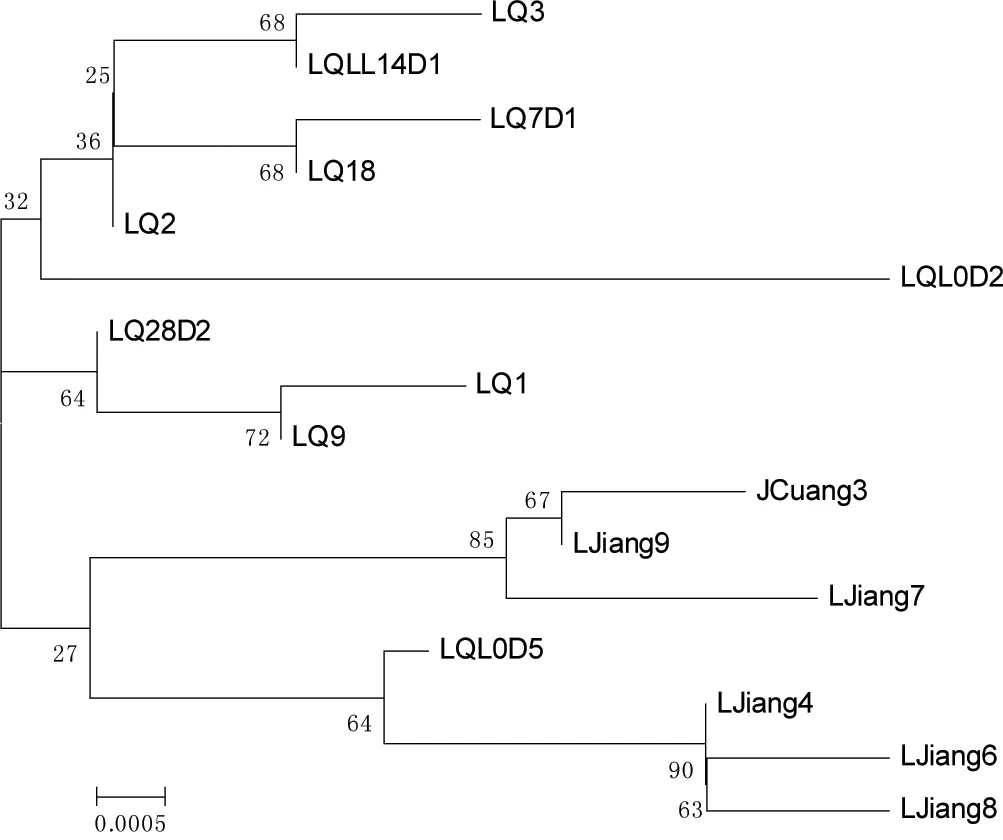
图3 基于D-loop基因部分序列构建的单倍型NJ树
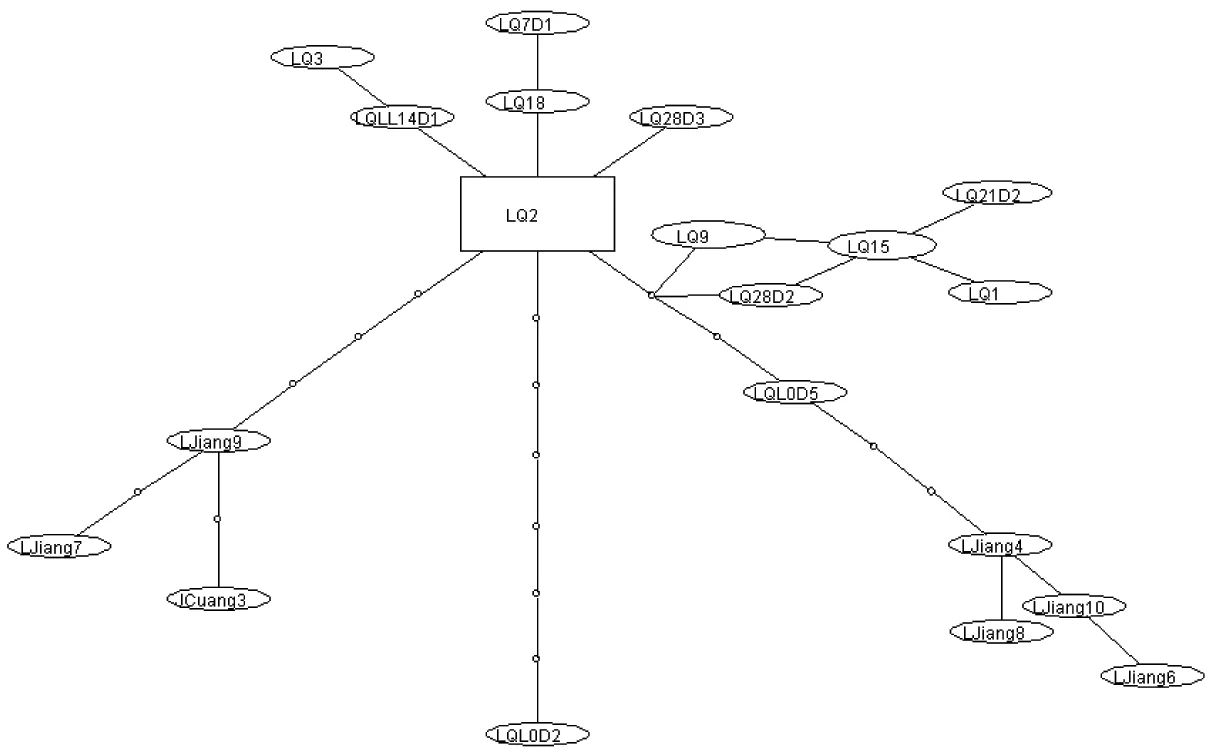
图4 基于D-loop基因部分序列构建单倍型间的进化网络关系
3 讨论
物种遗传多样性的高低与其生存能力等密切相关。衡量一个种群mtDNA的遗传变异有两个重要指标:单倍型间的平均遗传距离(P)和核苷酸多态性(Pi)。由于Pi值考虑了各种mtDNA单倍型在群体中所占的比例,因此在反映一个群体的mtDNA的多态程度时比单纯的遗传距离平均值要精确,Pi值较高则说明群体的遗传多样性较高。遗传多样性越高,其生存能力等越大。相反,较低的遗传多样性可导致其生存能力等降低[14]。研究表明同种哺乳动物个体之间的平均核苷酸顺序歧异值在0.3%到4%之间,最大时竟达10%以上[15]。在本研究中,无论利用Cytb基因还是利用D-loop基因均显示横断山种群(剑川、丽江种群)拥有更高的遗传多样性,这可能与横断山地区拥有特殊的生态环境相对应。在本研究中,无论利用Cytb基因全序列还是利用D-loop基因部分序列均显示横断山种群(剑川、丽江种群)拥有更高地遗传多样性,这可能与横断山地区拥有特殊的生态环境相对应。与其它保护物种相比,云南地区中缅树鼩的遗传多样性较低(表1),这种现象一方面可能与种类差异有关,另一方面可能与云南地区是中缅树鼩分布的北限,使其适应能力、生存能力降低,物种退化等有关。
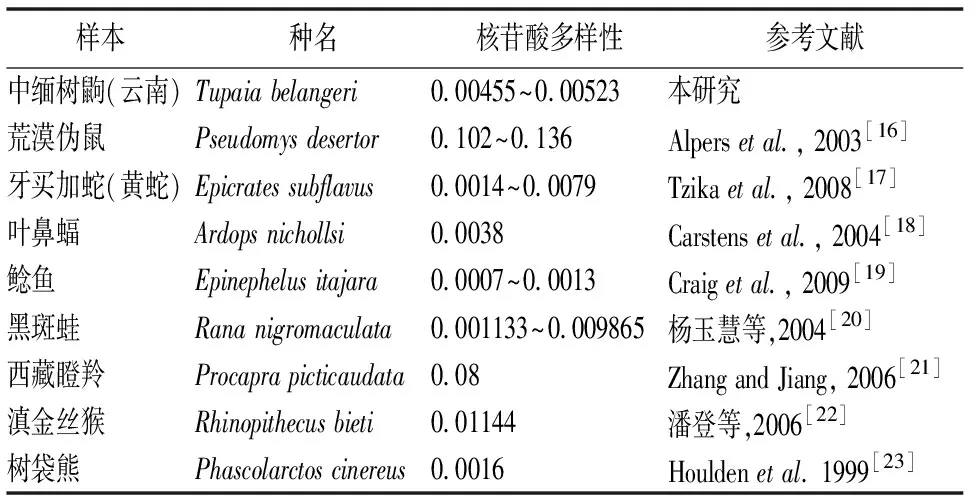
表1 不同物种mtDNA核苷酸多样性
Cytb基因的进化速率适中,适合于种群水平差异的检测,常常被用作为研究系统进化和分类的分子标记[24]。而D-loop基因,为不编码基因,其DNA序列的进化速度在线粒体基因组中最快,适合做种内群体遗传变异的研究。在本研究结果表明D-loop基因在分析种群的遗传多样性时更为有效。
关于现生树鼩的起源假说有两种:即“岛屿起源”假说和“大陆起源”假说。现生树鼩中有6种树鼩仅仅极限分布于婆罗洲及其附近岛屿,另外4种树鼩也分布于该岛屿及其附近地区,而且值得注意的是与树鼩(攀鼩目)为姐妹群的皮翼目也主要分布于婆罗洲等地区,这一分布特征强有力地支持“岛屿起源”假说[25-26]。然而,目前已经在大陆发现几种树鼩的化石记录,值得注意的是在中国元谋发现了Dendrogale和笔尾树鼩的近亲,遗憾的是仅仅发现牙齿,且没有给出详细的描述和图片,如果这两种树鼩的近亲确实存在于中国南部的中新世地层中,那么将强有力的支持“大陆起源”假说[27-29]。本研究结果显示中缅树鼩的变异方向很可能是从禄劝地区指向横断山地区,在一定程度上反应了中缅树鼩由南向北的扩散模式,支持树鼩“岛屿起源”这一假说。
参考文献:
[1]Zhang L, Liu P F, Zhu W L, et al. Variations in thermal physiology and energetics of the tree shrew (Tupaiabelangeri) in response to cold acclimation[J]. Journal of Comparative Physiology B., 2012, 182: 167-176.
[2]Zhu W L, Xie J, Lian X, et al. Thermogenic characteristics and evaporative water loss in the tree shrew(Tupaiabelangeri) [J]. Journal of Thermal Biology, 2010, 35: 290-294.
[3]Zhang L, Wang R, Zhu W L, et al. Adaptive thermogenesis of the liver in tree shrew (Tupaiabelangeri) during cold acclimation[J]. Animal Biology, 2011, 61: 385-401.
[4]Zhu W L, Jia T, Huang C M, et al. Changes of energy metabolism, thermogenesis and body mass in the tree shrew (Tupaiabelangerichinensis) during cold exposure[J]. Italian Journal of Zoology, 2012, 79: 175 181.
[5]Zhu W L, Zhang H, Wang Z K. Seasonal changes in body mass and thermogenesis in tree shrews (Tupaiabelangeri) the roles of photoperiod and cold[J]. Journal of Thermal Biology, 2012, 37:479-484.
[6]Nichols R A, Hewitt G M. The genetic consequences of long dispersal during colonization[J]. Heredity, 1994, 72: 312-317.
[7]Birky C W, Fuerst P, Maruyama T. Organelle gene diversity under migration, mutation, and drift: equilibrium expectations, approach to equilibrium, effects of heteroplasmic cells, and comparison to nuclear genes[J]. Genetics, 1989, 121: 613-627.
[8]Li M, Rao G, Wei F W. Population genetic structure and geographic subdivision of the red panda (Ailurusfulgens) [J]. Acta Zoologica Sinica, 2002, 48: 480-486.
[9]贾 婷, 杨晓密, 李宗瀚, 等. 基于细胞色素b基因探讨昆明禄劝地区树鼩的分类意义[J]. 动物学杂志, 2008, 43(4):26-33.
[10]Thompson J D, Gibson T J, Plewniak F. The clustal x windows interface: exible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 24: 4876-4882.
[11]Tamura K, Dudley J, Nei M. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24: 1596-1599.
[12]Kimura M. A simple method of estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences[J]. Mol Evol, 1980, 16: 111-120.
[13]Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4: 406-425.
[14]Liu F G R, Miyamoto M M, Freire N P. Molecular and morphological supertrees for eutherian (placental) mammals[J]. Science, 2001, 291: 1786-1789.
[15]Neigel J E, Avise J C. Application of a random walk model to geographic distributions of animal mitochondrial DNA variation[J]. Genetics, 1993. 135: 1209-1220.
[16]Alpers D L, Gaikhorst G, Lambert C. An extension to the known range of the desert mousePseudomysdesertorsouth into the Great Victoria Desert, Western Australia[J]. Australian Mammalogy, 2003, 25: 95-96.
[17]Tzika A C, Koenig S, Miller R. Population structure of an endemic vulnerable species, theJamaicanboa(Epicratessubflavus)[J]. Molecular Ecology, 2008, 17: 533-544.
[18]Carstens B C, Sullivan J, D valos L M. Exploring population genetic structure in three species of lesserAntilleanbats[J]. Molecular Ecology, 2004, 13: 2557-2566.
[19]Craig M T, Graham R T, Torres R A. How many species of goliath grouper are there Cryptic genetic divergence in a threatened marine fish and the resurrection of a geopolitical species[J]. Endangered Species Research, 2009, 7: 167-174.
[20]杨玉慧, 张德兴, 李义明. 中国黑斑蛙种群的线粒体DNA多样性和生物地理演化过程的初探[J]. 动物学报, 2004, 50: 193-201.
[21]Zhang F, Jiang Z. Mitochondrial phylogeography and genetic diversity of Tibetan gazelle (Procaprapicticaudata): implications for conservation[J]. Molecular Phylogenetics and Evolution, 2006, 41: 313-321.
[22]潘 登, 符云新, 张亚平. 基于线粒体控制区的滇金丝猴群体遗传学研究[J]. 遗传, 2006, 28: 432-436.
[23]Houlden B A, Costello B H, Sharkey D. Phylogeographic differentiation in the mitochondrial control region in the koala,Phascolarctoscinereus(Goldfuss 1817)[J]. Molecular Ecology, 1999, 8: 999-1011.
[24]Meyer A. Evolution of mitochondria DNA in fishes[J]. Biochem Mol Biol Fishes, 1993, 2: 1-36.
[25]Murphy W J, Eiziri E, Johnson W E. Molecular phylogenetics and the origins of placental mammals[J]. Nature, 2001, 409: 614-618.
[26]Olson L E, Sargis E J, Martin R D. Molecular phylogenetics and evolution intraordinal phylogenetics of tree shrews (Mammalia: Scandentia) based on evidence from the mitochondrial 12S rRNA gene[J]. Molecular Phylgenetics and Evolution, 2005, 35: 656-673.
[27]Murphy W J, Eizirik E, O′Brien S J. Resolution of the early placental mammal radiation using Bayesian phylogenetics[J]. Science, 2001, 294: 2348-2351.
[28]Sargis E J. Tree shrews[M]. Singer R. (Ed.) Encyclopedia of Paleontology. Fitzroy Dearborn, Chicago. 1999: 1286-1287.
[29]Sargis E J. New views on tree shrews: the role of tupaiids in primate supraordinal relationships[J]. Evol, Anthropol, 2004, 13: 56-66.
