基于ITS序列分析技术鉴定川牛膝与常见伪品麻牛膝
2014-03-10陈诗晴蒲沁琳陈小军宋瑱
陈诗晴,蒲沁琳,陈小军,宋瑱
·品种品质·
基于ITS序列分析技术鉴定川牛膝与常见伪品麻牛膝
陈诗晴1,蒲沁琳1,陈小军2,宋瑱1
目的:比较川牛膝与其常见伪品麻牛膝之间的ITS序列差异及规律。为川牛膝与麻牛膝的DNA条形码鉴别提供适合的分子标记。方法:收集川牛膝及麻牛膝成品药材并提取纯化其基因组DNA,经PCR扩增得到ITS序列(包括ITS1、5.8S nrDNA、ITS2)并进行T-A克隆后测序,分析两者序列差异。结果:PCR扩增获得两者ITS序列,经多序列对比分析得出川牛膝与伪品麻牛膝的ITS序列存在明显差异。结论:ITS序列分析可以用作鉴定川牛膝与麻牛膝药材。
川牛膝;麻牛膝;PCR;ITS序列;DNA测序
中药材川牛膝是苋科植物川牛膝(Cyathμla officinalis Kuan)的干燥根,为《中华人民共和国药典》1990年版收载,是四川省著名的道地药材,具有逐瘀通经、通利关节、利尿通淋等功能[1,2]。近年来,现代药理研究证明川牛膝多糖成分具有抗肿瘤、抗生育等新的作用[3],使其再次受到关注。但是与川牛膝同科植物的麻牛膝入药部位外观与川牛膝相似,用传统鉴别方法(如外观、形态、质地等)很难准确地把两种药材区别开来;中医临床经验认为麻牛膝的性味功效与川牛膝不同,不宜作川牛膝药用。因此,如何简便、快速和准确地鉴定川牛膝的真伪是我们广泛应用道地药材川牛膝面临的最大难题。
ITS序列是真核生物细胞基因组中的核糖体rRNA基因的转录间隔序列。18S nrDNA序列、ITS1间隔序列、5.8S nrDNA序列、ITS2间隔序列和26S nrDNA依次紧密连接在一起,形成一个完整的转录单位,该转录单位是典型的高度重复序列,在植物基因组中拷贝数可达500~40000。在技术上易于扩增,其中的ITS1序列和ITS2序列的可变性与物种遗传关系密切,成为了众多研究者对于药用植物的鉴别,揭示药用植物的遗传差异和鉴定中药材品种的道地性的新方法和手段[4~7]。

图1 ITS完整序列示意图
1 材料和方法
1.1 材料
1.1.1 药材样品 分别从四川雅安天全县、西昌、宝兴等地采集川牛膝药材10份,西昌会理采集及成都药材市场购买麻牛膝药材5份。道地药材为雅安川牛膝GAP基地产品。采集后置于干燥环境保存。
1.1.2 主要试剂 CTAB、Tris、蛋白酶K购自sigma公司;HOTSTART taq DNA聚合酶,pEASY-T5 zero质粒载体购自北京全式金公司;凝胶回收试剂盒购自北京博迈德公司,磁珠法细菌基因组DNA提取试剂盒及扩增引物购自上海生工公司。
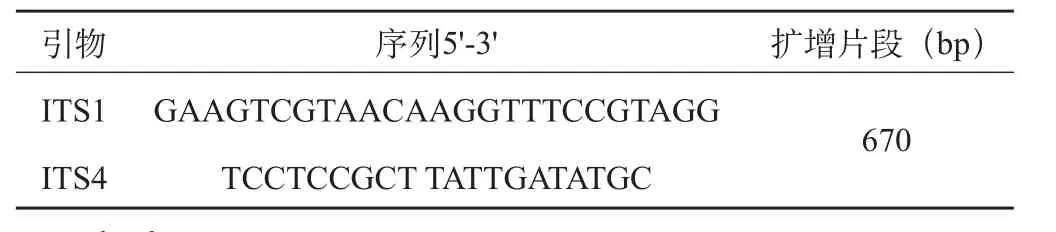
表1 扩增引物及其序列
1.2 方法
1.2.1 基因组DNA分离纯化 采用本实验室经过预试验改良后的磁珠法提取,以75%酒精消毒川牛膝及麻牛膝药材,剔去表面后液氮研磨成粉末,10%CTAB/NaCl裂解液裂解样品粉末后加入吸附性磁珠。以磁力架吸附包含DNA的磁珠,加入70%的乙醇洗涤磁珠,挥干乙醇后加入50μL洗脱液洗脱获得基因组DNA。将获得的DNA用0.7%琼脂糖凝胶电泳鉴定完整性,紫外分光光度仪检测OD260值计算浓度。
1.2.2 PCR扩增ITS序列 参照NCBI上牛膝属ITS完整序列(包括ITS1、5.8S nrDNA、ITS2,GenBank: DQ497186.1)设计引物ITS1及ITS4。以上一步所得的DNA为模板构建川牛膝ITS序列PCR反应体系(50μL):10×Buffer,200mM dNTP,0.25uM引物,1U HOTSTART taq DNA聚合酶,100ng模板,补超纯水至50μL。扩增反应参数为:94℃预变性5分钟;94℃变性30秒;52℃退火30秒;72℃延伸1分钟;循环30次后72℃延伸7分钟。1.5%琼脂糖凝胶电泳检测PCR产物。
1.2.3 PCR产物的T-A克隆及序列分析 将PCR产物经紫外分光定量后按摩尔比7: 1加入pEASY-T5 zero质粒载体,25℃连接10分钟,42℃热激30秒转化JM109感受态细胞,在含氨苄青霉素(100mg/L)的SOC平板上培养24小时后进行菌落PCR筛选,获得阳性克隆。将筛选后的阳性克隆送上海生工公司测序鉴定。
2 结果
2.1 川牛膝及麻牛膝药材(根茎)的基因组DNA的鉴定
取“1.2.1”中获得牛膝药材DNA溶液5μL上样于0.7%琼脂糖凝胶电泳结果见图2。图中可以分析出DNA分子完整,无明显降解,且浓度较高,可以满足下游PCR实验。紫外分光检测A260/A280均为1.7-2.0范围内,无明显蛋白污染。
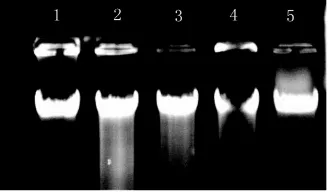
图2 川牛膝及麻牛膝基因组DNA的电泳鉴定结果
2.2 PCR扩增ITS序列的电泳结果
以1.2.2方法扩增16份样品ITS序列经电泳检测均获得明显的特异性产物,大小670bp。见图3。

图3 川牛膝及麻牛膝ITS序列PCR产物的电泳结果
2.3 川牛膝及麻牛膝ITS序列测序结果分析
经测序后,参照NCBI中苋科植物ITS序列经BlAST比对,以及DNAMAN软件多序列分析,确定11株川牛膝ITS序列长度约为591bp,G+C%约为53.3-53.7%。其中ITS1序列约232bp、5.8S nrDNA序列约162bp、ITS2序列约197bp。5株麻牛膝ITS序列长度约为587bp,G+C%约为51.6-52.1%。其中ITS1序列约230bp、5.8S nrDNA序列约162bp、ITS2序列约195bp。所有样品5.8S nrDNA序列都高度保守,ITS1及ITS2序列在同种间只有少量差异,在川牛膝与麻牛膝间有明显的差异30余处,见图4。

图4 川牛膝及麻牛膝ITS序列差异对比分析结果
3 结论与讨论
本次研究对于样品的采样部位是成品药材即根茎部位,而由于植物根茎的DNA分离纯化难度较高,所以现有众多研究者采样部位主要是采用了新鲜叶片提取DNA,方法以CTAB法为主[8,9]。但此方法不适合对药材成品的DNA提取。本研究采用了优化后的磁珠法。磁珠法提取生物样本DNA在方法上有快速、样品纯度及浓度高的优点,但对于植物根茎的细胞裂解不及CTAB法效率高。本次研究综合利用磁珠法和CTAB法的优点,先利用CTAB裂解液处理样品裂解细胞,再使用磁珠法收集纯化DNA,获得了良好的效果。
对11株川牛膝及5株麻牛膝ITS序列比对分析发现,在同种的川牛膝ITS序列中5.8S nrDNA序列高度保守,未发现有突变位点,ITS1及ITS2序列在随产地不同只有少数几个碱基有差异,道地药材和其余10株不同产地川牛膝没有明显稳定的差异。所以,ITS序列分析不能用作鉴别川牛膝道地药材,需要另寻其他分子标记。
在川牛膝和麻牛膝不同种牛膝间发现,其ITS1及ITS2序列在42位、50位、88位、90位等30多处位点有明显的碱基替换、缺失、插入等差异。并且这些差异具有较好的稳定性。经过重复双向测序验证,这些结果准确可靠。
通过本研究发现ITS序列测序后经过序列比对能较好的反映出川牛膝和麻牛膝的分子差异。因此可利用这种技术鉴定川牛膝和麻牛膝药材。
[1] 张敬杰,芦卫红,史旭霞.牛膝的草本学研究[J].现代中西结合杂志,1994,7(11):696.
[2] 胡世林.中国道地药材论丛[M].北京:北京中医古籍出版社, 1997:62.
[3] 陈红,刘友平.川牛膝多糖抗肿瘤作用初探[J].成都中医药大学学报,2001,24(1):16.
[4] Gottschling M,plotner J.plotner.Secondary structure models ofthe nuclear intemal -transcribed spacer regions and 5.8S rRNA in CaleiodinelIoideae(Peridiniaeeae) and other dinofagellates[J].Nucleic Acids Res,2004,32(1):307.
[5] Baldwin BG,Sandersonm J,Porter JM.TheITSIegion of nuclear ribosomal DNA:A valuable soμl'ce of evidence on angiosperm phylogeny[J].Ann Missouri Bot Gard,1995,82:247.
[6] Hyosig Won,Susanne S,Renner.The internal transcribed spacer of nuclear ribosomal DNA in the gymnosperm Gnetum[J]. Molecμlar Phylogenetics and Evolution,2005,36:581.
[7] 赵欢,吴卫.核糖体DNA ITS序列分析在药用植物研究中的应用[J].时珍国医国药,2009,20(4):959.
[8] 丁平,方琴.巴戟天与常见混伪品的rDNA-ITS序列分析及其分子鉴定[J].中草药,2005,36(6):908.
[9] 赵永亮,傅体华. 川牛膝(Cyathula oficinalis Kuan)RAPD和AFLP标记的多态性聚类分析[J].安徽农业科学, 2008,36(16): 6682.
(责任编辑:蒋淼)
The identifcation of Chuanniuxi and adulterants Maniuxi based on ITS sequence analysis
CHEN Shi-qing1,PU Qinglin1,CHEN Xiao-jun2,SONG Zhen1//(1. Chengdu University of Traditional Chinese Medicine, Chengdu 611137;2. Sichuan Lu Tian Hua Co., Ltd. Luzhou 646300)
Objective:To fnd the difference of ITS sequences and regularity between Chuanniuxi and its common adulterants Maniuxi, and provide suitable molecular markers for the DNA barcoding identification of Chuanniuxi and Maniuxi. Method:The samples of Chuanniuxi and Maniuxi were collected, and genomic DNA were extracted to get the ITS sequences by PCR amplification (including ITS1, 5.8S nrDNA and ITS2). Then ITS sequence and sequence divergence were analyzed after T-A cloning.Result:The ITS sequence were got after PCR amplifcation and signifcant differences between Chuanniuxi and Maniuxi was found by multiple sequence alignment analysis. Conclusion:ITS sequence analysis can be used to identify the raw material Chuanniuxiu and Maniuxi.
Chuanniuxi; Maniuxi; PCR; ITS sequences; DNA sequence analysis
R 282.5
A
1674-926X(2014)04-002-03
成都中医药大学科技发展基金(编号ZRYB2010);成都中医药大学大学生科研实践创新课题(编号2012-90)
1.成都中医药大学医学技术学院,四川 成都611137;2.四川省泸天化股份有限公司中化室,四川 泸州 646300
陈诗晴(1993-),女,本科,成都中医药大学医学技术学院生物技术专业Tel: 18215525190 Email:502590353@qq.com
宋瑱(1979-),男,医学硕士,主管检验技师,主要从事医学分子生物学研究Email:red_cell@qq.com
2013-10-24
