靶向转化生长因子-βⅡ型受体核酸适配子结合位点预测及相关验证研究*
2014-03-04黄阳玉朱晓燕
王 薇,黄阳玉,朱晓燕,陈 侠,许 多,肖 奕,谢 琳△
(1.第三军医大学大坪医院野战外科研究所眼科,重庆400042;2.华中科技大学物理学院生物分子物理与模建小组,武汉430074)
抗青光眼滤过手术是治疗原发性青光眼的主要方法之一,而滤过术后手术区的滤过通道瘢痕化是手术失败的主要原因[1]。因此,临床上常使用5-氟尿嘧啶、丝裂霉素等抗代谢药物来抑制术后疤痕形成,提高手术成功率,但其无法完全避免诸如滤过泡渗漏、低眼压、眼内炎、角膜损伤等并发症的发生。所以,寻求一种更为安全、有效、毒性小的药物作用于抗青光眼术后抑制瘢痕形成仍然是目前的研究方向。瘢痕的形成与转化生长因子-β(TGF-β)的活性有着密切关系[2-3],其中TGF-β与其Ⅱ型受体(TβRⅡ)的结合是导致组织瘢痕化的始动环节。因此,拮抗或者抑制TGF-β与TβRⅡ的结合,可为青光眼滤过术后抗瘢痕提供新的方法。本课题组前期应用指数富集的配基系统进化技术(SELEX)筛选出TβRⅡ的单链核酸适配子S58,并验证了S58可对TGF-β介导的人Tenon囊成纤维细胞(HTFs)分化产生抑制作用[4]。适配子作用药物从基础转向临床必须在稳定性上进行优化验证,甚至需要对基团修饰。而核酸适配子能够识别靶分子并结合紧密是基于其三维结构,单链核酸适配子结构灵活多变,能在溶液根据靶分子结构发生适用性折叠重构,形成几何构象匹配、热力学稳定的空间结构,比如与靶分子形成口袋、假结等结构[5]。因此,研究适配子S58的三维结构是有必要的,本研究建立S58的三维模型,旨在预测S58与TβRⅡ的结合部位,并进一步验证S58的高亲和力及特异靶向性,从而明确S58的结构稳定性及特异性。
1 材料与方法
1.1 材料 本研究中所用细胞由本课题组前期原代培养的HTFs传代而来[6],取第4~10代对数生长期的细胞用于实验。
1.2 主要试剂及仪器 DMEM培养基、胎牛血清、胰蛋白酶、双抗、Hanks平衡盐溶液(美国Hyclone公司);人重组TGF-β2蛋白(美国Peproteeh公司);鼠抗人α-平滑肌肌动蛋白(α-SMA)单克隆抗体(美国Abcam公司);鼠抗人单克隆β-actin抗体、辣根标记山羊抗兔Ig G、辣根标记山羊抗鼠Ig G(北京中杉公司);TβRⅡ胞外段蛋白(美国Rapidbio公司),核酸适配子S58(5′-ACA TTG CTG CGT GAT CGC CTC ACA TGG GTT TGT CTG GTC GAT TTG GAG GTG GTG GGT GGC-3′)及各剪切序列均由上海生工生物工程公司进行DNA合成。生物传感器(美国Ther mo公司);蛋白电泳及转膜设备(美国Bio-Rad公司);光学倒置相差显微镜(日本Oly mpas公司)。
1.3 方法
1.3.1 三维建模 目前没有直接的软件可用于预测单链DNA的三级结构,本研究采用3d RNA[7]先建立RNA的三级结构,3d RNA是一个基于RNA序列和二级结构的预测方法,再通过计算机测算翻译突变为DNA的三级结构。首先应用DNA序列转录成RNA序列,应用mfold[8]软件进行RNA的二级结构预测,再通过输入RNA序列和二级结构,3d RNA可以预测RNA的三级结构。得到RNA的三级结构之后,需要将其转化成对应的DNA结构。这个过程分为两步:(1)核苷酸突变;(2)优化结构。首先,对于RNA结构中的每一个核糖核苷酸分子,用对应的脱氧核糖核苷酸分子进行取代,即进行重叠操作,重叠的原子只包括碱基。通过上述的重叠操作,得到的DNA分子可能有部分共价键断裂或者原子重叠,所以需要进行结构的优化[9],本研究采用amber对其进行了优化。TβRⅡ胞外段蛋白可以直接通过蛋白质数据库搜索得到晶体结构。
1.3.2 分子对接预测结合位点 得到2个分子的三维结构后采用Hex Server程序[10]对2个分子进行对接。Hex Server是一个基于图像处理的进行蛋白质与蛋白质、DNA、RNA的对接服务器。Hex Server经过运行测算给出最有可能对接后的20个相互作用能量最低的复合物构象。在此基础上使用软件Bind N[11]预测受体蛋白可能的结合位点,Bind N是一个基于网络的根据氨基酸序列来有效预测蛋白质的DNA、RNA结合位点工具。其预测DNA的结合残基灵敏度为69.40%,特异度为70.47%,根据支持向量机大于0.372 5预测出24个与DNA的结合残基位点,这样又进一步缩小了结合位点的范围。最后进一步采用分子动力学模拟方法进行进一步细致的分子对接。
1.3.3 适配子ss DNA与TβRⅡ的亲和力检测 采用第三军医大学附属西南医院中心实验室建立的生物传感器技术检测S58及各剪切的DNA与TβRⅡ的亲和力大小。按照已经建立的方法包被TβRⅡ,S58及剪切的DNA各组均采用杜氏磷酸盐缓冲液溶解成200 mg/L,取5μL上样,检测与TβRⅡ的亲和力[12]。
1.3.4 蛋白免疫印迹法(Wester n blot)法检测人HTFs中α-SMA的表达 培养8瓶HTFs待贴壁并长至70%左右融合,将培养基换为无血清培养基再培养24 h,分别加以DMEM(DMEM对照 组)、TGF-β2、TGF-β2+S58、TGF-β2+S58(3′-5 bp)、TGF-β2+S58(3′-10 bp)、TGF-β2+S58(5′-5 bp)、TGF-β2+S58(5′-10 bp)、TGF-β2+S58(5′-15 bp)诱导HTFs 24 h,并以此分为不同的组别。各组TGF-β2浓度均为2 ng/mL,DNA浓度均为50 n mol/L;中止刺激后PBS清洗细胞3次,每次10 min,每瓶加入180μL含蛋白酶抑制剂的细胞裂解液,提取细胞总蛋白,考马斯亮蓝G-2250法测定每个样本中的蛋白浓度。根据蛋白浓度取30μL样品上样,质量分数10%的十二烷基硫酸钠-聚丙烯酰胺凝胶电泳进行电泳。蛋白质转膜180 min。质量分数5%脱脂牛奶室温封闭膜2 h。鼠抗人α-SMA单克隆抗体按1∶200比例加入到2 mL 5%脱脂牛奶中4℃孵育膜过夜。TBST洗膜3次,每次10 min,辣根标记山羊抗鼠Ig G按1∶5 000比例加入到2 mL 5%脱脂牛奶中室温孵育2 h,TBST洗膜3次。发光液作用于膜上,暗室内曝光,观察X线片上的条带,扫描后保存,Quantity One 4.6.2软件(美国Bio Rad公司)计算出胶片上各蛋白条带灰度值,计算出α-SMA相对表达量,并作出柱状图。
1.4 统计学处理 采用SPSS13.0统计软件进行分析,计量资料以±s表示,组间差异比较采用单因素方差分析,以P<0.05为差异有统计学意义。
2 结 果
2.1 适配子ss DNA S58及TβRⅡ胞外段的三维模型 RNA预测为一个带发卡结构的RNA,转换成单链DNA时,DNA自身适应性互补形成局部双螺旋结构。蛋白质数据库中搜索得到TRβⅡ胞外段蛋白1PLO,由122个氨基酸组成,有10种自然态结构,并且是以二聚体结构存在。比对TGF-β与其受体的复合晶体结构,找到10种自然态中最接近与配体结合时的结合态结构1PLO-3,其结构紧密,包括了9个β折叠。见图1。
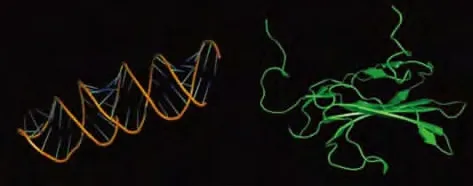
图1 适配子ss DNA S58(左)及TβRⅡ胞外段(右)的三维结构
2.2 适配子ss DNA S58及TβRⅡ胞外段蛋白的分子对接 人为去掉DNA 3′端游离的5个碱基及受体蛋白两端游离氨基酸部位,将优化后的S58结构与TβRⅡ胞外段蛋白进行分子对接,得到10个相互作用能量最低的对接构象,相互作用能量包括2个分子之间的静电力和范德华力,可以间接反映适配子DNA与TβRⅡ胞外段蛋白的结合情况,相互作用的能量越低,结合则越稳定。因此选择最低能量-502.65 kJ/mol的对接构象,见图2。对接位置位于DNA的中间部位,运用Cocomaps软件分析,使用8Å作为定义结合残基的距离阈值,即对于复合物中核酸的某个氨基酸残基,如果其包含的至少一个原子与复合物中核酸序列的任何一个原子间的距离小于8Å,那么这个氨基酸残基为核苷酸-结合残基。相对应的核酸结合位点分别包括位点Ⅰ(T4、T5、G6、C7)、位点Ⅱ(G13、A14、T15、C16、G17、C18)、位点Ⅲ(T31、G32、T33、C34)及位点Ⅳ(G40、A41、T42、T43、T44、G45、G46)。
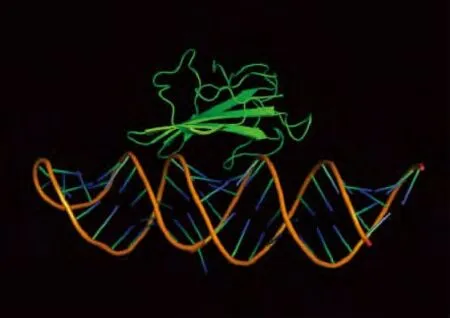
图2 适配子ss DNA S58与TβRⅡ胞外段对接位点
2.3 剪切S58与亲和力检测 根据S58与TβRⅡ胞外段的结合位于DNA中间部位,采取分别从DNA两端逐步剪切碱基,每次剪切5 bp。S58与TβRⅡ胞外段的结合力最强,其次为S58-5即3′端剪切5 bp,但不论从5′端还是3′端开始剪切DNA S58,得到的结构修饰后DNA亲和力与S58相比均没有明显增加,反而有所下降。见图3。
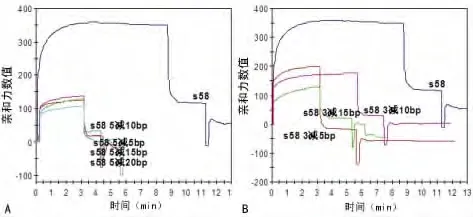
图3 S58-1~7与TβRⅡ胞外段的结合力检测
2.4 结构优化后DNA作用下细胞α-SMA的表达变化 α-SMA是肌成纤维细胞的标志性蛋白,是瘢痕收缩的物质基础。TGF-β2组刺激HTFs后α-SMA表达较DMEM对照组明显增加(P<0.001),TGF-β2+S58组刺激HTFs后α-SMA表达较TGF-β2组明显降低(P<0.001),所有结构优化后的DNA中只有TGF-β2+S58(3′-5 bp)组较TGF-β2组α-SMA表达有明显降低(P<0.001),但其降低程度不如TGF-β2+S58组,其他组与TGF-β2组比较α-SMA表达差异均无统计学意义(P>0.05),见图4。
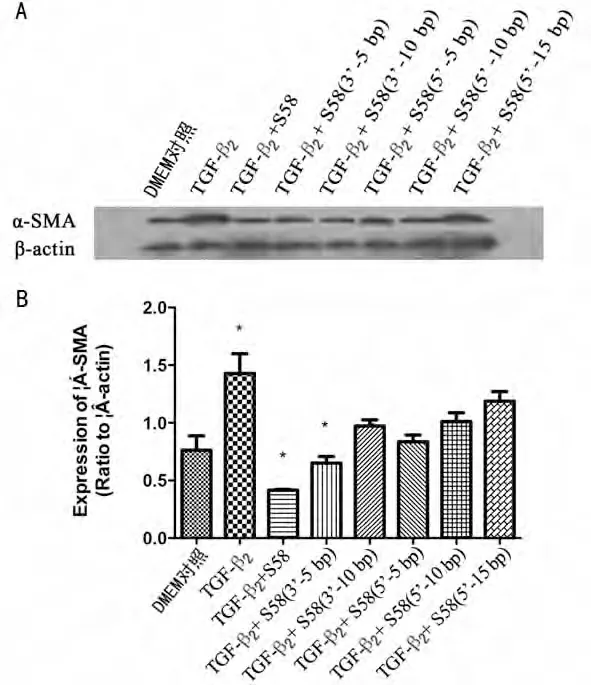
图4 各实验组刺激下HTFs表达α-SMA的变化
3 讨 论
适配子在眼科方面已经有很大进展,目前许多研究显示适配子如贝伐单抗、寡聚核RNA-寡聚苷酸、培加尼布等能通过抑制血管内皮生长因子作用从而明显降低眼部的血管形成。哌加他尼钠是第一个通过美国食品与药品管理局认证用于治疗年龄相关性黄斑变性的核酸适配子药物[13],还有其他治疗眼部新生血管的适配子药物在临床实验中。因此,能封阻TβRⅡ降低青光眼术后滤过区纤维化形成的适配子S58成为新的研究切入点。适配子应用于临床治疗的优点在于其亲和力高、稳定性好、易人工合成。所有的适配子能发挥高效、特异的作用,基于其特殊的、稳定的三维结构,与靶分子特异的作用位点结合,且具有较高的亲和力[14],所以,研究适配子与靶分子的结合位点及结合力是解释适配子发挥生物效应的理论根据,并且进一步验证适配子的稳定性可为适配子运用于临床药物提供理论依据。
随着计算机技术的飞速发展,计算机技术辅助研究蛋白质和核酸的相互作用成为当下的热点,特别是在药物分子的筛选和设计方面已经有很大进展[15]。其中分子对接是针对研究配体与受体分子之间的相互作用,基于结合的复合物构象,预测其结合模式与亲和力的一种重要理论方法。Luscombe等[16]利用氨基酸序列的保守性构建计算机算法来预测蛋白质/DNA复合体中DNA的结合位点。研究2个分子的结合位点,首先需要建立2个分子的三维结构。寡核苷酸适配子在溶液中可以形成假结、发夹、凸环、G-四分体等三维空间结构,通过氢键、范德华力、疏水堆积作用等与靶分子紧密结合。有研究表明,适配子与靶分子的结合是相互诱导的适应性识别,当靶分子存在时,单链DNA或RNA发生适应性折叠,与靶分子相互识别[17]。本实验中适配子S58即为60个碱基的寡核苷酸,与靶受体蛋白作用时遵循“锁钥原理”,发生适应性自身碱基互补情况,且DNA带负电荷,在与靶受体蛋白结合时会向带正电荷的碱性氨基酸靠近,DNA分子的灵活性使其能够形成的结构千变万化,单链DNA有局部自互补的情况,由于适配子的结构特殊性,其三维结构预测往往需要从二级结构着手,因此DNA的三维结构预测有一定难度。蛋白质的结构可以通过蛋白质晶体学得到或者从蛋白质数据库中找到其已知的结构,正如本实验中的TβRⅡ胞外段蛋白结构。分子对接的结果表明,DNA与TβRⅡ胞外段蛋白的结合位点均在空间结构上与受体相靠近或与受体存在氢键相互作用,这些位点在配体与受体结合、定位和水解过程中可能发挥重要作用,实验从适配子S58的三维建模研究切入,合理地运用大量目前广泛得到认可成功率较高的建模、分子对接软件,在理论上较为准确地预测到适配子S58与TβRⅡ胞外段蛋白的结合位点,极大地减少可能的结合位点数量,为进一步实验提供指导。
生物传感器是从分子水平检测生物分子活性力的技术手段,其检测快速、灵敏度高、选择性强,近年来在医学领域应用越来越广泛,特别是针对核酸与蛋白质的作用检测,Mccauley等[18]研制了基于蛋白芯片基础的适体生物传感器来对蛋白进行分析鉴定,这种传感器可以固定着针对不同蛋白质的DNA和RNA适体。本实验也运用生物传感性针对TβRⅡ胞外段蛋白,根据计算机预测的核酸适配子与TβRⅡ胞外段蛋白的结合位点提示5′端剪切掉5 bp会影响到结合位点Ⅰ(T4、T5、G6、C7),故5′端剪切进一步验证剪切后DNA S58~4与TβRⅡ胞外段蛋白的亲和力是下降的,细胞水平上也对TGF-β2刺激后的HTFs表达α-SMA无明显抑制作用,这与计算机预测的结果相符。又因为SELEX技术在筛选过程中是必须应用大容量的随机寡核苷酸文库,大约在1 010个随机RNA序列中才有一个序列与靶分子特异结合[18],且核酸文库中是由中间为一定长度的随机序列和5′、3′端的固定序列构成,固定序列是多聚酶链式反应及其他酶学反应相关引物的结合位点。因此,推测剪切掉的5′端序列是带有限制性内切酶位点的固定序列。但当同时选择剪切3′端序列时,当剪切掉5 bp时,DNA与TβRⅡ胞外段蛋白亲和力有所下降,尽管能抑制TGFβ2刺激后HTFsα-SMA的表达,但实验组α-SMA的表达仍较S58组强,抑制TGF-β2介导的HTFS转分化作用是较S58减弱的,并且在剪切3′端非预测的结合位点后,适配子与TβRⅡ胞外段蛋白亲和力同样也下降。推测因单链核苷酸在不同的溶液、p H、温度等因素下易形成多种空间构象,特别在剪切掉适配子序列后极有可能改变核苷酸局部的结构,从而无法与靶向TβRⅡ胞外段蛋白质的关键作用位点结合。因此,进一步论证了S58为靶向TβRⅡ的特异性分子,结构上具有高度稳定性,任何结构的改变均会降低与TβRⅡ的亲和力,为S58的体内实验提供了明确的理论基础。
[1]Yu DY,Morgan WH,Sun X,et al.The critical role of theconjunctiva in glaucoma filtration surgery[J].Prog RetinEye Res,2009,28(5):303-328.
[2]Cordeiro MF.Role of transforming growth factor beta inconjunctival scarring[J].Clin Sci(Lond),2003,104(2):181-187.
[3]Zhu X,Li L,Zou L,et al.A novel aptamer targeting TGFβreceptorⅡinhibits transdifferentiation of human tenon′s fibroblasts into myofibroblast[J].Invest OphthalmolVis Sci,2012,53(11):6897-6903.
[4]Zhan LS,Zhuo HL,Wang HZ,et al.Screening and characterization of aptamers of hepatitis C virus NS3 helicase[J].Progr Biochem Biophys,2005,32(3):245-250.
[5]朱晓燕,李磊,鲜光军,等.人眼Tenon囊成纤维细胞体外培养及生长特性的比较[J].局解手术学杂志,2012,21(3):233-235.
[6]Zhao Y,Huang Y,Gong Z,et al.Automated and fastbuilding of three-dimensional RNA structures[J].SciRep,2012,2(2012):734.
[7]Zuker M.Mfold web server for nucleic acid folding andhybridization prediction[J].Nucleic Acids Res,2003,31(13):3406-3415.
[8]Wang JM,Wolf RM,Caldwell JW,et al.Development andtesting of a general amber force field.[J].J ComputChem,2004,25(9):1157-1174.
[9]Macindoe G,Mavridis L,Venkatraman V,et al.HexServer:an FFT-based protein docking server powered bygraphics processors[J].Nucleic Acids Res,2010,38(WebServer issue):W445-449.
[10]Wang L,Brown SJ.Bind N:a web-based tool for efficientprediction of DNA and RNA binding sites in amino acidsequences[J].Nucleic Acids Res,2006,34(Web Server issue):W243-248.
[11]Genfa L,Jiang Z,Hong Z,et al.The screening and isolation of an effective anti-endotoxin monomer from radixpaeoniae rubra using affinity biosensor technology[J].IntImmunopharmacol,2005,5(6):1007-1017.
[12]Ng EW,Shima DT,Calias P,et al.Pegaptanib,a targetedanti-VEGF aptamer for ocular vascular disease[J].NatRev Drug Discov,2006,5(2):123-132.
[13]Navani NK,Li Y.Nucleic acid aptamers and enzymes assensors[J].Curr Opin Chem Biol,2006,10(3):272-281.
[14]Rohs R,Bloch I,Sklenar H,et al.Molecular flexibility inab initio drug docking to DNA:binding-site and bindingmode transitions in all-atom monte carlo simulations[J].Nucleic Acids Res,2005,33(22):7048-7057.
[15]Barbault F,Ren B,Rebehmed J,et al.Flexible computational docking studies of new aminoglycosides targetingRNA 16S bacterial ribosome site[J].Eur J Med Chem,2008,43(8):1648-1656.
[16]Luscombe NM,Thornton JM.Protein-DNA interactions:amino acid conservation and the effects of mutations onbinding specificity[J].J Mol Biol,2002,320(5):991-1009.
[17]Hermann T,Patel DJ.Adaptive recognition by nucleic acidaptamers[J].Science,2000,287(5454):820-825.
[18]Mccauley TG,Hamaguchi N,Stanton M.Aptamer-basedbiosensor arrays for detection and quantification of biological macromolecules[J].Anal Biochem,2003,19(2):244-250.
