大鼠粪便中细菌基因组DNA提取方法的比较
2014-02-25骞宇,赵欣
骞 宇,赵 欣
(重庆第二师范学院生物与化学工程系,重庆400067)
大鼠粪便中细菌基因组DNA提取方法的比较
骞 宇,赵 欣*
(重庆第二师范学院生物与化学工程系,重庆400067)
为获得高质量的肠道细菌基因组DNA,用于研究肠道菌群的多样性,本实验分别采用改良的溶菌酶法和两种试剂盒DNA提取法提取大鼠粪便中的细菌基因组DNA,通过DNA浓度和纯度的测定、PCR-变性梯度凝胶电泳技术(PCR-DGGE技术)对提取效果进行比较,以找到适合于粪便中细菌基因组DNA的提取方法。结果表明,改良的溶菌酶法提取的DNA质量好,纯度高,而且具有菌群多样性丰富等特点,更适合用于肠道微生物菌群后续分子生物学研究。
细菌基因组DNA,大鼠粪便,肠道菌群,变性梯度凝胶电泳
肠道菌群是机体的重要组成部分,其菌群结构数量和种类的变化与机体的健康和疾病有密切关系[1-3],因此对肠道菌群的研究也越来越受到人们的关注。随着分子生物学技术在肠道微生物研究中的广泛运用,提取到高质量的细菌基因组DNA对于研究肠道微生物菌群来说尤为关键。而DNA提取方法很多,传统的DNA提取方法和各种试剂盒法的应用都非常广泛,但是大多数传统方法繁琐复杂,费时又费力,现在提取哺乳动物肠道微生物菌群基因组DNA的主要方法是用传统的溶菌酶法、酚/氯仿抽提法等,而这些方法提取的DNA纯度低,含的蛋白质等杂质较多,严重影响后续的分子生物学方法的准确性。而很多DNA提取试剂盒的价格昂贵,提取出来的DNA产量相对较低,效果不理想。目前,很多学者都提出了针对不同实验样品的传统DNA提取法的改良方法[4-6],但是关于实验室常用实验大鼠粪便中细菌基因组DNA提取方法研究较少,而且现在还没有专门用于从大鼠肠道中提取微生物基因组DNA的试剂盒[7]。因此,寻找一种适用于大鼠肠道细菌基因组DNA的提取方法,对于后续的肠道微生物多样性的分子生物学研究非常有必要。
本实验分别采用改良的溶菌酶法,粪便基因组DNA试剂盒提取法和通用基因组DNA试剂盒提取法提取大鼠粪便中细菌基因组DNA,对DNA提取效果进行比较,探寻适合于提取大鼠肠道微生物菌群DNA的快捷、经济、高效的方法,为后续的分子生物学实验提供依据。
1 材料与方法
1.1 材料与仪器
样品采集 采集大鼠新鲜粪便0.2g,放入2mL的无菌离心管中(取2份样品),放置在-80℃冰箱中冷冻保存;粪便基因组DNA提取试剂盒(离心柱型) 北京天根生物技术有限公司;通用基因组DNA提取试剂盒 北京索莱宝科技有限公司;GelRed凝胶核酸染料 美国biotium公司;蛋白酶K、溶菌酶、过硫酸铵、TEMED 美国Sigma公司;丙烯酰胺、甲叉双丙烯酰胺 美国Genview公司;PCR的试剂 中国大连TaKaRa公司;引物 均由上海生工合成。
紫外分光光度计(MiNi) 日本岛津公司;制冰机(SIM-F140AY65) 日本三洋电器公司;凝胶成像系统、梯度PCR仪(S1000)、变性梯度凝胶电泳仪(DcodeSystemapparatus) 美国Bio-Rad公司。
1.2 粪便中细菌基因组DNA的提取
1.2.1 样品预处理[8]将200mg左右粪便样品放入2mL离心管,加入1mL磷酸盐缓冲液(PBS,0.1mol/L,pH7.4),20μL 20%PVPP,充分振荡混匀。室温下2000r/min离心6min,取上清液。沉淀中再加入1mL PBS混匀,离心,取上清液,合并两次上清液再3000r/min离心6min,取上清液,再12000r/min离心6min,收集沉淀。沉淀用PBS清洗一次。分别用下面的3种方法抽提DNA。
1.2.2 改良溶菌酶法 用传统的溶菌酶法提取的基因组DNA的质量少,而且含有的蛋白质杂质较多[8],故在传统溶菌酶法中,加入裂解液Ⅰ和Ⅱ,有利于细胞的裂解,可获得更多的DNA,并且用氯仿/异戊醇反复抽提,有利于去除蛋白质等杂质,具体方法如下:在预处理的样品中依次加入300μL裂解液Ⅰ(0.15mol/L NaCl,0.1mol/L Na2EDTA,pH8.0)、100mg/mL溶菌酶100μL、5μL蛋白酶K(10mg/mL),在37℃下225r/min振荡1h;加入300μL裂解缓冲液Ⅱ(10%SDS,0.1mol/L NaCl,0.5mol/L Tris-HCl,pH8.0),50μL 20% PVPP,65℃保温10min;加入750μL Tris-饱和酚∶氯仿∶异戊醇(25∶24∶1),混合振荡2min,13000r/min离心8min。上清液转入新的2mL离心管;加入等体积氯仿∶异戊醇(24∶1)。混合振荡2min,13000r/min离心8min,上清液转入另一2mL离心管;加入1/10体积的3mol/L醋酸钠和2倍体积无水乙醇,-20℃沉淀2h;15000g/min离心15min,沉淀用70%冰乙醇洗涤一次,自然风干;最后沉淀用50μL无菌去离子水溶解,-20℃保存。
1.2.3 粪便基因组DNA试剂盒提取法 将预处理的样品,采用北京天根生物技术有限公司的粪便基因组DNA提取试剂盒提取样品细菌基因组DNA。提取得到的DNA溶于50μL无菌去离子水中。
1.2.4 通用基因组DNA试剂盒提取法 将预处理的样品,采用北京索莱宝科技有限公司的通用基因组DNA提取试剂盒提取样品细菌基因组DNA。提取得到的DNA溶于50μL无菌去离子水中。
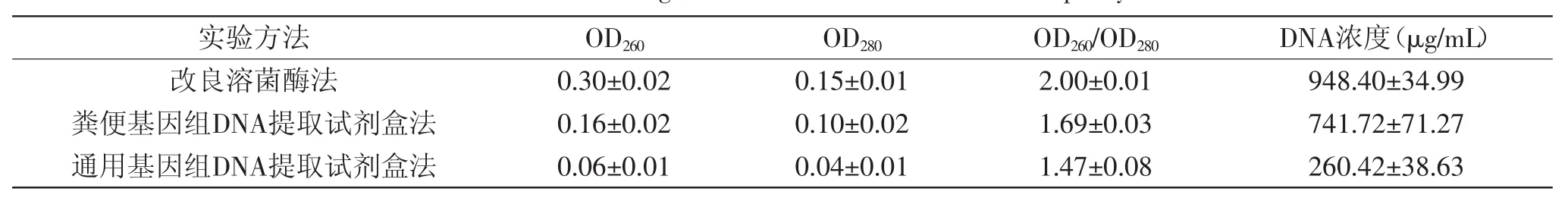
表1 细菌基因组DNA的浓度和纯度Table.1 Bacterial genomic DNA of concentration and purity
1.3 粪便中细菌基因组DNA的纯度和质量检测
1.3.1 DNA提取效果及质量的检测 分别取三种方法提取得到的DNA样品1μL,稀释10倍,用紫外分光光度计检测DNA的A260、A280和A260/A280值;同时取三种方法抽提获得的DNA各5μL,用0.8%琼脂糖凝胶电泳,GelRed染色,凝胶成像仪观察分析。
1.3.2 PCR扩增
1.3.2.1 细菌基因组16S rDNA PCR扩增 用16S rDNA通用引物[9-10]27f(5’-AGAGTTTGA TCCTGGCTCAG-3’)和1492r(5’-GGCTACCTTGTTACGACTT-3’)进行V3区扩增,扩增产物长度约为1500bp。25μL扩增体系:1μL DNA模板,正向和反向引物各0.5μL,12.5μL 2×Taq PCR Master Mix,灭菌的超纯水补足至25μL。反应程序为:95℃预变性5min,94℃变性50s,55℃退火1min,72℃延伸1min,30个循环,72℃延伸5min。扩增产物用1%琼脂糖凝胶电泳,GelRed染色,凝胶成像仪观察分析。
1.3.2.2 细菌巢式PCR扩增 为提高反应的特异性,用另一对引物特异性扩增位于第一次PCR产物内的一段更小的DNA片断,以保证扩增片段的准确性。以第一次PCR产物作为模板,用引物[10]338f(5’-ACT CCA CGG GAG GCA GCA G-3’)及518r(5’-ATT ACC GCG GCT GCT GG-3’),并在引物338f的5’末端加上40bp的GC夹子(CGC CCG CCG CGC GCG GCG GGC GGG GCG GGG GCA CGG GGG G),进行扩增。50μL扩增体系:2μL DNA模板,正向和反向引物各1.0μL,25μL 2×Taq PCR Master Mix,灭菌的超纯水补足至50μL。反应程序:94℃ 5min,94℃ 30s,65~55℃ 30s(每个循环降低0.5℃),72℃30s,20个循环,94℃30s,55℃30s,72℃30s,8个循环,72℃延伸7min。扩增产物用1.2%琼脂糖凝胶电泳,GelRed染色,凝胶成像仪观察分析。
1.3.3 DGGE电泳分析 参照朱阳玲等[11]的方法,采用Bio-Rad公司的DcodeTM通用突变检测系统对三种方法提取的细菌基因组DNA的巢式PCR扩增产物进行电泳分离分析。采用10%(w/v)的聚丙烯酰胺凝胶,变性剂浓度梯度范围为35%~60%,电泳温度60℃,恒压200V下电泳4h。电泳结束后取出胶放入配制好的GelRed染液中,浸染30min,再使用紫外凝胶成像系统观察拍照。
2 结果与分析
2.1 三种方法获得的细菌基因组DNA质量比较
2.1.1 细菌基因组DNA的浓度和纯度 由表1可知,用改良溶菌酶法得到的样品细菌基因组DNA浓度最高,通用基因组DNA试剂盒提取法得到的样品细菌基因组DNA浓度最低。两种试剂盒法提取的大鼠粪便细菌基因组DNA的OD260/OD280的值均小于1.7,改良溶菌酶法提取的基因组DNA的OD260/OD280的值在1.9~2.0之间。正常DNA样品的OD260/OD280值约为1.8~2.0,若OD260/OD280值小于1.8或大于2.0,说明可能有蛋白污染或RNA污染[12-13]。因此改良溶菌酶法提取的基因组DNA纯度较高,而两种试剂盒法提取的基因组DNA都有不同程度的蛋白质污染或RNA污染。
2.1.2 细菌基因组DNA的电泳 如图1所示,所得DNA样品均大于23kbp,说明这三种方法都能提取出细菌基因组DNA,1、2泳道条带清晰且亮度较高,说明改良后的溶菌酶法DNA提取效果最好;3、4泳道条带亮度稍弱,5、6泳道条带亮度相对最弱,表明用两种基因组DNA提取试剂盒法提取的样品基因组DNA纯度跟改良后的溶菌酶法提取相比稍差,且得率较低。这与表1所显示的信息是一致的,说明改良后的溶菌酶法提取样品细菌基因组DNA效果最好。
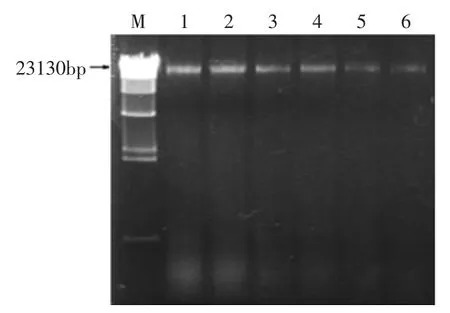
图10 .8%琼脂糖凝胶电泳图Fig.1 0.8%agarose gel electrophoresis figure
2.2 细菌基因组DNA的16S rDNA的PCR扩增结果
2.2.1 第一次PCR扩增结果 由图2可见,对细菌16S rDNA V3区进行第一次PCR扩增,阴性对照无条带,说明扩增无污染;1~6泳道均得到长度约1500bp的特异性扩增片段,符合预期扩增片段长度。而且3、4泳道亮度很强,说明扩增产物较多,效果较好。

图2 细菌16S rDNA第一次PCR扩增产物的琼脂糖凝胶电泳图谱Fig.2 Agarose gel electropherogram of First PCR amplication of16S rDNA of bacteria
2.2.2 巢式PCR扩增结果 如图3所示,阴性对照无条带,说明扩增无污染,1~6泳道均扩增出长度约200bp的片段,条带清晰,无杂带,证明PCR效果较好。亮度为5,6泳道>1,2泳道>3,4泳道,说明改良溶菌酶法提取的DNA质量最好。
本实验证明改良溶菌酶法提取粪便样品中的细菌基因组DNA的浓度和纯度均优于两种试剂盒法。
2.3 变性梯度凝胶电泳(DGGE)结果分析
如图4所示,1、2泳道显现出的条带最多,其次是5、6泳道,3、4泳道显现出的条带最少,说明改良溶菌酶法提取的DNA样品的菌群多样性丰富,粪便基因组DNA提取试剂盒法次之,通用基因组DNA提取试剂盒法效果不太理想。
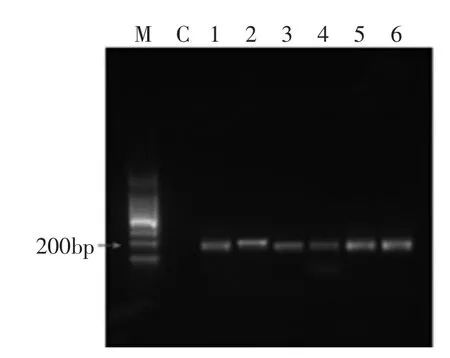
图3 细菌16S rDNA巢式PCR扩增产物的琼脂糖凝胶电泳图谱Fig.3 Agarose gel electropherogram of Nested-PCR amplication of 16S rDNA of bacteria
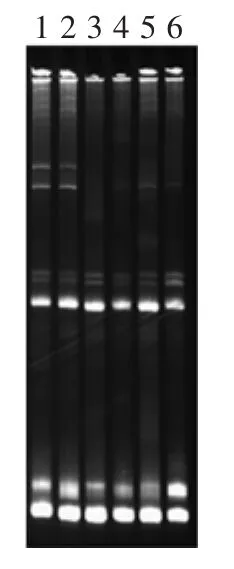
图4 细菌巢式PCR产物的水平变性梯度凝胶电泳图谱Fig.4 DGGE profile of bacterial Nested-PCR amplication
3 讨论
动物和人都是由宿主自身及其体内定植的微生物菌群共同组成的超级生物体。动物和人体的健康状况可由外界环境因子与机体自身及体内的微生物菌群共同相互作用决定,因此研究宿主与体内菌群之间的相作关系等具有重大的意义。粪便与肠道内容物相似,含有大量的微生物、植物和肠道脱落细胞等,将粪便微生物作为研究对象,有利于更好地研究肠道微生物[14]。而且粪便取样方便、无损伤。很多研究人员都选择从动物粪便中提取出细菌基因组DNA运用于肠道微生物的研究。但由于粪便成分复杂,不同的细菌细胞壁的组成成分和结构不一样,且粪便中伴有各种Taq酶抑制剂和胆红素、胆盐等DNA降解物[15],对不同提取方法的敏感度也不一样,所以不同的提取方法所获得的基因组DNA有差异。而样品DNA的提取是肠道微生物菌群后续分子研究中最关键、最重要的一步,提取时不仅要尽可能地将样品中所有细菌的基因组DNA提取出来,保证DNA的质量和数量,而且要尽量去除其中抑制分子克隆中多种酶活性的杂质。
本实验的目的是希望从大鼠粪便样品中获得高浓度、高纯度的,并且能够尽量全面的反映出样品中菌群多样性的细菌基因组DNA。根据紫外分光光度法、DNA样品的电泳图和PCR扩增图,发现改良溶菌酶法提取的细菌基因组DNA的浓度和纯度均高于其他两种方法提取的DNA。因DGGE图谱能够比较直观地反映细菌菌群的多样性,故采用PCR-DGGE技术从细菌多样性的角度评价DNA提取方法的优劣。通过3种方法得到的同一份粪便样品的DGGE图谱差异较大,说明在肠道菌群的分子生态研究中,DNA提取的方法会影响我们对菌群多样性的分析,甚至引起误差。实验结果说明改良溶菌酶法比粪便基因组DNA提取试剂盒法和通用基因组DNA提取试剂盒法提取的细菌基因组DN的纯度和质量更好,且能够观察到粪便中菌群的多样性更丰富,所以更合适肠道菌群多样性的研究。
4 结论
本实验中使用的三种方法均能够从大鼠粪便中提取到细菌基因组DNA,通过紫外分光光度法、PCRDGGE等方法检测,结果显示,相对于两种试剂盒DNA提取法,改良溶菌酶法提取的大鼠粪便细菌基因组DNA的纯度更高、质量更好,而且能够观察到更高的多样性,所以更合适肠道菌群后续分子生物学研究。
[1]吴高峰.应用PCR-DGGE技术对不同日龄仔猪肠道菌群分布规律的研究[D].郑州:河南农业大学,2009.
[2]季天荣,叶雪芳,陈庄.变性梯度凝胶电泳(DGGE)及其在动物肠道微生态研究中的应用[C].中国畜牧兽医学会动物微生态学分会第三届第八次学术研讨会论文集,广州:华南农业大学,2006:13-15.
[3]陈蓓,黄瑞.粪便标本中细菌DNA提取方法的比较[J].中国血液流变学杂志,2007,17(2):210-214.
[4]赵健元,李进华.一种高效的哺乳动物粪便DNA提取通用方法[J].激光生物学报,2008,17(5):695-700.
[5]Thakuria D,Schmidt O,Liliensiek A K.Field preservation and DNA extractionmethods for intestinalmicrobial diversity analysis in earthworms[J].Journal of Microbiological Methods,2008,10:1-8.
[6]杨德君,吴襟,刘毅,等.一种快速提取肠道微生物总DNA的方法[J].中国微生态学杂志,2006,18(2):91-93.
[7]李丽婷,许文涛,郭星,等.提取鼠肠道内微生物基因组DNA的方法研究[J].中国粮油学报,2010,25(1):122-127.
[8]LaMONTAGNE M G,MICHEL F C,Jr HOLDEN P A,et al. Evaluation of extraction and purification methods for obtaining PCR-amplifiable DNA from compost for microbial community analysis[J].Journal of Microbiology Methods,2002,49(3):255-264.
[9]Muyzer G,de Waal E C,Uitterlinden A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].Appl Environ Microbiol,1993,59(3):695-700.
[10]Kim T,Lee J,Kim S,et al.Analysis of microbial communities in doenjang,a Korean fermented soybean paste,using nested PCR-denaturing gradient gel electrophoresis[J].International Journal of Food Microbiology,2009,131(2-3):265-271.
[11]朱扬玲.采用PCR-DGGE方法研究浙江玫瑰醋酿造过程中的微生物多样性[D].杭州:浙江工商大学,2009.
[12]吴银宝,史金才,莫测辉,等.猪粪和土壤样品中微生物DNA提取方法的比较[J].农业工程学报,2006,22(增刊2):10-13.
[13]徐晓宇,阂航,刘和,等.土壤微生物总DNA提取方法的比较[J].农业生物技术学报,2005(3):377-381.
[14]徐敏娟.鹅肠道细菌多样性的分子生态学初步研究[D].扬州:扬州大学,2008.
[15]郑刚,陈己任,胡博文,等.基于DGGE分析的大鼠粪便及肠道细菌DNA提取方法研究[J].食品科学,2011,32(17):215-218.
A comparison of methods for the extraction of bacterial genomic DNA in rat feces
QIAN Yu,ZHAO Xin*
(Department of Biological and Chemical Engineering,Chongqing University of Education,Chongqing 400067,China)
In order to obtain high-quality bacterial genomic DNA,and to study the diversity of the intestinal flora,improved lysozyme method,and two kinds of DNA extraction kits were used to extract bacterial genomic DNA in rat feces.These extraction techniques were compared through measuring DNA concentration and purity,and PCR-DGGE technique to find the best method for extracting bacterial genomic DNA in rat feces.Results showed that bacterial genomic DNA was extracted by improved lysozyme method had better quality and purity,and diversity of flora.It was more suitable for the subsequent intestinal microflora molecular biology research.
bacterial genomic DNA;rat feces;gut microflora;denaturant gradient gel electrophoresis(DGGE)
TS201.3
A
1002-0306(2014)04-0166-04
2013-07-16 *通讯联系人
骞宇(1976-),女,博士,讲师,研究方向:食品科学。
重庆市科委自然科学基金项目(cstc2011jjA80001)。
