23株酿酒酵母ISSR指纹图谱分析及SCAR标记的建立
2014-01-18栾春艳李晓玲郑国斌
栾春艳,李晓玲,*,郑国斌,姚 娟,王 健
(1.三峡大学化学与生命科学学院,湖北 宜昌 443002;2.安琪酵母股份有限公司,湖北 宜昌 443003)
23株酿酒酵母ISSR指纹图谱分析及SCAR标记的建立
栾春艳1,李晓玲1,*,郑国斌2,姚 娟2,王 健2
(1.三峡大学化学与生命科学学院,湖北 宜昌 443002;2.安琪酵母股份有限公司,湖北 宜昌 443003)
目的:构建酿酒酵母菌株的简单重复序列间多态性指纹图谱数据库并建立序列特异性扩增区(sequence characterized amplifi ed region,SCAR)标记技术,为酿酒酵母菌株的分类、遗传亲缘关系鉴定及菌种专利保护提供可靠的DNA分子标记技术依据。方法:在简单重复序列间多态性(inter-simple sequence repeat,ISSR)指纹数据分析基础上进行聚类分析并对菌种进行分类鉴定,同时将酿酒酵母菌株9号和15号中扩增获得的ISSR特异性DNA带转化为可以直接用于菌株快速鉴定的SCAR分子标记。结果:构建23株酿酒酵母的ISSR指纹图谱,并在相似系数为0.85水平上将23个供试菌株分为3大类,其中,1、2、4、7、15、16、17、19、20、21、23聚为第一类;10、11、12、13、14、18号菌株聚为第二类且10号和11号菌为同一菌株;3、5、6、8、9、22号菌聚为第三类且属于同一菌株。此外,利用所获得的2个特异性条带成功转化为序列特异性扩增区分子标记。结论:在生产上酿酒酵母菌株遗传背景差异不大,常存在同物异名现象,而采用ISSR指纹及其SCAR分子标记技术快速鉴定酿酒酵母菌株在工业生产上具有重要意义。
酿酒酵母菌;简单重复序列间多态性;DNA指纹图谱;序列特异性扩增区
酵母菌是一类单细胞真菌,在自然界分布广泛,目前已知的酵母菌有1 000多种,主要生长在偏酸性潮湿的含糖环境中,如水果、蔬菜、蜜饯的内部和表面以及果园土壤中[1]。酵母菌除了应用于食品生产(如酒精饮料、酱油、食醋、馒头和面包的发酵等)中,其本身也具有很高的营养价值,除了蛋白质、碳水化合物、脂类以外,酵母还富含多种维生素、矿物质和酶类[1]。在发酵生产过程中,随着发酵环境的改变,有些酵母菌可能会发生变异退化而影响食品安全,有必要对食品生产、加工、保鲜和贮藏过程中酵母菌株的种属进行鉴定,以确定食品中哪些酵母菌是有益的,哪些是有害的,以及确定有着特定发酵特性的酵母菌是何种属。因此,快速准确的鉴定酵母菌种、分析发酵液遗传多样性、筛选有利菌种、对发酵过程进行有效控制,既可以提高产品质量,又能提高产量,增加经济效益具有重要意义。
传统的酵母菌鉴定一般要通过形态观察和生理生化实验,但这些表型性状只反映了很有限的遗传信息。由于酵母菌生长过程受到很多因素的控制,各种不同菌种之间生理生化差异不显著,而且同种菌株在鉴别时的结果也不稳定,常常导致鉴别出现误差。这些不足之处使传统的鉴定方法的应用受到了很大的限制。因此,有必要用快速、简单和可靠的鉴定方法确定酵母菌的种属。
近年来,随着分子生物学的发展和各项新技术的广泛应用,酵母菌分类鉴定工作有了飞速发展。为了弥补分类方法的不足,新的技术和方法不断地被引入酵母菌的分类研究中。其中核糖体DNA序列分析[2-3]、脉冲电场凝胶电泳技术[4]、染色体DNA的限制性片段长度多态性分析(restriction fragment length polymorphisma,RFLP)[5-6]、扩增片段长度多态性(Amplified fragment length polymorphism,AFLP)[7]、微卫星多态性分析(simple sequence repeat,SSR)[8]、基于随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)建立菌株的序列特异性扩增区(sequence characterized amplifi ed region,SCAR)[9]等方法开始广泛应用于不同酵母菌种的快速、准确的鉴定中。而简单重复序列间多态性(intersimple sequence repeat,ISSR)分子标记技术兼具SSR、RAPD、RFLP、AFLP等分子标记的优点,与SSR相比,ISSR不需要预先获知序列信息而使成本降低,且多态性更丰富;与RAPD相比,ISSR不仅具备RAPD的简便、易操作等特点,且具有较高的重复性和稳定性;与RFLP、AFLP相比,ISSR更快捷、成本较低、DNA用量小、安全性较高。基于基因组差异的ISSR及SCAR 等分子标记技术已被应用于葡萄座腔菌、黑粉菌株和土壤芽孢杆菌等[10-13]以及一些植物和真菌[14-20]的遗传多样性分析、指纹图谱构建以及菌株鉴定等方面,但尚未见应用于酵母菌种的遗传多样性分析、指纹图谱构建及快速分类鉴定的报道。本研究以安琪酵母股份有限公司菌株保藏中心保藏的23株酵母菌为研究对象,构建菌株ISSR分子标记指纹图谱并建立菌株的特异性SCAR标记,为酵母菌的遗传亲缘关系分析和菌种分类鉴定提供分子生物技术依据。
1 材料与方法
1.1 菌株与培养基
采用安琪酵母股份有限公司菌种保藏中心保藏的酵母菌,这些菌株是不同生产批次所保存的菌株和本实验室针对不同的产品需求从自然界中筛选的菌株,主要用于酿酒生产中,分别编号为1~23,采用YPD培养基进行培养活化。
1.2 酵母菌基因组的DNA提取
取新鲜培养的酵母菌体,使用北京三博远志酵母基因组DNA提取试剂盒(离心柱型)提取酵母基因组DNA。
1.3 ISSR多态性分析
引物参照哥伦比亚大学提供的序列(UBC801~UBC900),以部分供试菌DNA为模板,筛选出能扩增出清晰条带且条带具有多态性的23条引物进行ISSR分析,如表1所示。PCR反应体系(总体积25 μL):DNA 150 ng,Taq DNA 聚合酶1 U,10×PCR Buffer 2.5 μL,2.5 mmol/L dNTP 2 μL,20.0 mmol/L引物各0.5 μL,剩余体积用无菌水补齐。扩增程序:94 ℃预变性5 min,94 ℃变性30 s、52 ℃退火45 s、72 ℃延伸90 s、30个循环,最后72 ℃延伸10 min,4 ℃保存。PCR扩增产物用1.0%琼脂糖于100 V电压下电泳,在含有0.5 μg/mL溴化乙锭溶液中染色20~30 min,蒸馏水洗涤2~3次后在凝胶成像系统上观察、拍照记录。

表1 筛选出的ISSR引物扩增出的总条带数和多态性条带数Table 1 The total bands and the polymorphic bands amplified with the screened ISSR primers
1.4 聚类分析和DNA指纹图谱构建
用所选出的23条引物对23个样品基因组DNA进行PCR扩增反应。同一引物扩增出的产物,在同一位点上,有扩增条带为阳性,记录为1;没有条带为阴性,记录为0。从上到下逐条记录,转换成0、1矩阵,输入Excel表格中,用NTSYS-pc聚类分析软件进行非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)聚类分析,构建23个菌株的聚类树状图,并选择稳定的 ISSR多态性标记谱带用于构建供试菌株的ISSR 指纹图谱。
1.5 ISSR特异片段的回收 、克隆及测序
采用Biomiga的琼脂糖凝胶回收纯化试剂盒进行特异条带DNA的回收,纯化后参照pMD18-T Vector(TaKaRa D101A)试剂盒说明书进行连接实验,转化于E.coli DH5α感受态细胞,挑取适合的菌落进行PCR检测,反应体系及其扩增程序与1.3节ISSR分析相同。检测成功的样品经过含氨苄青霉素的LB液体培养基中活化送北京鼎科公司测序。
1.6 SCAR分析
根据ISSR特异性条带测序结果,应用引物设计软件Primer Premier5.0设计SCAR 标记引物对,并由生工生物工程(上海)股份有限公司合成引物对。采用设计的特异性引物对23个菌株进行 PCR 扩增,验证SCAR标记。SCAR标记的PCR 反应体系:总体积25 μL,DNA 150 ng,Taq DNA聚合酶1 U,10×PCR Buffer 2.5 μL,2.5 mmol/L dNTP 2 μL ,20.0 mmol/L正反引物各0.5 μL,剩余体积用无菌水补齐。
2 结果与分析
2.1 ISSR扩增多态性分析
从100条引物中筛选出23条有多态性的引物,23条引物共扩增出条带数351,其中多态性条带数263,多态性比率66.7%(UBC 807)~93.9%(UBC 896),平均为74.9%。不同引物扩增的总条带数和多态性带数有一定的差异,23条引物表现出较高的多态性。引物827和896的多态性比率最高为93.8%,引物817的多态性比率最低为54.5%。
2.2 ISSR聚类分析
根据ISSR标记的扩增结果,应用NTSYS软件对23个菌株进行聚类分析,得到的聚类图如图1所示。在相似系数为0.85水平上将23个供试菌株分为三大类。其中,1、4、2、7、15、16、19、20、17、21、23号菌株聚为第一类;10、11、12、13、14、18号菌株聚为第二类;3、5、6、8、9、22号菌聚为第三类。其中第一大类又分为两个亚类,一个亚类是1、4、2、7;另一个亚类是15、16、19、20、17、21、23。此外,从聚类分析结果也可看出10、11号菌株在100%水平聚为一类,可能为同一菌株;同样的3、5、6、8、9、22号菌在100%水平聚为第三类,说明它们的遗传背景相同,也可能为同一菌株。

图1 ISSR聚类分析图Fig.1 Dendrogram based on ISSR cluster analysis of the 23 S. cerevisiae strains
2.3 ISSR指纹图谱的构建
从100个引物中选择23个多态性标记谱带,依次排序,用来构建23个供试菌株ISSR指纹图谱,如表2所示。
表2 酿酒酵母23个菌株的计算机指纹图谱Table 2 Computerized ISSR fi ngerprinti ng of the 23 S. cerevisiae

表2 酿酒酵母23个菌株的计算机指纹图谱Table 2 Computerized ISSR fi ngerprinti ng of the 23 S. cerevisiae
菌株编号 计算机化指纹图谱 菌株编号 计算机化指纹图谱1 00000001000101100010010 13 00001010000000110000100 2 00001000100001000010010 14 00000000000001010000100 3 11110100110010001101001 15 00000001001101000010010 4 00001000000101100000000 16 00010001001101000010010 5 11110100110010001101001 17 00000000001101000110000 6 11110100110010001101001 18 00000010000100100100010 7 00000001000101100010000 19 00000001001101100110010 8 11110100110010001101001 20 00000011001101100110010 9 11110100110010001101001 21 00000001001101000110010 10 00010000000101100000000 22 11110100110010001101001 11 00010000000101100000000 23 00000001001101010110010 12 00000010000000110000100
2.4 SCAR标记的建立
对23个ISSR引物的扩增图进行分析,已经确定3、5、6、8、9、22号菌株具有相同的扩增带型,可视为同一个菌株,因此找寻特异性条带是将以上6个菌株归为同一种菌株,且以9号菌为代表。在所有扩增图中初步确定有2个特异性的DNA条带,分别出现在引物UBC855对9号菌株的扩增条带中(图2,箭头所示)和引物UBC827对15号菌株的扩增条带中(图3,箭头所示)。
用1.0%的琼脂糖胶检测所回收2个菌株特异性DNA带。根据DNA片段大小,初步判断检测到的DNA条带正是所需的目标DNA。通过菌落PCR电泳检测并挑取有效克隆的菌体,分别进行测序。据所测出的完整序列,应用引物设计软件Primer Premier 5.0设计相应2对特异性引物,分别编号为S9-1和S9-2;S15-1和S15-2,其序列分别为:5’-TCCAGGTGGTATCGCTTAT-3’,5’-A G T T C T G C T C C A A T C G T G-3’;5’-C C A C G C T C A T T A T T T G T T-3’,5’-GGCTACTTATGCCATTCC-3’。用所设计的4对引物对23个菌株的总DNA进行扩增获得预期结果:特异性引物只对特定的菌株有唯一的1条扩增带,通过比对大小可确定为所期望的目标条带,而在其他供试菌株中均无相应的扩增带(图4、5),这表明ISSR标记已成功转化为SCAR标记。
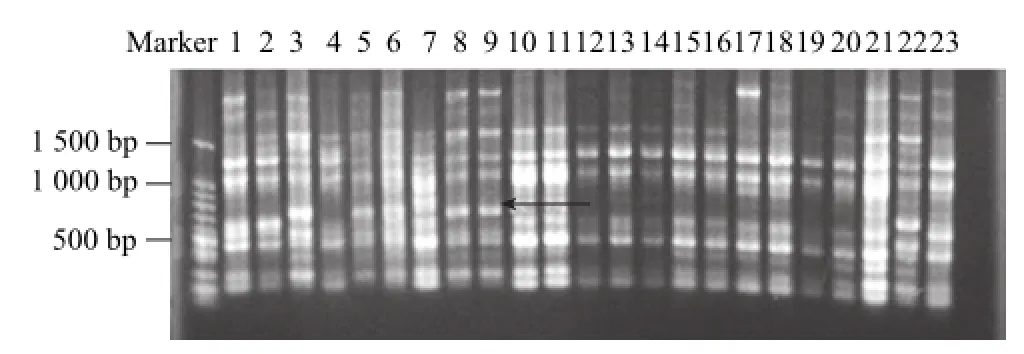
图2 引物UBC855对23个菌株的ISSR扩增检测结果Fig.2 The amplification results of the 23 S. cerevisiae strains by primer UBC855
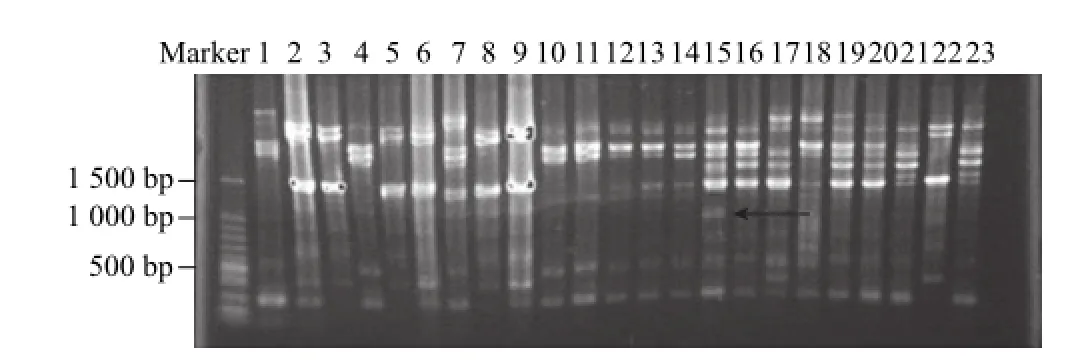
图3 引物UBC827对23个菌株的ISSR扩增检测结果Fig.3 The amplification results of the 23 S.cerevisiae strains by primer UBC827

图4 引物对S9-1和S9-2对23个菌株的特异检测结果Fig.4 The specificity of the primer pairs S3-1 and S3-2 for the 23 strains of S. cerevisiae
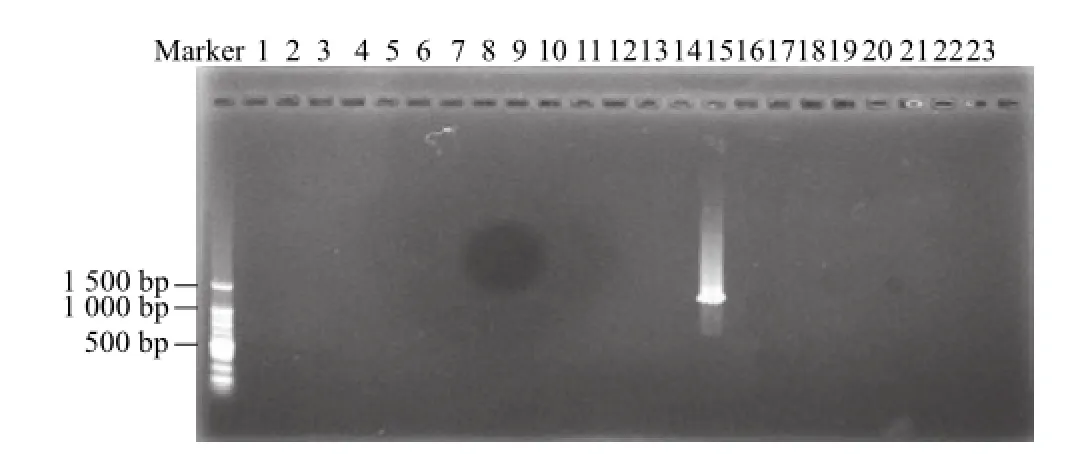
图5 引物对S15-1和S15-2对23个菌株的特异检测结果Fig. 5 The specificity of the primer pairs S15-1 and S15-2 for 23 S.cerevisiae strains
3 讨 论
本研究采用ISSR分子标记技术对23个酿酒酵母菌株进行指纹图谱构建,并进行了聚类分析,结果表明23个菌株的遗传背景相似度高,可能和选育过程中亲本来源含有共有的骨干亲本有关。此外,还鉴别出3、5、6、8、9和22号这6个菌株为同一菌株,10、11号菌为同一菌株,可能是同株不同名,当然还需要结合其他分子标记和表型形状数据进一步分析确定,这些菌株是否真正是异名同株,或者是在菌株运输、保存及使用过程中发生菌株混淆。
SCAR标记是在RAPD技术基础上发展起来的。SCAR标记是将目标RAPD片段进行克隆并对其末端测序,根据RAPD片段两端序列设计特异引物,对基因DNA 片段再进行PCR特异扩增,把与原RAPD片段相对应的单一位点鉴别出来。SCAR标记是共显性遗传,待检DNA 间的差异可直接通过有无扩增产物来显示。SCAR标记方便、快捷、可靠,可以快速检测大量个体,结果稳定性好,重现性高等特点。2001年,Hermosa等[13]报道用SCAR标记可对木霉菌种进行鉴定,2005年吴学谦等[21]报道用SCAR标记快速准确地鉴定香菇菌株的真伪。2011年,SCAR标记方法在酵母菌株鉴定中也开始得到应用,Wang Pinmei等[22]通过RAPD分析得到了用于酵母菌株鉴定的SCAR标记。本实验在构建23个酿酒酵母菌株ISSR指纹图谱的基础上,进一步构建9号和15号菌株的ISSRSCAR标记。9号和15号菌株是生产中常用的酿酒酵母菌株,在高渗环境下具有较强的耐高渗能力。在工业生产中使用耐高渗能力强的菌种会极大地提高生产效率。因此筛选并鉴定耐高渗能力强的菌株已成为酵母菌种选育的一个重要方向。因此基于ISSR指纹图谱的基础上本实验重点构建9号和15号菌株的ISSR-SCAR,分别选取9号和15号菌株的特异性条带回收、克隆及测序,然后设计特异性引物对23个菌株进行PCR扩增,出现了目标条带,这表明ISSR标记已成功转化为SCAR标记。
实际上本实验中共找到了6个特异性条带,但其中成功转化为SCAR标记的只有2个。虽然SCAR标记可以由RAPD,ISSR等标记转化而来,但这个转化过程确实相当复杂,本实验中转化率只有33%。转化失败的原因可能是回收纯化,引物设计或退火温度没有控制好等。在检测SCAR标记的过程中出现了所设计的特异性引物可以对多个菌株进行扩增的情况,可能是这些菌株的亲缘关系太近造成的。SCAR标记需要在较高的退火温度下实现特异扩增,一般比其Tm值高2~5 ℃,此外,特异引物的采用也排除了随机引物结合位点之间的竞争,稳定性和可重复性显著提高,使得SCAR标记的结果更加可靠。ISSR标记本就具有较高的重复性,再加上SCAR标记的快速、简便、低廉的特点,通过大量ISSR引物筛选获得每个菌株的特异 SCAR 标记。本研究建立的SCAR标记技术体系可有效区分目标菌株和其他菌株,再结合本实验室的脉冲电场凝胶电泳技术(另文发表),可以多技术互补,建立准确高效的酵母鉴定体系,为酵母菌种的专利保护、遗传育种、菌种改良和工业生产应用奠定强有力的技术基础。
[1] 杨静静, 孟镇, 钟其顶, 等. 分子生物学技术在酵母菌多相分类鉴定中的应用[J]. 中国酿造, 2011(4): 16-20.
[2] 徐艳文, 杨莹, 薛军侠, 等. 26S rDNA-RFLP分析在非酿酒酵母菌分类研究中的应用[J]. 微生物学杂志, 2007, 27(4): 29-32.
[3] TAKASHINMA M, NAKASE T. Four new species of the genus Sporobolomyce isolated from leaves in Thailand[J]. Mycoscience, 2001, 41: 357-369.
[4] 白逢彦, 贾建华. 脉冲电泳核型分析在酿酒酵母菌分类学研究中的应用[J]. 微生物学报, 2000, 40(1): 9-13.
[5] JOSEPA S, JOSEP C, BRAULIO E Z, et al. Isolation and identification of yeasts associated with vineyard and winery by RFLP analysis of ribosomal genes and mitochondrial DNA[J]. Microbiological Research, 2002, 157: 267-274.
[6] PILAR S T, PATROCINIO G, ROSA L, et al. Analysis of yeast population during spontaneous alcoholic fermentation: effect of the age of the cellar and the practice of inoculation[J]. International Journal of Food Microbiology, 2005,103: 49- 56.
[7] FLORES E P, ALBA J F. The uses of AFLP for detecting DNA polymorphism, genotype identification and genetic diversity between yeasts isolated from Mexican agave-distilled beverages and from grape musts[J]. Letters in Applied Microbiology, 2005, 41: 147-152.
[8] DORIT S, MARGARIDA C. The genetic structure of fermentative vineyard-associated Saccharomyces cerevisiae populations revealed by microsatellite analysis[J]. Antonie van Leeuw Enhoek, 2007, 91: 137-150.
[9] CLERCQ D D, COGNET S, PUJOL M, et al. Development of a SCAR marker and a semi-selective medium for specific quantification of Pichia anomala strain K on apple fruit surfaces[J]. Postharvest Biology and Technology, 2003, 29: 237-247.
[10] DEMETRIOU M C, THOMPSON G A, WRIGHT G C, et al. A molecular approach for the diagnosis of wood rotting disease in desert eitrus[J]. Myeologia, 2000, 92(6): 1214-1219.
[11] MENZIES J G, BAKKEREN G, MATHESON F. Use of inter-simple sequence repeats and amplified fragment length polymorphisms to analyze genetic relationships among small grain infecting species of Ustilago[J]. Ecology and Population Biology, 2003, 23(2): 67-175.
[12] 耿文义, 郭荣君, 李世东, 等. 用于土壤中生防芽孢杆菌B006和BH1检测的SCAR标记及其特异性[J]. 中国生物防治学报, 2011, 27(2): 233-240.
[13] HERMOSA M R, GRONDONA I, MINGUEZ J M D. Development of a strain- specific SCAR marker for the detection of Trichoderma atroviride 11, a biological control agent against soilborne fungal plant pathogens[J]. Current Genetics, 2001, 38(6): 343-350.
[14] 许美燕, 唐传红, 张劲松, 等. 利用SRAP和ISSR建立快速鉴定灵芝属菌株的SCAR标记[J]. 菌物学报, 2008, 27(5): 707-717.
[15] 宋春艳, 谭琦, 陈明杰. 香菇135菌SCAR标记的验证[J]. 食用菌学报, 2006, 13(3): 1-7.
[16] TERZOPOULOS P J, KOLANO B, BEBELI P J, et al. Identifi cation of Olea europaea L. cultivars using inter-simple sequence repeat markers[J]. Scientia Horticulturae, 2005, 105: 45-51.
[17] 唐利华, 肖扬, 边银丙. 中国黑木耳主要栽培菌株ISSR指纹分析及SCAR标记[J]. 菌物学报, 2008, 27(2): 243-251.
[18] 汪斌, 祁伟, 兰涛, 等. 应用 ISSR 分子标记绘制红麻种质资源 DNA指纹图谱[J]. 作物学报, 2011, 37(6): 1116-1123.
[19] 王海飞, 关建平, 马钰, 等. 中国蚕豆种质资源ISSR标记遗传多样性分析[J]. 作物学报, 2011, 37(4): 595-602.
[20] 易克, 徐向利, 卢向阳, 等. 利用SSR和ISSR标记技术构建西瓜分子遗传图谱[J]. 湖南农业大学学报: 自然科学版, 2008, 29(4): 333-337.
[21] 吴学谦, 李海波, 魏海龙, 等. SCAR分子标记技术在香菇菌株鉴定上的应用研究[J]. 菌物学报, 2005, 24(2): 259-266.
[22] WANG Pinmei, WU Xuechang, CHI X Q, et al. Development and application of RAPD-SCAR markers to identify intra-species hybrids of industrial Saccharomyces cerevisiae[J]. World Journal of Microbiology and Biotechnology, 2011, 27(1): 185-188.
ISSR Fingerprint Analysis and SCAR Marker of 23 Strains of Saccharomyces cerevisiae
LUAN Chun-yan1, LI Xiao-ling1,*, ZHENG Guo-bin2, YAO Juan2, WANG Jian2
(1. College of Chemistry and Life Science, China Three Gorges University, Yichang 443002, China; 2. Angel Yeast Co. Ltd., Yichang 443003, China)
Objective: To construct inter-simple sequence repeat (ISSR) DNA fi ngerprinting database of Saccharomyces cerevisiae and to establish corresponding specifi c sequence characterized amplifi ed region (SCAR) markers for use in the classifi cation, identifi cation and analysis of the genetic relationship among S. cerevisiae strains and patent protection. Methods: On the basis of ISSR DNA fi ngerprinting analysis, 23 Saccharomyces cerevisiae strains were clustered by UPGMA and their classifi cation and identifi cation were conducted. Then two specifi c ISSR bands from strains 9 and 15 were converted into two SCAR markers for rapid strain identifi cation. Results: The 23 strains could be divided into three categories. Of these, category I included strains 1, 2, 4, 7, 15, 16, 17, 19, 20, 21 and 23, category II included strains 10, 11, 12, 13, 14 and 18 in which strains 10 and 11 were the same, and category Ⅲincluded strains 3, 5, 6, 8, 9 and 22 which belonged to the same strain with a similarity coeffi cient of 0.85. Furthermore, two specifi c bands were converted into SCAR marker for strain identifi cation. Conclusion: The development of ISSR fi ngerprints and SCAR markers for rapid identifi cation of Saccharomyces cerevisiae strains is signifi cant for industrial production because there is no signifi cant difference in genetic background of Saccharomyces cerevisiae strains and it is often occurs that the same strains are called different names.
Saccharomyces cerevisiae; inter-simple sequence repeat (ISSR); DNA fingerprint; sequence characterized amplified region (SCAR)
Q939.97
A
1002-6630(2014)03-0163-05
10.7506/spkx1002-6630-201403033
2013-03-12
安琪酵母股份有限公司分子生物学技术平台建设项目(SDHZ20100138)
栾春艳(1988—),女,硕士研究生,研究方向为酵母分子生物学。E-mail:364595197@qq.com
*通信作者:李晓玲(1973—),女,副教授,博士,研究方向为分子生物学方法的应用。E-mail:lixiaolinggz@126.com
