MADS-box基因家族在蒺藜苜蓿的全基因组分析
2014-01-02张军宋丽莉郭东林郭长虹束永俊
张军,宋丽莉,郭东林,郭长虹,束永俊
(黑龙江省分子细胞遗传与遗传育种重点实验室 哈尔滨师范大学生命科学与技术学院,黑龙江 哈尔滨150025)
MADS-box基因家族是一类转录因子,广泛地存在于动物、植物和真菌等真核生物,它们在N端含有一个由58~60个氨基酸组成的保守结构域,称为 MADS-box结构域[1-2]。MADS-box是一个可以结合DNA序列的结构域,它可以识别结合CArG基序(CC[A/T]6GG),并激活下游基因的表达[3]。根据分子系统进化分析,MADS-box基因家族可以分为两大类:I型和Ⅱ型,其中:Ⅰ型主要是指含有SRF结构域,Ⅱ型主要含有MEF2类似结构域和植物中特异的MIKC类MADS-box基因。结合MADS-box基因的结构特征,可以将MADS-box家族分成5个小类:Ⅰ型的 Mα、Mβ和 Mγ;Ⅱ型的 MIKC和 MIKC*[1-2]。
在植物基因组中,MIKC类MADS-box转录因子的结构和功能研究比较清楚,它们通常含有4个结构域,分别为:MADS-box(M)、Intervening domain(I)、Kertain-like domain(K)和 C-terminal domain(C)[3]。这些转录因子在植物基因组特有的,在各种植物基因组之间是非常保守的,在植物生长、发育等过程起到重要的调控作用,比如:SOC1(SUPPRESSOR OF OVERESPRESSION OF CONSTANS1)、FLC1(FLOWERING LOCUS c),AGL24(AGAMOUS-LIKE GENE 24)、MAF1/FLM (MADS AFFECTING FLOWERING)和SVP (SHORT VEGETATIVE PHASE)等 MADS-box基因调控植物的开花时间[4-9];AP1(APETALA 1)、FUL(FRUITFUL)和CAL(CAULIFLOWER)等 MADS-box基因调控花芽组织的形成[10-12];AP1、SEP1-3(SEPALLATA 1-3)、AP3(APETALA 3)、PI(PISTILLATA)和 AG(AGAMOUS)等 MADS-box基因控制植物花器官的形成和种子的发育[13-15]。
根据分类学定义:苜蓿属含有4个亚属,56个种,其中最受科研人员关注的有:紫花苜蓿(Medicagosativa)和蒺藜苜蓿(Medicagotruncatula)两个种。紫花苜蓿是四倍体植物,具有优良的农艺性状,是全世界种植范围最广的牧草作物;蒺藜苜蓿是二倍体植物,其基因组较小(约470Mb),已经完成基因组测序,成为研究豆科,特别是苜蓿属(如紫花苜蓿)的模式植物[16-18]。在紫花苜蓿长期种植生产过程中,科研人员多重视苜蓿营养体性状,如产量、品质、抗性等,对苜蓿生殖过程性状关注较少,导致紫花苜蓿的种子生产水平一直低下,严重制约了紫花苜蓿的种植推广[17],同时,其他草类植物也存在类似的问题[19-20]。
本研究将对蒺藜苜蓿的基因组测序数据进行结构域搜索,鉴定MADS-box基因家族成员。通过序列比对和系统进化分析,完成MADS-box基因家族成员的分类,同时,根据MADS-box基因家族的染色体定位信息,明确其在基因组的分布特征。最后,结合蒺藜苜蓿的RNA-seq数据,分析MADS-box家族在蒺藜苜蓿心皮和花等生殖器官发育过程的表达谱,为解析蒺藜苜蓿中MADS-box基因家族的重要作用提供参考。
1 材料与方法
1.1 数据来源
蒺藜苜蓿基因组测序数据、基因转录序列、CDS序列、蛋白质序列及其注释信息[18](版本为:Mt4.0v1)均下载自JCVI(http://www.jcvi.org/medicago/)。
1.2 蒺藜苜蓿MADS-box基因家族的鉴定和分类
MADS-box基因的结构域信息(PF00319)下载自Pfam 数据库[21],利用软件 HMMER[22](V3.0)搜索蒺藜苜蓿的蛋白质序列,运行参数为:-E 0.01。将挖掘的MADS-box基因比对拟南芥的 MADS-box基因,根据拟南芥的MADS-box基因分类信息对蒺藜苜蓿MADS-box基因家族进行分类。同时,提取蒺藜苜蓿 MADS-box基因的注释信息,确定其内含子分布信息。
1.3 蒺藜苜蓿MADS-box基因家族的系统进化分析
提取蒺藜苜蓿MADS-box基因家族的蛋白质序列,利用ClustalW2[23]进行多重序列比较,比对结果采用MEGA4[24]进行系统进化分析,系统进化分析参数如下:1)建树方法为邻近法(neighbor-joining,NJ);2)遗传距离为泊松距离(Poisson correction);3)抽样次数为1000(bootstrap:1000replications)。
1.4 蒺藜苜蓿MADS-box基因家族的染色体定位分析
从蒺藜苜蓿基因组中提取MADS-box基因的基因组序列和CDS序列,利用BLAST[25]进行两两比对。当2个MADS-box基因的一致性超过85%时,则将这2个 MADS-box基因之间存在基因复制(gene duplication)。提取所有MADS-box基因在蒺藜苜蓿基因中的位置信息,结合MADS-box基因间的基因复制情况,利用软件CIRCOS[26]绘制MADS-box基因家族在蒺藜苜蓿基因组中的分布情况。
1.5 蒺藜苜蓿MADS-box基因家族的表达分析
蒺藜苜蓿的转录组测序(RNA-seq)数据[18]下载自 NCBI的SRA 数据库(http://www.ncbi.nlm.nih.gov,登录号为:SRR350517-SRR350521,SRR350538和SRR349692)。转录组数据包含蒺藜苜蓿的根部(root),根部结瘤(nodule),叶片(blade),芽(bud),心皮(seedpod)和花(flower)6个组织和部位。转录组数据采用 TopHat[27]和Cufflink[28]进行分析,获得蒺藜苜蓿基因的表达量(fragments per kilobase of exon per million fragments mapped,FPKM值)。利用MATLAB(R2008B)提取 MADS-box基因的表达量,去除表达量较低的 MADS-box基因(FPKM值小于1),然后,对剩下的 MADS-box基因表达量进行对数转换和标准化,最后,对蒺藜苜蓿MADS-box基因的表达情况进行聚类分析。
2 结果与分析
2.1 蒺藜苜蓿MADS-box基因家族的鉴定和分类
通过HMMER搜索,蒺藜苜蓿基因组总共鉴定出138个MADS-box基因家族成员,如表1所示。这些MADS-box基因主要分成两大类,即Ⅰ型和Ⅱ型MADS-box基因,其中:Ⅱ型MADS-box基因有46个,包含有MIKC(41个)和 MIKC*(5个)两类;Ⅰ型 MADS-box基因有92个,包含有 Mα(49个)、Mβ(7个)和 Mγ(36个)3类。两类MADS-box基因中,Ⅱ型MADS-box基因大多数都含有多个内含子,多数为6~8个,甚至超过10个,如MtMADS044,45和46都含有10~11个内含子;而Ⅰ型MADS-box基因大多数不含有内含子或者含有1个内含子。与其他植物相比,蒺藜苜蓿基因组中MADS-box基因家族成员总数差别不大,如拟南芥为107,水稻为75,大豆为106,但是,成员组成差异较大,蒺藜苜蓿Ⅱ型与Ⅰ型分别为46和92个,Ⅱ型占总MADS-box基因家族的33%,拟南芥为42%,水稻为57%[29],大豆为68%[30],蒺藜苜蓿的Ⅱ型MADS-box基因比例明显偏低。

表1 蒺藜苜蓿基因组中鉴定的MADS-box基因Table 1 The MADS-box genes identified in M. truncatula

续表1 Continued
2.2 蒺藜苜蓿MADS-box基因家族的系统进化分析
利用ClustalW2和MEGA进行系统进化分析,如图1所示。结果显示,在系统进化上,Ⅱ型和Ⅰ型MADS-box基因是各自独立系统演化,两种之间没有交叉。其中:Ⅱ型中的MIKC类保守性较好,独自分成一支;MIKC*类保守性稍微弱一些,分成两邻近的两支;Ⅰ型的3个类:Mα、Mβ和Mγ,总体上系统分类良好,大多数成员都可以正确的分类,只有MtMADS073、130和134这3个成员进化关系出现不一致。这也说明通过MADS-box基因在植物中保守性较强,可以通过拟南芥的分类信息鉴定蒺藜苜蓿MADS-box基因家族的分类情况。
2.3 蒺藜苜蓿MADS-box基因家族的染色体定位分析
通过提取蒺藜苜蓿MADS-box基因的染色体定位信息,发现4个(MtMADS001、47、48和96)定位在尚未完全组装的长片段上,剩下的134个成员定位在8条染色体上,如图2所示。每条染色体分布有5~27个MADS-box基因,其中:1号染色体最多为27个,其次为3号和4号染色体,分别为26和23个;6号染色体最少,只有5个。此外,MADS-box基因家族在蒺藜苜蓿染色体组上不是均匀分布,它们呈聚集形式分布,如1,3,4和5号染色体上都有多个MADS-box的基因簇。通过两两比对分析发现:多数蒺藜苜蓿MADS-box基因都拥有2个或以上的拷贝,即存在基因复制情况,其中:Ⅱ型MADS-box基因成员复制较少,如图2中红色(MIKC)和浅红色(MIKC*)线条所示,Ⅰ型的基因复制较多,如图2中蓝色(Mα)、浅蓝色(Mβ)和紫色(Mγ)线条所示。
2.4 蒺藜苜蓿MADS-box基因家族的表达分析
通过下载NCBI数据库中蒺藜苜蓿的RNA-seq数据,分析得到蒺藜苜蓿MADS-box基因家族在6种组织的表达谱。蒺藜苜蓿的表达谱显示,多数MADS-box基因(91/138,66%)FPKM都小于1,说明这些MADS-box基因在6种组织中的表达量极低或者不表达,其中:Ⅰ型MADS-box基因有75个,Ⅱ型MADS-box基因有26个。剩下47个MADS-box基因的表达谱进行聚类分析,如图3所示。根据表达谱信息,47个基因主要可以分成3组:A组含有13个基因,其中Ⅰ型8个,Ⅱ型5个,主要在蒺藜苜蓿的心皮和花等生殖器官中表达;B组含有16个基因,其中Ⅰ型9个,Ⅱ型7个,这些MADS-box基因虽然表达,但是在各种组织中表达量都不高;C组含有12个基因,其中Ⅰ型4个,Ⅱ型8个,主要在蒺藜苜蓿的根部、结瘤、叶片和芽中表达,在心皮和花组织中表达量较低。

图1 蒺藜苜蓿MADS-box基因家族的系统进化分析Fig.1 Phylogenetic tree of MADS-box gene family in M. truncatula
3 讨论
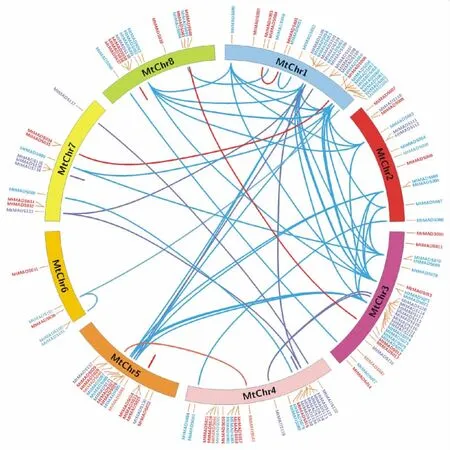
图2 蒺藜苜蓿MADS-box基因在染色体定位Fig.2 Chromosomal locations of MADS-box genes in M. truncatula

图3 蒺藜苜蓿MADS-box基因表达的聚类分析Fig.3 Heat map of MADS-box gene expression obtained from RNA-seq in M. truncatula
通过全基因组分析,从蒺藜苜蓿中鉴定了138个MADS-box基因,其中Ⅱ型MADS-box基因46个,这与拟南芥(Arabidopsisthaliana,45个)和水稻(Oryzasativa,43个)等植物的报导一致,但是比大豆(Glycinemax)中Ⅱ型MADS-box基因(72个)要少,这可能是由于大豆基因组发生加倍,是古四倍体造成。同时,Ⅱ型MADS-box基因一般含有多个内含子,Ⅰ型一般不含有或者只含有1个内含子,通常含有多个内含子的基因一般比较保守,而不含有内含子的基因保守性较差[1,9]。此外,蒺藜苜蓿的Ⅰ型和Ⅱ型MADS-box基因在基因组分布模式也有差异,Ⅱ型基本上遍布基因组各条染色体上(2~10个),比较均匀;Ⅰ型只是集中在少数染色体上,如:1号染色体(22个)和3号染色体(19个),其他染色体(6号染色体,3个)上极少,呈基因簇状分布。最后,比较基因组学和表达谱分析结果显示,蒺藜苜蓿的Ⅰ型MADS-box基因含有大量的复制基因,大多基因都不表达或者表达量极低;而Ⅰ型MADS-box基因的复制较少,且表达模式较为稳定。综合上面可以发现,在蒺藜苜蓿基因组中,Ⅰ型MADS-box基因处于积极复制的“扩张期”,虽然基因数量较多,但是参与调控的过程较少;而Ⅱ型MADS-box基因基本进入“稳定期”,基因复制较少,家族成员数量也较少,但是,这些基因保守性好,积极参与蒺藜苜蓿器官形成和发育等过程的调控。
通过蒺藜苜蓿的RNA-seq数据分析发现,大多数Ⅰ型MADS-box基因成员不表达或者表达量极低,而Ⅱ型MADS-box的表达量相对较高,这也为Ⅱ型MADS-box基因在蒺藜苜蓿的器官发育和形态建成过程中的重要调控作用奠定了基础。在蒺藜苜蓿MADS-box基因家族的表达谱中,A组基因(图3)主要是调控蒺藜苜蓿生殖器官花和心皮的发育和形成,其中Ⅱ型MADS-box基因有5个。通过同源搜索和系统进化分析发现,它们分别属于SEP(MtMADS012和 MtMADS020)、AP3/PI(MtMADS014和 MtMADS044)和 AP1(MtMADS039)等亚家族,在拟南芥、水稻等植物中,这3个亚家族也参与花等生殖器官的发生和形成,说明这些MADS-box基因的功能高度保守,在蒺藜苜蓿中也通过这些MADS-box基因的表达调控控制花等生殖器官的形态发生。此外,C组基因主要调控蒺藜苜蓿根部、叶片和芽等组织的分化和形态形成,其中Ⅱ型MADS-box基因有8个,分别属于SOC1(MtMADS017、MtMADS035和 MtMADS036)、ANR1(MtMADS019、MtMADS023和 MtMADS029)以及SVP(MtMADS018和MtMADS028),它们在各个组织中表达量都较高,参与植物各个器官的发育,这与其他植物中的报道类似,这就意味着蒺藜苜蓿的Ⅱ型MADS-box基因无论从结构上,还是表达模式上,甚至是生物学功能上都非常保守[1,3,29-30]。
4 结论
本研究采用结构域搜索的方法,在蒺藜苜蓿基因组中鉴定了MADS-box基因家族的全部基因成员,并通过序列比对和系统进化方法,确定了MADS-box基因家族的分类和进化关系。通过染色体定位分析,研究了蒺藜苜蓿中MADS-box基因家族的演化特点。同时,结合RNA-seq的表达谱,阐述了MADS-box基因家族在植物器官发育,特别是生殖器官发育过程中的重要调控作用,这将为揭示蒺藜苜蓿种子形成机制提供参考,也为解析紫花苜蓿种子生长过程提供重要的借鉴作用。
[1]Theiβen G,Becker A,Di Rosa A,etal.A short history of MADS-box genes in plants[J].Plant Molecular Biology,2000,42(1):115-149.
[2]Becker A,Winter K-U,Meyer B,etal.MADS-box gene diversity in seed plants 300million years ago[J].Molecular Biology and Evolution,2000,17(10):1425-1434.
[3]De Bodt S,Raes J,Van de Peer Y,etal.And then there were many:MADS goes genomic[J].Trends in Plant Science,2003,8(10):475-483.
[4]Michaels S D,Amasino R M.FLOWERING LOCUS C encodes a novel MADS domain protein that acts as a repressor of flowering[J].The Plant Cell,1999,11(5):949-956.
[5]Hartmann U,Hhmann S,Nettesheim K,etal.Molecular cloning of SVP:a negative regulator of the floral transition inArabidopsis[J].The Plant Journal,2000,21(4):351-360.
[6]Samach A,Onouchi H,Gold S E,etal.Distinct roles ofCONSTANStarget genes in reproductive development ofArabidopsis[J].Science,2000,288:1613-1616.
[7]Scortecci K C,Michaels S D,Amasino R M.Identification of a MADS-box gene,FLOWERING LOCUS M,that represses flowering[J].The Plant Journal,2001,26(2):229-236.
[8]Michaels S D,Ditta G,Gustafson-Brown C,etal.AGL24acts as a promoter of flowering inArabidopsisand is positively regulated by vernalization[J].The Plant Journal,2003,33(5):867-874.
[9]Kaufmann K,Melzer R,Theiβen G.MIKC-type MADS-domain proteins:structural modularity,protein interactions and network evolution in land plants[J].Gene,2005,347(2):183-198.
[10]Alejandra Mandel M,Gustafson-Brown C,Savidge B,etal.Molecular characterization of theArabidopsisfloral homeotic geneAPETALA1[J].Nature,1992,360:273-277.
[11]Bowman J L,Alvarez J,Weigel D,etal.Control of flower development inArabidopsisthalianabyAPETALA1and interacting genes[J].Development,1993,119(3):721-743.
[12]Gu Q,Ferrandiz C,Yanofsky M F,etal.The FRUITFULL MADS-box gene mediates cell differentiation duringArabidopsisfruit development[J].Development,1998,125(8):1509-1517.
[13]Pelaz S,Ditta G S,Baumann E,etal.B and C floral organ identity functions require SEPALLATA MADS-box genes[J].Nature,2000,405:200-203.
[14]Liljegren S J,Ditta G S,Eshed Y,etal.SHATTERPROOF MADS-box genes control seed dispersal inArabidopsis[J].Nature,2000,404:766-770.
[15]Nesi N,Debeaujon I,Jond C,etal.The TRANSPARENTTESTA16locus encodes the ARABIDOPSIS BSISTER MADS domain protein and is required for proper development and pigmentation of the seed coat[J].The Plant Cell,2002,14(10):2463-2479.
[16]江腾,林勇祥,刘雪,等.苜蓿全基因组 WRKY转录因子基因的分析[J].草业学报,2011,20(3):211-218.
[17]刘志鹏,张吉宇,王彦荣.紫花苜蓿配子体发育遗传调控的研究进展[J].草业学报,2011,20(4):270-278.
[18]Young N D,Debelle F,Oldroyd G E,etal.TheMedicagogenome provides insight into the evolution of rhizobial symbioses[J].Nature,2011,480:520-524.
[19]吕奉菊,崔美辰,陈明林.蚕茧草的繁殖生物学研究[J].草业学报,2013,22(3):196-203.
[20]黄利春,金樑,张树振,等.蝶形花亚科植物花粉释放机制[J].草业学报,2013,22(6):305-314.
[21]Finn R D,Mistry J,Schuster-Bckler B,etal.Pfam:clans,web tools and services[J].Nucleic Acids Research,2006,34(S1):247-251.
[22]Finn R D,Clements J,Eddy S R.HMMER web server:interactive sequence similarity searching[J].Nucleic Acids Research,2011,39(S2):29-37.
[23]Thompson J D,Higgins D G,Gibson T J.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22(22):4673-4680.
[24]Tamura K,Dudley J,Nei M,etal.MEGA4:molecular evolutionary genetics analysis(MEGA)software wersion 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[25]Altschul S F,Madden T L,Schaffer A A,etal.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Res,1997,25(17):3389-3402.
[26]Krzywinski M I,Schein J E,Birol I,etal.Circos:An information aesthetic for comparative genomics[J].Genome Research,2009,19(9):1639-1645.
[27]Trapnell C,Pachter L,Salzberg S L.TopHat:discovering splice junctions with RNA-Seq[J].Bioinformatics,2009,25(9):1105-1111.
[28]Trapnell C,Williams B A,Pertea G,etal.Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J].Nat Biotech,2010,28(5):511-515.
[29]Zhao Y,Li X,Chen W,etal.Whole-genome survey and characterization of MADS-box gene family in maize and sorghum[J].Plant Cell,Tissue and Organ Culture,2011,105(2):159-173.
[30]Shu Y,Yu D,Wang D,etal.Genome-wide survey and expression analysis of the MADS-box gene family in soybean[J].Molecular Biology Reports,2013,40(6):3901-3911.
