转基因玉米种子快速筛查方法研究与应用
2013-12-23吴明生云晓敏宋歌牛茜彭瑞迪
吴明生 云晓敏 宋歌 牛茜 彭瑞迪
(北京市种子管理站,北京 100088)
据国际农业生物技术应用服务组织(ISAAA)发布的最新资料显示[1],2011年全球商业化种植的转基因作物面积持续增加,达到1.6亿hm2,实现连续15年(1996-2011年)的增长。其中转基因玉米种植面积达到5 100万hm2,占到总面积的32%。而与此同时,我国也把转基因作物研发作为一项国家战略加以推进,新育成转基因作物品种越来越多,转基因玉米、水稻等主粮作物的商业化也将被提上日程。
然而科学界对于转基因植物的安全性备存争议,使其存在一定的不确定性。普遍观点认为转基因食品安全以及生态安全性还有待长期、持续的跟踪研究。因此,我国目前一方面在加大转基因研发力度,同时也在强化对转基因的监管,严控农业转基因污染、保障我国转基因产业健康发展。而快速、高效、准确的转基因检测技术研究及应用是加大农业转基因监管力度,提高科学管理水平的重要手段之一。
DNA快速提取和高效的DNA扩增技术是影响转基因种子快速检测的关键环节。本研究将热碱法[2]用于玉米种子粉末样品的DNA提取,实现了DNA的快速提取。同时根据中国转基因作物检测与监测网和转基因作物数据库(GM Crop Database)提供的信息,对目前商业化的转基因玉米品种的分子特征进行了分析,统计了常用的外源基因在这些品种的分布情况(表1)。选择覆盖面广,使用频率高的3个外源基因,即CaMV35S启动子、NOS终止子和Bar基因作为检测的靶标基因形成组合,通过体系优化,形成复合荧光PCR体系,建立了高效的转基因玉米筛查系统。该方法特异性强、灵敏度高、重现性好,适用于转基因玉米成分的大规模筛查检测,而且应用实时荧光PCR方法可大大提高检测效率,降低检测过程中样品交叉污染和环境的污染。

表1 商业化转基因玉米转化体外源基因应用频数分析
1 材料与方法
1.1 材料
1.1.1 试验样品 转基因玉米阳性参照样品BT176和BT11由中国农业科技发展中心提供;阴性玉米样品农大108和郑单958由本实验室收集,所有样品经粉碎后4℃保存备用。
1.1.2 生化试剂 定量反应用配套试剂盒TaqMan Universal PCR Master Mix 购自美国应用生物系统公司(Applied Biosystems,ABI)。植物新型基因组DNA提取试剂盒、Taq酶、dNTPs、DNA Marker购自北京天根生化科技有限公司,实时荧光PCR所用的探针和引物序列见表2,分别采用HEX,FAM,Cy3三种不同荧光标记对3个基因的进行标记,由上海英骏生物技术有限公司合成。其余试剂由北京化学试剂公司提供。
1.2 方法
1.2.1 DNA提取与质量检测
1.2.1.1 快速提取法 称取50 mg玉米粉末样品加入1.5 mL Eppendorf 离心管中,加入200 μL NaOH溶液(0.1 mol/L),涡旋震荡30 s,将离心管放在沸水浴中加热5 min后加入200 mL Tris-HCl溶液(10 mmol/L、pH2.0),涡旋震荡10 s,上清液即可作为DNA模板用于扩增。
1.2.1.2 试剂盒法 参照试剂盒使用说明书。
1.2.1.3 质量检测 采用1.0%琼脂糖凝胶对两种方法提取的DNA进行电泳检测。

表2 引物和探针序列信息
1.2.2 实时荧光PCR 实时荧光PCR扩增采用ABI公司提供的试剂盒,反应在20 μL的体系中进行,其中2×TaqMan Universal PCR Master mix 10 μL,DNA模板1 μL,3个基因的正反向引物各0.25 μL(10-20 μmol/L),探 针0.5 μL(5-10 μmol/L)。扩 增 在ABI7300型实时荧光定量PCR仪上进行,扩增程序采用两步法:95℃预变性10 min;95℃变性20 s,60℃退火1 min,40个循环。
2 结果
2.1 DNA提取质量分析
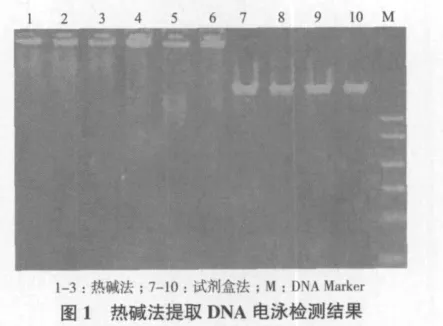
琼脂糖凝胶电泳检测结果(图1)显示,热碱法提取的DNA与试剂盒法提取的DNA相比,DNA提取的量较少,没有明显的亮带,弥散现象较为严重,这主要是因为DNA在热碱条件下存在降解。但是以两种方法提取的DNA为模板,通过实时荧光PCR方法对玉米内源基因zSSIIb进行检测,结果(图2)显示,两种DNA模板扩增效率差异不大,CT值相差约2个循环,这主要可能因为荧光PCR方法扩增的DNA片段小(100 bp左右),而且方法的灵敏性好,对DNA模板用量的要求不高。
2.2 复合荧光PCR体系的建立
为降低不同引物之间的竞争以及荧光背景干扰,将3个被检基因的引物和探针浓度设置不同梯度组合,通过组合之间对比优化分析,最终确立3个被检基因的引物及探针合适浓度。其中CaMV35S启动子的引物和探针终浓度都为200 nmol/L;NOS终止子和BAR基因的引物终浓度都为250 nmol/L,探针终浓度都为300 nmol/L。优化好的体系可对含量为1%转基因阳性参照(BT11和BT176混合样)的3个基因进行有效扩增,CT值都位于30-34之间(图3)。
2.3 特异性和灵敏度检测
为检测复合荧光PCR的特异性和灵敏度,利用热碱法提取阳性参照样品的DNA,并通过梯度稀释获得的转基因含量0%、0.01%、0.05%、0.1%和0.5%的DNA模板,用复合荧光PCR法进行扩增。结果显示,该方法特异性好,阴性样品均没有扩增曲线,阳性样品含有的外源基因都获得扩增,如BT11的CaMV35S启动子和NOS终止子,BT176的CaMV35S启动子和Bar基因。灵敏度分析结果(表3)显示,含量≥0.1%样品的3个目的基因都得到有效扩增(CT值<38),且不同重复间曲线重合性好,含量小于0.1%样品扩增曲线出现不稳定,不同重复间曲线重合性差。


2.4 样品的实际检测
利用建立的快速方法,对2011年在北京区域试验和预备试验的235份玉米品种进行了复测,检测结果(未显示)与先前常规方法(试剂盒提取DNA,普通PCR检测)检测结果完全一致,而且弱阳性结果情况发生概率小(2%),复检的样品少,省事省力,说明该方法具有很强的实用性,适合大批量样品的快速检测。
3 讨论
随着我国以及全球转基因研究的不断深入,批准进行商业化种植或作为加工原料进口的转基因产品将持续增加。为确保转基因产品监管到位,保障农业转基因生物安全,加大转基因产品抽检力度势在必行,涉及转基因检测的样品量将逐年递增,因此对转基因检测技术提出更高的要求,急需建立快速高效的检测方法。
提高转基因检测的效率,首先需要从试验方法上进行改进。同其它分子检测一样,DNA成功提取是进行检测的关键,而DNA提取因方法不同,繁琐程度差异较大[7,8],从而影响整个检测的效率,因此根据检测的需求选择合适的DNA提取方法显得非常重要。本研究在利用热碱法快速提取玉米种子胚DNA并成功进行SSR-PCR检测的研究基础上[2],建立了玉米种子粉末DNA快速提取法,虽然该方法提取的DNA质量不高,但是针对荧光PCR灵敏度高、扩增特异性强以及扩增片段小的特点,完全可以满足后续检测的质量要求。另外,该方法成本低廉,相对于试剂盒方法,DNA提取费用可以忽略不计。

表3 实时荧光复合PCR特异性与灵敏度检测
其次要从策略上进行改进,针对目前繁多的外源基因和转化体,对于大批量样品的检测,不可能对其进行所有外源基因和转化体的鉴定,只有系统分析转化体的分子特征,筛选使用频率高的一些外源基因作为靶标基因进行筛查,对于筛查结果为阳性的样品再进行转化体鉴定将大大提高检测的效率。本研究通过分析目前商业化转基因玉米转化体的分子特征,确定CaMV35S启动子、NOS终止子和Bar 3个基因为筛查基因,能覆盖目前所有商业化的玉米转化体,并且通过体系优化,建立同时检测3个基因的复合荧光PCR方法,仅通过一次PCR扩增,只要有一个基因检测结果为阳性,就可以确定该样品含转基因成分,进而采用二分法进行品系鉴定,方便快捷,适合大量样品快速检测。
4 结论
将DNA热碱提取法和复合荧光PCR技术相结合,建立起玉米种子转基因成分快速筛查方法,实现了DNA快速提取和PCR高效扩增有效结合,不仅能保证检测准确性和灵敏性,而且还大大降低假阳性发生,适合大批量玉米种子样品的快速筛查检测,为农业转基因安全监管提供重要的技术支撑。
[1] Jame C. 2011全球生物技术/转基因作物商业化发展态势[J].中国生物工程杂志, 2012, 32(1):1-14.
[2] 吴明生, 贾希海, 田雷, 等.利用SSR技术快速准确鉴定杂交玉米种子纯度[J].分子植物育种, 2006, 4(3):381-384.
[3] Tomoaki Y, Hideo K, Takeshi M, Takashi K. Applicability of quantification of genetically modified organisms to foods processed from maize and soy[J]. J Agric Food Chem, 2005, 53(6):2052-2059.
[4] Hideo K, Yoichiro S, Takeshi M, et al. Novel reference molecules for quantitation of genetically modified maize and soybean[J]. J AOAC Int, 2002, 85(5):1077.
[5] Julien P, Maher C, Laetitia C, Marcel R. Development of two screening duplex PCR assays for genetically modified organism quantification using multiplex real-time PCR master mixes[J]. Eur Food Res Technol, 2011, 232:327-334.
[6] Grohmann L, Brunen-Nieweler C, Nemeth A, Waiblinger HU. Collaborative trial validation studies of real-time PCR-based GMO screening methods for detection of the bar gene and the ctp2-cp4epsps construct[J]. J Agric Food Chem, 2009, 57(19):8913-8920.
[7] Porcar M, Ramos S, Latorre A. A simple DNA extraction method suitable for PCR detection of genetically modified maize[J]. J Sci Food Agric, 2007, 87(14):2728-2731.
[8] 陶李, 张富丽, 宋君.转基因玉米检测中4种DNA提取方法比较[J].现代农业科技, 2011, 12:53-56.
