盐生植物耐盐分子机制的研究进展
2013-12-23付畅孙玉刚傅桂荣
付畅 孙玉刚 傅桂荣
(哈尔滨师范大学生命科学与技术学院,哈尔滨 150025)
盐胁迫是影响农业生产和生态环境的重要非生物胁迫之一。迄今为止,人们已在转录水平以及翻译水平上对植物耐盐分子机制作了大量的研究。植物的耐盐性是多基因性状,目前揭示植物耐盐分子机制的主流方法是转录组分析,如DNA 微阵列(DNA microarray)、表达序列标签(expressed sequence tags,ESTs)、抑制性消减杂交(suppressed subtractive hybridization,SSH)以及新兴的RNA测序技术(RNA sequencing,RNA seq)和蛋白质组分析,可以全面、系统地反映植物的耐盐分子机制。分析耐盐性较强的盐生植物在盐胁迫下的转录组及蛋白质组是研究植物耐盐分子机制和鉴定耐盐相关基因的重要策略之一[1,2]。比较仅抗逆性存在差异的相近物种或基因型在逆境胁迫下的转录组及蛋白质组是鉴定抗逆基因和分析抗逆分子机制的另外一个重要策略[1]。比较耐盐性有差别的近缘种,如小盐芥(Thellungiella halophila)和拟南芥,不同品种(如耐盐的水稻品种和不耐盐的水稻品种)、不同生态型(如干旱区生态型和湿润区生态型)在盐胁迫应答中的差别,有助于揭示植物的耐盐分子机制。本文主要介绍盐生植物耐盐分子机制的研究进展。
1 盐生植物响应盐胁迫的转录因子
当植物受到逆境胁迫时,转录因子会与相应的顺式作用元件结合,启动相应基因的转录表达,调控并减轻逆境胁迫给植物带来的伤害。转录组学的研究发现,大量转录因子在盐胁迫条件下被诱导表达[3-5],这些被盐胁迫诱导表达的转录因子与植物对盐胁迫的适应性之间具有密切关系。目前已经鉴定出多个转录因子家族的成员参与了盐生植物对盐胁迫的应答反应(表1),这些转录因子以不同的应答反应模式在植物对盐胁迫的适应过程中发挥重要作用。大部分盐胁迫诱导的转录因子家族的成员具有不同的表达模式,一些成员在胁迫早期产生应答,而有些成员仅在随后的时间点上被诱导;有些转录因子则呈现出较大的空间表达差异,这与盐胁迫下植物中高度复杂的调控网络的存在是一致的[3,5]。有些转录因子基因对不同的胁迫具有普遍性应答的特性,而另外一些转录因子对盐胁迫具有特异性应答的特性[5]。
2 盐胁迫下盐生植物的渗透平衡调节
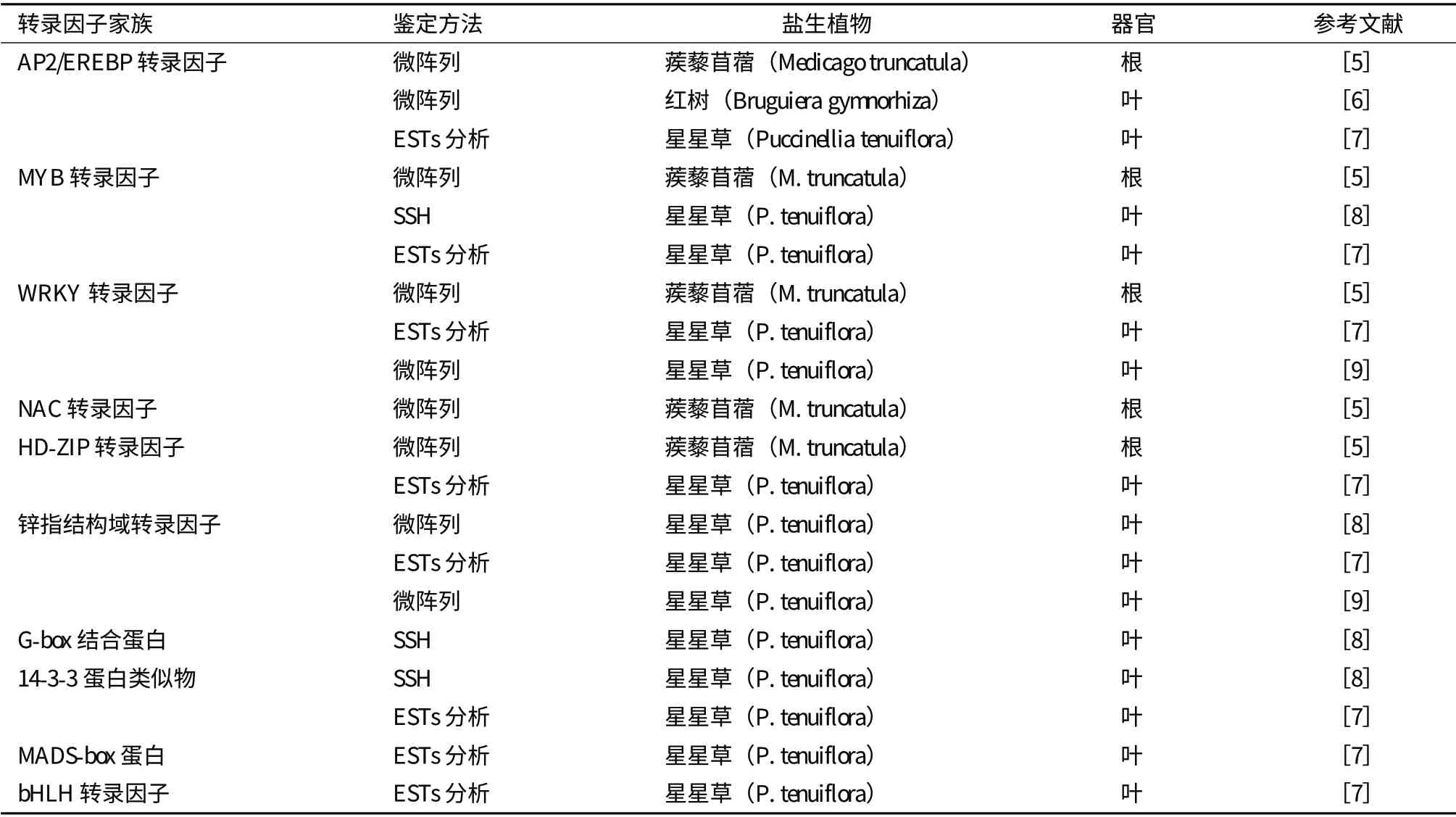
表1 盐生植物影响盐胁迫的转录应答因子
高盐会引起渗透胁迫,植物需要维持渗透平衡。盐生植物可以利用无机离子和有机渗透调节剂调节渗透平衡[6,10]。
2.1 离子的渗透调节作用
盐生植物Hordeum maritimum可以在根和地上部积累比K+更多的Na+却没有明显的毒害症状。在中等盐胁迫浓度下Hordeum maritimum利用无机离子作为渗透调节剂,而有机物和大部分K+用于代谢活性。有机物只在高盐胁迫下作为渗透调节剂[11]。红树(B. gymnorhiza)具有较高耐盐性主要是因为具有较强的吸收Na+和K+作为渗透调节剂的能力,以及维持K+平衡的能力,而不是由于可溶性糖的生物合成[6]。可见,非盐生植物受到盐胁迫后以有机物作为主要的渗透调节剂,而盐生植物在低盐胁迫时先以无机离子作为渗透调节剂,受到高盐胁迫时才以有机物作为渗透调节剂,这可能是盐生植物的耐盐性高于非盐生植物的重要原因之一。
2.2 脯氨酸的渗透调节作用
脯氨酸的积累和催化脯氨酸合成最后一步的△1-吡咯啉-5-羧酸合成酶(Δ1-pyrroline-5- carboxylate synthetases,P5CS)基因的表达与盐胁迫应答反应密切相关。盐胁迫下脯氨酸的积累可能与P5CS的表达有关[12,13]。无论在正常生长条件下还是在高盐胁迫下,盐芥中的脯氨酸含量均高于拟南芥[10]。目前关于盐胁迫下脯氨酸的作用存在争议。一种观点认为,P5CS基因的表达和脯氨酸的积累对植物的耐盐性有利,脯氨酸积累的作用是参与渗透调节、保护酶和细胞结构以及作为活性氧清除剂。支持依据之一是,对耐盐滨藜施加外源脯氨酸提高了耐盐滨藜的耐旱性[14]。另一种观点则认为,脯氨酸的积累与盐胁迫的伤害程度密切相关。脯氨酸的积累可能是盐胁迫引起的受害症状,而不是耐盐性指标[15]。该观点的依据之一是,施加外源的脯氨酸导致了盐诱导的伤害[16]。依据之二是耐盐的水稻品种中自由脯氨酸的积累量低于盐敏感的水稻品种[17]。依据之三是,脯氨酸的渗透调节作用可能是不足的[17]。P5CS基因在胁迫应答中的主要作用可能不是脯氨酸的代谢,而是发挥了比脯氨酸积累更重要的作用[18]。
2.3 甘氨酸甜菜碱的渗透调节作用
甘氨酸甜菜碱是植物中重要的渗透调节剂之一。耐盐滨藜在高盐胁迫下积累更多的甘氨酸甜菜碱,耐旱滨藜则在缺水胁迫下积累更多的脯氨酸,向耐旱滨藜施加外源甘氨酸甜菜碱,耐旱滨藜的耐盐性得到提高[14]。甘氨酸甜菜碱可能在海滨碱蓬(Suaeda maritima)的耐盐性中起着至关重要的作用[2]。甘氨酸甜菜碱在盐胁迫中的渗透调节作用可能比脯氨酸更重要。
2.4 其他有机渗透调节剂的渗透调节作用
NaCl胁迫拟南芥后相当大一部分海藻糖合成途径中的基因被诱导转录,一些脱水素基因也被诱导表达[3]。尽管海藻糖被认为在细菌和真菌中作为渗透保护剂,在植物中被认为由于其在细胞内的浓度太低以至于不足以成为有效的渗透保护剂,因此更被视为植物中的信号分子[19]。
3 盐胁迫下盐生植物的离子平衡调节
盐胁迫会对植物产生离子胁迫,植物需要重建离子平衡。主要通过减少Na+的流入、增加Na+的排出、将Na+在液泡中区隔化,以及增加K+的吸收从而提高K+/Na+比来减轻Na+的毒害。植物质膜上的Na+/H+反向转运蛋白负责将进入细胞内的Na+再排到细胞外,需要以质膜H+-ATPase产生的跨膜电化学势梯度作为推动力,液泡膜上的Na+/H+反向转运蛋白负责将Na+在液泡中区隔化,需要以液泡膜H+-ATP酶和H+-焦磷酸酶产生的质子电化学势梯度作为推动力。在植物中Na+是通过K+转运蛋白进入植物细胞的,K+转运蛋白分为低亲和性K+转运蛋白和高亲和K+转运蛋白两类[20]。
3.1 盐生植物的离子平衡基因具有较高的本底表达水平和盐诱导表达水平
编码质膜Na+/H+反向转运蛋白的SOS1(salt overly sensitive 1)基因、编码丝氨酸/苏氨酸蛋白激酶的SOS2(salt overly sensitive 2)基因、编码液泡膜Na+/H+反向转运蛋白的NHX1基因和K+高亲和系统基因HKT1是重要的离子平衡调节基因,它们在盐芥中的表达水平均高于拟南芥[21]。SOS1基因在盐芥中的本底表达水平和盐诱导表达水平均高于拟南芥[22]。盐地碱蓬(Suaeda salsa)的SsHKT1基因在盐胁迫后上调表达[23],而盐胁迫下在红树(B. gymnorhiza)中没有检测到K+通道或转运蛋白基因以及Na+/H+反向转运蛋白基因的上调表达,K+平衡的机制尚不清楚,Na+/H+反向转运蛋白被认为可能是通过转录后水平和翻译水平的调节响应盐胁迫[6]。NaHCO3胁迫下星星草中质膜H+-ATP酶的丰度较高,表明其在调节离子平衡中具有重要作用[7]。NaHCO3胁迫下液泡膜H+-ATP酶,叶绿体ATP合成酶、质膜H+-ATP酶和液泡膜和Na+/H+反向转运蛋白基因NHX基因在刚毛柽柳中表达,表明刚毛柽柳具有高效的离子平衡调节途径[24]。重要的离子平衡调节基因保持较高的本底表达水平和盐诱导表达水平可能是盐生植物抵御盐胁迫的重要策略之一。
3.2 利用离子作为渗透调节剂
如2.1部分所述,盐生植物具有较强的吸收Na+和K+作为渗透调节剂的能力[6],可以有效地将Na+在细胞内区隔化,进行渗透调节,因而可在根中和地上部积累较多的Na+却没有明显的毒害症状[11]。
3.3 在地上部积累Na+
盐胁迫下盐芥和拟南芥中的Na+含量都会增加,但盐芥有控制Na+在地上部积累的能力[25]。NaCl能明显促进盐地碱蓬(S. salsa)地上部的生长,而没有促进根的生长。Na+和Cl+主要在盐地碱蓬的地上部积累。NaCl明显提高了根中V型-ATP酶的活性,地上部V型-ATP酶的活性则提高得更多[26]。将盐离子运输并积累于地上部在盐生植物抵御盐胁迫的过程中发挥重要作用。
3.4 维持较高的K+/Na+比
盐芥的耐盐性与其维持较高的K+/Na+比有关[25]。红树在非胁迫条件和胁迫条件下都能维持较高的K+水平。这是红树与其他甜土植物的明显区别[6]。星星草的耐盐性可能主要是由于根对K+的高选择性吸收从而限制Na+的流入,而不是根Na+的外流或叶片泌盐[27]。
4 盐胁迫下盐生植物的氧化还原平衡调节
盐胁迫导致植物积累的活性氧(reactive oxygen species,ROS)和其他毒性物质会破坏膜系统,蛋白质和核酸分子[28],具有解毒作用的酶可以提高耐盐性,拥有有效的活性氧清除机制对植物来说至关重要[24]。NaHCO3胁迫下的星星草SSH文库中与活性氧清除有关的ESTs有31个[9],NaHCO3胁迫下刚毛柽柳中cDNA文库中有高比例的有关活性氧清除的ESTs[24]。星星草响应NaHCO3胁迫的活性氧清除基因呈现不同的表达模式[8]。这些结果表明,活性氧清除基因积极参与盐生植物抵御NaHCO3胁迫的应答反应,在转录水平上的调控具有一定的复杂性。
4.1 过氧化氢酶(catalase,CAT)
CAT具有重要的抗氧化作用。NaHCO3胁迫下CAT基因在星星草中表达[7]。在盐胁迫早期阶段星星草中CAT基因的表达明显上调[8,9]。CAT在星星草耐受NaHCO3胁迫中发挥重要作用。
4.2 谷胱甘肽转移酶(glutathione S-transferase,GST)
GST能够解毒ROS,也参与胁迫应答。NaHCO3胁迫下星星草GST和CAT具有相似的表达模式,表明这两个基因在对盐胁迫的应答中可能协同调节[9]。
4.3 金属硫蛋白(metallothionein ,MT)基因和硫氧还蛋白(thioredoxin,TRX)
MT能在非生物胁迫下清除活性氧,TRX作为植物抗氧化网络的组分参与避免氧化伤害。MT和TRX基因在NaHCO3胁迫24 h后在刚毛柽柳中上调表达,表明它们在NaHCO3胁迫下刚毛柽柳清除活性氧的过程中发挥重要作用[24]。
活性氧清除基因积极参与盐生植物抵御NaHCO3胁迫的应答反应。ROS清除的转录调节可能是星星草适应NaHCO3胁迫的重要机制之一。
5 盐胁迫下盐生植物光合作用的调节
离子胁迫、水分胁迫、叶绿体基质的减少、活性氧分子的产生都可以抑制光合作用。光合作用调节是逆境胁迫下的重要调节途径[24]。NaHCO3胁迫下星星草光合作用基因差异表达[9],表明光合作用调节参与盐生植物对盐胁迫的应答。
5.1 盐胁迫下盐生植物光系统的变化
叶绿素II a/b结合蛋白是主要的天线复合物,可以捕获光能并将其传递到光反应中心。几种不同类型的结合蛋白在胁迫后的刚毛柽柳中或上调或下调表达,表明盐碱胁迫强烈影响植物捕获和传递光的能力[24]。短期盐胁迫会影响植物的气体交换和光合作用的电子传递。在亚致死的盐胁迫浓度处理下,拟南芥光系统II(PSII)的电子传递受到抑制,光系统Ⅰ的电子流增加,而盐芥的光系统Ⅰ不受影响,光系统II 的电子流有大幅增加[29]。高盐胁迫没有影响盐生植物Arthrocnemum macrostachyum的PSII[30]。NaHCO3胁迫下刚毛柽柳PSII的相关基因呈现不同的表达模式,表明它们并非协同调节[24]。PSII对盐胁迫的耐性在盐生植物对盐胁迫的应答中发挥重要作用,其基因表达调控具有一定复杂性。
5.2 盐胁迫下盐生植物CO2固定的变化
1,5-二磷酸核酮糖羧化酶/加氧酶(Ribulose bisphosphate carboxylase oxygenase,Rubisico)催化植物光合作用中CO2的固定。编码刚毛柽柳Rubisico小亚基的基因在NaHCO3胁迫后显著变化,表明NaHCO3胁迫强烈影响刚毛柽柳中CO2的固定[24]。适度的盐胁迫可以提高滨海湿生盐土植物Atriplex portulacoides的羧化能力[31]。因此,适度的盐胁迫可以促进盐生植物CO2的固定。
5.3 盐胁迫下盐生植物光合速率的变化
盐胁迫会导致碳同化速率的下降,但极耐盐植物维持叶面积的能力可以抵消高盐胁迫下碳同化速率的下降[31]。净光合速率对盐胁迫的响应,主要是制约气孔导度和细胞内CO2浓度。高盐胁迫提高了盐生植物Arthrocnemum macrostachyum的净光合速率,但没有影响PSII,提高净光合作用速率似乎是盐生植物适应盐胁迫的机制之一[30]。制约滨海湿生盐土植物Atriplex portulacoides 光合作用的主要因素似乎是气孔导度和细胞内的CO2浓度,而不是对PS II的影响[31]。盐生植物在盐胁迫下保持或提高净光合速率是对盐胁迫的积极应答策略之一。
6 盐胁迫下盐生植物的代谢变化
盐胁迫下大多数代谢产物在盐芥中的水平高于拟南芥。盐芥地上部的含水量低于拟南芥,盐芥具有失去更多水分的能力,这有助于维持土壤和植物之间的水势梯度。盐芥通过代谢配置耐受脱水,抵御渗透胁迫[32]。
6.1 盐胁迫下盐生植物蛋白质的合成与降解
半胱氨酸蛋白酶基因参与许多生物过程并介导植物对环境胁迫的应答[9]。半胱氨酸蛋白酶抑制剂(cystatin)基因也参与植物对环境胁迫的应答[33]。星星草半胱氨酸蛋白酶基因的激活可能在NaHCO3胁迫后被瞬时抑制[9]。盐生植物Cakile maritima的半胱氨酸蛋白酶抑制剂基因CmC在非胁迫植株组成型表达,但在不同生态型表达水平不同。高盐胁迫下,干旱地区生态型以外的生态型中CmC的表达水平上升[33]。我们在盐碱地星星草根系的SSH文库中同时检测到半胱氨酸蛋白酶基因Legumain和半胱氨酸蛋白酶抑制剂基因(未发表),表明盐碱地逆境胁迫下蛋白质的合成与降解存在动态平衡,其时空表达特性值得深入研究。
6.2 盐胁迫下盐生植物的糖代谢
一般在逆境胁迫下,植物体内的糖代谢均受影响。高等植物有两类不同的甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH),一类存在于叶绿体中并参与卡尔文循环;另一类存在于细胞质中并参与糖酵解过程[34]。同非胁迫的对照相比,NaHCO3胁迫下星星草叶中GAPDH基因在转录水平上差异表达[35]。Na2CO3胁迫下星星草叶中GAPDH在蛋白质水平上上调表达[36]。研究发现,同非胁迫的对照相比,盐碱地星星草根系中GAPDH基因差异表达(未发表),表明其在星星草抵御盐胁迫中发挥作用。
植物大多需将叶中的光合产物(葡萄糖)转化成非还原性糖(如蔗糖)才能转运到其他器官,为其生长提供养分。蔗糖还具有信号功能,植物中存在一条特异的蔗糖信号传导途径[37]。蔗糖代谢水平常被用来衡量环境对植物胁迫的程度以及植物对环境的适应性。蔗糖代谢关键酶主要有转化酶(invertase,Ivr)、蔗糖合成酶(sucrose synthase,SS)、蔗糖磷酸合成酶(sucrose phosphate synthase,SPS)。SPS是合成蔗糖的酶,Ivr是分解蔗糖的酶,SS既能催化合成蔗糖的反应,也能催化分解蔗糖的反应,主要作用是水解蔗糖。蔗糖积累现象是植物对逆境胁迫的一种保护性反馈[37,38]。NaHCO3胁迫后星星草中蔗糖合成酶基因的表达量明显高于对照[35]。研究发现,同对照相比,盐碱地星星草根系的蔗糖合成酶基因差异表达,盐碱地星星草根系可能通过蔗糖合成酶基因的表达调节蔗糖代谢从而帮助星星草抵御盐碱胁迫。
7 总结
盐生植物通过响应盐胁迫的转录因子激活或抑制相关基因的表达;先以无机离子作为主要的渗透调节剂调节渗透平衡,随着盐浓度的提高,再以有机物作为渗透调节剂,其中甘氨酸甜菜碱似乎是最重要的有机渗透调节剂;通过将离子在液泡中区隔化并作为渗透调节剂,将离子在地上部积累,维持较高的K+/Na+比等方式维持离子平衡;通过CAT、GST、MT和TRX基因的高效表达清除活性氧以维持氧化还原平衡;光合作用系统通过保持光系统II的功能不被影响或少被影响,利用适度的盐胁迫促进CO2的固定,维持光合作用的叶面积、保持或提高净光合速率等策略响应盐胁迫;蔗糖代谢和蛋白质动态的合成与降解积极参与对盐胁迫的应答反应。虽然盐生植物的耐盐分子机制取得了较大进展,但对于植物耐盐性的复杂性而言,仍然是不足的。盐生植物应答盐胁迫的系统化网络尚未建立,目前还任重道远。盐生植物耐盐分子机制的研究中,衡量植物是否处于积极防御状态的评价指标还需确认,如脯氨酸是作为盐胁迫的伤害指标还是抗性指标需要进一步证实。但我们相信随着生物学技术手段的进步,这些问题终将被解答。盐生植物耐盐分子机制研究中取得的重要进展为植物耐盐分子机制的全面揭示奠定了很好的基础。目前已从盐生植物中分离出来的耐盐相关基因,如bZIP转录因子基因、金属硫蛋白基因、谷胱甘肽转移酶基因、甘油醛-3-磷酸脱氢酶基因等则有望在植物耐盐基因工程中发挥重要作用。
[1] Deyholos MK. Making the most of drought and salinity transcriptomics[J]. Plant, Cell and Environ, 2010, 33(4):648-654.
[2] Sahu BB, Shaw BP. Isolation, identification and expression analysis of salt-induced genes in Suaeda maritima, a natural halophyte using PCR-based suppression subtractive hybridization[J]. BMC Plant Biol, 2009, 9:69.
[3] Jiang Y, Deyholos MK. Comprehensive transcriptional profiling of NaCl-stressed Arabidopsis roots reveals novel classes of responsive genes[J]. BMC Plant Biology, 2006, 6(1):25.
[4] Udvardi MK, Kakar K, Wandrey M, et al. Legume transcription factors:global regulators of plant development and response to the environment[J]. Plant Physiol, 2007, 144(2):538-549.
[5] Gruber V, Blanchet S, Diet A, et al. Identification of transcription factors involved in root apex responses to salt stress in Medicago truncatula[J]. Mol Genet and Genomics, 2009, 281(1):55-66.
[6] Miyama M, Tada Y. Transcriptional and physiological study of the response of Burma mangrove(Bruguiera gymnorhiza)to salt and osmotic stress[J]. Plant Mol Biol, 2008, 68(1-2):119-129.
[7] Wang Y, Chu Y, Liu G, et al. Identification of expressed sequence tags in an alkali grass(Puccinellia tenuiflora)cDNA library[J]. J Plant Physiol, 2007, 164(1):78-89.
[8] Wang Y, Yang C, Liu G, et al. Microarray and suppression subtractive hybridization analyses of gene expression in Puccinellia tenuiflora after exposure to NaHCO3[J]. Plant Sci, 2007, 173:309-320.
[9] Wang Y, Yang C, Liu G, et al. Development of a cDNA microarray to identify gene expression of Puccinellia tenuiflora under saline-alkali stress[J]. Plant Physiol Biochem, 2007, 45(8):567-576.
[10] Taji T, Seki M, Satou M, et al. Comparative genomics in salt tolerance between Arabidopsis and Arabidopsis-related halophyte salt cress using Arabidopsis microarray[J]. Plant Physiol, 2004, 135(3):1697-1709.
[11] Yousfi S, Rabhi M, Hessini K, et al. Differences in efficient metabolite management and nutrient metabolic regulation between wild and cultivated barley grown at high salinity[J]. Plant Biol(Stuttg), 2010, 12(4):650-658.
[12] Kim DW, Shibato J, Agrawal GK, et al. Gene transcription in the leaves of rice undergoing salt-induced morphological changes(Oryza sativa L.)[J]. Mol Cells, 2007, 24(1):45-59.
[13] Igarashi Y, Yoshiba Y, Sanada Y, et al. Characterization of the gene for Δ1-pyrroline-5-carboxylate synthetase and correlation between the expression of the gene and salt tolerance in Oryza sativa L.[J]. Plant Mol. Biol, 1997, 33(5):857-865.
[14] Ben Hassine A, Ghanem ME, Bouzid S, et al. An inland and a coastal population of the Mediterranean xero-halophyte species Atriplex halimus L. differ in their ability to accumulate proline and glycinebetaine in response to salinity and water stress[J]. J Exp Bot, 2008, 59(6):1315-1326.
[15] Lutts S, Majerus V, Kinet JM. NaCl effects on proline metabolism in rice(Oryza sativa)seedlings[J]. Physiologia Plantarum, 1999, 105(3):450-458.
[16] Garcia AB, de Almeida Engler J, lyer S, et al. Effects of osmoprotectants upon NaCl stress in rice[J]. Plant Physiol, 1997, 115(1):159-169.
[17] Lutts S, Kinet JM, Bouharmont J. Effects of salt stress on growth, mineral nutrition and proline accumulation in relation to osmotic adjustment in rice(Oryza saliva L.)cultivars differing in salinity resistance[J]. Plant Growth Regul, 1996, 19(3):207-218.
[18] Hur J, Jung KH, Lee CH, et al. Stress inducible OsP5CS2 gene is essential for salt and cold tolerance in rice[J]. Plant Sci, 2004, 167:417-426.
[19] Avonce N, Leyman B, Mascorro-Gallardo JO, et al. The Arabidopsis trehalose-6-P synthase AtTPS1 gene is a regulator of glucose, abscisic acid, and stress signaling[J]. Plant Physiol, 2004, 136(3):3649-3659.
[20] Blumwald E, Aharon GS, Apse MP. Sodium transport in plant cells[J]. Biochim Biophys Acta, 2000, 1465(1-2):140-151.
[21] Taji T, Komatsu K, Katori T, et al. Comparative genomic analysis of 1047 completely sequenced cDNAs from an Arabidopsis-related model halophyte, Thellungiella halophila[J]. BMC Plant Biol, 2010, 10:261.
[22] Oh DH, Dassanayake M, Haas JS, et al. Genome structures and halophyte-specific gene expression of the extremophile Thellungiella parvula in comparison with Thellungiella salsuginea(Thellungiella halophila)and Arabidopsis[J]. Plant Physiol, 2010, 154(3):1040-1052.
[23] Shao Q, Zhao C, Han N, et al. Cloning and expression pattern of SsHKT1 encoding a putative cation transporter from halophyte Suaeda salsa[J]. DNA Seq, 2008, 19(2):106-114.
[24] Gao C, Wang Y, Liu G, et al. Expression profiling of salinity-alkali stress responses by large-scale expressed sequence tag analysis in Tamarix hispid. Plant Mol Biol, 2008, 66(3):245-258.
[25] Ghars MA, Parre E, Debez A, et al. Comparative salt tolerance analysis between Arabidopsis thaliana and Thellungiella halophila, with special emphasis on K+/Na+selectivity and proline accumulation[J]. J Plant Physiol, 2008, 165(6):588-599.
[26] Yang MF, Song J, Wang BS. Organ-specific responses of vacuolar H-ATPase in the shoots and roots of C3 halophyte Suaeda salsa to NaCl[J]. J Integr Plant Biol, 2010, 52(3):308-314.
[27] Wang CM, Zhang JL, Liu XS, et al. Puccinellia tenuiflora maintains a low Na+level under salinity by limiting unidirectional Na+in flux resulting in a high selectivity for K+over Na+[J]. Plant Cell Environ, 2009, 32(5):486-496.
[28] Hasegawa PM, Bressan RA, Zhu JK, et al. Plant cellular and molecular responses to high salinity[J]. Annu Rev Plant Physiol Plant Mol Biol, 2000, 51:463-469.
[29] Stepien P, Johnson GN. Contrasting responses of photosynthesis to salt stress in the glycophyte Arabidopsis and the halophyte thellungiella:role of the plastid terminal oxidase as an alternative electron sink[J]. Plant Physiol, 2009, 149(2):1154-1165.
[30] Redondo-Gómez S, Mateos-Naranjo E, Figueroa ME, et al. Salt stimulation of growth and photosynthesis in an extreme halophyte, Arthrocnemum macrostachyum[J]. Plant Biol(Stuttg), 2010, 12(1):79-87.
[31] Redondo-Gómez S, Mateos-Naranjo E, Davy AJ, et al. Growth and photosynthetic responses to salinity of the salt-marsh shrub Atriplex portulacoides[J].Ann Bot, 2007, 100(3):555-563.
[32] Lugan R, Niogret MF, Leport L, et al. Metabolome and water homeostasis analysis of Thellungiella salsuginea suggests that dehydration tolerance is a key response to osmotic stress in this halophyte[J]. Plant J, 2010, 64(2):215-229.
[33] Megdiche W, Passaquet C, Zourrig W, et al. Molecular cloning and characterization of novel cystatin gene in leaves Cakile maritima halophyte[J]. J Plant Physiol, 2009, 166(7):739-749.
[34] Martin W, Brinkmann H, Savonna C, et al. Evidence for a chimeric nature of nuclear genomes:eubacterial origin of eukaryotic glyceraldehyde-3-phosphate dehydrogenase genes[J]. Proc Natl Acad Sci USA, 1993, 90(18):8692-8696.
[35] 张国栋. 抑制性消减杂交和cDNA 微阵列技术研究星星草耐盐机理[D]. 哈尔滨:东北林业大学, 2006:29-60.
[36] 石永丽. 星星草(Puccinellia teniflora)响应Na2CO3胁迫差异表达蛋白质组学研究[D]. 哈尔滨:哈尔滨师范大学, 2009:19-34.
[37] Sheen J, Zhou L, Jang JC. Sugar as signaling molecules[J]. Curr Opin Plant Biol, 1999, 2(5):410-418.
[38] Gibson SI. Plant sugar-response pathways. Part of a complex regulatory web[J]. Plant Physiol, 2000, 124(4):1532-1539.
