中国林蛙核糖核酸酶Rdrlec的原核表达及抗菌活性检测
2013-12-23尹雪薇彭艳丽冉帅陶凤云
尹雪薇 彭艳丽 冉帅 陶凤云
(北京联合大学生物化学工程学院,北京 100023)
近年来,抗菌及抗肿瘤的核糖核酸酶作为多功能分子引起了广泛的关注,这方面的研究相当活跃,成为新药开发的热点[1-4]。来源于家鸡(Gallus gallus)的两个RNase A超家族成员RNase A-1和A-2具有81%氨基酸序列一致性,等电点分别是10.2和11.0,其中具有较高等电点的RNase A-2具有显著的杀菌活性,而RNase A-1没有杀菌活性。进一步的研究发现,RNase A-2中的两个结构域,Domain II(TTRRHFRIT)和Domain III(RYSGNQFNRRVRVGCRG)对该分子的杀菌活性具有重要贡献,可以不依赖于分子骨架而以单独肽段形式发挥杀菌活性[5]。来源于鱼类的多个RNase A超家族成员都发现具有杀菌活性[6,7],大肠杆菌外膜蛋白酶OmpT可以特异性地裂解斑马鱼ZF-RNase-3分子中的Arg-Arg肽键,释放出2个肽段,其中的大片段(氨基酸31-124)是有效的杀菌剂[8]。中国林蛙核糖核酸酶Rdronc分子中处于正选择压力下的带有正电荷的氨基酸残基对于杀菌活性具有重要作用[2]。由于核糖核酸酶A(ribonuclease A,RNase A)超家族成员数量众多,氨基酸序列变异很大,有假说认为该超家族成员可能起源于宿主防御功能[6],验证这一假说需要来源于不同物种的多种RNase A超家族成员具有杀菌活性的试验数据。
中国林蛙核糖核酸酶Rdrlec基因是本实验室从中国林蛙基因组中PCR扩增得到的495 bp的核苷酸片段,包含信号肽序列和完整的成熟蛋白编码区序列。Blast检索显示此序列与多条来源于蛙属的核糖核酸酶同源,其中与来源于日本蛙的核糖核酸酶Rjlec (protein ID:P18839),具有最高的氨基酸序列一致性(81%),我们将新基因命名为Rdrlec,提交到GenBank (登录号EU384704)。
为了进一步开发利用这一新的基因资源,探索其编码的蛋白产物的生物学活性,我们将此基因按照大肠杆菌偏好的密码子进行优化后,人工合成基因,定向克隆到pET-28a(+)构建重组表达载体,在大肠杆菌BL21(DE3)中诱导表达融合蛋白。通过包涵体复性、肠激酶切割融合蛋白、Ni-NTA亲和层析纯化等步骤获得了野生型Rdrlec重组蛋白,并检测其抗微生物活性,旨在为该分子的进一步开发应用奠定基础。
1 材料与方法
1.1 材料
表达载体pET-28a (+)、大肠杆菌DH5α、BL21(DE3) 、药敏测试大肠杆菌和金黄色葡萄球菌均由北京联合大学生物化学工程学院生物工程实验室保藏;Pfu DNA 聚合酶、DNA 片段纯化试剂盒均购自北京赛百盛基因技术有限公司;T4 DNA 连接酶、限制性内切酶EcoR I和Hind III等购自宝生物工程(大连)有限公司;质粒提取试剂盒、琼脂糖胶回收试剂盒、异丙基硫代-β-D-半乳糖苷(IPTG)购自天根生化科技(北京)有限公司;LB 培养基购自美国BD公司;Ni-NTA介质购自Qiagen;重组肠激酶购自Novagen公司。TritonX-100 购自Sigma 公司;其他试剂为国产分析纯。
1.2 方法
1.2.1 Rdrlec基因序列的优化、合成与PCR扩增 根据大肠杆菌密码子的偏好性及密码子的合理搭配设计Rdrlec的基因序列,在基因的N端添加肠激酶裂解位点,在C端添加连续2个终止密码子,在上述序列的两端分别引入EcoR I和Hind III限制性内切酶识别位点。设计的核苷酸序列由生工生物工程(上海)有限公司合成,并克隆在pUC57质粒上,命名为pUC57-Rdrlec,保存在大肠杆菌DH5α中。
根据以上的序列设计两条特异性PCR扩增Rdrlec的引物:5'-CCCGAATTCGACGATGATGACAAACAG-3'和5'-GGCAAGCTTGTCATTAACAGCTACCG-3',下划线部分依次为EcoR I和Hind III限制性内切酶位点,其两侧添加了保护性碱基以提高限制酶切割效率。引物由北京赛百盛基因技术有限公司合成。
以pUC57-Rdrlec为模板,PCR扩增Rdrlec基因片段,PCR条件为:94℃ 预变性5 min;94℃30 s、55℃ 30 s、72℃ 50 s,共进行35 个循环;最后72℃延伸10 min。将PCR产物进行3. 0% 琼脂糖凝胶电泳,用凝胶回收试剂盒回收目标基因片段。
1.2.2 重组原核表达载体的构建 将纯化回收的PCR产物和pET-28a (+)载体均用EcoR I和Hind III双酶切,酶切产物电泳分离并切胶纯化后,使用T4 DNA 连接酶在16℃连接过夜,连接产物转化大肠杆菌BL21(DE3)感受态细胞,卡那霉素筛选阳性克隆。提取质粒,分别用EcoR I单酶切重组表达载体,及用EcoR I和Hind III双酶切重组表达载体,进行酶切验证后,重组质粒命名为pET28a-Rdrlec,送北京三博远志生物技术有限公司进行测序验证。
1.2.3 重组融合蛋白的诱导表达 将测序正确的表达质粒转化大肠杆菌BL21(DE3)感受态细胞,获得工程菌BL21(DE3)/ pET28a-Rdrlec。挑单菌落接种于LB培养液(50 μg/mL卡那霉素),37℃振荡培养过夜,按10%转接到含有卡那霉素的新鲜液体培养基中,37℃振荡培养至OD600为0.8时,加入IPTG使其终浓度为0.5 mmol/L,降低培养温度至34℃,诱导6 h后,8 000 r/min离心15 min收集菌体,用PBS洗3次,取少量样品采用SDS-PAGE (5%分离胶,15%浓缩胶)检测重组蛋白表达情况。
1.2.4 重组融合蛋白的纯化 离心收集细胞,洗涤去除培养基,将菌体重悬于裂解液(20 mmol/L Tris·HCl (pH8.0),0.5 mmol/L EDTA,50 mmol/L NaCl,0.5% Triton X-100)中,在冰浴条件下超声破碎菌体(超声5 s,间隔5 s,50 次循环,功率300 W),接着4℃、15 000 r/min离心30 min收集包涵体。将包涵体用洗涤液[1%(V/V)Triton X-100,1 mol/L NaCl]充分洗涤后,溶于尿素变性缓冲液(8 mol/L Urea,50 mmol/L Tris,pH7.5,25 mmol/L DTT)中,室温下溶解2 h。离心除去沉淀,将蛋白浓度控制在50 μg/mL,在复性缓冲液(含有 0.10 mol/L NaCl,3.0 mmol/L 还原型谷胱甘肽,0.6 mmol/L 氧化型谷胱甘肽的 0.10 mol/L Tris-乙酸缓冲液,pH8.0)中4℃复性40 h。
离心收集上清,用超滤管浓缩(截流相对分子质量3 kD)后,透析,进行Ni-NTA亲和层析,用60 mmol/L咪唑充分洗去杂蛋白后,用500 mmol/L咪唑洗脱目标融合蛋白。
1.2.5 肠激酶切割融合蛋白释放Rdrlec 将纯化的融合蛋白透析后,利用肠激酶进行酶切,以释放Rdrlec。反应体系为:10×rEK Cleavage buffer 5 μL,融合蛋白50 μg,重组肠激酶4 μL(1 U/μL),去离子水补充至总体积50 μL,25℃温浴酶切反应24 h,取样SDS-PAGE分析酶切效果。将酶切后的溶液上样于Ni-NTA柱,载体序列由于带有6×His标签,特异性吸附到层析介质上,而Rdrlec存在于穿过液中。收集Rdrlec部分,透析并冻干浓缩后,再次进行Sephadex G75层析纯化Rdrlec。进行15% SDSPAGE检测酶切和纯化效果。
1.2.6 Rdrlec的酶活性 参考文献方法[2,9],酶活性的检测基于核糖核酸酶降解酵母总RNA的反应。将纯化的酵母总RNA底物溶解在反应缓冲液(40 mmol/L乙酸钠,pH6.0)中。反应总体积为0.8 mL,含有0.8 mg RNA底物。将重组Rdrlec加入到反应体系中,37℃反应4 h,然后添加0.5 mL冰冷的20 mmol/L硝酸镧和3%高氯酸终止反应。在室温下13 000 r/min离心除去未降解的RNA,通过OD260值检测溶解于上清液中的RNA降解产物的量,分别以RNase A和乙酸钠缓冲液作为酶活性检测的阳性对照和阴性对照。
1.2.7 Rdrlec的抗菌活性 将斜面保存的Escherichia coli和Staphylococcus auteus分别进行LB固体平板划线,挑取单菌落接种到LB液体培养基中,置于37℃摇床中培养至指数生长期后,连续10倍稀释涂平板进行菌落计数,调整菌液浓度为(1-5)×106作为测试菌液。取不同浓度的重组蛋白用10 mmol/L pH7.4的磷酸钠缓冲液连续10倍稀释后取5 μL 加入到20 μL 菌悬液中,对照组加入等量的磷酸钠缓冲液。37℃培养4 h,使重组蛋白发挥杀菌作用。处理后的菌悬液连续10倍稀释,涂LB琼脂平板,做3个重复,37℃过夜培养后计数菌落形成单位,计算细菌存活数,并绘制曲线。
2 结果
2.1 Rdrlec基因的优化与PCR扩增
由于Rdrlec基因(GenBank EU384704)来源于两栖动物蛙类含有大肠杆菌稀有密码子,会影响其在大肠杆菌中的高效表达,首先使用稀有密码子计算 器(Rare Codon Calculator,http://nihserver.mbi.ucla.edu/RACC/)对Rdrlec成熟肽对应的基因序列进行分析,发现序列中共有10个稀有密码子(5个精氨酸稀有密码子,3个异亮氨酸稀有密码子和2个脯氨酸稀有密码子),如图1中下划线所示。为了提高重组蛋白的表达量,对该序列进行了优化,原则是首先根据表达物种的密码子频率进行调整,以使基因序列的密码子使用频率尽可能的与物种的最优密码子频率一致,去掉了频率在10%以下的密码子;然后尽可能降低序列中的碱基重复结构,使RNA二级结构相对简单、稳定;同时使整个DNA序列的G+C含量尽可能接近50%。优化后的序列与优化前序列比对结果如图1。
分别在优化后的Rdrlec基因的N端添加肠激酶裂解位点 (DDDDK)及在C端添加连续2个终止密码子 (TAATGA),可以使表达出的融合蛋白经过肠激酶裂解后得到具有天然N 末端和C末端的重组蛋白;在上述序列的两端分别引入EcoR I和Hind III限制性内切酶识别位点,可使该基因片段定向连接到pUC57载体相应的多克隆位点处。以pUC57-Rdrlec为模板,利用高保真Pfu DNA聚合酶和目的基因特异性引物PCR扩增出目的基因片段,其分子量大约为370 bp(图2),与理论值相符。

图1 密码子优化前后的序列比对

图2 PCR扩增目标基因
2.2 重组表达载体的构建
1%琼脂糖凝胶电泳检测结果(图3)显示,重组表达载体pET28a-Rdrlec大小约为5 700 bp,双酶切后得到约5 350 bp的载体大片段和约360 bp的目的基因片段,片段大小与理论值相符,初步表明表达载体构建成功。测序结果显示载体中的基因序列和设计序列完全一致,且阅读框架正确,重组表达载体pET28a-Rdrlec构建成功。

图3 EcoR I 和Hind III双酶切鉴定重组表达载体pET28a-Rdrlec
2.3 重组融合蛋白的诱导表达与纯化
将测序正确的重组表达载体pET28a-Rdrlec转化到表达宿主菌BL21(DE3),在含有50 μg/mL卡那霉素的LB液体培养基中培养至OD600达到0.8时,加入终浓度为0.5 mmol/L的IPTG,并降低诱导温度至34℃诱导重组融合蛋白表达,分别取诱导0 h和6 h的菌液,SDS-PAGE分析表达产物(图4),诱导6 h后的工程菌在约17 kD处有一条明显的新生的重组蛋白表达条带,与重组融合蛋白的预期分子量值(16.7 kD)相符,而未诱导的工程菌在相应位置处无相应的蛋白条带。分析菌体经超声波裂解后的上清和沉淀(图4中泳道6,7),可见重组蛋白大部分分布在沉淀中,以包涵体形式表达。
SDS-PAGE显示(图4中泳道3),Ni-NTA亲和层析后重组融合蛋白得到了有效的纯化。
将纯化后的融合蛋白用肠激酶裂解,释放Rdrlec蛋白,电泳结果(图4中泳道4)显示,酶切效果理想,释放出分子量约12.3 kD的Rdrlec蛋白和分子量约4.4 kD的载体融合肽。肠激酶裂解产物再次进行Ni-NTA亲和层析,由于载体融合肽带有6×His标签而结合在层析介质上,而Rdrlec蛋白则流出层析柱。将流出液中的Rdrlec蛋白透析后冻干,溶解于pH6.0 的40 mmol/L乙酸钠缓冲液中,电泳显示(图4中泳道5),Rdrlec重组蛋白得到了有效的纯化。

图4 重组蛋白表达及纯化产物电泳检测
2.4 Rdrlec的酶活性
重组Rdrlec具有酶活性,其酶活性弱于RNase A,但是显著高于乙酸钠阴性对照组(图5)。由于核糖核酸酶的活性依赖于分子正确的空间结构,因此可以推测Rdrlec重组蛋白已经正确折叠。
2.5 Rdrlec重组蛋白的抗菌活性
Rdrlec重组蛋白对测试的两种菌均具有显著的抗菌活性(图6),其中对大肠杆菌抑制作用最强,在Rdrlec重组蛋白浓度为2 μmol/L时,菌落数下降约5个数量级。对金黄色葡萄球菌的抑制作用其次,菌落数下降约3个数量级。
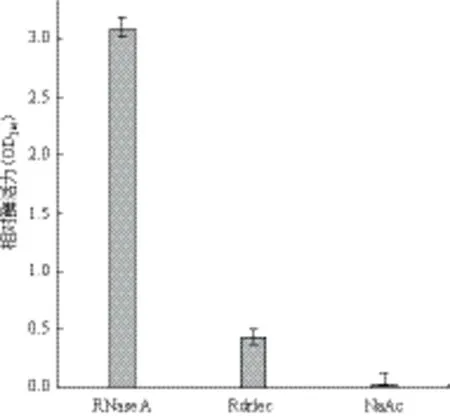
图5 Rdrlec重组蛋白的酶活性与RNase A比较
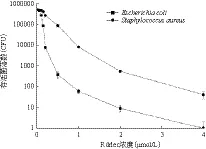
图6 Rdrlec重组蛋白的抗菌性
3 讨论
为了探索Rdrlec这一新基因编码的蛋白产物的功能,我们利用基因工程技术在大肠杆菌中表达重组蛋白。由于有活性的重组蛋白在表达过程中可能对宿主细胞有毒害,因此采用包涵体形式表达。包涵体主要由目的蛋白构成的,但是由于错误折叠形成无活性、不溶的蛋白聚集体,包涵体与细胞质成分明显分开,细胞破碎之后仍以颗粒形式存在。包涵体内绝大多数成分是蛋白质,杂质主要是一些外膜蛋白、质粒DNA、核糖体元件、脂质、肽聚糖和脂多糖等。包涵体的形成对于重组蛋白的生产提供了几个有利的因素:包涵体具有高密度,易于通过低速离心分离纯化;包涵体有较强的抗酸碱、抗蛋白酶的能力,有利于保护重组蛋白免受外界强烈作用的破坏而降解损失;可用于生产天然构象时对宿主细胞有毒害的蛋白质。
由于基因表达水平与偏好密码子的使用程度之间存在强的相关性,因此为了提高重组蛋白的表达效率,我们采用大肠杆菌偏好密码子进行了基因全序列优化和人工合成。同时,由于天然N 端对抗菌肽的生物活有决定性作用,为了保证所表达的重组蛋白具有天然N 端,我们在抗菌肽基因的N 端添加了肠激酶识别序列。
本研究表达出的中国林蛙核糖核酸酶Rdrlec重组蛋白具有明显的抗菌作用,对革兰氏阳性菌和阴性菌表现出不同的抗菌活性,这可能与细菌细胞的表面结构组成有关。不同的细菌外膜组成上存在差异,使表面的负电荷表现差异,对同一种蛋白的结合能力造成差异[10]。抗菌蛋白必需穿过胞外基质、细胞壁肽聚糖层等到达细胞质膜。按照膜裂解型或非膜裂解型方式作用质膜[11],目前发现的绝大多数抗菌蛋白是通过损伤质膜的膜裂解型方式作用于细胞。抗菌蛋白的正电荷启动与质膜的结合,双亲α螺旋或β折叠的结构进一步实现对膜脂双层的插入或破坏。
中国林蛙核糖核酸酶Rdrlec作为一种有效的抗菌蛋白,为抗微生物感染提供了新的候选药物分子,在医药、食品防腐和动物饲料添加剂等领域可能具有应用的潜力。
4 结论
按照大肠杆菌偏好的密码子合成Rdrlec基因,并在目的基因的N端添加肠激酶切割位点,同时在C端添加终止密码子,通过EcoR I和Hind III限制性内切酶位点将目的基因定向克隆到pET-28a(+)表达载体中,在大肠杆菌BL21(DE3)中表达出带有6×His标签的融合蛋白,利用Ni-NTA亲和层析纯化融合蛋白后,经过肠激酶切割,获得具有抗菌活性的不带有额外氨基酸序列残留的Rdrlec重组蛋白,重组蛋白具有水解RNA的酶活性,说明形成了正确的空间结构,重组蛋白对对测试的革兰氏阳性金黄色葡萄球菌和革兰氏阴性大肠杆菌均具有显著的抗菌活性,提示中国林蛙核糖核酸酶Rdrlec可能在抗微生物感染中发挥作用。
[1] 陶凤云, 赵伟, 马润宇. 核糖核酸酶A超家族成员杀菌作用研究进展[J]. 中国抗生素杂志, 2009, 34(6):321-325, 379.
[2] Tao F, Fan M, Zhao W, et al. A novel cationic ribonuclease with antimicrobial activity from Rana dybowskii[J]. Biochem Genet, 2011, 49 (5-6):369-384.
[3] 陶凤云, 赵伟, 林强, 等. 中国林蛙卵核糖核酸酶的分离纯化及其抗肿瘤作用[J]. 中国生物工程杂志, 2010, 30(5):103-109.
[4] Lee I. Ranpirnase (Onconase), a cytotoxic amphibian ribonuclease, manipulates tumor physiological parameters as a selective killer and a potential enhancer for chemotherapy and radiation in cancer therapy[J]. Expert Opin Biol Ther, 2008, 8 (6):813-827.
[5] Nitto T, Dyer KD, Czapiga M, et al. Evolution and function of leukocyte RNase A ribonucleases of the avian species, Gallus gallus[J]. J Biol Chem, 2006, 281:25622-25634.
[6] Cho S, Zhang J. Zebrafish ribonucleases are bactericidal:implications for the origin of the vertebrate RNase A superfamily[J]. Mol Biol Evol, 2007, 24 (5):1259-1268.
[7] Pizzo E, Varcamonti M, Di Maro A, et al. Ribonucleases with angiogenic and bactericidal activities from the Atlantic salmon[J]. FEBS J, 2008, 275 (6):1283-1295.
[8] Zanfardino A, Pizzo E, Di Maro A, et al. The bactericidal action on Esch-erichia coli of ZF-RNase-3 is triggered by the suicidal action of the bacterium OmpT protease[J]. FEBS J, 2010, 277:1921-1928.
[9] Rosenberg HF. Recombinant human eosinophil cationic protein. Ribonuclease activity is not essential for cytotoxicity[J]. J Biol Chem, 1995, 270 (14):7876-7881.
[10] Papo N, Shai Y. Can we predict biological activity of antimicrobial peptides from their interactions with model phospholipid membranes? [J]. Peptides, 2003, 24 (11):1693-1703.
[11] Powers JP, Hancock RE. The relationship between peptide structure and antibacterial activity[J]. Peptides, 2003, 24 (11):1681-1691.
